2. 南京师范大学江苏省石斛兰产业化技术工程中心, 江苏 南京 210023
2. Jiangsu Provincial Engineering Research Center for Technical Industrialization for Dendrobium, Nanjing Normal University, Nanjing 210023, China
石豆兰属(Bulbophyllum Thouars), 隶属于兰科(Orchidaceae), 约2 200种, 主要分布于热带和亚热带地区。我国约有105种, 广泛分布于贵州、广西、云南和福建等地[1]。石豆兰属植物不仅具有观赏价值, 还具备极高的药用价值, 可以清热润燥、化痰消肿。近年来, 对石豆兰属植物的研究主要集中在组织培养、化学成分和传粉生物学等方面, 并取得了较大进展[2-4], 但对其物种鉴别的研究却难以顺利开展, 主要原因在于: ①复杂的环境因素和传粉者的选择压力导致石豆兰属植物之间产生了较多相似的形态特征, 使它们难以区分; ②加工后的石豆兰药材与石斛属(Dendrobium Sw.)、石仙桃属(Pholidota Lindl. ex Hook.)、金石斛属(Flickingeria Hawkes)药材极为相似; ③缺乏能有效鉴别石豆兰属药用植物的DNA分子标记。因此, 亟需建立一种快速准确的石豆兰属药用植物的鉴别方法[5]。
叶绿体是半自主性细胞器, 拥有自己特有的基因组。叶绿体基因组具有“环形”结构、DNA裸露、以操纵子为单元进行转录等特征[6]。陆生植物的叶绿体基因组较为保守, 大小为100~170 kbp, 可分为4个部分:大单拷贝区(large single copy, LSC)、小单拷贝区(small single copy, SSC)和两个反向重复区(inverted repeat, IRB和IRA)。叶绿体基因组约有120个基因, 主要包含3类:与基因表达相关的基因、与光合作用相关的基因和RNA基因。与核基因组和线粒体基因组相比, 叶绿体基因组具有结构保守、单亲遗传不存在重组、序列变异度适中等特点, 是分子生物学研究中的重要工具[7]。
叶绿体基因组在长期的进化过程中, 结构上发生了一些变异[8], 如IR收缩[9]、倒位[10, 11]、基因和内含子的丢失[12]等。这些结构变异可以用于探讨物种间叶绿体基因组的进化关系。叶绿体基因组在序列上的变异主要集中在非编码区, 这些非编码区序列不仅可以应用于系统地理和系统发育研究, 还可以有效应用于物种鉴定研究[13]。例如:高变序列ndhF-rpl32、rpl32-trnL、ndhC-trnV、5′rps16-trnQ、psbE-petL、trnT-psbD、petA-psbJ和rpl16 intron, 可用于被子植物的系统发育研究[14]。Shaw等[14, 15]基于大量的数据统计, 筛选出可应用于物种鉴定的叶绿体基因组3个高变区域: ① ccsA到ndhF的区域, 其中包含两个最易变的非编码区ndhF-rpl32和rpl32-trnL; ② matK到3′trnG的一个较大区域, 其中包含几个变异度较高的区域, 包括matK、5′trnK-3′rps16、5′rps16-trnQ和trnS-5′trnG等; ③ rpoB到psbD的一个较小的区域, 其中包括trnT-psbD和rpoB-trnC。围绕这些区域筛选出的有效片段已成功应用于石斛属、兰属、蝴蝶兰属等药用植物的鉴定研究中。
近年来随着第二代测序技术的迅猛发展[16], 测序成本不断降低, 利用比较叶绿体基因组的方法, 筛选高变序列用于物种鉴定研究已变得可行[17-22]。因此, 本研究利用第二代测序技术, 测序、组装获得了3个石豆兰属药用植物叶绿体基因组, 结合其他兰科植物叶绿体基因组进行了比较, 分析了其属内叶绿体基因组的结构变化, 明确了系统发育关系, 并筛选出适合石豆兰属药用植物鉴定的叶绿体基因组高变序列。这些高变序列可以应用于石豆兰属药用植物的鉴定研究。
材料与方法植物材料和DNA提取 本研究测序的3个石豆兰属药用植物(赤唇石豆兰Bulbophyllum affine Lindl、长足石豆兰Bulbophyllum pectinatum Finet、富宁卷瓣兰Bulbophyllum funingense Z. H. Tsi et S. C. Chen)保存在南京师范大学生命科学学院植物所温室。上述石豆兰属植物每个个体取2 g新鲜叶片, 利用试剂盒(DNeasy Plant Mini Kit, Qiagen, Germany)提取总DNA。
DNA测序和叶绿体基因组的组装注释 提取的满足质量标准的DNA样本(浓度 > 300 ng·μL-1, A260/A230 > 1.7, A260/A280值间于1.8到2.0)用于高通量测序。测序平台为Illumina Hiseq 4000, 每个DNA样本获得约7.3 Gb raw data。用CLC Genomics Workbench 6.0.1 (CLC Bio, Aarhus, Denmark)组装。去除coverage < 50×的序列, 以Dendrobium devonianum Paxt.叶绿体基因组为参照, 对3个石豆兰叶绿体基因组进行组装。使用DOGMA和在线tRNA-scan进行基因注释[23, 24]。通过BLAST和3个石豆兰叶绿体基因组的多序列比对, 确定每个注释基因的边界。
GC含量统计和mVISTA分析 利用Vector NTI版本信息提取出叶绿体基因组中每个基因的位置信息。用基因位置信息整理LSC/IRB/SSC/IRA的节点信息。统计3个石豆兰属植物叶绿体基因组的LSC/IRB/SSC/IRA 4个区域及整体的GC含量。将各个叶绿体基因组的基因位置文件和序列文件提交到在线mVISTA网站上进行分析[25-29]。
SC/IR节点图谱的绘制 参考Zhu等[30]的文章中对LSC/IRB/LSC/IRA区域的4个节点的绘制方法, 基于新测序的3个石豆兰属植物(B. affine、B. pectinatum、B. funingense)、已有的2个石斛属植物(Dendrobium officinale Kimura et Migo、Dendrobium tosaense Makino)和1个蝴蝶兰属植物[Phalaenopsis equestris (Schauer) Rchb.]绘制节点图谱。
SSR分析 利用MISA软件对石豆兰属植物叶绿体基因组进行分析[18, 19, 23, 31], 参数设置为:单核苷酸重复次数 > 8, 二核苷酸重复次数 > 6, 三核苷酸、四核苷酸、五核苷酸和六核苷酸重复次数 > 5; 2个SSR之间的最小距离设置为100 bp。
系统进化分析 从NCBI上下载兰科植物的叶绿体全基因组[D. officinale、D. tosaense、Dendrobium primulinum Lindl.、Dendrobium hercoglossum Rchb. F.、Dendrobium falconeri Hook.、Phalaenopsis equestris、Bletilla striata (Thunb. ex Murray) Rchb. F.、Goodyera fumata Thw.、Cymbidium ensifolium (L.) Sw.、Cymbidium longibracteatum Y. S. Wu et S. C. Chen、Oncidium Gower Ramsey、Oncidium sphacelatum Lindl.]。利用Mafft 7.220对以上15个兰科植物叶绿体基因组进行比对, 将G. fumata作为外群, 利用RAxML构建Maximum Likelihood (ML)系统发育树[21, 22]。
序列变异度分析和高变区的筛选与验证 将基因的位置信息整理成非编码区的位置信息, 从序列FASTA文件中提取出每个非编码区的序列。用MEGA 5.0逐个比对每一个非编码区, 去除两端空缺。使用DnaSP 5.10统计出3个石豆兰属植物叶绿体基因组非编码区的SNP、InDel、保守位点、GC含量等信息[21, 32]。去除其中的保守位点数不足100的非编码区。利用公式: Sequence Variability = (SNP + InDel) / (SNP + InDel + Conserved site)计算非编码区的变异度[33]。基于非编码区的变异度和GC含量绘制分布图。
将15个兰科植物的前5变异度非编码区比对后串联, 将G. fumata作为外群, 利用RAxML构建Maximum Likelihood (ML)系统发育树。
结果与分析 1 石豆兰叶绿体基因组基本特征新测序获得的3个石豆兰叶绿体基因组(赤唇石豆兰Bulbophyllum affine LC556091、长足石豆兰Bulbophyllum pectinatum LC556092、富宁卷瓣兰Bulbophyllum funingense LC556093)为环形双链DNA, 大小为147 169~148 230 bp (表 1和图 1)。叶绿体基因组包含一对反向重复区域(inverted repeat): IRA和IRB, 和两个单拷贝区域(single copy): LSC和SSC (图 1)。LSC、SSC和IR区域的大小分别为77 478~78 178 bp、17 094~17 529 bp和26 081~26 403 bp。总GC含量为37.86%~38.01%, LSC的GC含量为35.83%~36.01%, SSC的GC含量为30.38%~30.87%, IR的GC含量为43.25%~43.37%。石豆兰属植物叶绿体基因组拥有74个蛋白质编码基因、30个tRNA基因和4个rRNA基因。
| Table 1 The basic information of chloroplast genomes of Bulbophyllum orchids |

|
Figure 1 Chloroplast genome map of Bulbophyllum orchids. The length of the chloroplast genome is shown in the circle. The different colors on the circle represent different types of genes |
基于B. affine叶绿体基因组, 利用mVISTA软件对新测序的3个石豆兰属植物叶绿体基因组进行了比较分析(图 2)。结果表明, ① tRNA与rRNA编码基因在石豆兰属植物中极其保守; ②基因编码区比非编码区保守, 变异较高的区域基本集中在LSC和SSC区域中。这与之前的研究结论一致[34-36]。
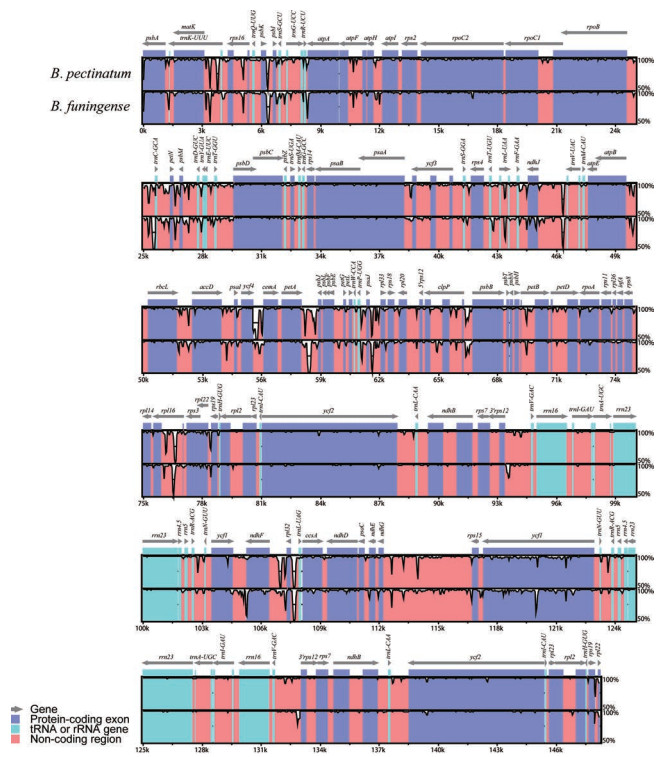
|
Figure 2 mVISTA analysis results of Bulbophyllum orchids. The reference chloroplast genome is the chloroplast genome of B. affine |
基于节点图谱(图 3)可见, 6个植物的LSC/IRB节点位于rpl22基因中, SSC/IRA节点则位于ycf1基因中, 这使得IRB/SSC节点处保留了部分ycf1基因片段, IRA/LSC节点处保留了部分rpl22基因片段。在前期研究中, 节点图谱主要有3种类型: ① SSC/IRA节点在(-) ycf1中, LSC/IRB节点在rpl22和rps19之间; ② SSC/IRA节点在(-) ycf1中, LSC/IRB节点在rps19中; ③ SSC/IRA节点在(-) ycf1中, LSC/IRB节点在rpl22中。第一种和第三种主要包含单子叶植物[22, 23, 26, 30], 而第二种则包含双子叶植物和单子叶植物[21, 24, 32]。本研究中, 石豆兰属植物叶绿体基因组节点处变化不大, 结构较为保守, 属于常见的第三种类型, 即SSC/IRA节点在(-) ycf1中, LSC/IRB节点在rpl22中, 与兰科其他属植物的节点图谱类型相近。
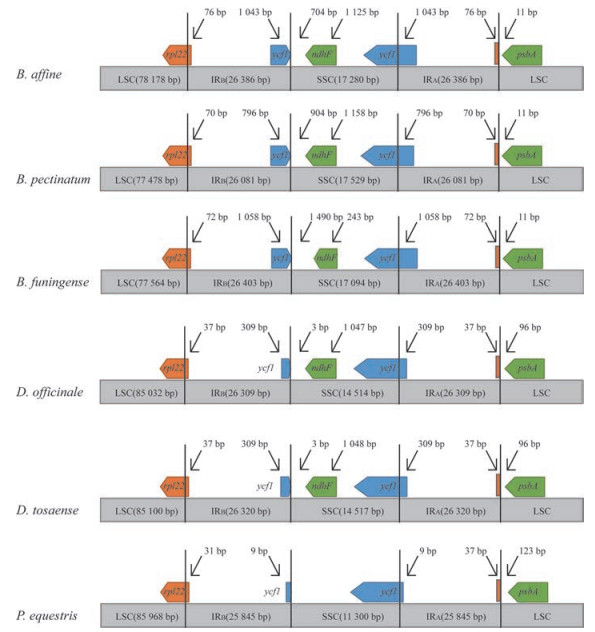
|
Figure 3 Boundary expansion and contraction diagram of the chloroplast genomes of Bulbophyllum orchids. The figure shows the length of each region and the location of the genes along the border |
利用MISA软件对石豆兰属植物叶绿体基因组进行分析, 在B. affine、B. funingense、B. pectinatum这3种石豆兰中分别检测到95、98、100个SSR位点(图 4A), 大部分位于基因间隔区(intergenic spacer, IGS)。检测到的SSR位点包括单核苷酸重复、双核苷酸重复以及复合型SSR (图 4B), 其中单核苷酸重复最多, 在这3种石豆兰中分别有75、75、82个, 并且主要分布在LSC区域中。由图 4可知, 石豆兰属植物叶绿体基因组中的SSR主要是由A和T组成的, 其中大部分是以A/T碱基构成的单核苷酸重复, 其次是由AT/TA构成的二核苷酸重复。
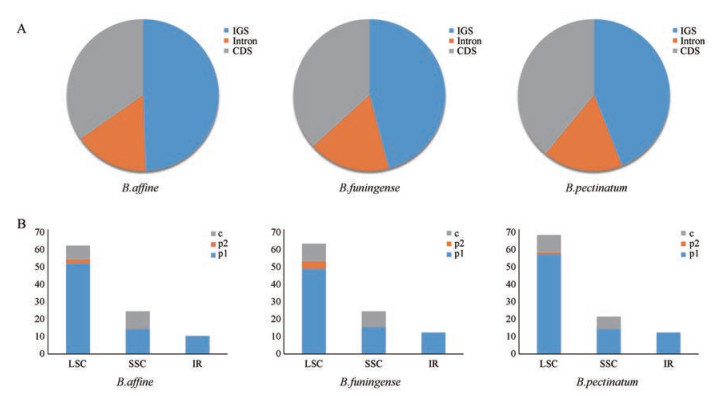
|
Figure 4 SSR distribution of chloroplast genome of Bulbophyllum orchids. A: Distribution of SSR in the coding region, intron region, and intergenic region of the gene; B: Distribution of various types of SSRs in different regions |
基于全叶绿体基因组构建的ML系统发育树可知(图 5A), 在石豆兰属内, B. affine和B. funingense的亲缘关系更近。在整个兰科植物属间关系上, 与石豆兰属植物亲缘关系最近的属是石斛属植物。

|
Figure 5 ML phylogenetic tree of chloroplast genomes. The figure shows a ML phylogenetic tree constructed from the complete chloroplast genomes (A) and a ML phylogenetic tree constructed from the non-coding regions which have the top five sequence variability (B) |
基于石豆兰属植物叶绿体基因组非编码区的GC含量和变异度分布图可知(图 6), IR区域的非编码区变异度明显低于LSC (P < 0.001)和SSC (P < 0.001)区域。同时, IR区域的GC含量明显高于LSC (P < 0.001)和SSC (P < 0.001)区域。可见, GC含量高的区域变异度相对较低[33, 37]。图 6中突出标记了前10变异度非编码区。同时, 列举了前10变异度非编码区的基本信息(表 2), 分别是psbI-trnS、psbC-trnS、clpP-ex1-psbB、psaJ-rpl33、rpl33-rps18、trnP-psaJ、rpl16-ex1-rps3、trnT-trnL-ex1、accD-psaI、atpH-atpI。它们全部分布在LSC区域中。在基于前5变异度非编码区构建的ML系统发育树中(图 5B), 拓扑结构与基于全叶绿体基因组构建的ML系统发育树相同。由此可见, 前5变异度非编码区不仅能够正确区分石豆兰属植物与兰科其他属植物, 而且也能正确区分石豆兰属中的不同植物。最后, psbI-trnS + psbC-trnS + clpP-ex1-psbB + psaJ-rpl33 + rpl33-rps18的片段组合具有最高的变异度, 推荐用于石豆兰属植物的物种鉴定研究。
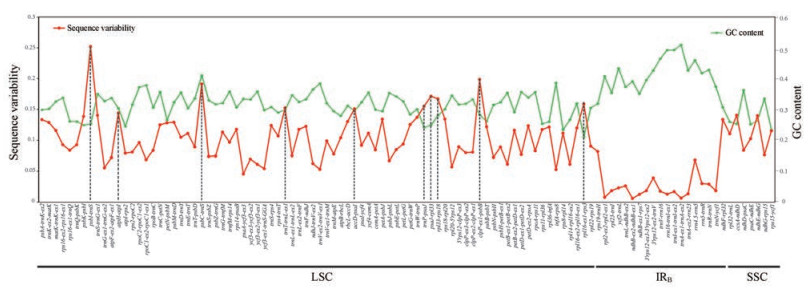
|
Figure 6 Distribution of sequence variability and GC content of the non-coding regions in chloroplast genomes of Bulbophyllum orchids. The red part shows the distribution of sequence variability; The green part shows the distribution of GC content |
| Table 2 Basic information of non-coding regions which have top ten sequence variability |
石豆兰属植物具有极高的观赏价值和药用价值。目前, 对石豆兰属植物的研究在组织培养、化学成分和传粉生物学等方面取得了较大进展[2-4], 但对其物种鉴别研究却成果甚微, 因此开发出石豆兰属药用植物的专属DNA条形码将会带来巨大的价值。本研究基于二代高通量测序技术和比较基因组学研究方法对3种石豆兰属药用植物进行了分析研究。结果显示, 石豆兰属植物叶绿体基因组在基因组结构和基因序列方面相当保守。本研究还统计并计算了所有非编码区的GC含量和变异度。结果显示, 石豆兰属植物叶绿体基因组的高变序列具有多样性和聚集性。最后, 筛选了5个变异度最高的高变序列用于石豆兰属药用植物的物种鉴定。
前期研究表明, 叶绿体基因组在基因组结构和基因序列方面相当保守[38-40]。在序列长度方面, 叶绿体基因组总长度差异在1 200 bp以内, LSC、SSC和IR各区域的长度差异分别在700、500和400 bp以内。此外, LSC、SSC和IR区域的节点处高度保守, 表明石豆兰属植物的叶绿体基因组结构高度保守。mVISTA分析表明, 3个石豆兰属植物叶绿体基因组的基因序列高度相似。此外, LSC、SSC、IR各个区域的GC含量明显不同, 变化幅度都在0.5%以内, 可见石豆兰属植物叶绿体基因组各个区域明显不同, 并且在属内各个区域的序列十分相似。因此, 石豆兰属植物叶绿体基因组在基因序列方面也相当保守。
叶绿体基因组的高变序列具有多样性与聚集性, 例如: ①在Phalaenopsis属植物中trnS-trnG、psaC-ndhE、clpP-psbB、rpl16 intron、rpoB-trnC、trnT-psbD、rbcL-accD、rpl32-trnL、ccsA-ndhD和ndhC-trnV具有最高的变异度, 而在Cymbidium属植物中rpl32-trnL、trnE-trnT、trnH-psbA、trnK-rps16和trnT-trnL具有最高的变异度[14], 表明兰科植物叶绿体基因组的高变序列存在多样性; ②前期研究表明, 被子植物的高变序列主要存在于3个区域中, Niu等[15]进一步针对石斛属植物叶绿体基因组高变区进行分析, 总结出4个高变序列聚集区。本研究筛选的最高变的10个非编码区序列中只有psbI-trnS和trnT-trnL-ex1在其他兰科植物中出现, 表明叶绿体基因组的高变序列具有多样性。石豆兰属植物的高变序列全部集中在LSC区域, 并且出现了明显的聚集, 主要分为两个聚集区(psbI-trnS和accD-psbB区域)。石豆兰属植物与亲缘关系较近的石斛属植物的高变序列聚集区有较大的差异, 可见兰科不同属植物的高变序列聚集区也具有特异性。因此, 有必要针对兰科重要属筛选高变序列, 用于属内植物的有效鉴定。
高变序列适合用于构建DNA条形码。前期研究表明, 对叶绿体基因组进行比较基因组学研究筛选出的高变序列, 能够有效地应用于种间鉴别[41, 42]和系统发育研究[14]。例如: ①非编码区片段: rpl32-trnL、trnE-trnT、trnH-psbA、trnK-rps16和trnT-trnL在Cymbidium属植物中具有较高的序列变异度, 在Cymbidium属植物的种间鉴别中有巨大潜力; ② trnS-trnG、psaC-ndhE、clpP-psbB、rpl16 intron、rpoB-trnC、trnT-psbD、rbcL-accD、rpl32-trnL、ccsA-ndhD和ndhC-trnV这10个非编码区片段因具有最高的序列变异度可以有效应用于Phalaenopsis属的物种鉴别[14]。高变序列由于其序列短的特点, 其能作为DNA条形码经济、快速地区分不同属种的植物。ML系统发育树分析表明, 石豆兰属植物与石斛属植物亲缘关系十分接近, 然而两个属的高变序列存在较大差异。石豆兰属包含大量药用植物, 因此筛选其属内特异性DNA条形码意义重大。因此, 基于本研究结果, 推荐石豆兰属植物叶绿体基因组中最高变的5个非编码区(psbI-trnS、psbC-trnS、clpP-ex1-psbB、psaJ-rpl33、rpl33-rps18)组合用于设计鉴定石豆兰属药用植物的专属DNA条形码。
作者贡献:牛志韬、丁小余和杨嘉鹏负责研究设计; 杨嘉鹏、朱紫乐和陈粤珺负责实验操作; 杨嘉鹏、朱紫乐、范雅娟和朱菲负责数据分析; 杨嘉鹏负责论文撰写。所有的作者都对原稿的最终版本做出了贡献。
利益冲突:无相关利益冲突。
| [1] |
Ya JD, Guo YJ, Liu C, et al. Bulbophyllum reflexipetalum (Orchidaceae, Epidendroideae, Malaxideae), a new species from Xizang, China[J]. PhytoKeys, 2019, 130: 33-39. DOI:10.3897/phytokeys.130.34153 |
| [2] |
Teixeira S, Borba E, Semir J. Lip anatomy and its implications for the pollination mechanisms of Bulbophyllum species (Orchidaceae)[J]. Ann Bot, 2004, 93: 499-505. DOI:10.1093/aob/mch072 |
| [3] |
Fang YS, Yang MH, Cai L, et al. New phenylpropanoids from Bulbophyllum retusiusculum[J]. Arch Pharm Res, 2018, 41: 1074-1081. DOI:10.1007/s12272-018-1067-6 |
| [4] |
Gamisch A, Staedler YM, Schönenberger J, et al. Histological and micro-CT evidence of stigmatic rostellum receptivity promoting auto-pollination in the madagascan orchid Bulbophyllum bicoloratum[J]. PLoS One, 2013, 8: e72688. DOI:10.1371/journal.pone.0072688 |
| [5] |
Kim YK, Jo S, Cheon SH, et al. Corrigendum:plastome evolution and phylogeny of Orchidaceae, with 24 new sequences[J]. Front Plant Sci, 2020, 11: 322. DOI:10.3389/fpls.2020.00322 |
| [6] |
Pfalz J, Bayraktar OA, Prikryl J, et al. Site-specific binding of a PPR protein defines and stabilizes 5' and 3' mRNA termini in chloroplasts[J]. EMBO J, 2009, 28: 2042-2052. DOI:10.1038/emboj.2009.121 |
| [7] |
Jansen RK, Cai ZQ, Raubeson LA, et al. Analysis of 81 genes from 64 plastid genomes resolves relationships in angiosperms and identifies genome-scale evolutionary patterns[J]. Proc Natl Acad Sci U S A, 2007, 104: 19369-19374. DOI:10.1073/pnas.0709121104 |
| [8] |
Dyall SD, Brown MT, Johnson PJ. Ancient invasions:from endosymbionts to organelles[J]. Science, 2004, 304: 253-257. DOI:10.1126/science.1094884 |
| [9] |
Palmer JD, Nugent JM, Herbon LA. Unusual structure of geranium chloroplast DNA:a triple-sized inverted repeat, extensive gene duplications, multiple inversions, and two repeat families[J]. Proc Natl Acad Sci U S A, 1987, 84: 769-773. DOI:10.1073/pnas.84.3.769 |
| [10] |
Kim KJ, Lee HL. Widespread occurrence of small inversions in the chloroplast genomes of land plants[J]. Mol Cells, 2005, 19: 104-113. |
| [11] |
Greiner S, Wang X, Rauwolf U, et al. The complete nucleotide sequences of the five genetically distinct plastid genomes of Oenothera, subsection Oenothera:I. sequence evaluation and plastome evolution[J]. Nucleic Acids Res, 2008, 36: 2366-2378. DOI:10.1093/nar/gkn081 |
| [12] |
Magee AM, Aspinall S, Rice DW, et al. Localized hypermutation and associated gene losses in legume chloroplast genomes[J]. Genome Res, 2010, 20: 1700-1710. DOI:10.1101/gr.111955.110 |
| [13] |
Niu ZT, Xue QY, Zhu SY, et al. The complete plastome sequences of four Orchid species:insights into the evolution of the Orchidaceae and the utility of plastomic mutational hotspots[J]. Front Plant Sci, 2017, 8: 715. DOI:10.3389/fpls.2017.00715 |
| [14] |
Shaw J, Shafer HL, Leonard OR, et al. Chloroplast DNA sequence utility for the lowest phylogenetic and phylogeographic inferences in angiosperms:the tortoise and the hare IV[J]. Am J Bot, 2014, 101: 1987-2004. DOI:10.3732/ajb.1400398 |
| [15] |
Niu ZT, Pan JJ, Xue QY, et al. Plastome-wide comparison reveals new SNV resources for the authentication of Dendrobium huoshanense and its corresponding medicinal slice (Huoshan Fengdou)[J]. Acta Pharm Sin B, 2018, 8: 466-477. DOI:10.1016/j.apsb.2017.12.004 |
| [16] |
Nock CJ, Waters DL, Edwards MA, et al. Chloroplast genome sequences from total DNA for plant identification[J]. Plant Biotechnol J, 2011, 9: 328-333. DOI:10.1111/j.1467-7652.2010.00558.x |
| [17] |
Li XW, Yang Y, Henry RJ, et al. Plant DNA barcoding:from gene to genome[J]. Biol Rev Camb Philos Soc, 2015, 90: 157-166. DOI:10.1111/brv.12104 |
| [18] |
Sun JY, Dong XF, Cao QH, et al. A systematic comparison of eight new plastome sequences from Ipomoea L[J]. PeerJ, 2019, 7: e6563. DOI:10.7717/peerj.6563 |
| [19] |
Bi Y, Zhang MF, Xue J, et al. Chloroplast genomic resources for phylogeny and DNA barcoding:a case study on Fritillaria[J]. Sci Rep, 2018, 8: 1184. DOI:10.1038/s41598-018-19591-9 |
| [20] |
Gichira AW, Avoga S, Li ZZ, et al. Comparative genomics of 11 complete chloroplast genomes of Senecioneae (Asteraceae) species:DNA barcodes and phylogenetics[J]. Bot Stud, 2019, 60: 17. DOI:10.1186/s40529-019-0265-y |
| [21] |
Xie DF, Yu Y, Deng YQ, et al. Comparative analysis of the chloroplast genomes of the Chinese endemic genus Urophysa and their contribution to chloroplast phylogeny and adaptive evolution[J]. Int J Mol Sci, 2018, 19: 1847. DOI:10.3390/ijms19071847 |
| [22] |
Gao BM, Yuan L, Tang TL, et al. The complete chloroplast genome sequence of Alpinia oxyphylla Miq. and comparison analysis within the Zingiberaceae family[J]. PLoS One, 2019, 14: e0218817. DOI:10.1371/journal.pone.0218817 |
| [23] |
Li DM, Zhao CY, Liu XF. Complete chloroplast genome sequences of Kaempferia galanga and Kaempferia elegans:molecular structures and comparative analysis[J]. Molecules, 2019, 24: 474. DOI:10.3390/molecules24030474 |
| [24] |
Meng J, Li XP, Li HT, et al. Comparative analysis of the complete chloroplast genomes of four Aconitum medicinal species[J]. Molecules, 2018, 23: 1015. DOI:10.3390/molecules23051015 |
| [25] |
Park I, Yang SY, Kim WJ, et al. Sequencing and comparative analysis of the chloroplast genome of Angelica polymorpha and the development of a novel Indel marker for species identification[J]. Molecules, 2019, 24: 1038. DOI:10.3390/molecules24061038 |
| [26] |
Wang W, Chen SY, Zhang XZ. Whole-genome comparison reveals divergent IR borders and mutation hotspots in chloroplast genomes of herbaceous bamboos (Bambusoideae:Olyreae)[J]. Molecules, 2018, 23: 1537. DOI:10.3390/molecules23071537 |
| [27] |
Liu ML, Fan WB, Wang N, et al. Evolutionary analysis of plastid genomes of seven Lonicera L. species:implications for sequence divergence and phylogenetic relationships[J]. Int J Mol Sci, 2018, 19: 4039. DOI:10.3390/ijms19124039 |
| [28] |
Yang QQ, Jiang M, Wang LQ, et al. Complete chloroplast genome of Allium chinense:comparative genomic and phylogenetic analysis[J]. Acta Pharm Sin (药学学报), 2019, 54: 173-181. |
| [29] |
Zhang MY, Wang XF, Gao J, et al. Complete chloroplast genome of Paeonia mairei H. Lév.:characterization and phylogeny[J]. Acta Pharm Sin (药学学报), 2020, 55: 168-176. |
| [30] |
Zhu SY, Niu ZT, Xue QY, et al. Accurate authentication of Dendrobium officinale and its closely related species by comparative analysis of complete plastomes[J]. Acta Pharm Sin B, 2018, 8: 969-980. DOI:10.1016/j.apsb.2018.05.009 |
| [31] |
Qiao YG, He JX, Wang YF, et al. Analysis of chloroplast genome and its characteristics of medicinal plant Sophora flavescens[J]. Acta Pharm Sin (药学学报), 2019, 54: 2106-2112. |
| [32] |
Yan M, Zhao XQ, Zhou JQ, et al. The complete chloroplast genomes of Punica granatum and a comparison with other species in Lythraceae[J]. Int J Mol Sci, 2019, 20: 2886. DOI:10.3390/ijms20122886 |
| [33] |
Niu ZT, Zhu SY, Pan JJ, et al. Comparative analysis of Dendrobium plastomes and utility of plastomic mutational hotspots[J]. Sci Rep, 2017, 7: 2073. DOI:10.1038/s41598-017-02252-8 |
| [34] |
Tyagi S, Jung J, Kim JS, et al. Comparative analysis of the complete chloroplast genome of mainland Aster spathulifolius and other Aster species[J]. Plants (Basel), 2020, 9: 568. |
| [35] |
Cheng Y, Zhang LM, Qi JM, et al. Complete chloroplast genome sequence of Hibiscus cannabinus and comparative analysis of the Malvaceae family[J]. Front Genet, 2020, 11: 227. DOI:10.3389/fgene.2020.00227 |
| [36] |
Munyao JN, Dong X, Yang JX, et al. Complete chloroplast genomes of Chlorophytum comosum and Chlorophytum gallabatense:genome structures, comparative and phylogenetic analysis[J]. Plants (Basel), 2020, 9: 296. |
| [37] |
Yang JB, Tang M, Li HT, et al. Complete chloroplast genome of the genus Cymbidium:lights into the species identification, phylogenetic implications and population genetic analyses[J]. BMC Evol Biol, 2013, 13: 84. DOI:10.1186/1471-2148-13-84 |
| [38] |
Yu XY, Zuo LH, Lu DD, et al. Comparative analysis of chloroplast genomes of five Robinia species:genome comparative and evolution analysis[J]. Gene, 2019, 689: 141-151. DOI:10.1016/j.gene.2018.12.023 |
| [39] |
Zhang X, Rong CX, Qin L, et al. Complete chloroplast genome sequence of Malus hupehensis:genome structure, comparative analysis, and phylogenetic relationships[J]. Molecules, 2018, 23: 2917. DOI:10.3390/molecules23112917 |
| [40] |
Wang XM, Zhou T, Bai GQ, et al. Complete chloroplast genome sequence of Fagopyrum dibotrys:genome features, comparative analysis and phylogenetic relationships[J]. Sci Rep, 2018, 8: 12379. DOI:10.1038/s41598-018-30398-6 |
| [41] |
Ahmed I, Matthews PJ, Biggs PJ, et al. Identification of chloroplast genome loci suitable for high-resolution phylogeographic studies of Colocasia esculenta (L.) Schott (Araceae) and closely related taxa[J]. Mol Ecol Resour, 2013, 13: 929-937. DOI:10.1111/1755-0998.12128 |
| [42] |
Hsu CY, Wu CS, Chaw SM. Ancient nuclear plastid DNA in the yew family (taxaceae)[J]. Genome Biol Evol, 2014, 6: 2111-2121. DOI:10.1093/gbe/evu165 |
 2020, Vol. 55
2020, Vol. 55


