2. 江苏海岸药业有限公司, 江苏 苏州 215215
2. Jiangsu Coastal Pharmaceutical Co., Ltd., Suzhou 215215, China
基因编辑技术(genome editing)可以精确地破坏、插入或替换基因组中特定位点的DNA序列, 在基因功能的研究和遗传疾病的治疗中发挥着巨大的作用。人工核酸内切酶(engineered endonuclease, EEN)介导的基因编辑技术极大地改善了早期基于同源重组的基因打靶技术(gene targeting)效率低的问题, 使得科研工作者可以高效地对各种细胞类型和生物的任何基因进行编辑[1]。EEN主要包括:锌指核酸酶(zinc finger nucleases, ZFN)、转录激活因子样效应物核酸酶(transcription activator-like effector nucleases, TALEN)及CRISPR/Cas技术[1]。
1 CRISPR/Cas9基因编辑技术简介及作用机制成簇的规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)是广泛存在于细菌和古细菌中的一组DNA序列[2], 由重复(repeats)序列和间隔(spacer)序列组成。其中间隔序列与外源性DNA具有同源性, 是噬菌体、病毒等侵入性核酸留下的痕迹, 在随后的感染中可以检测和破坏类似的DNA。Cas序列位于CRISPR序列的附近, 编码与CRISPR功能密切相关的蛋白。CRISPR/Cas系统是细菌和古细菌在抵御病毒和质粒的不断入侵中演化来的一种获得性免疫防御机制[3-6]。
根据使用Cas蛋白的种类, CRISPR/Cas系统分为两类:第一类CRISPR/Cas系统功能的实现需要多种Cas蛋白; CRISPR/Cas9属于第二类中的Ⅱ型CRISPR/Cas系统, 仅使用具有多个功能域的一种Cas蛋白, 即CRISPR相关蛋白9 (CRISPR-associated protein 9, Cas9)[2, 7]。CRISPR/Cas9系统由两部分组成: ① crRNA (CRISPR RNA, crRNA)和反式激活CRISPR RNA (tracrRNA), crRNA以CRISPR序列为模板转录而来, 与tracrRNA通过碱基互补配对形成的嵌合RNA (tracrRNA/crRNA)可以引导Cas9结合到特定DNA位点; ② Cas9由Cas序列编码的一种核酸内切酶, 以tracrRNA/crRNA作为向导并识别特定的前间隔序列邻近基序(protospacer-adjacent motif, PAM)后, Cas9的HNH结构域切割和tracrRNA/crRNA互补的DNA链, RuvC结构域切割另一条DNA链形成双链断裂(double strand break, DSB)[8]。科研人员将这两条RNA融合成一条单链向导RNA (single-guide RNA, sgRNA), Cas9与sgRNA结合后也能够识别并切割与sgRNA互补配对的DNA。对sgRNA序列进行设计, 可以切割不同的目的基因。基因组特定位点产生DSB后, 受损的基因组可以通过非同源末端连接(non-homologous end joining, NHEJ)形成随机的多个碱基的插入(insertions)或删除(deletions), 从而使目的基因失活; 也可以通过同源重组修复(homology directed repair, HDR)机制, 在模板(donor template)存在的条件下, 实现定点的基因替换(replacements) (图 1)[8-10]。由于Cas9的核酸酶活性高、sgRNA识别机制的特点, 基因编辑效率和特异性都很高, 该技术已广泛应用于基因编辑, 涉及各个领域, 包括生命科学、生物技术、医学和农业, 在新药研发过程中, 对靶基因的定点编辑使得构建动物模型或细胞系模型更加便捷, 极大地加速了药物靶点的筛选验证及新药的研发[11]。
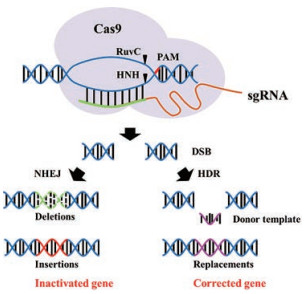
|
Figure 1 Mechanism of genome editing by CRISPR/Cas9. CRISPR: Clustered regularly interspaced short palindromic repeats; Cas9: CRISPR-associated protein 9; PAM: Protospacer-adjacent motif; sgRNA: Single-guide RNA; DSB: Double strand break; NHEJ: Non-homologous end joining; HDR: Homology directed repair |
CRISPR/Cas9来源于原核生物, 对靶细胞的基因进行编辑需要将功能组分递送到胞内。目前, 在靶细胞实现基因编辑的方式有3种(图 2): ①递送编码Cas9蛋白和sgRNA的质粒; ②递送Cas9 mRNA和sgRNA; ③递送Cas9蛋白和sgRNA, 可以分别递送Cas9蛋白和sgRNA, 也可以递送二者结合形成的核糖核蛋白复合物(ribonucleoprotein complexes, RNP)。CRISPR/Cas9系统递送方法包括:物理方法(微注射、电穿孔、核感染和膜变形)、病毒载体和非病毒载体。物理方法主要用于体外或离体细胞和组织的基因编辑[12, 13]; 病毒载体目前已广泛应用于CRISPR/Cas9系统的体内外给药, 但其基本缺点, 如致癌风险大、插入体积小、免疫应答和难以大规模生产等, 严重限制了进一步应用[14]; 适合体内递送CRISPR/Cas9系统的高效非病毒载体亟待研究。

|
Figure 2 Genome editing by three forms of CRISPR/Cas9: ① pCas9/sgRNA, ② mRNA and sgRNA, ③ Cas9 and sgRNA |
pX330是最常用于真核细胞基因组的CRISPR/Cas9二合一质粒, 8 963 bp、分子质量5.55×103 kDa, 具有1.79×104个磷酸根, 单个质粒带1.79×104个负电荷[15, 16]。递送CRISPR/Cas9质粒进行基因编辑, 质粒可以同时编码Cas9蛋白、sgRNA和HDR模板, 操作简单, 成本低, 且双链DNA的稳定性比Cas9蛋白和Cas9 mRNA更好。但pDNA尺寸过大, 负电荷很强, 给负载和包埋都带来了较大的困难, 显著增加了CRISPR/Cas9系统递送和表达的难度[17]; 此外, pDNA需要进入细胞核进行转录, 转录的Cas9 mRNA需要转移到细胞质中进行翻译, 并与sgRNA组装后进入细胞核才能发挥功能, 整个过程需要较长的时间; pDNA可能会随机整合到细胞的基因组中, 导致Cas9蛋白长时间存在于细胞中, 这可能会导致更高的脱靶效应和强烈的免疫反应[18]; 对于不断分裂的细胞, 质粒在核膜解体时扩散到细胞核中, 对于非分裂细胞, 需要额外的核定位信号(nuclear localization sequence, NLS)或阳离子细胞穿透肽(cell-penetrating peptides, CPPs)才能将质粒转运到细胞核中[19-21]。
pCas9/sgRNA递送系统应具备以下特点: ①具有足够的正电荷, 阳离子脂质或具有丰富氨基的聚合物适合用于制备递送系统, 但是高正电荷会引起细胞毒性, 必须综合考虑; ②尽管pX330的双链结构相对稳定, 但血清和体液富含核酸酶, 高表达Toll样受体的先天免疫细胞能够识别DNA并引起炎症[22], 递送系统需要保护pX330免受降解, 同时降低pX330的免疫原性; ③将pX330选择性地递送到靶细胞中, 由于不同组织细胞的基因组相同, pX330一旦表达, 可以非选择性地编辑每个细胞的基因组, 具有细胞靶向能力的递送系统可以使脱靶效应和潜在的安全风险最小化。
2.2 递送Cas9 mRNA和sgRNACas9 mRNA和sgRNA通常使用dsDNA模板通过体外转录得到, dsDNA模板的序列源自pCas9。将Cas9 mRNA和sgRNA递送到靶细胞后, Cas9 mRNA翻译表达成Cas9蛋白, 随后Cas9蛋白与sgRNA在细胞内形成RNP发挥基因编辑作用。Cas9 mRNA为单链线性RNA, 长度约为4 300 nt, 分子质量约为1.38×103 kDa, 含约4 300个负电荷, sgRNA长度约100 nt。基因编辑过程中, Cas9 mRNA无需入核, 进入细胞质即可翻译加工成Cas9蛋白, Cas9蛋白的表达是瞬时的, 产生的脱靶效应比CRISPR/Cas9质粒更低, 起效更快, 没有整合风险, 不会引发免疫反应, 对于临床治疗更安全。但Cas9 mRNA在细胞质中的半衰期很短, 24 h内会降解, 极不稳定, 对递送系统要求更高, 瞬时表达虽然减少了脱靶效应和免疫反应, 但会导致效率降低。与基于质粒的CRISPR/Cas9可以利用特定的启动子来驱动Cas9的特异性表达[23]不同, Cas9 mRNA在翻译成Cas9核酸酶后可以非特异性地编辑每种细胞。
鉴于Cas9 mRNA自身的长度及其与sgRNA长度的巨大差异, 理想的非病毒载体需具有较强的正电荷, 能同时封装长和短RNA且有效压缩Cas9 mRNA。在改善单链RNA (ssRNA)稳定性方面, 科研人员已尝试通过对Cas mRNA和sgRNA进行化学修饰来抵抗核酸酶的降解, 提高其稳定性和基因编辑效率[24-26], 设计非病毒载体时也可以将二者封装在纳米粒内部, 避免RNase的降解和免疫系统的识别。递送系统需要选择性地进入靶细胞, 减少脱靶效应; 进入细胞后, 递送系统应能在内含体中保持完整, 并迅速成功逃逸进入细胞质发挥功能。
2.3 递送Cas9蛋白和sgRNACas9蛋白可直接购买, 但价格昂贵且无法设计定制。使用pET-28b质粒表达Cas9蛋白也是获得Cas9蛋白的一种方式, sgRNA可以通过体外转录制备, Cas9蛋白和sgRNA体外孵育得到RNP[27]。Cas9蛋白带有22个正电荷, 而sgRNA带有约100个负电荷, RNP的总电荷为负, 大小约10 nm。由于无需转录和翻译, 该递送方式能快速实现基因编辑。RNP进入细胞后迅速降解, 只能短暂地发挥功能, 可以减少脱靶效应、毒性和免疫反应。但是Cas9蛋白(约160 kDa)分子质量巨大且带正电荷, sgRNA呈现强负电荷, 要实现Cas9/sgRNA RNPs的高效递送较为困难。因为成本和细菌内毒素污染问题, 想要获得大量高活性的Cas9蛋白也存在一定难度。与主要诱导先天性免疫应答的质粒和RNA不同, RNP可以引起适应性免疫应答。78%成年人中检测到了针对金葡菌Cas9复合物(Staphylococcus aureus, SaCas9)的IgG抗体, 而58%成年人中携带了针对化脓性链球菌Cas9 (Streptococcus pyogenes, SpCas9)的IgG抗体[28]。在67%人类供体中检测到了抗SpCas9 T细胞[28]。抗Cas9抗体和T细胞在人类中已经存在。抗体可以直接与Cas9 RNP结合, 而当靶细胞表面存在Cas9 RNP时, 抗Cas9 T细胞可以攻击靶细胞[29]。
设计和制备递送系统时需要重点考虑如何维持Cas9的核酸酶活性, 保护RNP抵抗血清和体液中的蛋白酶、核酸酶、抗体和T细胞的识别。一旦进入靶细胞, 递送系统应帮助RNP从内含体快速逃逸到细胞质中, 以便于RNP在Cas9末端NLS的帮助下进入细胞核中。此外, 递送系统上修饰CPPs或NLS也有助于RNP进入细胞核发挥功能。
3 非病毒载体的研究进展 3.1 基于脂质、类脂质的非病毒载体阳离子脂质和类脂质载体可以借助静电相互作用装载核酸, 已被广泛用于microRNA、siRNA和shRNA等的递送, 同理可递送基于质粒DNA、mRNA、RNP的CRISPR/Cas9系统。虽然Cas9蛋白带22个正电荷, 但与sgRNA结合或与带负电荷的绿色荧光蛋白(GFP)融合表达后会阴离子化, 局部注射后, 商用转染试剂Lipofectamine 2000可将RNP递送到小鼠内耳毛细胞中[30]。此外, 很多结构新颖、性能优良的脂质和类脂质材料被设计出来用于载体构建, 这些载体往往需要PEG修饰以屏蔽载体正电荷, 减少蛋白吸附, 提高循环稳定性, 使其达到体内给药的要求。
常用的合成磷脂分子有两条相同的疏水链, 一些非常规的脂质和类脂质也可以高效递送CRISPR/Cas9系统。Li等[31]利用N1, N3, N5-三(2-氨基乙基)苯-1, 3, 5三甲酰胺(TT)的3个活性氨基, 合成了有3个疏水尾巴的类脂质, 建立了一个类脂纳米粒(lipid-like nanoparticles, LLN)库。Jiang等[32]在此基础上对TT3 LLN进行处方优化, 静脉给药后可以消除小鼠肝细胞中的乙型肝炎病毒(HBV), 显著降低前蛋白转化酶枯草溶菌素9 (proprotein convertase subtilisin/kexin type 9, PCSK9)蛋白水平。Dharmalingam等[33]使用食品级棕榈硬脂酸和甘油制备了具有两条不对称疏水链的脂质体。体外实验表明, 相比对称脂质体(P-Lips), 不对称脂质体(PS-Lips)可以更有效地转染pCas。Schuh等[34]用疏水链含有6~12个碳原子的中链甘油三酯(medium chain triglycerides, MCT)制备的阳离子纳米乳以吸附和封装两种方式装载pCRISPR/Cas9和供体DNA, 可以成功转染成纤维细胞, 提高α-艾杜糖醛酸酶(alpha-L-iduronidase, IDUA)的活性, 为治疗Ⅰ型黏多糖病提供了一种可能的方法。
为了提高递送效率, Miller等[35]设计了一类两性离子氨基脂质(zwitterionic amino lipids, ZALs), 制备的纳米粒称为ZNP (ZAL nanoparticles), 其中基于环氧结构的ZAL (ZA3-Ep10)递送效率最高。ZNP递送低剂量(15 nmol·L-1) sgRNA即可将细胞蛋白表达降低超过90%。静脉注射ZNP可对小鼠肝、肾和肺等器官进行基因编辑。为促进载体内含体逃逸, 提高基因编辑效率, Liu等[36]设计、筛选出一种生物还原性脂质BAMEA-O16B (含有3个二硫键), 其制备的脂质纳米粒(lipid nanoparticle, LNP)进入细胞后, 可以响应还原环境迅速释放Cas9 mRNA, 对HEK (人胚胎肾细胞)-EGFP (增强绿色荧光蛋白)细胞EGFP的敲除效率达到90%;静脉注射可以将小鼠血清PCSK9水平敲低至对照组的20%。脂质体或LNP递送RNP的缺点是大多数RNP被吸附在载体表面, 无法保护RNP在全身注射后不受核酸酶降解和免疫识别。将RNP封装在载体内部有利于提高循环稳定性, 也是对载体胞内迅速释放RNP能力的考验, 利用肿瘤细胞胞内环境的特点设计环境响应型载体是解决问题的一个思路。除了递送Cas9 mRNA, Wang等[37]合成并筛选出的还原响应型脂质X-O14B(X代表胺的个数)可以作为RNP的高效载体。Li等[38]改变磷脂的亲水头部并在亲水头和脂肪尾之间引入了酰胺键, 研发出20种安全性良好的还原响应型脂质。
脂质材料和其他材料(如鱼精蛋白、聚合物、金纳米粒等)结合使用可以设计出高效的载体。Zhang等[39]在硫酸软骨素(chondroitin sulfate)的辅助下, 将pCas9/sgRNA与鱼精蛋白(protamine, PS)复合, 得到负电三元核心, 外层包覆阳离子脂质体保护质粒免受核酸降解。最后, 将DSPE-PEG通过后插入法修饰在LNP表面, 得到PLNP (PEG LNP)。瘤内注射pCas9/gPLK-1 PLNP可以破坏A375细胞的PLK-1 (poll样激酶-1), 对肿瘤生长的抑制率达67%。
脂质载体经PEG修饰实现长循环, 通过被动靶向作用可蓄积于肿瘤组织, 对其进行配体修饰或对脂质进行结构改造可以增强载体的靶向性, 促进细胞摄取, 提高递送效率。He等[40]构建了叶酸受体靶向脂质体(F-LP), 体内递送pCRISPR/Cas靶向阻断DNA甲基转移酶1基因, 对紫杉醇敏感和紫杉醇耐药的乳腺癌都有抑制作用。Li等[41]使用了多功能肽R8-dGR (整合素αvβ3和neuropilin-1的配体)修饰阳离子脂质体, 将脂质体和紫杉醇同时静脉注射后, 可阻断缺氧诱导因子-1α, 抑制肿瘤转移, 延长胰腺癌小鼠的生存期。Guo等[42]制备了细胞间黏附分子-1 (intercellular cell adhesion molecule-1, ICAM1)抗体修饰的非阳离子纳米脂质凝胶系统(tumor targeted nanolipogel system, tNLG), 全身给药tNLG可以有效敲除三阴性乳腺癌(TNBC)中的脂质运载蛋白2 (lipocalin 2, Lcn2), 对TNBC的编辑效率达81%, 生长抑制率达77%。
除了修饰主动靶向配体增强载体的主动靶向性, Dong等[43]受脂蛋白启发, 设计了一种脂肽纳米粒(lipopeptide nanoparticles, LPN)用于siRNA的选择性递送。脂肽是以氨基酸、寡肽或多肽作为亲水头部连接脂质的疏水性尾巴得到的新的脂质。在众多脂肽中, cKK-E12对肝细胞显示出极高的特异性, 在动物模型中具有强大的沉默效应。通过微流控技术制备cKK-E12 LPN装载经化学修饰的Cas9 mRNA/gRNA阻断PCSK9[44]。静脉注射Cas9 mRNA/gPCSK9 LNP可显著降低高胆固醇血症模型小鼠的血清胆固醇水平。
类脂化合物在体内无法降解, 会带来严重的不良反应, 设计生物可降解的脂质或类脂化合物可以进一步提高载体安全性。Zhang等[45]设计并合成了一系列生物可降解的类脂化合物, 两个先导材料:具有线性酯链结构的MPA (N-甲基-1, 3丙二胺)-A和具有分枝酯链结构的MPA-Ab制备的LLN可以在体内外高效递送Cas9 mRNA/gRNA用于基因编辑。Finn等[46]合成了一种可电离脂质LP01, 其生物可降解、相容性好。LNP-INT01 (LP01制备的脂质纳米粒)单次给药后可以高效阻断小鼠肝细胞中转甲状腺素蛋白(transthyretin, Ttr)基因, 血清转甲状腺素蛋白水平降低97%, 效果持续时间≥12个月。
以上阳离子脂质和类脂质非病毒载体介导CRISPR系统递送见表 1。
| Table 1 Examples of lipid and lipid-like non-viral vector-mediated CRISPR delivery. DOPE: 2-Dioleoyl-sn-glycero-3-phosphoethanolamine; Chol: Cholesterol; EGFP: Enhanced green fluorescence protein; DMG-PEG: 1, 2-Dimyristoyl-sn-glycerol-methoxypolyethylene glycol; DOTAP: 1, 2-Dioleoyl-sn-glycero-3-trimethylammonium propane; DSPE-PEG: 1, 2-Distearoyl-sn-glycero-3-phosphoethanolamine-N-[methoxy(polyethylene glycol)]; DOPC: 1, 2-Dioleoyl-sn-glycero-3-phosphocholine; DSPC: 1, 2-Dioctadecanoyl-sn-glycero-3-phophocholine |
聚乙烯亚胺(polyethylenimine, PEI)作为基因递送的“金标准”, 可以递送不同大小核酸。研究表明, 支化PEI (25 kDa)可以用作pCas9/sgRNA载体, 支化PEI以细胞毒性较低的N/P比(阳离子聚合物的氮原子与DNA的磷酸基团的浓度比)转染时与脂质体转染的效率没有显著性差异[47]; β-环糊精修饰可以降低PEI (25 kDa)的毒性, 有利于实现多次给药[48]; 除了高分子量PEI, 二油酰磷脂酰乙醇胺(PE)修饰的低分子量PEI (2 kDa)也可以压缩pCas[49]。
PEI也被用于递送RNP, 静电作用是负电物质常用的装载策略, Cho等[50]将PEI和Cas9 RNP混合形成带正电的复合物, 然后将复合物通过静电相互作用装载到卵磷脂脂质体中。静脉注射后可以有效破坏二肽基肽酶-4 (dipeptidyl peptidase-4, DPP-4)基因, 改善db/db小鼠的2型糖尿病(type-2 diabetes, T2D)症状; 通过共价修饰和融合表达使Cas9蛋白获得理想的表面电荷也是RNP递送时常用的方法, Kang等[51]用支化PEI (bPEI)共价修饰Cas9蛋白增强其正电荷, Cas9-bPEI与gRNA混合形成纳米复合物可以逆转细菌耐药性。
上述的PEI递送pCas9研究应用限于细胞水平, 体内递送pCas9需要提高PEI的循环稳定性和肿瘤靶向性: PEG修饰、靶向配体修饰、透明质酸(HA)、环境敏感型外壳包覆和氟化物修饰都是可行的方法。Liang等[52]合成PEG-PEI-胆固醇(PPC)聚合物用于pCas9递送, 骨肉瘤(OS)细胞特异性适体LC09作为靶头, 静脉注射后LC09可以促进pCas9选择性地分布在OS肿瘤中, 载体阻断血管内皮生长因子A (VEGFA), 降低OS恶性程度及其肺转移。研究表明, 用氟化物修饰后阳离子聚合物可以有效地封装核酸[53], 受此启发, Li等[54]设计了一种核壳纳米粒, 以七氟丁酸氟化的PEI (1.8 kDa)压缩质粒作为核心, RGD-R8-PEG-HA作为外壳, HA屏蔽了核心的正电荷, PEG提高了载体稳定性, RGD-R8增强载体靶向性。纳米粒装载pCas9/sgMTH1腹腔给药后对小鼠卵巢癌转移瘤的生长有显著的抑制作用。Liu等[55]用2, 3-二甲基马来酸酐(DMMA)修饰PEG-b-聚赖氨酸得到mPEG-b-PLys/DMMA, 作为环境响应外壳, 包覆苯硼酸(PBA)-PEI, 得到肿瘤组织弱酸性微环境响应型的核壳结构纳米粒。在肿瘤组织酸性环境中, DMMA降解, 外壳表面电荷变正, 离开正电荷核心, PBA通过与肿瘤表面的唾液酸结合增强细胞摄取。该递送系统可以递送pCRISPR-dCas9 (失活Cas9)激活miR-524表达从而抑制MDA-MB-231肿瘤的生长。
此外, 支化富羟基聚阳离子(ARP-F)[56]、聚精氨酸微囊[57]、乙醇胺胺化的聚(甲基丙烯酸缩水甘油酯)[58]、聚(β-氨基酯) (PBAE)[59]、季铵封端的聚环氧丙烷(PPO-NMe3)、两亲性普朗尼克F127自组装成的胶束[60]和刷状半导体聚合物[61]等也被用于pCas9/sgRNA的体内外递送。
聚合物载体递送RNP也取得了一系列进展, Liu等[62]为了找到简便高效的蛋白递送方式, 合成并筛选出PBA修饰的树状大分子P1-P4, 用于包括Cas9 RNP在内共13种蛋白的递送。P4可以通过硼-氮络合、阳离子-π键相互作用、正负电荷间的离子相互作用与蛋白质复合。P4与Cas9 RNP形成直径300 nm左右的单分散纳米粒, 其对人肾上皮细胞(293T)的EGFP可实现40%敲除, 对选取的两个靶位点的编辑效率都可以达到20%左右。单体原位发生自由基聚合反应形成聚合物胶囊封装Cas9 RNP, 有利于解决RNP表面电荷分布不均的问题。Chen等[63]将含咪唑的单体、谷胱甘肽(GSH)敏感的交联剂和PEG可通过氢键和范德华力吸附到RNP的表面, 单体之间发生丙烯酸酯自由基聚合反应, 形成聚合物纳米胶囊。得到的GSH敏感、易修饰的纳米胶囊在细胞摄取后可以保护内含体中Cas9 RNP, 逃逸到细胞质后被GSH迅速降解, 释放RNP发挥基因编辑功能。
Xu等[64]设计的阳离子脂质辅助聚合物纳米粒(cationic lipid-assisted polymeric nanoparticles, CLAN)最初用于siRNA递送, 已证明其能够递送各种核酸, 包括siRNA、miRNA、CpG寡脱氧核苷酸、mRNA和质粒。鉴于阳离子脂质在该递送系统中起到辅助聚合装载核酸的作用, 被归类到基于聚合物的非病毒载体。慢性髓系白血病(CML)是由断点簇区域(BCR)基因与Abelson小鼠白血病病毒癌基因同源物(ABL)基因的融合引起的。基于CRISPR/Cas9切割靶基因形成DSB破坏基因功能的作用机制, Liu等[65]对CML小鼠静脉注射CLAN pCas9/gBCR-ABL有效地阻断了血液和骨髓中CML细胞的BCR-ABL基因, 提高CML小鼠的存活率。
如何提高CLAN递送CRISPR/Cas9的特异性, 研究人员给出了两种解决办法:一是不断优化CLAN的性质, 实现了对中性粒细胞[66]、B细胞[67]、巨噬细胞[68]和树突状细胞[69]的选择性递送; 二是将pCas/sgRNA的启动子更换为细胞特异性的启动子, 例如: Luo等[23]对pCas进行改造, 以CD68启动子替代pX330和pX458的鸡β-肌动蛋白启动子得到pM330和pM458。CD68启动子可以启动巨噬细胞和单核细胞前体细胞中CD68蛋白的表达, 改造后的pCas/sgRNA在巨噬细胞中特异性启动Cas9核酸酶表达, 从而提高基因编辑的特异性, 降低脱靶效应。
3.3 基于囊泡的非病毒载体外泌体是天然形成的囊泡, 具有生物相容性高和免疫原性低的优点, 且已被证明具有细胞定位能力, 可以携带各种生物分子(质粒、siRNA和miRNA)。与来源于上皮细胞的外泌体相比, 来源于肿瘤细胞的外泌体可以借助自身的细胞嗜性高效靶向递送pCas9/sgRNA到肿瘤组织。Kim等[70]纯化了SKOV3 (人卵巢癌细胞)的外泌体(SKOV3-Exo), 静脉注射后SKOV3-Exo选择性地蓄积在SKOV3肿瘤中, 通过阻断聚(ADP-核糖)聚合酶-1 (PARP-1)基因抑制肿瘤的生长。Chen等[71]将HPV或HBV特异性CRISPR/Cas9表达质粒分别转染到HeLa细胞和HuH7细胞中, 发现靶向HPV或HBV的gRNA和Cas9蛋白可以分别封装到两种细胞产生的内源性外泌体中。内源性的外泌体或可用作CRISPR/Cas9系统一种安全有效的递送载体, 但也可能使基因编辑技术的脱靶效应和安全性问题更加复杂。
提高外泌体对CRISPR/Cas9系统的包封率有两种方法:制备杂化外泌体或引入可以富集CRISPR/Cas9的成分, 这些成分可以通过蛋白-RNA、蛋白-蛋白相互作用将mRNA和RNP高效装载到外泌体。Lin等[72]将pCas9和阳离子脂质体复合, 再与外泌体共孵育制备杂化外泌体, 提高了pCas9的负载量, 对于不能单独使用脂质体转染的间充质干细胞也有较好的转染效果。dCas9 mRNA的长度近5 000 nt, 难以通过电穿孔或其他方法封装到外泌体中, Li等[73]将外泌体表面标志物CD9和人抗原R (HuR)融合表达, 在dCas9 mRNA序列中增加3个富AU元件(AU rich element, ARE)。HuR是一种RNA结合蛋白, 与RNA序列中ARE具有高度亲和力, 如此可将dCas9 mRNA和gRNA高效装载到CD9-HuR功能化的外泌体中。静脉给药后, CD9-HuR外泌体可递送dCas9 mRNA和gRNA至肝脏, 实现了基因沉默。Wang等[74]设计了一种功能化的细胞外囊泡(extracellular vesicles, EV), 即抑制蛋白结构域蛋白1 (arrestin domain containing protein 1, ARRDC1)介导的微囊泡(ARMMs)。ARRDC1与WW结构域[约40个氨基酸组成的结构域, 含有两个色氨酸(W)残基]之间存在相互作用, Cas9蛋白与WW结构域(来源于ITCH蛋白)融合后可以被募集到ARMM。结果表明, ARMM可以显著减少EGFP阳性细胞, 除了递送Cas9 RNP, 还可以递送蛋白和RNA。
此外, 来源于红细胞(red blood cells, RBC)的EV可以用于基于质粒和RNA的CRISPR/Cas9转染, 而且对Cas9 mRNA/gRNA的递送更高效[75]; Campbell等[76]制备纳米囊泡(gesicles)时, 将水疱性口炎病毒G (VSV-G)和红色荧光蛋白(cherry picker red)质粒转染到293T细胞, 可以促进囊泡的形成及Cas9 RNP的装载。
3.4 基于金纳米粒的非病毒载体金纳米粒(AuNPs)与带巯基(-SH)的物质很容易通过Au-S键交联, 从而改变其表面电荷、亲水性和靶向性, 已被用于pCas9和RNP的递送。
AuNPs表面修饰阳离子肽后, 可以通过静电作用吸附pCas9, Wang等[77]将具有细胞核靶向能力的TAT肽(C端为半胱氨酸)修饰在AuNPs的表面, pCas9通过静电相互作用被压缩在AuNPs的表面, 外层包覆PEG修饰的阳离子脂质体。静脉给药后, 载体蓄积在黑色素瘤中, 进入细胞后, pCas9通过AuNPs的激光触发的热效应释放, 阳离子TAT肽可以引导pCas9/sgPLK-1进入细胞核, 破坏PLK-1基因以抑制肿瘤生长。
与质粒相比, Cas9 RNP表面电荷分布更为复杂, 将正电荷单体、负电荷单体吸附到RNP表面后形成原位聚合纳米胶囊[63]是解决问题的一种策略, Mout等[78]提出另一种思路, 即增强RNP的负电荷从而增强RNP和AuNPs之间的静电作用。他们将谷氨酸多肽(Etag)连接到Cas9的N端来增强负电荷, 将NLS连接到C端促进Cas9入核, 得到Cas9En (n为谷氨酸个数)可以通过氢键/静电相互作用与精氨酸化的AuNPs (ArgNPs)形成纳米组装体。该纳米组装体可在HeLa细胞上进行跨膜转移, 然后将Cas9En RNP递送到细胞核中。
AuNPs表面修饰寡核苷酸后, 可以利用Cas9核酸酶对特定DNA和crRNA的亲和力将CRISPR/Cas9系统装载到AuNPs。Lee等[79]设计了CRISPR-Gold, AuNPs表面偶联硫醇寡核苷酸(DNA-Thiol), DNA-Thiol可与供体ssDNA杂交, 通过ssDNA与Cas9核酸酶的亲和力装载Cas9 RNP, 外层包覆可以破坏细胞内含体的阳离子聚合物PAsp (DET), 肌内注射后可以修复突变的肌营养不良蛋白基因, 减少mdx小鼠的肌肉纤维化。颅内注射后CRISPR-Gold还可以编辑脑中大多数类型的细胞[80]。Shahbazi等[81]将AuNPs核心与SH-间隔子[18个单体的寡聚乙二醇(OEG)]-crRNA偶联, Cas9通过与crRNA天然的亲和力装载到纳米粒, 复合物用PEI包覆, 供体ssDNA通过静电相互作用吸附在表面, 体外条件下载体可以在造血干细胞和祖细胞(HSPCs)中实现了靶向HDR。
3.5 基于多肽、蛋白的非病毒载体功能性的阳离子多肽CPP和NLS肽已被证明可有效辅助核酸的递送[82]。CPPs可以通过共价连接、静电吸附与Cas9蛋白、gRNA形成阳离子纳米粒, 在多种细胞系实现高效基因阻断[83]。内含体溶解肽(ppTG21)通过静电作用吸附在唾液酸糖蛋白受体配体(ASGPrL)修饰的Cas9 RNP外层, 可以促进Cas9-ASGPrL RNP的内含体逃逸[84]。Cas9与低分子量鱼精蛋白、NLS融合表达后与crRNA/tracrRNA形成三复合物, 天然穿膜肽鱼精蛋白和NLS可以介导复合物进入细胞和细胞核, 瘤内注射该三元复合物可以破坏KRAS基因, 抑制小鼠A549肿瘤的生长[85]。鱼精蛋白通过静电吸附装载pCas9/sgRNA, 外层包覆内溶肽(KALA)和适体AS1411修饰的羧甲基壳聚糖, 得到的多功能纳米粒可以将肿瘤细胞中周期蛋白依赖性激酶11 (cyclin-dependent kinases11, CDK11)表达减少了75%以上, 并进一步调节其他肿瘤发展、转移和免疫相关蛋白的表达, 有利于逆转肿瘤免疫抑制和肿瘤的发展[86]。
其他具有螺旋结构和正电荷的多肽, 如: α-螺旋结构阳离子多肽PPABLG[21]、Endo-Porter (EP)肽[87]、两亲性R7L10肽[88]、两亲性穿膜肽[89]递送pCas9或RNP也可以促进载体内吞和内含体逃逸, 提高递送效率和基因编辑效率。
运用生物工程手段或仿生技术装载Cas9蛋白和pCas9也是一种递送策略。受噬菌体P22笼状结构的启发, Qazi等[90]制备了病毒样颗粒(VLP)装载RNP作为细胞特异性递送系统, 体外与质粒共孵育后可以切割目标双链DNA。受染色体结构的启发, Killian等[91]通过盐梯度透析法将pCas9与组蛋白组装在一起, 制备了人工染色质, 染色质和双特异性抗体连接后可在抗体介导下高效地将pCas9转染到细胞中, 转染效率达90%。
正电荷蛋白可以通过静电作用吸附质粒或RNP, 负电荷蛋白通过正电荷聚合物改性后也可用于递送CRISPR/Cas9系统。负电荷红色荧光蛋白(red fluorescence protein, RFP)被阳离子聚合物壳聚糖(chitosan, CS)包覆后和Cas9 RNP与供体ssDNA通过静电相互作用自组装成较大的纳米组装体, 可以被多种细胞内吞并发挥基因编辑功能[92]。
3.6 其他非病毒载体沸石咪唑酯骨架(zeolitic imidazolate framework, ZIF)生物相容性好、孔径可调节, 适于递送Cas9蛋白。Alsaiari等[93]使用ZIF-8封装Cas9 RNP, 酸性条件(pH 5.0或6.0)下, Cas9 RNP从ZIF-8中快速释放, 有利于内含体逃逸; 给药4天后表达EGFP的中国仓鼠卵巢细胞(EGFP-CHO)荧光强度降低37%。Yang等[94]设计了一种ATP响应的ZIF-90, ZIF-90可以封装包括GFP、RNase A-NBC和Cas9在内的多种蛋白, 并且这些蛋白可以在ATP存在时迅速释放, 递送Cas9 RNP到HeLa细胞, GFP敲除效率达35%。
AuNPs递送系统可借助供体DNA与Cas9蛋白的亲和力装载RNP[79], DNA也可以直接装载RNP。Sun等[95]通过滚环扩增(RCA)合成了DNA纳米线团(NC), NC与sgRNA通过碱基互补配对完成Cas9 RNP的装载, 复合物的外层包覆PEI, 以增强内含体逃逸。NC可以高效地递送Cas9 RNP到U2OS (人骨肉瘤细胞)中, 实现EGFP敲除; 瘤内注射后NC对U2OS-EGFP荷瘤小鼠的EGFP敲除率达25%。
控制释放递送系统可以在特定区域快速释放装载的药物或基因, 有助于提高递送效率, 减少脱靶。已有研究人员将上转换纳米粒(UCNPs)用于Cas9 RNP的递送, 近红外(NIR)光照射后, UCNPs-Cas9@PEI局部发射紫外光触发4-(羟甲基)-3-硝基苯甲酸(ONA)裂解, 从而释放Cas9 RNP。瘤内注射靶向PLK-1的UCNPs-Cas9@PEI可以敲除PLK-1基因, 抑制小鼠A549肿瘤的生长[96]。Zhu等[97]制备了一种DNA酶控制的CRISPR/Cas9纳米递送系统。Mn2+存在时, Cas9 RNP可以通过DNA酶介导的底物DNA断裂完成释放过程。结果表明, 该纳米系统可以将Cas9 RNP递送到细胞核, 实现基因编辑。
二维材料如:氧化石墨烯(graphene oxide, GO)和黑磷(black phosphorus, BP)纳米片也可用于Cas9蛋白的递送。GO具有良好的生物相容性和安全性, 经PEG和PEI修饰后通过物理吸附和π-堆积力装载Cas9 RNP。GO-PEG-PEI可以在保留Cas9活性的同时将RNP快速转运到人源细胞中, 基因编辑效率达39%[98]。BP通过静电吸附装载Cas9 RNP, 在内含体酸性环境下可以迅速降解为高度生物相容的无机磷酸盐, 实现由渗透压改变所引起的内含体破裂, 释放Cas9 RNP, 该递送系统可在较低浓度下对不同细胞株及动物荷瘤模型实现高效的基因编辑和基因沉默[99]。
4 总结CRISPR/Cas9系统的出现简化了基因编辑的步骤, 给基础研究和临床基因疾病、癌症治疗带来了便利和希望, 但面临问题也有很多, 如递送效率低、脱靶效应和免疫反应, 这些问题的解决依赖于研究的不断深入和发展。CRISPR/Cas9系统的递送方式有许多, 但物理方法不适用于体内; 病毒载体装载能力有限、免疫原性高、有致癌风险; 非病毒载体包载能力强, 免疫原性低, 易于组装, 虽然递送效率较低, 但它是最可能用于体内给药的递送方式。因此, 许多研究人员都致力于设计开发安全高效的非病毒递送系统。CRISPR/Cas9的递送本质上是核酸、蛋白质或核糖核蛋白递送的一个综合领域, 此前核酸和蛋白质递送载体的发展为此提供重要参考和经验, CRISPR/Cas9的递送为日渐完善的递送系统设计理论提供了用武之地。目前很多非病毒载体都是基于不同类别材料的杂合体, 综合各种材料的优势提高递送效率, 阳离子脂质、阳离子聚合物、多肽、蛋白、金纳米粒、囊泡、金属材料和DNA纳米线团等递送系统已经在体内、体外研究中取得了一系列进展。这些非病毒载体借助静电吸附、范德华力、氢键和共价键装载CRISPR/Cas9系统, 为适应全身给药, 载体需要修饰PEG或包覆其他负电材料(如HA、环境响应性外壳)以增强载体循环稳定性, 局部给药的基因编辑范围局限, 安全性较好, 不会引发大规模的脱靶效应, 由于无需做到长循环, 载体外层可以包覆PEI、PAsp (DET)等材料促进细胞摄取和内含体逃逸。CRISPR/Cas9技术具有广阔的应用前景, 非病毒载体递送效率的不断提高必将助力CRISPR/Cas9技术的临床转化, 为更多患者缓解甚至治愈疾病。
作者贡献: 张学农提出构想; 王钰负责整理文献、撰写初稿; 黄归负责检索文献、修改手稿; 杨菡确定手稿框架。所有作者参与手稿的阅读与修改。
利益冲突: 作者声明不存在利益冲突。
| [1] |
Gaj T, Gersbach CA, Barbas CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends Biotechnol, 2013, 31: 397-405. DOI:10.1016/j.tibtech.2013.04.004 |
| [2] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2015, 13: 722-736. DOI:10.1038/nrmicro3569 |
| [3] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315: 1709-1712. DOI:10.1126/science.1138140 |
| [4] |
Fineran PC, Charpentier E. Memory of viral infections by CRISPR-Cas adaptive immune systems:acquisition of new information[J]. Virology, 2012, 434: 202-209. DOI:10.1016/j.virol.2012.10.003 |
| [5] |
Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327: 167-170. DOI:10.1126/science.1179555 |
| [6] |
Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482: 331-338. DOI:10.1038/nature10886 |
| [7] |
John VDO, Westra ER, Jackson RN, et al. Unravelling the structural and mechanistic basis of CRISPR-Cas systems[J]. Nat Rev Microbiol, 2014, 12: 479-492. DOI:10.1038/nrmicro3279 |
| [8] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337: 816-821. DOI:10.1126/science.1225829 |
| [9] |
Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339: 823-826. DOI:10.1126/science.1232033 |
| [10] |
Sung P, Klein H. Mechanism of homologous recombination:mediators and helicases take on regulatory functions[J]. Nat Rev Mol Cell Biol, 2006, 7: 739-750. DOI:10.1038/nrm2008 |
| [11] |
Lu D, Li L, Deng XM. CRISPR/Cas9 and its application in drugs development[J]. Acta Pharm Sin (药学学报), 2018, 53: 11-20. |
| [12] |
Wells DJ. Gene therapy progress and prospects:electroporation and other physical methods[J]. Gene Ther, 2004, 11: 1363-1369. DOI:10.1038/sj.gt.3302337 |
| [13] |
Maurisse R, Semir DD, Emamekhoo H, et al. Comparative transfection of DNA into primary and transformed mammalian cells from different lineages[J]. BMC Biotechnol, 2010, 10: 1-9. |
| [14] |
Yin H, Kanasty RL, Eltoukhy AA, et al. Non-viral vectors for gene-based therapy[J]. Nat Rev Genet, 2014, 15: 541-555. DOI:10.1038/nrg3763 |
| [15] |
Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8: 2281-2308. DOI:10.1038/nprot.2013.143 |
| [16] |
Fritz J, Cooper EB, Gaudet S, et al. Electronic detection of DNA by its intrinsic molecular charge[J]. Proc Natl Acad Sci U S A, 2002, 99: 14142-14146. DOI:10.1073/pnas.232276699 |
| [17] |
Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9[J]. Nature, 2015, 520: 186-191. DOI:10.1038/nature14299 |
| [18] |
Hughes TS, Langer SJ, Virtanen SI, et al. Immunogenicity of intrathecal plasmid gene delivery:cytokine release and effects on transgene expression[J]. J Gene Med, 2009, 11: 782-790. DOI:10.1002/jgm.1364 |
| [19] |
Brandén LJ, Mohamed A, Smith CIE. A peptide nucleic acid-nuclear localization signal fusion that mediates nuclear transport of DNA[J]. Nat Biotechnol, 1999, 17: 784-787. DOI:10.1038/11726 |
| [20] |
Copolovici DM, Langel K, Eriste E, et al. Cell-penetrating peptides:design, synthesis, and applications[J]. ACS Nano, 2014, 8: 1972-1994. DOI:10.1021/nn4057269 |
| [21] |
Wang HX, Song ZY, Lao YH, et al. Nonviral gene editing via CRISPR/Cas9 delivery by membrane-disruptive and endosomolytic helical polypeptide[J]. Proc Natl Acad Sci U S A, 2018, 115: 4903-4908. DOI:10.1073/pnas.1712963115 |
| [22] |
Hemmi H, Takeuchi O, Kawai T, et al. A Toll-like receptor recognizes bacterial DNA[J]. Nature, 2000, 408: 740-745. DOI:10.1038/35047123 |
| [23] |
Luo YL, Xu CF, Li HJ, et al. Macrophage-specific in vivo gene editing using cationic lipid-assisted polymeric nanoparticles[J]. ACS Nano, 2018, 12: 994-1005. DOI:10.1021/acsnano.7b07874 |
| [24] |
Vaidyanathan S, Azizian KT, Haque A, et al. Uridine depletion and chemical modification increase Cas9 mRNA activity and reduce immunogenicity without HPLC purification[J]. Mol Ther Nucleic Acids, 2018, 12: 530-542. DOI:10.1016/j.omtn.2018.06.010 |
| [25] |
Anderson BR, Muramatsu H, Jha BK, et al. Nucleoside modifications in RNA limit activation of 2'-5'-oligoadenylate synthetase and increase resistance to cleavage by RNase L[J]. Nucleic Acids Res, 2011, 39: 9329-9338. DOI:10.1093/nar/gkr586 |
| [26] |
Bak RO, Hendel A, Clark JT, et al. Chemically modified guide RNAs enhance CRISPR-Cas genome editing in human primary cells[J]. Hum Gene Ther, 2015, 26: A11-A12. |
| [27] |
Kim S, Kim D, Cho SW, et al. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins[J]. Genome Res, 2014, 24: 1012-1019. DOI:10.1101/gr.171322.113 |
| [28] |
Charlesworth CT, Deshpande PS, Dever DP, et al. Identification of preexisting adaptive immunity to Cas9 proteins in humans[J]. Nat Med, 2019, 25: 249-254. DOI:10.1038/s41591-018-0326-x |
| [29] |
Crudele JM, Chamberlain JS. Cas9 immunity creates challenges for CRISPR gene editing therapies[J]. Nat Commun, 2018, 9: 3497. DOI:10.1038/s41467-018-05843-9 |
| [30] |
Zuris JA, Thompson DB, Shu Y, et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo[J]. Nat Biotechnol, 2015, 33: 73-80. DOI:10.1038/nbt.3081 |
| [31] |
Li B, Luo X, Deng BB, et al. An orthogonal array optimization of lipid-like nanoparticles for mRNA delivery in vivo[J]. Nano Lett, 2015, 15: 8099-8107. DOI:10.1021/acs.nanolett.5b03528 |
| [32] |
Jiang C, Mei M, Li B, et al. A non-viral CRISPR/Cas9 delivery system for therapeutically targeting HBV DNA and pcsk9 in vivo[J]. Cell Res, 2017, 27: 440-443. DOI:10.1038/cr.2017.16 |
| [33] |
Dharmalingam P, Rachamalla HKR, Lohchania B, et al. Green transfection:cationic lipid nanocarrier system derivatized from vegetable fat, palmstearin enhances nucleic acid transfections[J]. ACS Omega, 2017, 2: 7892-7903. DOI:10.1021/acsomega.7b00935 |
| [34] |
Schuh RS, de Carvalhob TG, Giugliani R, et al. Gene editing of MPS I human fibroblasts by co-delivery of a CRISPR/Cas9 plasmid and a donor oligonucleotide using nanoemulsions as nonviral carriers[J]. Eur J Pharm Biopharm, 2018, 122: 158-166. DOI:10.1016/j.ejpb.2017.10.017 |
| [35] |
Miller JB, Zhang SY, Kos P, et al. Non-viral CRISPR/Cas gene editing in vitro and in vivo enabled by synthetic nanoparticle co-delivery of Cas9 mRNA and sgRNA[J]. Angew ChemInt Ed, 2017, 56: 1059-1063. DOI:10.1002/anie.201610209 |
| [36] |
Liu J, Chang J, Jiang Y, et al. Fast and efficient CRISPR/Cas9 genome editing in vivo enabled by bioreducible lipid and messenger RNA nanoparticles[J]. Adv Mater, 2019, 31: e1902575. DOI:10.1002/adma.201902575 |
| [37] |
Wang M, Zuris JA, Meng F, et al. Efficient delivery of genome-editing proteins using bioreducible lipid nanoparticles[J]. Proc Natl Acad Sci U S A, 2016, 113: 2868-2873. DOI:10.1073/pnas.1520244113 |
| [38] |
Li Y, Justin B, Yingjie Y, et al. Intracellular delivery and biodistribution study of CRISPR/Cas9 ribonucleoprotein loaded bioreducible lipidoid nanoparticles[J]. Biomater Sci, 2019, 7: 596-606. DOI:10.1039/C8BM00637G |
| [39] |
Zhang LM, Wang P, Feng Q, et al. Lipid nanoparticle-mediated efficient delivery of CRISPR/Cas9 for tumor therapy[J]. NPG Asia Mater, 2017, 9: e441. DOI:10.1038/am.2017.185 |
| [40] |
He ZY, Zhang YG, Yang YH, et al. In vivo ovarian cancer gene therapy using CRISPR-Cas9 system[J]. Hum Gene Ther, 2018, 29: 223-233. DOI:10.1089/hum.2017.209 |
| [41] |
Li M, Xie HB, Liu YK, et al. Knockdown of hypoxia-inducible factor-1 alpha by tumor targeted delivery of CRISPR/Cas9 system suppressed the metastasis of pancreatic cancer[J]. J Control Release, 2019, 304: 204-215. DOI:10.1016/j.jconrel.2019.05.019 |
| [42] |
Guo P, Yang J, Huang J, et al. Therapeutic genome editing of triple-negative breast tumors using a noncationic and deformable nanolipogel[J]. Proc Natl Acad Sci U S A, 2019, 116: 18295-18303. DOI:10.1073/pnas.1904697116 |
| [43] |
Dong YZ, Love KT, Dorkin JR, et al. Lipopeptide nanoparticles for potent and selective siRNA delivery in rodents and nonhuman primates[J]. Proc Natl Acad Sci U S A, 2014, 111: 3955-3960. DOI:10.1073/pnas.1322937111 |
| [44] |
Yin H, Song CQ, Suresh S, et al. Structure-guided chemical modi-fication of guide RNA enables potent non-viral in vivo genome editing[J]. Nat Biotechnol, 2017, 35: 1179-1187. DOI:10.1038/nbt.4005 |
| [45] |
Zhang XF, Li B, Luo X, et al. Biodegradable amino-ester nanomaterials for Cas9 mRNA delivery in vitro and in vivo[J]. ACS Appl Mater Interfaces, 2017, 9: 25481-25487. DOI:10.1021/acsami.7b08163 |
| [46] |
Finn JD, Smith AR, Patel MC, et al. A single administration of CRISPR/Cas9 lipid nanoparticles achieves robust and persistent in vivo genome editing[J]. Cell Rep, 2018, 22: 2227-2235. DOI:10.1016/j.celrep.2018.02.014 |
| [47] |
Ryu N, Kim MA, Park D, et al. Effective PEI-mediated delivery of CRISPR-Cas9 complex for targeted gene therapy[J]. Nanomedicine, 2018, 14: 2095-2102. DOI:10.1016/j.nano.2018.06.009 |
| [48] |
Zhang Z, Wan T, Chen Y, et al. Cationic polymer-mediated CRISPR/Cas9 plasmid delivery for genome editing[J]. Macromol Rapid Commun, 2018, 40: 1800068. |
| [49] |
Yan M, Wen J, Liang M, et al. Modulation of gene expression by polymer nanocapsule delivery of DNA cassettes encoding small RNAs[J]. PLoS One, 2015, 10: e0127986. DOI:10.1371/journal.pone.0127986 |
| [50] |
Cho EY, Ryu JY, Lee HAR, et al. Lecithin nano-liposomal particle as a CRISPR/Cas9 complex delivery system for treating type 2 diabetes[J]. J Nanobiotechnol, 2019, 17: 19. DOI:10.1186/s12951-019-0452-8 |
| [51] |
Kang YK, Kwon K, Ryu JS, et al. Nonviral genome editing based on a polymer-derivatized CRISPR nanocomplex for targeting bacterial pathogens and antibiotic resistance[J]. Bioconjugate Chem, 2017, 28: 957-967. DOI:10.1021/acs.bioconjchem.6b00676 |
| [52] |
Liang C, Li FF, Wang LY, et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma[J]. Biomaterials, 2017, 147: 68-85. DOI:10.1016/j.biomaterials.2017.09.015 |
| [53] |
Wang MM, Liu HM, Li L, et al. A fluorinated dendrimer achieves excellent gene transfection efficacy at extremely low nitrogen to phosphorus ratios[J]. Nat Commun, 2014, 5: 3053. DOI:10.1038/ncomms4053 |
| [54] |
Li L, Song LJ, Liu XW, et al. Artificial virus delivers CRISPR-Cas9 system for genome editing of cells in mice[J]. ACS Nano, 2017, 11: 95-111. DOI:10.1021/acsnano.6b04261 |
| [55] |
Liu Q, Zhao K, Wang C, et al. Multistage delivery nanoparticle facilitates efficient CRISPR/dCas9 activation and tumor growth suppression in vivo[J]. Adv Sci, 2019, 6: 1801423. DOI:10.1002/advs.201801423 |
| [56] |
Qi Y, Song HQ, Xiao HH, et al. Fluorinated acid-labile branched hydroxyl-rich nanosystems for flexible and robust delivery of plasmids[J]. Small, 2018, 14: e1803061. DOI:10.1002/smll.201803061 |
| [57] |
Timin AS, Muslimov AR, Lepik KV, et al. Efficient gene editing via non-viral delivery of CRISPR-Cas9 system using polymeric and hybrid microcarriers[J]. Nanomedicine, 2018, 14: 97-108. DOI:10.1016/j.nano.2017.09.001 |
| [58] |
Zhang XP, Xu C, Gao SJ, et al. CRISPR/Cas9 delivery mediated with hydroxyl-rich nanosystems for gene editing in aorta[J]. Adv Sci, 2019, 6: 1900386. DOI:10.1002/advs.201900386 |
| [59] |
Zhu D, Shen H, Tan SW, et al. Nanoparticles based on poly (β-amino ester) and HPV16-targeting CRISPR/shRNA as potential drugs for HPV16-related cervical malignancy[J]. Mol Ther, 2018, 26: 2443-2455. DOI:10.1016/j.ymthe.2018.07.019 |
| [60] |
Lao YH, Li MQ, Gao MA, et al. HPV oncogene manipulation using nonvirally delivered CRISPR/Cas9 or Natronobacterium gregoryi argonaute[J]. Adv Sci, 2018, 5: 1700540. DOI:10.1002/advs.201700540 |
| [61] |
Li L, Yang Z, Zhu SJ, et al. A rationally designed semiconducting polymer brush for NIR-Ⅱ imaging-guided light-triggered remote control of CRISPR/Cas9 genome editing[J]. Adv Mater, 2019, 31: 1901187. DOI:10.1002/adma.201901187 |
| [62] |
Liu CY, Wan T, Wang H, et al. A boronic acid-rich dendrimer with robust and unprecedented efficiency for cytosolic protein delivery and CRISPR-Cas9 gene editing[J]. Sci Adv, 2019, 5: eaaw8922. DOI:10.1126/sciadv.aaw8922 |
| [63] |
Chen GJ, Abdeen AA, Wang YY, et al. A biodegradable nanocapsule delivers a Cas9 ribonucleoprotein complex for in vivo genome editing[J]. Nat Biotechnol, 2019, 14: 974-980. |
| [64] |
Xu CF, Iqbal S, Shen S, et al. Development of "CLAN" nanomedicine for nucleic acid therapeutics[J]. Small, 2019, 15: e1900055. DOI:10.1002/smll.201900055 |
| [65] |
Liu Y, Zhao G, Xu CF, et al. Systemic delivery of CRISPR/Cas9 with PEG-PLGA nanoparticles for chronic myeloid leukemia targeted therapy[J]. Biomater Sci, 2018, 6: 1592-1603. DOI:10.1039/C8BM00263K |
| [66] |
Liu Y, Cao ZT, Xu CF, et al. Optimization of lipid-assisted nanoparticle for disturbing neutrophils-related inflammation[J]. Biomaterials, 2018, 172: 92-104. DOI:10.1016/j.biomaterials.2018.04.052 |
| [67] |
Li M, Fan YN, Chen ZY, et al. Optimized nanoparticle-mediated delivery of CRISPR-Cas9 system for B cell intervention[J]. Nano Res, 2018, 11: 6270-6282. DOI:10.1007/s12274-018-2150-5 |
| [68] |
Xu CF, Lu ZD, Luo YL, et al. Targeting of NLRP3 inflammasome with gene editing for the amelioration of inflammatory diseases[J]. Nat Commun, 2018, 9: 4092. DOI:10.1038/s41467-018-06522-5 |
| [69] |
Zhang Y, Shen S, Zhao G, et al. In situ repurposing of dendritic cells with CRISPR/Cas9-based nanomedicine to induce transplant tolerance[J]. Biomaterials, 2019, 217: 119302. DOI:10.1016/j.biomaterials.2019.119302 |
| [70] |
Kim SM, Yang Y, Oh SJ, et al. Cancer-derived exosomes as a delivery platform of CRISPR/Cas9 confer cancer cell tropism-dependent targeting[J]. J Control Release, 2017, 266: 8-16. DOI:10.1016/j.jconrel.2017.09.013 |
| [71] |
Chen R, Huang HX, Liu H, et al. Gene editing:friend or foe? Evidence indicates endogenous exosomes can deliver functional gRNA and Cas9 protein[J]. Small, 2019, 5: 1902686. |
| [72] |
Lin Y, Wu JH, Gu WH, et al. Exosome-liposome hybrid nanoparticles deliver CRISPR/Cas9 system in MSCs[J]. Adv Sci, 2018, 5: 1700611. DOI:10.1002/advs.201700611 |
| [73] |
Li ZL, Zhou XY, Wei MY, et al. In vitro and in vivo RNA inhibition by CD9-HuR functionalized exosomes encapsulated with miRNA or CRISPR/dCas9[J]. Nano Lett, 2019, 19: 19-28. DOI:10.1021/acs.nanolett.8b02689 |
| [74] |
Wang QY, Yu JJ, Kadungure T, et al. ARMMs as a versatile platform for intracellular delivery of macromolecules[J]. Nat Commun, 2018, 9: 960. DOI:10.1038/s41467-018-03390-x |
| [75] |
Usman WM, Pham TC, Kwok YY, et al. Efficient RNA drug delivery using red blood cell extracellular vesicles[J]. Nat Commun, 2018, 9: 2359. DOI:10.1038/s41467-018-04791-8 |
| [76] |
Campbell LA, Coke LM, Richie CT, et al. Gesicle-mediated delivery of CRISPR/Cas9 ribonucleoprotein complex for inactivating the HIV provirus[J]. Mol Ther, 2019, 27: 151-163. DOI:10.1016/j.ymthe.2018.10.002 |
| [77] |
Wang P, Zhang LM, Zheng WF, et al. Thermo-triggered release of CRISPR-Cas9 system by lipid-encapsulated gold nanoparticles for tumor therapy[J]. Angew Chem Int Ed, 2018, 57: 1491-1496. DOI:10.1002/anie.201708689 |
| [78] |
Mout R, Ray M, Tonga GY, et al. Direct cytosolic delivery of CRISPR/Cas9 ribonucleoprotein for efficient gene editing[J]. ACS Nano, 2017, 11: 2452-2458. DOI:10.1021/acsnano.6b07600 |
| [79] |
Lee K, Conboy M, Park HM, et al. Nanoparticle delivery of Cas9 ribonucleoprotein and donor DNA in vivo induces homology-directed DNA repair[J]. Nat Biomed Eng, 2017, 1: 889-901. DOI:10.1038/s41551-017-0137-2 |
| [80] |
Lee B, Lee K, Panda S, et al. Nanoparticle delivery of CRISPR into the brain rescues a mouse model of fragile X syndrome from exaggerated repetitive behaviours[J]. Nat Biomed Eng, 2018, 2: 497-507. DOI:10.1038/s41551-018-0252-8 |
| [81] |
Shahbazi R, Sghia-Hughes G, Reid JL, et al. Targeted homology-directed repair in blood stem and progenitor cells with CRISPR nanoformulations[J]. Nat Mater, 2019, 18: 1124-1132. DOI:10.1038/s41563-019-0385-5 |
| [82] |
Lehto T, Ezzat K, Wood MJA, et al. Peptides for nucleic acid delivery[J]. Adv Drug Deliv Rev, 2016, 106: 172-182. DOI:10.1016/j.addr.2016.06.008 |
| [83] |
Ramakrishna S, Dad ABK, Beloor J, et al. Gene disruption by cell-penetrating peptide-mediated delivery of Cas9 protein and guide RNA[J]. Genome Res, 2014, 24: 1020-1027. DOI:10.1101/gr.171264.113 |
| [84] |
Rouet R, Thuma BA, Roy MD, et al. Receptor-mediated delivery of CRISPR-Cas9 endonuclease for cell type specific gene editing[J]. J Am Chem Soc, 2018, 140: 6596-6603. DOI:10.1021/jacs.8b01551 |
| [85] |
Kim SM, Shin SC, Kim EE, et al. Simple in vivo gene editing via direct self-assembly of Cas9 ribonucleoprotein complexes for cancer treatment[J]. ACS Nano, 2018, 12: 7750-7760. DOI:10.1021/acsnano.8b01670 |
| [86] |
Liu BY, He XY, Zhuo RX, et al. Tumor targeted genome editing mediated by a multi-functional gene vector for regulating cell behaviors[J]. J Control Release, 2018, 291: 90-98. DOI:10.1016/j.jconrel.2018.10.018 |
| [87] |
Shen YF, Cohen JL, Nicoloro SM, et al. CRISPR-delivery particles targeting nuclear receptor-interacting protein 1(Nrip1) in adipose cells to enhance energy expenditure[J]. J Biol Chem, 2018, 293: 17291-17305. DOI:10.1074/jbc.RA118.004554 |
| [88] |
Park H, Oh JJ, Shim GY, et al. In vivo neuronal gene editing via CRISPR-Cas9 amphiphilic nanocomplexes alleviates deficits in mouse models of Alzheimer's disease[J]. Nat Neurosci, 2019, 22: 524-528. DOI:10.1038/s41593-019-0352-0 |
| [89] |
Lostalé-Seijo I, Louzao I, Juanes M, et al. Peptide/Cas9 nanostructures for ribonucleoprotein cell membrane transport and gene edition[J]. Chem Sci, 2017, 8: 7883-8466. DOI:10.1039/C7SC90074K |
| [90] |
Qazi S, Miettinen HM, Wilkinson RA, et al. Programmed self-assembly of an active P22-Cas9 nano carrier system[J]. Mol Pharm, 2016, 13: 1191-1196. DOI:10.1021/acs.molpharmaceut.5b00822 |
| [91] |
Killian T, Buntz A, Herlet T, et al. Antibody-targeted chromatin enables effective intracellular delivery and functionality of CRISPR/Cas9 expression plasmids[J]. Nucleic Acids Res, 2019, 47: e55. DOI:10.1093/nar/gkz137 |
| [92] |
Qiao J, Sun WL, Lin SY, et al. Cytosolic delivery of CRISPR/Cas9 ribonucleoproteins for genome editing using chitosan-coated red fluorescent protein[J]. Chem Commun, 2019, 55: 4707-4710. DOI:10.1039/C9CC00010K |
| [93] |
Alsaiari SK, Patil S, Alyami M, et al. Endosomal escape and delivery of CRISPR/Cas9 genome editing machinery enabled by nanoscale zeolitic imidazolate framework[J]. J Am Chem Soc, 2018, 140: 143-146. DOI:10.1021/jacs.7b11754 |
| [94] |
Yang XT, Tang Q, Jiang Y, et al. Nanoscale ATP-responsive zeolitic imidazole framework-90 as a general platform for cytosolic protein delivery and genome editing[J]. J Am Chem Soc, 2019, 141: 3782-3786. DOI:10.1021/jacs.8b11996 |
| [95] |
Sun WJ, Ji WY, Hall JM, et al. Self-assembled DNA nanoclews for the efficient delivery of CRISPR-Cas9 for genome editing[J]. Angew Chem Int Ed, 2015, 54: 12029-12033. DOI:10.1002/anie.201506030 |
| [96] |
Pan YC, Yang JJ, Luan XW, et al. Near-infrared upconversion-activated CRISPR-Cas9 system:a remote-controlled gene editing platform[J]. Sci Adv, 2019, 5: eaav7199. DOI:10.1126/sciadv.aav7199 |
| [97] |
Zhu XL, Lv MM, Liu JW, et al. DNAzyme activated protein-scaffolded CRISPR-Cas9 nanoassembly for genome editing[J]. Chem Commun, 2019, 55: 6511-6514. DOI:10.1039/C9CC03172C |
| [98] |
Yue HH, Zhou XM, Cheng M, et al. Graphene oxide-mediated Cas9/sgRNA delivery for efficient genome editing[J]. Nanoscale, 2018, 10: 1063-1071. DOI:10.1039/C7NR07999K |
| [99] |
Zhou W, Cui H, Ying L, et al. Enhanced cytosolic delivery and releases of CRISPR/Cas9 by black phosphorus nanosheets for genome editing[J]. Angew Chem Int Ed, 2018, 57: 10268-10272. DOI:10.1002/anie.201806941 |
 2020, Vol. 55
2020, Vol. 55


