2. Department of Systems Biology, the University of Texas MD Anderson Cancer Center, Houston, TX 77030, USA
2. Department of Systems Biology, the University of Texas MD Anderson Cancer Center, Houston, TX 77030, USA
多种内源性和外源性因素可不断诱发DNA损伤, 如细胞代谢产生的活性氧、紫外线和遗传毒性化学物质等。为应对基因组完整性受到的威胁, 细胞具有一系列复杂的信号网络, 称为“DNA损伤应答(DNA damage response, DDR)”[1]。DNA损伤应答检测DNA损伤, 启动检查点并阻滞细胞周期, 同时激活特定的修复通路。当DNA损伤的程度超过其修复能力时, DNA损伤应答将激活衰老或凋亡信号通路, 清除受损的细胞[2]。
精确的DNA损伤应答与修复对于维持正常细胞基因组完整性至关重要, 其关键基因的功能性丧失可促进肿瘤的发生和发展[3]。癌基因诱发复制压力增加和内源性DNA损伤水平升高, 使肿瘤细胞更加依赖于DNA损伤修复机制以维持生存和增殖。传统的抗肿瘤治疗方法大多通过诱导DNA损伤而产生细胞毒性, 如放射治疗和烷化剂等化疗药物。而肿瘤细胞常通过上调残存的修复通路来清除DNA损伤, 导致肿瘤对放化疗的敏感性降低并产生耐药性[4]。
DNA双链断裂是最具致命性的DNA损伤, 也是电离辐射和多种化疗药物产生细胞毒性的主要原因。若没有得到及时且正确的修复, 双链断裂可造成染色体的缺失、融合和异位, 最终导致细胞死亡[5]。高等真核生物具有两种主要的DNA双链断裂修复机制, 即同源重组(homologous recombination, HR)和非同源末端连接(non-homologous end joining, NHEJ)。非同源末端连接不需要DNA之间具有同源性, 以非保真的方式直接连接断裂的末端。由于断裂的末端在连接前可能被部分降解, 且不同DNA断裂产生的末端也可能被连接在一起, 这种修复方式可导致基因突变或染色体重排, 是一种易错(error-prone)的修复方式。相比之下, 同源重组修复以具有序列同源性的姐妹染色单体为模板进行修复, 具有高度的保真性(error-free), 是正确修复DNA双链断裂的重要方式[6]。家族性乳腺癌、卵巢癌等同源重组修复缺陷的肿瘤表现出对放化疗的敏感性增强, 提示靶向同源重组修复可以成为增敏放化疗的有效策略。而聚ADP-核糖聚合酶[poly(ADP-ribose) polymerase, PARP]抑制剂的成功应用及“chemical HRDness”概念的提出, 也使靶向同源重组修复成为了新的抗肿瘤研究热点。
本文将介绍PARP抑制剂的发展与靶向同源重组修复的必要性, 总结靶向同源重组修复通路与其调节通路的临床前和临床研究进展, 讨论其与PARP抑制剂、放化疗、免疫检查点抑制剂联用的优势与目前亟待解决的问题, 并对其在抗肿瘤治疗中的应用前景进行展望。
1 同源重组修复缺陷同源重组修复是一种高保真的DNA双链断裂修复方式, 利用具有同源序列的姐妹染色单体为模板, 在细胞的S期和G2期高度活跃。乳腺癌易感基因1/2 (breast cancer susceptibility gene 1/2, BRCA1/2)是同源重组修复通路中的关键组分, 也被认为是肿瘤抑制因子, 在家族性乳腺癌和卵巢癌中经常发生突变。BRCA1/2突变失活会导致同源重组修复缺陷, 增加基因组的不稳定性并促进肿瘤的发生和发展。在BRCA1/2突变的卵巢癌或乳腺癌中, PARP抑制剂的应用则取得了巨大的成功, 已成为靶向治疗领域的重大成果之一, 而这种应用是基于“合成致死”效应[7]。
1922年, 遗传学家Calvin Bridges发现了一种有趣的现象:某两个特定的基因同时突变失活会导致果蝇死亡; 而这两个基因单独任何一个突变失活, 都不会给果蝇带来致命的伤害。随后, Theodosius Dobzhansky将这种现象命名为“合成致死(synthetic lethality)”效应。在这一概念沉寂了近百年后, 两个独立的研究团队于2005年“背靠背”在《Nature》发表了重要研究成果, 首次证明PARP抑制剂与BRCA1/2突变之间存在“合成致死”效应[8, 9], 由此开启了应用“合成致死”治疗肿瘤的新大门。PARP是DNA单链断裂的感受器, 催化NAD+合成聚ADP-核糖[poly(ADP-ribose), PAR]链, 将其连接于组蛋白, 松弛染色质并募集修复蛋白。PARP抑制剂通过与NAD+竞争结合PARP, 抑制PAR链的合成, 从而抑制修复蛋白募集和PARP从染色质的解离。此时, 未修复的单链断裂会在复制的过程中进一步转化为双链断裂。由于PARP抑制剂还可将PARP捕获于断裂处, 造成复制叉的停滞与崩溃, 同样产生双链断裂。若由于BRCA1/2突变导致了肿瘤细胞同源重组修复缺陷, 双链断裂无法得到高保真的修复, 从而持续产生致命性DNA损伤, 发生染色体的融合和易位, 最终导致细胞凋亡[10] (图 1)。
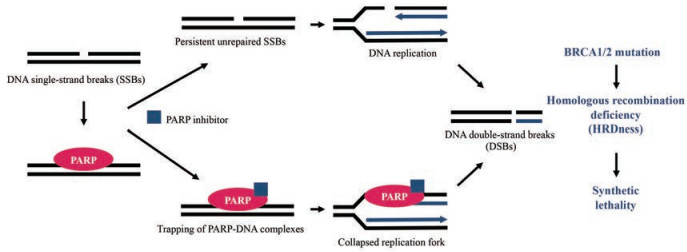
|
Figure 1 Schematic diagram of the mechanisms of synthetic lethality in BRCA1/2 mutant cancers. PARP: Poly ADP-ribose polymerase; BRCA1/2: Breast cancer susceptibility gene 1/2 |
2014年, olaparib成为首个成功上市的PARP抑制剂, 被美国食品药品监督管理局(Food and Drug Administration, FDA)和欧洲药品管理局(European Medicines Agency, EMA)批准用于治疗对3种以上既往治疗方案无效且BRCA1/2突变的晚期卵巢癌。2016年, rucaparib获批上市, 用于治疗对2种以上既往治疗方案无效且BRCA1/2突变的晚期卵巢癌。2017年, niraparib被批准用于复发性铂敏感性上皮卵巢癌、输卵管癌或腹膜癌的维持治疗[1, 7]。2018年, olaparib获批用于治疗HER2阴性且BRCA1/2突变的转移性乳腺癌[11]。2019年底, olaparib获批成为BRCA1/2突变的转移性胰腺癌[12]的一线维持疗法。2020年, rucaparib获批用于治疗携带有害BRCA基因突变的转移性去势抵抗性前列腺癌成人患者。此外, 许多扩大PARP抑制剂适应症的临床试验也在紧锣密鼓地开展中。
PARP抑制剂的抗肿瘤谱在不断扩大, 临床应用的生物标志物也从生殖系或体细胞BRCA1/2突变, 扩展到与BRCA突变具有相似分子特征的“BRCAness”肿瘤, 以及包括其他DNA损伤修复相关基因, 如共济失调毛细血管扩张突变基因(ataxia telangiectasia-mutated gene, ATM)和共济失调毛细血管扩张与RAD3相关基因(ataxia telangiectasia and Rad3 related gene, ATR)等突变的“HRDness”肿瘤(图 2)。但是, 仍然存在大量的肿瘤患者缺乏这些突变基因而无法受益于PARP抑制剂的单药治疗。此外, 许多原本对PARP抑制剂敏感的肿瘤患者由于相关基因出现回复突变而恢复同源重组修复功能, 对PARP抑制剂产生耐药性。

|
Figure 2 Schematic diagram of predictive biomarkers of response to PARP inhibitors. HRD: Homologous recombination repair; DDR: DNA damage response; ATM: Ataxia telangiectasia mutated gene |
近年来, 研究人员提出“chemical HRDness”的概念, 即利用分子靶向药物造成同源重组修复缺陷的表型, 使耐药的患者重新对PARP抑制剂敏感, 并使同源重组修复功能正常的肿瘤患者也能受益于PARP抑制剂的治疗(图 2)。因此, 如何靶向同源重组修复以造成“HRDness”的表型, 如何优化联合用药策略以降低毒性并维持疗效, 如何通过生物标志物在临床应用中评估患者的同源重组修复能力, 都是值得深入研究的问题。
2 同源重组修复通路同源重组修复具体可分为3个阶段:联会前(presynapsis)、联会(synapsis)和联会后(postsynapsis)[6]。在联会前阶段, MRN (MRE11-RAD50-NBS1)复合物识别并结合到DNA双链断裂部位, 与CtIP一起对断裂双链的5'端进行少量核苷酸的切割。随后在BRCA1的促进下, 核酸酶(EXO1和DNA2)能够对5'端进行充分地切割, 在3'端产生一长段RPA包裹的单链DNA (single stranded DNA, ssDNA)。在联会阶段, BRCA1-PALB2-BRCA2复合物将重组酶RAD51装载到ssDNA, 形成联会前丝。RAD52也与RPA发生相互作用, 促进RAD51置换ssDNA上包裹的RPA。随后, 联会前丝侵入其他双链DNA寻找同源序列。一旦发现了同源区域, 修复进入联会后阶段。侵入产生的置换环(D环)将依赖于多种不同的方式(如第二末端捕获、合成后链退火、多重入侵与合成等)完成修复, 产生交换或非交换的重组双链DNA[13] (图 3)。
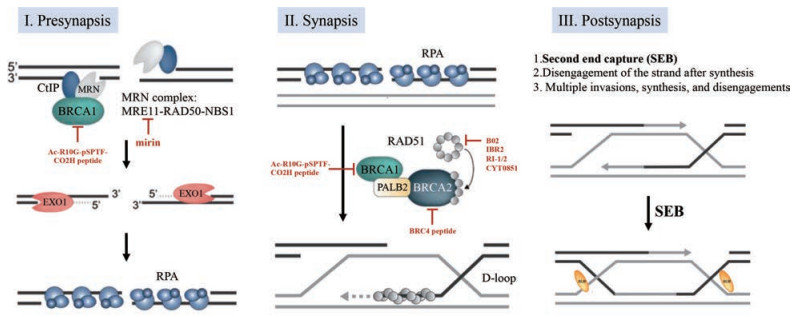
|
Figure 3 Schematic diagram of HR repair pathway and inhibitors directly targeting HR components. HR: Homologous recombination; EXO1: Exonuclease 1; RPA: Replication protein A |
同源重组修复是一个受到严格调控的过程, 其功能很大程度上取决于多种调节通路的活性, 包括细胞周期与检查点调控通路、PI3K信号转导通路、表观遗传与染色质重塑通路等(图 4)。许多研究表明, 除了直接抑制同源重组修复通路的关键组分, 干扰其调节通路也可造成“HRDness”的表型, 为靶向同源重组修复提供了更多的选择。
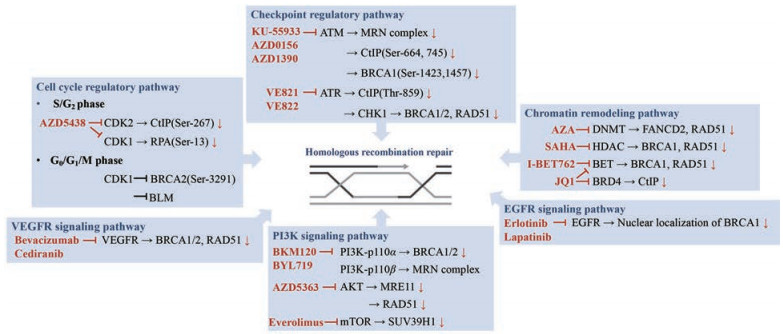
|
Figure 4 Schematic diagram of regulatory pathways of HR and potential therapeutic targets. ATR: Ataxia telangiectasia and Rad3 related gene; CHK1: Checkpoint kinase 1; CDK1/2: Cyclin-dependent kinase 1/2; PI3K: Phosphatidylinositol 3-kinase; mTOR: Mammalian target of rapamycin; DNMT: DNA methyltransferase; HDAC: Histone deacetylase; BET: Bromodomain and extraterminal protein; BRD4: Bromodomain containing 4; EGFR: Epidermal growth factor receptor; VEGFR: Vascular endothelial growth factor receptor |
同源重组修复与细胞周期密切相关, 在很大程度上受到周期蛋白依赖性激酶(cyclin-dependent kinases, CDKs)的调控[14]。在联会前阶段, CDKs调控DNA末端切割, 参与双链断裂修复机制的选择。一旦断裂处进行了末端切割, 则只能进行同源重组修复, 因为切割产生的ssDNA不能作为非同源末端连接修复的底物。研究发现, CDK2磷酸化CtIP, 促进CtIP、BRCA1和MRN复合物之间的相互作用[15], 而CDK2的缺失或抑制则会降低同源重组修复能力[16]。而CDK1-cyclin B介导RPA的磷酸化, 维持3' ssDNA的稳定性。RPA中CDK磷酸化位点的突变会导致细胞内DNA双链断裂增加, 修复蛋白向损伤部位的募集减少[17]。但在细胞有丝分裂期内, CDKs却介导了同源重组修复的负性调节, 从而将修复限制在细胞S期和G2期[18]。例如, CDK1介导BRCA2中C末端结构域Ser-3291的磷酸化, 阻碍BRCA2-RAD51相互作用。此外, CDK1磷酸化BLM解旋酶, 使其从核基质中解离, 从而抑制同源重组修复后期Holliday连接体的拆分[14]。此外, CDK12是一种RNA聚合酶II C末端结构域激酶, 最近发现其作为转录调控因子, 影响多个DNA损伤应答和修复相关基因的转录[19]。Johnson等[20]发现dinaciclib作为一种强效的CDK12抑制剂, 能在BRCA野生型的三阴乳腺癌细胞中抑制BRCA1/2和RAD51等同源重组修复关键基因的转录, 与veliparib联用协同抑制耐药的三阴乳腺癌异种移植瘤生长。
ATM和ATR是DNA损伤应答中的关键组分, 分别在双链断裂修复和单链断裂修复中起着中心调节作用[21]。当检测到DNA双链断裂时, MRN复合物募集并激活ATM。ATM向断裂处募集更多的MRN复合物, 同时磷酸化其他同源重组修复通路组分, 包括BRCA1和CtIP[22]。ATM基因的失活突变抑制了电离辐射诱导的Rad51焦点(foci)的形成, 提示同源重组修复功能受损[23]。全基因组研究表明, 转移性去势抵抗前列腺癌患者体内ATM突变频率很高, 已成为预测BRCA1/2野生型前列腺癌患者对PARP抑制剂敏感性的生物标志物[24]。与双链断裂激活ATM相平行, ATR响应于ssDNA而被激活。在同源重组修复中, ATR被末端切割产生的3' ssDNA激活, 并积极参与了修复过程。例如, CtIP在断裂处的积累和5'端的充分切割需要ATR依赖的CtIP过度磷酸化[25]。最近的研究还发现, 肿瘤细胞内高水平的复制应激持续激活ATR/CHK1通路, 并通过CHK1依赖性转录促进同源重组修复通路关键组分(BRCA1/2和RAD51等)的表达[26]。此外, ATM和ATR通过不同但存在交互作用的ATM/CHK2和ATR/CHK1通路调节细胞周期检查点[21]。ATR/CHK1和ATM/CHK1激活G2检查点, 而ATM/CHK1也依赖于p53激活G1检查点。由于大多数肿瘤细胞p53突变, 导致G1检查点失活, 使肿瘤细胞会更加依赖于G2检查点。此时, ATM和ATR的激活会产生G2期阻滞, 为同源重组修复提供了更多时间[27]。
PI3K信号转导通路在肿瘤细胞的生存、增殖、转移及血管生成中发挥重要作用[28]。基因组学研究发现, 众多肿瘤类型RTK/PI3K信号转导分子异常(包括RTK和PI3K的变异以及PTEN缺失等), 从而导致PI3K/AKT通路被持续性激活[29]。PI3K是一类脂激酶, 可通过磷酸化PIP2为PIP3, 激活AKT和下游效应物, 调节细胞分裂和胚胎发育。研究发现, PI3K还参与细胞对DNA双链断裂的应答[30], 维持同源重组修复的稳定状态[31]。在BRCA1/2野生型的三阴乳腺癌细胞中, PIK3CA基因敲减或抑制其激酶活性均可下调BRCA1/2表达并诱导“HRDness”表型, 增加DNA损伤和代偿性PARP活化。对其机制的初步研究表明, PI3K抑制导致MEK/ERK通路代偿性激活, 使转录抑制因子ETS1与BRCA1/2启动子结合, 从而抑制BRCA1/2表达[31]。AKT位于PI3K的下游, 在乳腺癌、结直肠癌和胰腺癌等多种肿瘤中出现基因扩增、过度表达和异常激活。研究发现, 活化的AKT可通过GSK3β/β-catenin/LEF通路上调MRN复合物中Mre11的表达, 而MRN复合物募集并磷酸化ATM, ATM又通过RNF168磷酸化AKT, 形成正反馈调节环路[32]。此外, AKT1基因敲减下调了RAD51的表达, 诱导“HRDness”表型[33]。
表观遗传学的最新研究揭示了DNA损伤后染色质修饰的动态变化, 证明了表观遗传和染色质重塑因子是DNA损伤应答与修复网络中的重要组成部分[34]。例如, 组蛋白去乙酰化酶(histone deacetylase, HDAC)催化组蛋白去乙酰化, 导致染色质浓缩并抑制基因表达, 在肿瘤中介导抑癌基因的转录抑制[34]。除组蛋白乙酰化外, DNA甲基化也是真核细胞基因表达调控的主要表观遗传学形式。DNA甲基转移酶(DNA methyltransferase, DNMT)催化DNA甲基化, 使DNA的构象、稳定性及与蛋白的相互作用方式产生变化, 进而调节基因转录。研究表明, HDAC和DNMT的活性显著影响同源重组修复相关基因(BRCA1、RAD51和FANCD2等)的表达[35-38], 这为使用表观遗传与染色质重塑因子抑制剂诱导“HRDness”表型提供了理论基础。
4 靶向同源重组修复药物与PARP抑制剂的联用 4.1 靶向同源重组修复通路关键组分的药物MRE11具有核酸内切酶和核酸外切酶活性, 切割DNA末端并将双链断裂引入同源重组修复通路。Mirin是采用化学遗传筛选法发现的第一个MRE11抑制剂, 可抑制其核酸外切酶活性, 从而抑制5'端切割和MRN依赖性ATM激活[39]。Ying等[40]发现MRE11在BRCA2突变或PARP抑制的肿瘤细胞中被过度激活, 而mirin与PARP抑制剂联用显著降低了BRCA2野生型卵巢癌和骨肉瘤细胞的存活率。此外, 研究还发现抑制MRE11内切酶活性会促进非同源末端连接替代同源重组修复, 而抑制外切酶活性可产生更强的DNA双链断裂修复缺陷[41], 为MRE11抑制剂的研发提供了理论基础。
BRCA1和BRCA2是同源重组修复的关键蛋白。BRCA1既能抵消53BP1对末端切割的抑制活性, 还通过募集PALB2和BRCA2将重组酶RAD51装载到ssDNA来启动同源重组[42]。虽然BRCA1/2在同源重组修复中充当多种蛋白-蛋白相互作用的关键枢纽, 但却难以采用基于结构的药物研究策略开发小分子靶向抑制剂, 因为它们都是由短的功能结构域组成, 中间穿插长且无序的连接结构[43]。Pessetto等[44]发现细胞渗透性肽Ac-R10G-pSPTF-CO2H可通过与BRCA1中BRCT结构域结合, 抑制其与CtIP等磷酸化蛋白的相互作用, 模拟了M177R/K BRCA1突变的表型, 增强了olaparib对BRCA1野生型HeLa细胞和MDA-MB-231细胞的增殖抑制作用。同样, 与BRCA2结构相似的BRC4肽破坏了BRCA2-RAD51相互作用, 与PARP抑制剂联用显著降低了BRCA2野生型HeLa细胞的克隆形成率[45]。研究人员正基于肽库发现更多可造成“BRCAness”表型的BRCA1/2选择性抑制剂[46]。此外, 利用小分子抑制剂靶向BRCA1中RING结构域介导的泛素连接酶活性, 也可作为抑制同源重组修复的有效方法[47]。
靶向RAD51的小分子抑制剂(如B02、IBR2和RI-1/2)可通过抑制RAD51单体低聚、与ssDNA结合及形成联会前丝, 诱导“HRDness”表型。RAD51抑制剂B02、PARP抑制剂veliparib和p38 MAPK抑制剂LY2228820的三联疗法在原位异种移植模型中显著抑制三阴乳腺癌的生长[48]。同样, IBR2抑制了乳腺癌异种移植瘤生长和携带BCR-ABLT315I突变的慢性粒细胞白血病模型中肿瘤细胞的增殖[49]。RI-1抑制子宫颈癌异种移植瘤的生长[50], 与veliparib联用具有协同抗三阴乳腺癌作用[51]。这些临床前研究表明, RAD51抑制剂可诱导同源重组修复缺陷, 并有潜力研发为新型广谱抗肿瘤药物。Cyteir公司研发的口服RAD51抑制剂CYT-0851能有效抑制同源重组修复, 与PARP抑制剂联用具有很强的协同作用, 目前正在针对B细胞瘤和多种实体瘤开展Ⅰ/Ⅱ期临床试验(NCT03997968) (表 1)。
| Table 1 Ongoing trials with inhibitors targeting HR. AR: Androgen receptor |
ATM抑制剂对同源重组修复具有显著的负性调节作用。KU-55933是第一种选择性ATM抑制剂, 在针对不同肿瘤的临床前研究中被广泛应用, 能显著抑制同源重组修复功能并增敏放化疗, 但高度的亲脂性限制了其在体内的应用[52, 53]。在此基础上开发了多种选择性强、生物利用度高的口服ATM抑制剂, 如AZD0156[54]、AZD1390[55]等。ATM抑制剂与PARP抑制剂联用的临床研究仍处于早期阶段, 目前正在开展AZD0156与olaparib或氟尿嘧啶等化疗药物联用治疗晚期实体瘤的Ⅰ期临床试验(NCT0258105) (表 1)。
ATR抑制剂VE821通过下调BRCA1/2、RAD51的表达抑制同源重组修复, 与olaparib联用协同抑制多种BRCA1/2野生型的肿瘤细胞增殖[26]。ATR抑制剂VE822与olaparib联用显著升高了胶质母细胞瘤干细胞原位移植瘤小鼠的存活率(P < 0.01)[56]。ATR抑制剂M6620 (VX-970)和AZD6738治疗晚期实体瘤的安全性和有效性已在Ⅰ期临床试验中进行了研究。M6620单药[57]耐受性良好, 未观察到剂量限制性毒性。但AZD6738在连续给药的第一个周期后观察到骨髓抑制的不良反应, 研究人员正在优化给药方案以改善其长期耐受性[58]。此前的Ⅰ期临床试验也研究了AZD6738与卡铂、olaparib或durvalumab的联合疗法[59]。AZD6738和olaparib联用具有良好的耐受性, 尽管骨髓抑制仍是报道的主要不良反应。在两名三阴性乳腺癌患者和一名雌激素受体阳性的乳腺癌患者中观察到靶病灶部分缓解, 即靶病灶最大径之和减少≥30%, 且维持4周以上。目前, 正在开展口服ATR抑制剂BAY1895344和M4344单药或与PARP抑制剂联用治疗晚期实体瘤的Ⅰ期临床试验(NCT03188965和NCT0278250) (表 1)。
4.3 PI3K/AKT/mTOR抑制剂靶向PI3K激酶的抑制剂对于抗肿瘤治疗具有积极作用[60], 目前已有4种PI3K抑制剂被FDA批准上市, 并有40多种化合物进入临床研究阶段[61]。在BRCA1/2野生型的三阴乳腺癌细胞中, PI3K泛抑制剂buparlisib (BKM120)可通过下调BRCA1/2表达诱导“HRDness”表型, 与olaparib联用显著抑制三阴乳腺癌异种移植瘤生长[31]。PI3K抑制剂和PARP抑制剂在体内的协同作用也在BRCA1突变的乳腺癌[62]、PIK3CA突变的卵巢癌[63]和PTEN突变的子宫内膜癌[64]等裸鼠移植瘤模型中被进一步验证。在70例晚期、高级别浆液性卵巢癌或乳腺癌患者中进行了buparlisib联合olaparib的Ⅰ期临床试验[65]。卵巢癌患者的客观缓解率为29%, 乳腺癌患者的客观缓解率为28%, 与对铂类药物的敏感性无关。恶心和疲劳是最常见的毒性反应, 剂量限制性毒性包括转氨酶升高和抑郁。由于毒性限制了buparlisib的使用剂量, 随后研究了olaparib与PI3K-p110α选择性抑制剂alpelisib (BYL719)的联合疗法[66]。在28例晚期卵巢癌患者中(其中有26例对铂类药物耐药), 客观缓解率为36%, 中位缓解时间为167天。值得注意的是, 客观缓解率在BRCA1/2生殖系突变或野生型的患者之间差别很小。
由于AKT的激活促进肿瘤细胞对PARP抑制剂产生耐药性[61], Ⅰ期临床试验研究了AKT抑制剂capivasertib (AZD5363)与olaparib治疗晚期实体瘤的联合疗法。联用具有良好的耐受性, 抗肿瘤活性与BRCA1/2状态或之前是否接受过铂类药物、PARP抑制剂、PI3K通路抑制剂治疗无关[67]。
基于转录组和通路分析发现, mTOR抑制剂也可诱导“HRDness”表型。在BRCA1/2野生型乳腺癌细胞和异种移植瘤模型中, mTOR抑制剂everolimus和talazoparib的联用比单用任一药物具有更大的疗效。不同的是, mTOR抑制剂并没有改变同源重组修复关键组分(如BRCA1/2或RAD51)的表达水平, 而是通过下调参与DNA双链断裂修复的组蛋白甲基转移酶SUV39H1的表达, 诱导同源重组修复缺陷[68]。
4.4 表观遗传与染色质重塑因子抑制剂通过对多种不同特征的表观遗传药物进行筛选, Yang等[69]发现溴结构域和末端外结构域(bromodomain and extraterminal, BET)抑制剂JQ1、I-BET762和OTX015等在同源重组修复功能正常的肿瘤中与olaparib联用具有协同作用。BET抑制剂可与BET蛋白溴结构域可逆性地结合, 阻止其与乙酰化组蛋白和转录因子相互作用。由于BET抑制剂常影响具有超级增强子的一小部分基因, 其对基因表达的调节具有一定的特异性。溴结构域中有序、疏水的口袋, 为小分子抑制剂的研发提供了有利的结合位点, 目前已有多种BET抑制剂进入了临床研究阶段, 用于治疗睾丸癌、急性白血病和多发性骨髓瘤[70]。研究发现, BET抑制剂可直接抑制同源重组修复关键基因BRCA1和RAD51的表达, 在同源重组修复功能正常的乳腺癌和卵巢癌细胞中造成“HRDness”表型[69, 71]。此外, JQ1还可下调G2/M检查点调节蛋白WEE1和DNA损伤应答蛋白TOPBP1的表达, 与olaparib联用使携带DNA损伤的细胞提前进入有丝分裂期, 导致有丝分裂灾难和细胞凋亡[72]。
在BET蛋白(BRD2、BRD3、BRD4和BRDT)中, BRD4可直接与RNA聚合酶II相互作用调节基因转录, 促进肿瘤细胞的增殖和存活[73, 74]。BRD4在高级别浆液性卵巢癌患者中经常扩增并与预后不良相关。Sun等[75]发现BRD4抑制剂可通过阻碍BRD4与CtIP的启动子和增强子结合, 抑制CtIP表达, 进而抑制断裂处5'端切除和3' ssDNA的形成, 造成同源重组修复缺陷。在多种BRCA1/2野生型的肿瘤细胞系和小鼠移植瘤模型中, BRD4抑制剂与olaparib联用具有协同作用。
DNMT抑制剂5-azacytidine (AZA)通过抑制FANCD2的表达和RAD51的募集, 诱导“HRDness”表型, 同时通过增强PARP1与损伤处染色质的紧密结合, 升高DNA双链断裂产生的频率, 与talazoparib联用显著抑制BRCA1/2野生型的非小细胞肺癌、急性髓细胞性白血病和乳腺癌异种移植瘤生长[37, 38]。HDAC抑制剂vorinostat (SAHA)能下调BRCA1和RAD51表达, 诱导同源重组修复缺陷, 与olaparib联用具有协同抗前列腺癌和三阴乳腺癌作用[35, 36]。Ⅰ/Ⅱ期临床试验中, vorinostat与化疗或放疗联用治疗晚期实体瘤具有很好的耐受性并显示出积极疗效[76], 而与PARP抑制剂的联用正在开展Ⅰ期临床试验(NCT0372245) (表 1)。
4.5 泛素化/类泛素化信号通路抑制剂泛素化修饰是真核生物中一种常见的翻译后修饰方式, 参与调控细胞内的众多生物学过程, 包括DNA损伤应答。当检测到DNA双链断裂时, 组蛋白H2AX可被ATM磷酸化为γ-H2AX。γ-H2AX通过MDC1募集E3泛素连接酶RNF8和RNF168, 对组蛋白H2A和γ-H2AX进行泛素化修饰。泛素化信号在募集BRCA1和53BP1等DNA损伤应答下游信号分子中发挥关键作用[77]。去泛素化酶USP13同样被ATM磷酸化激活, 通过对RAP80进行去泛素化而促进RAP80和BRCA1募集至双链断裂处。Li等[78]发现USP13选择性抑制剂Spautin-1通过增加RAP80的泛素化水平, 阻滞同源重组修复过程, 与olaparib联用显著抑制卵巢癌异种移植瘤的生长(P < 0.05)。此外, Song等[79]发现去泛素化酶UCHL3抑制剂perifosine通过抑制RAD51去泛素化, 阻滞RAD51与BRCA2相互作用, 与olaparib联用显著抑制三阴乳腺癌异种移植瘤的生长(P < 0.01)。
最近的研究还发现, 泛素样蛋白NEDD8同样被募集至DNA双链断裂处, 通过对组蛋白H4进行类泛素化修复, 促进RNF168募集和后续修复过程[80]。Guo等[81]发现类泛素化抑制剂MLN4924通过抑制BRCA1募集至双链断裂处, 在非小细胞肺癌中阻滞同源重组修复过程, 与PARP抑制剂联用有潜力发挥协同抗非小细胞肺癌作用。
4.6 肿瘤代谢抑制剂代谢改变是肿瘤的重要特征之一。为了维持细胞增殖, 肿瘤细胞常改变自身代谢与营养获得的方式。靶向肿瘤的异常代谢已成为抗肿瘤药物研发领域备受关注的研究方向之一, 针对不同代谢酶的多个小分子抑制剂正处在临床前和临床研究阶段。近年来的研究发现, 肿瘤的异常代谢与DNA损伤修复和基因组不稳定间存在着密切的联系[82], 因而代谢抑制剂与PARP抑制剂的联用也逐渐受到人们的广泛关注。
磷酸甘油酸变位酶1 (phosphoglycerate mutase 1, PGAM1)是糖酵解中的关键酶, 在包括乳腺癌、肺癌和胶质母细胞瘤等多种肿瘤中表达上调。PGAM1抑制剂PGMI-004A能通过耗竭细胞内的dNTP, 激活p21-APC/C-Cdh1通路, 促进CtIP以蛋白酶体途径降解, 从而阻滞同源重组修复, 增加了olaparib对BRCA野生型乳腺癌异种移植瘤的抑瘤率[83]。
缺乏VHL抑癌基因的肿瘤细胞常利用谷氨酰胺通过α-酮戊二酸的还原羧化生成柠檬酸盐和脂质, 同时在三羧酸循环中通过谷氨酰胺酶(glutaminase, GLS)生成天冬氨酸, 作为嘧啶核苷酸从头合成的碳源。Okazaki等[84]发现GLS1抑制剂BPTES可抑制嘧啶的从头合成, 升高细胞内ROS水平, 诱导复制应激, 与olaparib联用显著抑制肾癌UMRC3细胞异种移植瘤的生长(P < 0.05)。
此外, Fang等[85]利用CRISPR/Cas9技术在基因组范围内进行筛选, 发现C12orf5基因缺失显著影响A2780细胞对PARP抑制剂的反应性, 其编码TP53诱导的糖酵解和凋亡调节蛋白(TP53 induced glycolysis and apoptosis regulator, TIGAR)。敲除TIGAR可阻滞戊糖磷酸途径进而升高细胞内ROS水平, 并通过下调BRCA1/2和RAD51表达诱导同源重组修复缺陷, 增强了olaparib对卵巢癌细胞的增殖抑制作用, 为相关靶向抑制剂与PARP抑制剂的联用提供了理论基础。
4.7 其他PARP抑制可选择性地靶向低氧肿瘤细胞, 增加肿瘤的血管灌注[86]。临床前研究发现多种抗血管生成药物与PARP抑制剂联用具有协同作用, 部分是由于抗血管生成药物引起低氧状态, 并通过下调关键修复基因表达诱导“HRDness”表型[87]。Ⅲ期临床试验表明, 靶向血管内皮生长因子受体(VEGFR)的贝伐单抗和olaparib的联用, 与olaparib单用相比显著改进了卵巢癌患者的中位无进展生存期(22.9个月/16.6个月), 在同源重组修复缺陷的患者中联用效果更加显著(37.2个月/17.7个月)[88]。目前, 贝伐单抗与olaparib的联合疗法已被FDA批准作为一线维持疗法, 治疗接受铂类化疗药物治疗后完全或部分缓解且同源重组修复缺陷的卵巢癌患者。小分子VEGFR抑制剂西地尼布可通过抑制VEGFR酪氨酸激酶活性, 下调RAD51和BRCA1/2表达[89]。西地尼布与olaparib联用治疗复发性铂敏感卵巢癌患者的Ⅱ期临床研究显示, 联用比olaparib单药更能延长患者的中位无进展生存期(16.5个月/8.2个月), 并有改善总生存期的趋势[90]。目前, 正在开展西地尼布和olaparib联用治疗BRCA1/2突变卵巢癌的Ⅲ期临床试验(NCT02446600) (表 1)。
表皮生长因子受体(EGFR)信号通路在肿瘤中经常通过突变或扩增而被过度激活[91], 其部分致癌性也与促进DNA损伤修复有关。在同源重组修复中, EGFR与BRCA1形成复合物, 促进BRCA1移位至细胞核双链断裂处。EGFR抑制剂erlotinib和lapatinib可抑制BRCA1的核定位[92], 干扰Rad51募集到DNA损伤部位从而抑制同源重组修复, 与PARP抑制剂联用显著降低乳腺癌细胞的存活率[92, 93]。
最近的研究还表明, 雄激素受体(AR)信号通路在调节同源重组修复中具有重要作用。AR通路抑制可导致“HRDness”表型, 与PARP抑制剂联用在前列腺癌细胞中导致合成致死[94, 95]。AR抑制剂abiraterone和olaparib联用治疗去势抵抗性前列腺癌的Ⅱ期临床实验表明, 联用与abiraterone单药相比, 显著改善了患者放射治疗后的中位无进展生存期(13.8个月/8.2个月)[96]。目前, AR抑制剂enzalutamide和olaparib的联用正在开展Ⅱ期临床试验(NCT03012321) (表 1)。
此外, 多种靶向细胞周期调节(如CDK1/2和Cyclin D1等)和蛋白稳定性调控(如HSP90等)的药物也可导致“HRDness”表型, 其与PARP抑制剂的联合用药正在开展临床前和临床研究[14]。虽然靶向同源重组修复的调节通路可造成“HRDness”表型, 但由于其作用可能具有多重性, 仍需深入了解其他通路受到的影响和通路之间的交互作用, 以避免出现额外的不良反应。若能够通过合适的联用策略, 使肿瘤细胞长期保持“HRDness”表型, 还可延缓耐药性的产生。
5 靶向同源重组修复药物的其他应用除了与PARP抑制剂联用造成“合成致死”外, 人们最早认为靶向同源重组修复可以增强放射治疗和烷化剂等化疗药物的抗肿瘤效果, 因为这类传统的肿瘤治疗方法均通过诱导DNA损伤而产生细胞毒性。由于发现在同源重组修复缺陷的肿瘤中, 基因组不稳定性可增加肿瘤细胞的免疫原性, 提示靶向同源重组修复与免疫检查点抑制剂联用将具有很大的潜力。
5.1 放疗增敏尽管放射疗法在临床中得到了广泛的应用, 已成为实体瘤治疗的重要手段之一, 但放疗抗性仍然是治疗失败的主要原因, 导致肺癌、胰腺癌和胶质母细胞瘤等肿瘤在放疗后的无进展生存期依然很短[32]。电离辐射可使细胞产生碱基损伤、DNA单链断裂和双链断裂, 其中最严重的损伤为DNA双链断裂。辐射引起的损伤激活DNA损伤应答与修复网络, 介导辐射抗性的产生并降低临床疗效。许多化疗药物除了具有细胞毒性外, 还具有放疗增敏作用。尽管其增敏放疗的机制尚未研究透彻, 但是许多放疗增敏剂被发现能够抑制同源重组修复活性, 如吉西他滨、TAS-106、吉美嘧啶和己酮可可碱等, 提示靶向同源重组修复可成为放疗增敏的一种有效策略[97]。
由于放射治疗能够在肿瘤部位造成局部的DNA损伤, 靶向同源重组修复的抑制剂与放射治疗的联用受到了广泛的研究。Tu等[98]发现ATR抑制剂M6620可作为三阴乳腺癌的特异性放疗增敏剂。Pal等[99]发现HDAC和PI3K双重抑制剂CUDC-979与电离辐射的联合治疗具有极强抗小儿高级神经胶质瘤和弥漫性桥脑神经胶质瘤作用。HDAC抑制剂vorinostat与放疗联用治疗晚期实体瘤也在Ⅰ/Ⅱ期临床试验中表现出很好的耐受性和积极的治疗效果[76]。此外, ATM抑制剂[100]、AKT抑制剂[101]、CDK抑制剂[102]和热疗[103]等都被证明可通过抑制同源重组修复产生放疗增敏作用。
然而也存在许多靶向同源重组修复的抑制剂没有表现出显著和一致的增敏作用。研究发现, 传统光子辐射造成的DNA损伤多通过非同源末端连接进行修复, 而重离子(碳、铁)或高能质子辐射可引起更密集的DNA双链断裂, 需要更多同源重组修复的参与[104, 105]。因此, 靶向同源重组修复的抑制剂与重离子或高能质子辐射的联用将更具吸引力。
5.2 与烷化剂联用烷化剂是问世最早、种类最多、应用最广泛的一类化疗药物, 通过与DNA富电基团发生共价结合, 造成碱基损伤、错配、链间和链内交联等DNA损伤。烷化剂造成的DNA损伤最终在DNA复制等过程中可转化为致命性双链断裂, 是产生细胞毒性的关键因素。而同源重组修复参与此类损伤的清除, 是造成肿瘤细胞对烷化剂不敏感并产生耐药性的重要原因之一。研究发现, 同源重组修复缺陷(BRCA2、RAD51或XRCC3突变)的细胞对烷化剂尼莫司汀高度敏感, 而非同源末端连接缺陷(Ku80或DNA-PK突变)的细胞则对此药不敏感或仅轻度敏感[106], 提示靶向同源重组修复可作为增敏烷化剂的有效策略。
替莫唑胺是治疗胶质母细胞瘤的一线用药, 造成的碱基损伤包括O6-甲基鸟嘌呤、N7-甲基鸟嘌呤和N3-甲基腺嘌呤。在MGMT酶缺失的情况下, 不能被直接修复的O6-甲基鸟嘌呤可与胞嘧啶或胸腺嘧啶配对, 产生的错配则可被错配修复机制识别。错配修复并不能修复DNA模板链上的O6-甲基鸟嘌呤, 反而在新生链中产生DNA单链断裂, 并在进一步复制过程中转化为双链断裂。研究表明, 胶质母细胞瘤患者在长期使用替莫唑胺治疗后, 同源重组修复功能增强并产生获得性耐药[107, 108]。Gil Del Alcazar等[107]利用CDK1/2抑制剂阻断同源重组修复通路, 增强了替莫唑胺对胶质母细胞瘤细胞的增殖抑制作用。
洛莫司汀是一种临床广泛应用的烷化剂, 用于治疗神经胶质瘤、黑色素瘤、霍奇金淋巴瘤以及多种来源的脑转移性肿瘤。洛莫司汀产生的DNA交联损伤可阻碍复制叉的移动, 在修复过程中被核酸内切酶切割形成DNA双链断裂。Berte等[106]发现MRE11抑制剂mirin或RAD51抑制剂B02、RI-1与洛莫司汀联用增加了染色体畸变水平, 在体外和体内发挥协同抗胶质母细胞瘤作用。
但是多项临床试验发现, 靶向同源重组修复的抑制剂和烷化剂联用时会产生额外的剂量限制性毒性, 耐受性较差。例如, ATR抑制剂AZD6738与卡铂联用的Ⅰ期临床试验发现, 联用导致的多种毒性反应使患者需要延迟治疗并减小药物剂量, 包括3级以上的血小板减少症、中性粒细胞减少症和贫血[59]。如何优化联合用药方案(包括给药剂量、时间和顺序等), 在降低毒性的同时维持疗效, 是靶向同源重组修复在与烷化剂联用时亟待解决的问题。
5.3 与免疫检查点抑制剂联用免疫疗法的发展改变了许多肿瘤的治疗模式。免疫检查点抑制剂已在多种肿瘤的治疗中得到了成功的应用, 如抗PD-1/PD-L1单抗和抗CTLA4单抗等。研究发现, DNA修复通路的缺陷可增加免疫检查点抑制剂的活性。例如, 错配修复缺陷的肿瘤具有微卫星不稳定性表型, 特点是肿瘤突变负荷高, 免疫微环境活化, 肿瘤细胞、免疫细胞和基质细胞PD-1或PD-L1表达增加。FDA已批准抗PD-1单抗pembrolizumab用于治疗错配修复缺陷的晚期肿瘤[109]。
多项研究也证明了同源重组修复与肿瘤免疫之间存在关联, 为靶向同源重组修复的抑制剂与免疫检查点抑制剂联用提供了理论基础。首先, 在由BRCA1/2或FANCD2突变导致DNA损伤应答缺陷的乳腺癌中, S期特异性DNA双链断裂可导致细胞质内DNA损伤的积累, 激活STING介导的I型干扰素释放, 从而促进先天性免疫识别并激活T细胞[110]。其次, 同源重组修复缺陷常会引入大量的插入突变, 增加肿瘤的突变负担, 导致新抗原表位的产生和T细胞抗肿瘤活性的增强[111, 112]。这些研究表明, 通过靶向药物造成“HRDness”表型可在肿瘤中造成免疫弱点, 与免疫检查点抑制剂联用有潜力发挥协同抗肿瘤作用。
但是DNA双链断裂介导的免疫激活常伴随免疫抑制信号的增强, 如PD-L1表达上调。Sato等[113]发现由DNA双链断裂激活的ATM/ATR可通过STAT1/STAT3/IRF3通路上调肿瘤细胞内PD-L1的表达。ATR抑制剂AZD6738可抑制辐射诱导的PD-L1表达上调, 增加CD8+ T细胞的抗肿瘤活性[114, 115]。Ⅰ期临床试验也研究了ATR抑制剂AZD6738与抗PD-L1单抗durvalumab联用治疗晚期非小细胞肺癌或头颈鳞癌。患者每天先接受AZD6738作为导入治疗, 随后接受durvalumab治疗, 以28天为一个治疗周期。试验表明联用的耐受性良好, 并初步显示出协同的治疗效果[59]。
由于二者几乎没有重叠的毒性反应, 靶向同源重组修复的抑制剂与免疫检查点抑制剂联用具有良好的耐受性, 在临床应用中将会具有很大的潜力。
6 总结与展望目前, 随着分子生物学技术的不断发展和对同源重组修复机制认识的逐步深入, 靶向同源重组修复的抗肿瘤药物研究取得了长足的进展, 越来越多选择性高、作用效果强的药物在临床前研究和临床试验中涌现。利用分子靶向药物造成“HRDness”表型, 可使耐药的患者重新对PARP抑制剂敏感, 并使同源重组修复功能正常的肿瘤患者也能受益于PARP抑制剂的治疗。除了与PARP抑制剂联用造成“合成致死”外, 靶向同源重组修复还为传统的放化疗和新兴的免疫疗法提供了更大的发展空间。
为优化患者的选择, 显著改善患者的预后, 发展动态的生物标志物和检测方法成为当前的迫切需要。CDx-PCR测序平台是FDA较早批准的检测方法, 用于识别BRCA1/2编码区或内含子/外显子边界区存在有害生殖系突变的肿瘤患者[116]。随后研发的基于二代测序技术的myChoice HRD诊断方法, 不仅可以检测BRCA1/2突变, 而且能识别具有BRCA1/2突变分子特征的“BRCAness”的肿瘤患者, 包括杂合性缺失、大规模易位和端粒等位基因不平衡等[117, 118]。通过对血液中循环DNA (cfDNA)的监测, 可提供治疗过程中基因组变化的动态信息[119], 及时发现相关基因的回复突变[120]。目前, 基于差异表达基因转录组图谱建立的HRD评分系统[121], 有望实现对靶向药物诱导的“chemical HRDness”进行监测。
此外, 虽然多种分子靶向药物可造成“HRDness”的表型, 其与PARP抑制剂、放化疗或免疫检查点抑制剂的联用已被证明具有极大的临床潜力, 但临床试验也观察到联用具有不同程度的毒性反应, 部分情况下患者需要延缓治疗或减少给药剂量。优化联合的给药方案, 确定每种药物的给药顺序、剂量和时间间隔, 以尽量减少不良反应, 同时最大限度地提高治疗效果, 也是目前研究的重点。Fang等[122]发现, PARP抑制剂诱导的DNA损伤和周期检查点的激活在药物清除后持续存在, 与ATR抑制剂和WEE1抑制剂采用序贯疗法(sequential therapy)的给药方式在维持疗效的同时, 显著降低联用的毒性。这种序贯疗法的给药方式为优化此类药物的临床使用, 最大限度地提高患者的耐受性提供了新的思路。
DNA损伤应答与修复是一个复杂的信号网络, 涉及众多信号通路的参与, 且不同通路之间多存在交互作用。要想最大限度地发挥靶向同源重组修复抑制剂的作用, 需要深入地掌握不同DNA损伤类型的转换、修复通路之间的代偿和调控机制, 全面地了解不同组织和分子亚型的肿瘤细胞对抗基因组不稳定的动态机制, 以及这种不稳定性如何影响先天性免疫和获得性免疫。总之, 靶向同源重组修复在抗肿瘤治疗中具有极其重要的意义, 虽然目前仍有一些问题亟待解决, 相信在未来将会有很大的发展和应用空间。
作者贡献:钟振兴查阅部分文献, 撰写草稿; 彭鑫查阅部分文献、修改草稿; 孔德新提供撰写思路、修改草稿。
利益冲突:所有作者均无相关利益冲突。
| [1] |
Pilie PG, Tang C, Mills GB, et al. State-of-the-art strategies for targeting the DNA damage response in cancer[J]. Nat Rev Clin Oncol, 2019, 16: 81-104. |
| [2] |
Roos WP, Thomas AD, Kaina B. DNA damage and the balance between survival and death in cancer biology[J]. Nat Rev Cancer, 2016, 16: 20-33. |
| [3] |
Ma J, Setton J, Lee NY, et al. The therapeutic significance of mutational signatures from DNA repair deficiency in cancer[J]. Nat Commun, 2018, 9: 3292. |
| [4] |
Brown JS, O'Carrigan B, Jackson SP, et al. Targeting DNA repair in cancer: beyond PARP inhibitors[J]. Cancer Discov, 2017, 7: 20-37. |
| [5] |
Trenner A, Sartori AA. Harnessing DNA double-strand break repair for cancer treatment[J]. Front Oncol, 2019, 9: 1388. |
| [6] |
Srivastava M, Raghavan SC. DNA double-strand break repair inhibitors as cancer therapeutics[J]. Chem Biol, 2015, 22: 17-29. |
| [7] |
Lord CJ, Ashworth A. PARP inhibitors: synthetic lethality in the clinic[J]. Science, 2017, 355: 1152-1158. |
| [8] |
Bryant HE, Schultz N, Thomas HD, et al. Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase[J]. Nature, 2005, 434: 913-917. |
| [9] |
Farmer H, McCabe N, Lord CJ, et al. Targeting the DNA repair defect in BRCA mutant cells as a therapeutic strategy[J]. Nature, 2005, 434: 917-921. |
| [10] |
Ashworth A, Lord CJ. Synthetic lethal therapies for cancer: what's next after PARP inhibitors[J]. Nat Rev Clin Oncol, 2018, 15: 564-576. |
| [11] |
Le D, Gelmon KA. Olaparib tablets for the treatment of germ line BRCA-mutated metastatic breast cancer[J]. Expert Rev Clin Pharmacol, 2018, 11: 833-839. |
| [12] |
Zhu H, Wei M, Xu J, et al. PARP inhibitors in pancreatic cancer: molecular mechanisms and clinical applications[J]. Mol Cancer, 2020, 19: 49. |
| [13] |
Sun Y, McCorvie TJ, Yates LA, et al. Structural basis of homologous recombination[J]. Cell Mol Life Sci, 2020, 77: 3-18. |
| [14] |
Krajewska M, Fehrmann RS, de Vries EG, et al. Regulators of homologous recombination repair as novel targets for cancer treatment[J]. Front Genet, 2015, 6: 96. |
| [15] |
Chen L, Nievera CJ, Lee AY, et al. Cell cycle-dependent complex formation of BRCA1.CtIP.MRN is important for DNA double-strand break repair[J]. J Biol Chem, 2008, 283: 7713-7720. |
| [16] |
Buis J, Stoneham T, Spehalski E, et al. Mre11 regulates CtIP-dependent double-strand break repair by interaction with CDK2[J]. Nat Struct Mol Biol, 2012, 19: 246-252. |
| [17] |
Anantha RW, Vassin VM, Borowiec JA. Sequential and synergistic modification of human RPA stimulates chromosomal DNA repair[J]. J Biol Chem, 2007, 282: 35910-35923. |
| [18] |
Ovejero S, Bueno A, Sacristan MP. Working on genomic stability: from the S-phase to mitosis[J]. Genes, 2020, 11: 225. |
| [19] |
Liang K, Gao X, Gilmore JM, et al. Characterization of human cyclin-dependent kinase 12 (CDK12) and CDK13 complexes in C-terminal domain phosphorylation, gene transcription, and RNA processing[J]. Mol Cell Biol, 2015, 35: 928-938. |
| [20] |
Johnson SF, Cruz C, Greifenberg AK, et al. CDK12 inhibition reverses de novo and acquired PARP inhibitor resistance in BRCA wild-type and mutated models of triple-negative breast cancer[J]. Cell Rep, 2016, 17: 2367-2381. |
| [21] |
Jin MH, Oh DY. ATM in DNA repair in cancer[J]. Pharmacol Ther, 2019, 203: 107391. |
| [22] |
Cai MY, Dunn CE, Chen W, et al. Cooperation of the ATM and Fanconi anemia/BRCA pathways in double-strand break end resection[J]. Cell Rep, 2020, 30: 2402-2415. |
| [23] |
Kocher S, Rieckmann T, Rohaly G, et al. Radiation-induced double-strand breaks require ATM but not artemis for homologous recombination during S-phase[J]. Nucleic Acids Res, 2012, 40: 8336-8347. |
| [24] |
Criscuolo D, Morra F, Giannella R, et al. Identification of novel biomarkers of homologous recombination defect in DNA repair to predict sensitivity of prostate cancer cells to PARP-inhibitors[J]. Int J Mol Sci, 2019, 20: 3100. |
| [25] |
Peterson SE, Li Y, Wu-Baer F, et al. Activation of DSB processing requires phosphorylation of CtIP by ATR[J]. Mol Cell, 2013, 49: 657-667. |
| [26] |
Kim D, Liu Y, Oberly S, et al. ATR-mediated proteome remodeling is a major determinant of homologous recombination capacity in cancer cells[J]. Nucleic Acids Res, 2018, 46: 8311-8325. |
| [27] |
Smith J, Tho LM, Xu N, et al. The ATM-Chk2 and ATR-Chk1 pathways in DNA damage signaling and cancer[J]. Adv Cancer Res, 2010, 108: 73-112. |
| [28] |
Zhao W, Qiu Y, Kong D. Class I phosphatidylinositol 3-kinase inhibitors for cancer therapy[J]. Acta Pharm Sin B, 2017, 7: 27-37. |
| [29] |
Massacesi C, Di Tomaso E, Urban P, et al. PI3K inhibitors as new cancer therapeutics: implications for clinical trial design[J]. Onco Targets Ther, 2016, 9: 203-210. |
| [30] |
Kumar A, Fernandez-Capetillo O, Carrera AC. Nuclear phosphoinositide 3-kinase beta controls double-strand break DNA repair[J]. Proc Natl Acad Sci U S A, 2010, 107: 7491-7496. |
| [31] |
Ibrahim YH, Garcia-Garcia C, Serra V, et al. PI3K inhibition impairs BRCA1/2 expression and sensitizes BRCA-proficient triple-negative breast cancer to PARP inhibition[J]. Cancer Discov, 2012, 2: 1036-1047. |
| [32] |
Toulany M. Targeting DNA double-strand break repair pathways to improve radiotherapy response[J]. Genes, 2019, 10: 25. |
| [33] |
Mueck K, Rebholz S, Harati MD, et al. Akt1 stimulates homologous recombination repair of DNA double-strand breaks in a Rad51-dependent manner[J]. Int J Mol Sci, 2017, 18: 2473. |
| [34] |
Klinakis A, Karagiannis D, Rampias T. Targeting DNA repair in cancer: current state and novel approaches[J]. Cell Mol Life Sci, 2020, 77: 677-703. |
| [35] |
Chao OS, Goodman OB. Synergistic loss of prostate cancer cell viability by coinhibition of HDAC and PARP[J]. Mol Cancer Res, 2014, 12: 1755-1766. |
| [36] |
Min A, Im SA, Kim DK, et al. Histone deacetylase inhibitor, suberoylanilide hydroxamic acid (SAHA), enhances anti-tumor effects of the poly (ADP-ribose) polymerase (PARP) inhibitor olaparib in triple-negative breast cancer cells[J]. Breast Cancer Res, 2015, 17: 33. |
| [37] |
Muvarak NE, Chowdhury K, Xia L, et al. Enhancing the cytotoxic effects of PARP inhibitors with DNA demethylating agents - a potential therapy for cancer[J]. Cancer Cell, 2016, 30: 637-650. |
| [38] |
Abbotts R, Topper MJ, Biondi C, et al. DNA methyltransferase inhibitors induce a BRCAness phenotype that sensitizes NSCLC to PARP inhibitor and ionizing radiation[J]. Proc Natl Acad Sci U S A, 2019, 116: 22609-22618. |
| [39] |
Dupre A, Boyer-Chatenet L, Sattler RM, et al. A forward chemical genetic screen reveals an inhibitor of the Mre11-Rad50-Nbs1 complex[J]. Nat Chem Biol, 2008, 4: 119-125. |
| [40] |
Ying S, Hamdy FC, Helleday T. Mre11-dependent degradation of stalled DNA replication forks is prevented by BRCA2 and PARP1[J]. Cancer Res, 2012, 72: 2814-2821. |
| [41] |
Hengel SR, Spies MA, Spies M. Small-molecule inhibitors targeting DNA repair and DNA repair deficiency in research and cancer therapy[J]. Cell Chem Biol, 2017, 24: 1101-1119. |
| [42] |
Prakash R, Zhang Y, Feng W, et al. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins[J]. Cold Spring Harb Perspect Biol, 2015, 7: a016600. |
| [43] |
Chen CC, Feng W, Lim PX, et al. Homology-directed repair and the role of BRCA1, BRCA2, and related proteins in genome integrity and cancer[J]. Ann Rev Cancer Biol, 2018, 2: 313-336. |
| [44] |
Pessetto ZY, Yan Y, Bessho T, et al. Inhibition of BRCT(BRCA1)-phosphoprotein interaction enhances the cytotoxic effect of olaparib in breast cancer cells: a proof of concept study for synthetic lethal therapeutic option[J]. Breast Cancer Res Treat, 2012, 134: 511-517. |
| [45] |
Trenner A, Godau J, Sartori AA. A short BRCA2-derived cell-penetrating peptide targets RAD51 function and confers hypersensitivity toward PARP inhibition[J]. Mol Cancer Ther, 2018, 17: 1392-1404. |
| [46] |
White ER, Sun L, Ma Z, et al. Peptide library approach to uncover phosphomimetic inhibitors of the BRCA1 C-terminal domain[J]. ACS Chem Biol, 2015, 10: 1198-1208. |
| [47] |
Densham RM, Morris JR. The BRCA1 ubiquitin ligase function sets a new trend for remodelling in DNA repair[J]. Nucleus, 2017, 8: 116-125. |
| [48] |
Wiegmans AP, Miranda M, Wen SW, et al. RAD51 inhibition in triple negative breast cancer cells is challenged by compensatory survival signaling and requires rational combination therapy[J]. Oncotarget, 2016, 7: 60087-60100. |
| [49] |
Zhu J, Zhou L, Wu G, et al. A novel small molecule RAD51 inactivator overcomes imatinib-resistance in chronic myeloid leukaemia[J]. EMBO Mol Med, 2013, 5: 353-365. |
| [50] |
Chen Q, Cai D, Li M, et al. The homologous recombination protein RAD51 is a promising therapeutic target for cervical carcinoma[J]. Oncol Rep, 2017, 38: 767-774. |
| [51] |
Shi Y, Jin J, Wang X, et al. DAXX, as a tumor suppressor, impacts DNA damage repair and sensitizes BRCA-proficient TNBC cells to PARP inhibitors[J]. Neoplasia, 2019, 21: 533-544. |
| [52] |
Herrero AB, Gutierrez NC. Targeting ongoing DNA damage in multiple myeloma: effects of DNA damage response inhibitors on plasma cell survival[J]. Front Oncol, 2017, 7: 98. |
| [53] |
Nadkarni A, Shrivastav M, Mladek AC, et al. ATM inhibitor KU-55933 increases the TMZ responsiveness of only inherently TMZ sensitive GBM cells[J]. J Neurooncol, 2012, 110: 349-357. |
| [54] |
Pike KG, Barlaam B, Cadogan E, et al. The identification of potent, selective, and orally available inhibitors of ataxia telangiectasia mutated (ATM) kinase: the discovery of AZD0156[J]. J Med Chem, 2018, 61: 3823-3841. |
| [55] |
Durant ST, Zheng L, Wang Y, et al. The brain-penetrant clinical ATM inhibitor AZD1390 radiosensitizes and improves survival of preclinical brain tumor models[J]. Sci Adv, 2018, 4: eaat1719. |
| [56] |
Ning JF, Stanciu M, Humphrey MR, et al. Myc targeted CDK18 promotes ATR and homologous recombination to mediate PARP inhibitor resistance in glioblastoma[J]. Nat Commun, 2019, 10: 2910. |
| [57] |
O'Carrigan B, Luken DM, Jose M, et al. Phase Ⅰ trial of a first-in-class ATR inhibitor VX-970 as monotherapy (mono) or in combination (combo) with carboplatin (CP) incorporating pharmacodynamics (PD) studies[J]. J Clin Oncol, 2016, 34: 2504. |
| [58] |
Dillon MT, Espinasse A, Ellis S, et al. A phase Ⅰ dose-escalation study of ATR inhibitor monotherapy with AZD6738 in advanced solid tumors (PATRIOT Part A)[J]. Cancer Res, 2017, 77: CT084. |
| [59] |
Yap TA, Krebs MG, Postel-Vinay S, et al. Phase Ⅰ modular study of AZD6738, a novel oral, potent and selective ataxia telangiectasia Rad3-related (ATR) inhibitor in combination (combo) with carboplatin, olaparib or durvalumab in patients (pts) with advanced cancers[J]. Eur J Cancer, 2016, 69: S2. |
| [60] |
Tao H, Huifang L, Rongkun L, et al. Synthesis and biological evaluation of phosphoinositide 3-kinase (PI3K) inhibitors based on a quinoxaline scaffold[J]. Acta Pharm Sin (药学学报), 2020, 55: 96-105. |
| [61] |
Gallyas F, Sumegi B, Szabo C. Role of Akt activation in PARP inhibitor resistance in cancer[J]. Cancers, 2020, 12: 532. |
| [62] |
Juvekar A, Burga LN, Hu H, et al. Combining a PI3K inhibitor with a PARP inhibitor provides an effective therapy for BRCA1-related breast cancer[J]. Cancer Discov, 2012, 2: 1048-1063. |
| [63] |
Wang D, Wang M, Jiang N, et al. Effective use of PI3K inhibitor BKM120 and PARP inhibitor olaparib to treat PIK3CA mutant ovarian cancer[J]. Oncotarget, 2016, 7: 13153-13166. |
| [64] |
Philip CA, Laskov I, Beauchamp MC, et al. Inhibition of PI3K-AKT-mTOR pathway sensitizes endometrial cancer cell lines to PARP inhibitors[J]. BMC Cancer, 2017, 17: 638. |
| [65] |
Matulonis UA, Wulf GM, Barry WT, et al. Phase Ⅰ dose escalation study of the PI3kinase pathway inhibitor BKM120 and the oral poly (ADP ribose) polymerase (PARP) inhibitor olaparib for the treatment of high-grade serous ovarian and breast cancer[J]. Ann Oncol, 2017, 28: 512-518. |
| [66] |
Konstantinopoulos PA, Barry WT, Birrer M, et al. Phase Ⅰ study of the alpha specific PI3-kinase inhibitor BYL719 and the poly (ADP-Ribose) polymerase (PARP) inhibitor olaparib in recurrent ovarian and breast cancer: analysis of the dose escalation and ovarian cancer expansion cohort[J]. Cancer Res, 2017, 77: CT008. |
| [67] |
Michalarea V, Roda D, Drew Y, et al. Phase Ⅰ trial combining the PARP inhibitor olaparib (Ola) and AKT inhibitor AZD5363 (AZD) in germline (g) BRCA and non-BRCA mutant (m) advanced cancer patients (pts) incorporating noninvasive monitoring of cancer mutations[J]. Cancer Res, 2016, 76: CT010. |
| [68] |
Mo W, Liu Q, Lin CC, et al. mTOR inhibitors suppress homologous recombination repair and synergize with PARP inhibitors via regulating SUV39H1 in BRCA-proficient triple-negative breast cancer[J]. Clin Cancer Res, 2016, 22: 1699-1712. |
| [69] |
Yang L, Zhang Y, Shan W, et al. Repression of BET activity sensitizes homologous recombination-proficient cancers to PARP inhibition[J]. Sci Transl Med, 2017, 9: eaal1645. |
| [70] |
Stathis A, Bertoni F. BET proteins as targets for anticancer treatment[J]. Cancer Discov, 2018, 8: 24-36. |
| [71] |
Mio C, Gerratana L, Bolis M, et al. BET proteins regulate homologous recombination-mediated DNA repair: BRCAness and implications for cancer therapy[J]. Int J Cancer, 2019, 144: 755-766. |
| [72] |
Karakashev S, Zhu H, Yokoyama Y, et al. BET bromodomain inhibition synergizes with PARP inhibitor in epithelial ovarian cancer[J]. Cell Rep, 2017, 21: 3398-3405. |
| [73] |
Duan Y, Guan Y, Qin W, et al. Targeting Brd4 for cancer therapy: inhibitors and degraders[J]. MedChemComm, 2018, 9: 1779-1802. |
| [74] |
Donati B, Lorenzini E, Ciarrocchi A. BRD4 and cancer: going beyond transcriptional regulation[J]. Mol Cancer, 2018, 17: 164. |
| [75] |
Sun C, Yin J, Fang Y, et al. BRD4 inhibition is synthetic lethal with PARP inhibitors through the induction of homologous recombination deficiency[J]. Cancer Cell, 2018, 33: 401-416. |
| [76] |
Ramalingam SS, Kummar S, Sarantopoulos J, et al. Phase Ⅰ study of vorinostat in patients with advanced solid tumors and hepatic dysfunction: a national cancer institute organ dysfunction working group study[J]. J Clin Oncol, 2010, 28: 4507-4512. |
| [77] |
Su D, Ma S, Shan L, et al. Ubiquitin-specific protease 7 sustains DNA damage response and promotes cervical carcinogenesis[J]. J Clin Invest, 2018, 128: 4280-4296. |
| [78] |
Li Y, Luo K, Yin Y, et al. USP13 regulates the RAP80-BRCA1 complex dependent DNA damage response[J]. Nat Commun, 2017, 8: 15752. |
| [79] |
Song Z, Tu X, Zhou Q, et al. A novel UCHL3 inhibitor, perifosine, enhances PARP inhibitor cytotoxicity through inhibition of homologous recombination-mediated DNA double strand break repair[J]. Cell Death Dis, 2019, 10: 398. |
| [80] |
Ma T, Chen Y, Zhang F, et al. RNF111-dependent neddylation activates DNA damage-induced ubiquitination[J]. Mol Cell, 2013, 49: 897-907. |
| [81] |
Guo ZP, Hu YC, Xie Y, et al. MLN4924 suppresses the BRCA1 complex and synergizes with PARP inhibition in NSCLC cells[J]. Biochem Biophys Res Commun, 2017, 483: 223-229. |
| [82] |
Sulkowski PL, Corso CD, Robinson ND, et al. 2-Hydroxyglutarate produced by neomorphic IDH mutations suppresses homologous recombination and induces PARP inhibitor sensitivity[J]. Sci Transl Med, 2017, 9: eaal2463. |
| [83] |
Qu J, Sun W, Zhong J, et al. Phosphoglycerate mutase 1 regulates dNTP pool and promotes homologous recombination repair in cancer cells[J]. J Cell Biol, 2017, 216: 409-424. |
| [84] |
Okazaki A, Gameiro PA, Christodoulou D, et al. Glutaminase and poly(ADP-ribose) polymerase inhibitors suppress pyrimidine synthesis and VHL-deficient renal cancers[J]. J Clin Invest, 2017, 127: 1631-1645. |
| [85] |
Fang P, De Souza C, Minn K, et al. Genome-scale CRISPR knockout screen identifies TIGAR as a modifier of PARP inhibitor sensitivity[J]. Commun Biol, 2019, 2: 335. |
| [86] |
Borst GR, Kumareswaran R, Yücel H, et al. Neoadjuvant olaparib targets hypoxia to improve radioresponse in a homologous recombination-proficient breast cancer model[J]. Oncotarget, 2017, 8: 87638-87646. |
| [87] |
Bindra RS, Schaffer PJ, Meng A, et al. Down-regulation of Rad51 and decreased homologous recombination in hypoxic cancer cells[J]. Mol Cell Biol, 2004, 24: 8504-8518. |
| [88] |
Ray-Coquard I, Pautier P, Pignata S, et al. Olaparib plus bevacizumab as first-line maintenance in ovarian cancer[J]. N Engl J Med, 2019, 381: 2416-2428. |
| [89] |
Kaplan AR, Gueble SE, Liu Y, et al. Cediranib suppresses homology-directed DNA repair through down-regulation of BRCA1/2 and RAD51[J]. Sci Transl Med, 2019, 11: eaav4508. |
| [90] |
Liu JF, Barry WT, Birrer M, et al. Overall survival and updated progression-free survival outcomes in a randomized phase Ⅱ study of combination cediranib and olaparib versus olaparib in relapsed platinum-sensitive ovarian cancer[J]. Ann Oncol, 2019, 30: 551-557. |
| [91] |
Wang C, Chen SZ. Advances in the mechanisms of acquired resistance to EGFR-tyrosine kinase inhibitors in non-small cell lung cancer[J]. Acta Pharm Sin (药学学报), 2019, 54: 1364-1371. |
| [92] |
Nowsheen S, Cooper T, Stanley JA, et al. Synthetic lethal interactions between EGFR and PARP inhibition in human triple negative breast cancer cells[J]. PLoS One, 2012, 7: e46614. |
| [93] |
Li L, Wang H, Yang ES, et al. Erlotinib attenuates homologous recombinational repair of chromosomal breaks in human breast cancer cells[J]. Cancer Res, 2008, 68: 9141-9146. |
| [94] |
Asim M, Tarish F, Zecchini HI, et al. Synthetic lethality between androgen receptor signalling and the PARP pathway in prostate cancer[J]. Nat Commun, 2017, 8: 374. |
| [95] |
Virtanen V, Paunu K, Ahlskog JK, et al. PARP inhibitors in prostate cancer-the preclinical rationale and current clinical development[J]. Genes, 2019, 10: 565. |
| [96] |
Clarke N, Wiechno P, Alekseev B, et al. Olaparib combined with abiraterone in patients with metastatic castration-resistant prostate cancer: a randomised, double-blind, placebo-controlled, phase 2 trial[J]. Lancet Oncol, 2018, 19: 975-986. |
| [97] |
Mladenov E, Magin S, Soni A, et al. DNA double-strand break repair as determinant of cellular radiosensitivity to killing and target in radiation therapy[J]. Front Oncol, 2013, 3: 113. |
| [98] |
Tu X, Kahila MM, Zhou Q, et al. ATR inhibition is a promising radiosensitizing strategy for triple-negative breast cancer[J]. Mol Cancer Ther, 2018, 17: 2462-2472. |
| [99] |
Pal S, Kozono D, Yang X, et al. Dual HDAC and PI3K inhibition abrogates NFκB- and FOXM1-mediated DNA damage response to radiosensitize pediatric high-grade gliomas[J]. Cancer Res, 2018, 78: 4007-4021. |
| [100] |
Tang S, Li Z, Yang L, et al. A potential new role of ATM inhibitor in radiotherapy: suppressing ionizing radiation-activated EGFR[J]. Int J Radiat Biol, 2020. DOI:10.1080/09553002.2020.1707325 |
| [101] |
Gol TM, Rodemann HP, Dittmann K. Depletion of Akt1 and Akt2 impairs the repair of radiation-induced DNA double strand breaks via homologous recombination[J]. Int J Mol Sci, 2019, 20: 6316. |
| [102] |
Gottgens EL, Bussink J, Leszczynska KB, et al. Inhibition of CDK4/CDK6 enhances radiosensitivity of HPV negative head and neck squamous cell carcinomas[J]. Int J Radiat Oncol Biol Phys, 2019, 105: 548-558. |
| [103] |
Mei X, Cate RT, van Leeuwen CM, et al. Radiosensitization by hyperthermia: the effects of temperature, sequence, and time interval in cervical cell lines[J]. Cancers, 2020, 12: 582. |
| [104] |
Willers H, Dahm-Daphi J, Powell SN. Repair of radiation damage to DNA[J]. Br J Cancer, 2004, 90: 1297-1301. |
| [105] |
Bhattacharya S, Srinivasan K, Abdisalaam S, et al. RAD51 interconnects between DNA replication, DNA repair and immunity[J]. Nucleic Acids Res, 2017, 45: 4590-4605. |
| [106] |
Berte N, Piee-Staffa A, Piecha N, et al. Targeting homologous recombination by pharmacological inhibitors enhances the killing response of glioblastoma cells treated with alkylating drugs[J]. Mol Cancer Ther, 2016, 15: 2665-2678. |
| [107] |
Gil Del Alcazar CR, Todorova PK, Habib AA, et al. Augmented HR repair mediates acquired temozolomide resistance in glioblastoma[J]. Mol Cancer Res, 2016, 14: 928-940. |
| [108] |
Erasimus H, Gobin M, Niclou S, et al. DNA repair mechanisms and their clinical impact in glioblastoma[J]. Mutat Res Rev Mutat Res, 2016, 769: 19-35. |
| [109] |
Mouw KW, Konstantinopoulos PA. From checkpoint to checkpoint: DNA damage ATR/Chk1 checkpoint signalling elicits PD-L1 immune checkpoint activation[J]. Br J Cancer, 2018, 118: 933-935. |
| [110] |
Parkes EE, Walker SM, Taggart LE, et al. Activation of sting-dependent innate immune signaling by S-phase-specific DNA damage in breast cancer[J]. J Natl Cancer Inst, 2016, 109: djw199. |
| [111] |
McGrail DJ, Federico L, Li Y, et al. Multi-omics analysis reveals neoantigen-independent immune cell infiltration in copy-number driven cancers[J]. Nat Commun, 2018, 9: 1317. |
| [112] |
Turajlic S, Litchfield K, Xu H, et al. Insertion-and-deletion-derived tumour-specific neoantigens and the immunogenic phenotype: a pan-cancer analysis[J]. Lancet Oncol, 2017, 18: 1009-1021. |
| [113] |
Sato H, Niimi A, Yasuhara T, et al. DNA double-strand break repair pathway regulates PD-L1 expression in cancer cells[J]. Nat Commun, 2017, 8: 1751. |
| [114] |
Vendetti FP, Karukonda P, Clump DA, et al. ATR kinase inhibitor AZD6738 potentiates CD8+ T cell-dependent antitumor activity following radiation[J]. J Clin Invest, 2018, 128: 3926-3940. |
| [115] |
Sun L, Yang R, Li C, et al. Inhibition of ATR downregulates PD-L1 and sensitizes tumor cells to T cell-mediated killing[J]. Am J Cancer Res, 2018, 8: 1307-1316. |
| [116] |
Kim G, Ison G, McKee AE, et al. FDA approval summary: olaparib monotherapy in patients with deleterious germline BRCA-mutated advanced ovarian cancer treated with three or more lines of chemotherapy[J]. Clin Cancer Res, 2015, 21: 4257-4261. |
| [117] |
Mirza MR, Monk BJ, Herrstedt J, et al. Niraparib maintenance therapy in platinum-sensitive, recurrent ovarian cancer[J]. N Engl J Med, 2016, 375: 2154-2164. |
| [118] |
Hodgson DR, Dougherty BA, Lai Z, et al. Candidate biomarkers of PARP inhibitor sensitivity in ovarian cancer beyond the BRCA genes[J]. Br J Cancer, 2018, 119: 1401-1409. |
| [119] |
Goodall J, Mateo J, Yuan W, et al. Circulating cell-free DNA to guide prostate cancer treatment with PARP inhibition[J]. Cancer Discov, 2017, 7: 1006-1017. |
| [120] |
Xu C, Cao H, Shi C, et al. The role of circulating tumor DNA in therapeutic resistance[J]. Onco Targets Ther, 2019, 12: 9459-9471. |
| [121] |
Peng G, Chun-Jen Lin C, Mo W, et al. Genome-wide transcriptome profiling of homologous recombination DNA repair[J]. Nat Commun, 2014, 5: 3361. |
| [122] |
Fang Y, McGrail DJ, Sun C, et al. Sequential therapy with PARP and WEE1 inhibitors minimizes toxicity while maintaining efficacy[J]. Cancer Cell, 2019, 35: 851-867. |
 2020, Vol. 55
2020, Vol. 55


