2. 中国科学院上海药物研究所药物发现与设计中心, 上海 201203;
3. 中国科学院大学药学院, 北京 100049
2. Drug Discovery and Design Center, Shanghai Institute of Materia Medica, Chinese Academy of Sciences, Shanghai 201203, China;
3. School of Pharmacy, University of Chinese Academy of Sciences, Beijing 100049, China
冠状病毒得名于其显微镜下呈日冕状的棘突。自1966年分离得到第一株人类冠状病毒HCoV-229E, 人们已陆续发现了7种致病性冠状病毒, 即HCoV-229E、HCoV-OC43、HCoV-HKU1、HCoV-NL63、SARS-CoV、MERS-CoV和SARS-CoV-2。其中前4种冠状病毒会引发较轻微症状的普通感冒, 而后3种会导致严重病症且传染性强, 甚至引发病毒性肺炎致死。例如, SARS-CoV引发的严重急性呼吸综合征(非典型肺炎, SARS)是一种急性传染性呼吸系统疾病, 自2002年11月于中国广东出现首例患者, 迅速蔓延至32个国家和地区, 累计造成感染病例8 422例, 死亡率10%~15%。而中东呼吸综合征病毒(MERS-CoV)自2012年9月于沙特出现首例患者以来, 累计造成感染病例1 401例, 死亡率高达39%, 严重威胁了人类生命健康。SARS-CoV-2引发的新型冠状病毒肺炎(COVID-19)自2019年12月发现以来, 蔓延全球, 传染性极强, 以至现今疫情仍未结束且形势严峻, 迫切需要开发特效药。尽管人们针对这些致病性冠状病毒的药物研发投入巨大, 也取得了一些进展, 但迄今为止仍没有上市抗冠状病毒特效药, 目前COVID-19的治疗手段仍是常规的抗病毒和抗菌药物。
COVID-19疫情的暴发催生了前赴后继的抗冠状病毒药物研发的热潮。不断报道的活性化合物不仅给人们带来曙光, 也为后续药物研究开发提供可借鉴的思路。2020年1月, Pillaiyar等[1]总结了靶向18种涉及SARS-CoV和MERS-CoV感染过程的潜在靶标的活性分子研究进展。同年2月, 刘千勇等[2]总结了针对冠状病毒和宿主细胞的关键成药靶点, 选取总结了75个临床在研的靶向药物, 其中瑞德西韦和克力芝正启动针对COVID-19的III期临床试验; 王赫然等[3]聚焦COVID-19化学药、中药和生物制品并阐述研究进展情况。张超等[4]对14种注册用于新冠肺炎治疗临床试验的化学药物进行了总结和分析。由此可见, COVID-19疫情受到世界各国的广泛关注, SARS-CoV-2病毒相关药物的信息整理正为尽早控制疫情贡献重要力量。我们注意到, 针对SARS-CoV-2的药物研发工作进展迅速, 特效药需求的紧迫性也与日俱增, 更加完全的信息整理可以帮助科研工作者掌握最新动态, 开阔思路, 探索新颖靶标。因此, 覆盖COVID-19所有已知相关靶标的药物研发最新进展的信息整理毫无疑问是此次科研攻关所需要的。为助力广大科研工作者系统地了解COVID-19相关活性化合物的研究进展, 本综述分类总结了近20年来报道的冠状病毒活性化合物, 对于结构相似的化合物则以活性强者为代表进行归纳, 共列举了146个活性化合物, 同时总结了与这些活性化合物作用的蛋白质(潜在靶标蛋白)共26个。对那些具有抗冠状病毒活性但机制不明确的化合物, 本研究组通过COVID-19靶标预测平台D3Docking预测了它们的潜在靶标, 以期为药理及药化研究工作者提供参考信息。
1 抗冠状病毒药物的相关靶标蛋白冠状病毒是一类具有囊膜结构的单链RNA病毒, 直径约80~120 nm, 由结构蛋白、非结构蛋白和辅助蛋白组成。这三类蛋白与病毒的入侵和复制密切相关, 因此被认为是抗冠状病毒药物的重要靶标, 如刺突糖蛋白和主蛋白酶等。另一方面, 在病毒感染的过程中, 一些宿主蛋白也扮演着不可或缺的角色, 如病毒刺突糖蛋白需要与宿主血管紧张素转换酶2 (ACE2)等结合才能顺利入侵宿主, 因此, 这些宿主蛋白也成为设计抗冠状病毒药物的突破方向。在收集整理抗冠状病毒活性化合物的同时, 也系统地开展文献和数据库检索, 整理出了26个与这些活性化合物作用的蛋白质, 它们与冠状病毒的侵入、复制和释放等过程紧密相关, 是潜在的靶标蛋白, 其中包括9个病毒编码蛋白(表 1)和17个病毒感染相关的宿主蛋白(表 2)。
| Table 1 Virus-encoded proteins and the active compounds |
| Table 2 Human proteins related to virus invasion, replication and transmission and the active compounds |
刺突糖蛋白(spike glycoprotein, S protein)可以在病毒膜表面形成突出的同源三聚体, 每个单体可被蛋白酶切割成两个功能亚基。其中, S1亚基具有受体结合区域, 负责与宿主细胞表面上如ACE2受体结合识别宿主。S2亚基负责病毒与宿主细胞膜的融合, 因此刺突糖蛋白是病毒侵入宿主的关键蛋白, 也被认为是重要的抗冠状病毒靶标[5]。据报道, 化合物1 (SSAA09E2, 图 1)可抑制SARS-CoV刺突糖蛋白与ACE2的结合, 在293T细胞假病毒试验中的EC50 = 3.1 ± 0.2 μmol·L-1, CC50值大于100 μmol·L-1 [6]。化合物2 (pradimicin A, 普那米星A, 图 1)是从放线菌中分离得到的一种抗真菌非肽类抗生素, Van等[7]研究报道它能够与SARS-CoV刺突糖蛋白结合, EC50达到2.0~5.7 μmol·L-1。另外, 研究表明普那米星A也可能与M蛋白结合, 因此它可能是一种多靶标天然化合物。化合物3 (dihydrotanshinone, 二氢丹参酮, 图 1)是Kim等[8]利用MERS-CoV刺突糖蛋白进行高通量筛选得到的一种亲酯化合物, 在 > 2 μg·mL-1时表现出强的抗MERS-CoV作用。
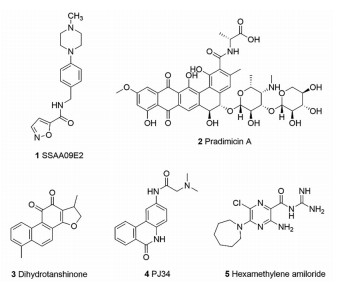
|
Figure 1 Representative compounds targeting coronavirus S pro‐ tein, N protein and E protein |
核衣壳蛋白(nucleocapsid phosphoprotein, N protein)是一种冠状病毒结构蛋白, 位于病毒颗粒的核心部分, 与病毒的复制和转录有关[9]。核衣壳蛋白具有3个不同且高度保守的结构域:N端结构域和C端结构域, 以及中央无序区域(RNA结合域)。其N端结合域可与RNA形成核糖核蛋白复合物, 通过与病毒基因组和膜蛋白的相互作用, 在病毒体装配过程中发挥着重要作用[10]。化合物4 (PJ34, 图 1)由Lin等[11]通过HCoV-OC43核衣壳蛋白N端结构域虚拟筛选获得, 实验证明其可以特异性结合在核衣壳蛋白N端结构域的核糖核酸结合口袋中, 在10 μmol·L-1时可有效抑制冠状病毒的复制过程。
2.3 包膜蛋白包膜蛋白(envelope protein, E protein)包含一个短的亲水性氨基末端、一个疏水区域和一个亲水性羧基末端[12], 主要存在于内质网-高尔基体之间, 参与病毒形态发生和组装, 此外也参与调节细胞应激反应和细胞凋亡[13]。包膜蛋白可充当病毒孔蛋白, 并在宿主膜中自组装, 形成允许离子运输的五聚体离子通道, 膜的脂质组成也会影响该离子通道的活性[14, 15]。Pervushin等[16]发现化合物5 (hexamethylene amiloride, 六亚甲基酰胺, 图 1)可有效抑制SARS-CoV与HCoV-229E包膜蛋白, 作用于包膜蛋白离子通道从而抑制病毒复制, 对于HCoV-229E的抑制活性EC50达到1 μmol·L-1。并通过分子对接进一步提出了六亚甲基酰胺在包膜蛋白中的两种可能结合模式, 一种是在包膜蛋白N15附近, 其氧原子与N15形成氢键; 另一种是在包膜蛋白C端, 与T35存在范德华相互作用。
2.4 主蛋白酶主蛋白酶(main protease, Mpro), 又称3-糜样蛋白酶(3-chymotrypsin-like protease, 3CLpro), 可以剪切病毒编码的复制酶多聚蛋白使其组装成复制酶复合物, 帮助病毒转录与复制, 因此也是抗冠状病毒的理想靶标[17, 18]。冠状病毒基因组包含两个开放阅读框ORF1a和ORF1b, 它们被宿主核糖体翻译成病毒复制酶多聚蛋白pp1a和pp1ab。主蛋白酶通过剪切pp1a多聚蛋白上的11个位点, 从而释放病毒复制和转录需要的多种非结构蛋白, 包括RNA依赖的RNA多聚酶和解旋酶等[19]。
天然产物是新药创造的源泉, 多种天然产物表现出SARS-CoV主蛋白酶的抑制活性。从雷公藤甲醇(95%)提取物中分离出的一系列三萜类化合物6~9 (celastrol, IC50 = 2.6 μmol·L-1; pritimerrin, IC50 = 9.9 μmol·L-1; tingenone, IC50 = 5.5 μmol·L-1; iguesterin, IC50 = 10.3 μmol·L-1, 图 2), 均表现出对SARS-CoV主蛋白酶的抑制活性[20]。萜类天然产物化合物10 (betulinic acid, IC50 = 10 μmol·L-1, 图 2)和化合物11 (savinin, IC50 = 25 μmol·L-1, 图 2)也被证明具有SARS-CoV主蛋白酶抑制活性[21]。另外, 从香椿中提取双黄酮的化合物12 (biflavone amentoflavone, IC50 = 8.3 μmol·L-1, 图 2)是一种SARS-CoV主蛋白酶的非竞争性抑制剂[22], 而从褐藻中提取的化合物13二鹅掌菜酚(dieckol, IC50 = 2.7 μmol·L-1, 图 2), 具有更强的SARS-CoV主蛋白酶抑制活性[23]。
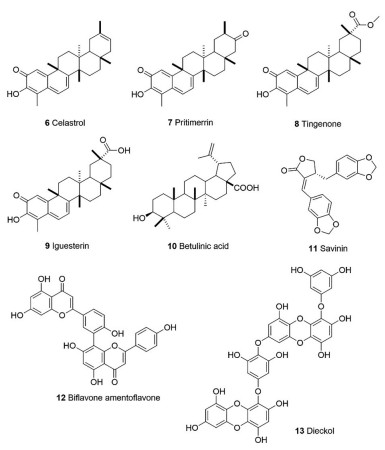
|
Figure 2 Representative natural products targeting coronavirus 3CLpro |
冠状病毒主蛋白酶的生物学研究给药物研究提供了强有力的理论指导。例如, 基于SARS-CoV主蛋白酶的催化二联体C145和H41这两个关键的活性位点, 共价结合C145以及与H41形成弱相互作用是很多活性化合物的设计方向。Yang等[24]设计合成的化合物14 (图 3)是一种新型的SARS-CoV主蛋白酶醛肽抑制剂, 通过与C145形成共价作用和氢键作用, 与F140、G143、H163、E166、Q189存在氢键和疏水作用(图 4a), 其Ki达到53 nmol·L-1。Ghosh等[25]设计合成了一系列含有迈克尔受体(Michael acceptor)的类肽抑制剂(15~20, 图 3)。相比化合物16 (Kincat = 0.045 min-1)能与SARS-CoV主蛋白酶C145共价结合, 以及与H164和E166形成氢键作用(图 4b), 化合物17 (Kincat = 0.0025 min-1)羟基取代羰基氧原子不能与E166形成氢键而活性下降; 化合物19 (IC50 = 80 μmol·L-1)与C145共价结合, 与H163和E166形成氢键作用(图 4c), 结构由异噁唑替换成Boc-丝氨酸活性增强[26]。由此可见, 除了C145参与的共价作用, E166参与的氢键作用对这类化合物的活性也是至关重要的。Shie等[27]通过对接研究发现化合物21 (nitroanilide peptide, 硝基苯胺肽, 图 3)的硝基能与SARS-CoV主蛋白酶上H41和A46形成氢键, 二甲胺-苯基结构又与P68、M165、Q189和Q192存在相互作用, 因此硝基苯胺肽对SARS-CoV主蛋白酶的抑制具有高选择性(IC50 = 0.06 μmol·L-1, Ki = 0.03 μmol·L-1)。Ghosh等[28]合成的5-氯吡啶酯衍生物22 (GRL-001, 图 3)与SARS-CoV主蛋白酶C145形成共价相互作用, 与H41形成π-π堆叠相互作用, 能高效抑制SARS-CoV, IC50达到30 nmol·L-1, EC50为6.9 μmol·L-1。Kao等[29]从50 240个小分子中高通量筛选出SARS-CoV主蛋白酶抑制剂化合物23 (MP576, 图 3), AutoDock显示通过与主蛋白酶的C145和H41形成氢键作用而结合在活性位点, 其IC50达到2.5 μmol·L-1, 体外测试具有较强的抗病毒活性(EC50 < 10 μmol·L-1)。另有文献报道, SARS-CoV主蛋白酶会在R188和G189位点处被降解, 因此Konno等[30-32]发现了抑制主蛋白酶R188I突变体的丝氨酸衍生物化合物24 (SK23, IC50 = 30 μmol·L-1, 图 3)、苯基异丝氨酸衍生物化合物25 (SK40, IC50 = 43 μmol·L-1, 图 3)和化合物26 (IC50 = 26 μmol·L-1, 图 3)均对主蛋白酶有抑制活性。这些重要的生物信息对计算机辅助药物设计也是不可或缺的。从化合物库中筛选得到化合物27、28 (aza-peptide epoxide、aziridine peptide, 图 3)能与C145共价结合, 抑制SARS-CoV主蛋白酶, 其中化合物27的Kincat/Ki值为~1 900 (mol·L-1)-1·s-1, 化合物28在100 μmol·L-1有54%的抑制活性[33, 34]。基于含8 000多种现有药物结构信息的数据库, Chen等[35]通过虚拟筛选获得化合物29 (cinanserin, 肉桂硫胺, 图 3), 实验证明其对SARS-CoV主蛋白酶和HCoV-229E主蛋白酶的抑制活性IC50分别达到4.92和4.68 μmol·L-1。
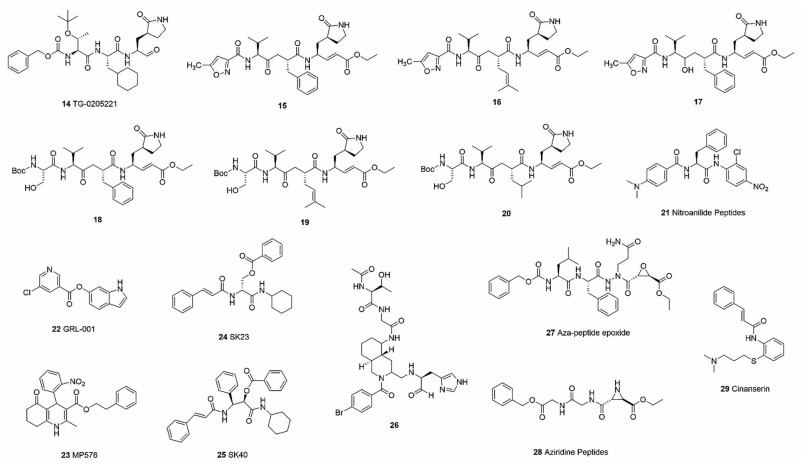
|
Figure 3 Representative compounds targeting coronavirus 3CLpro |

|
Figure 4 Binding mode of compound 14 (PDB ID: 2GX4), 16 (PDB ID: 2ALV) and 19 (PDB ID: 2QIQ) to SARS-CoV 3CLpro. The com‐ pounds are shown in green sticks, the crucial residues with ligands are shown in blue sticks |
目前多篇针对SARS-CoV主蛋白酶抑制剂的报道还得益于相似蛋白酶结构的抑制剂研究。例如, Shao等[36]基于HIV蛋白酶对称肽抑制剂进行结构改造, 得到的化合物30和31 (图 5)能有效抑制SARS-CoV主蛋白酶, 其Ki分别为0.34和0.073 μmol·L-1, 并且化合物31对SARS-CoV主蛋白酶的抑制还具有强特异性。基于鼻病毒主蛋白酶抑制剂isatin, Chen等[37]设计其衍生物化合物32、33 (图 5), 实验表明它们在低微摩尔范围内能抑制SARS-CoV主蛋白酶, IC50均在1 μmol·L-1以内。基于神经氨酸酶抑制剂骨架, Kumar等[38]合成化合物34~36 (图 5)具有SARS-CoV和MERS-CoV主蛋白酶低微摩尔抑制活性(34: SARS-CoV IC50 = 6.0 ± 1.2 μmol·L-1、MERS-CoV IC50 = 7.3 ± 2.1 μmol·L-1; 35: SARS-CoV IC50 = 5.8 ± 1.5 μmol·L-1、MERS-CoV IC50 = 7.4 ± 2.2 μmol·L-1; 36: SARS-CoV IC50 = 8.6 ± 2.1 μmol·L-1、MERS-CoV IC50 = 7.7 ± 2.2 μmol·L-1)。基于肠道病毒71 (EV71)蛋白酶的类肽抑制剂, Kumar等[39]设计了化合物37~39 (图 5)能有效抑制MERS-CoV主蛋白酶, 其IC50分别达到2.4、4.7和1.7 μmol·L-1, EC50值均为0.6~1.4 μmol·L-1, 对SARS-CoV主蛋白酶有更好的抑制活性, IC50分别达到0.7、0.5和0.2 μmol·L-1, 并进一步指出化合物37~39具有对α和β类CoV病毒的广谱抗病毒活性。由此可见, 其他冠状病毒蛋白酶的抑制剂研究也能为这次疫情攻关提供重要参考。Kim等[40]设计的化合物40 (GC376, 图 5)对MERS-CoV和SARS-CoV主蛋白酶均有抑制作用, 其IC50值为1.56和4.35 μmol·L-1。基于GC376的结构改造, Galasiti kankanamalage等[41]获得了化合物41 (GC813, 图 5)对MERS-CoV活性有进一步提高(40与41的EC50分别为0.9和0.5 μmol·L-1), 以及一系列含哌啶结构的活性化合物(42: SARS-CoV IC50 = 5.1 μmol·L-1、MERS-CoV IC50 = 0.4 μmol·L-1; 43: SARS-CoV IC50 = 6.3 μmol·L-1、MERS-CoV IC50 = 0.7 μmol·L-1; 44: SARS-CoV IC50 = 5.5 μmol·L-1、MERS-CoV IC50 = 6.1 μmol·L-1; 45: SARS-CoV IC50 = 3.2 μmol·L-1、MERS-CoV IC50 = 0.6 μmol·L-1, 图 5), 其中化合物40和41的CC50 > 100 μmol·L-1。Karypidou等[42]发现1, 2, 3-三唑衍生物46 (图 5)对HCoV-229E主蛋白酶有抑制活性, EC50 = 8.95 μmol·L-1, 也有可能对其他冠状病毒主蛋白酶有抑制活性。但利弊相生, 这类抑制剂的发现也可能伴随着选择性的问题。
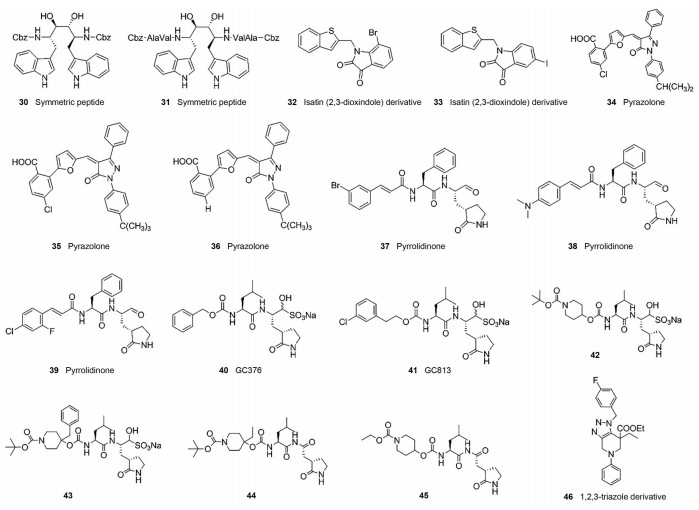
|
Figure 5 Representative compounds targeting coronavirus 3CLpro |
此外, 安全性和有效性也是药物研发的追求目标。Wu等[43]发现嘧硫草醚衍生物47 (图 6)与SARS-CoV主蛋白酶结合存在多种相互作用, 与T26、L27、F140、L141、G143、H163、E166和H172形成疏水相互作用, 与N142、S144和C145存在氢键作用, 因此能有效抑制SARS-CoV, IC50达到4.47 μmol·L-1, 并且对哺乳动物293T细胞具有低细胞毒性。Zhang等[44]发现化合物48和化合物49 (图 6)不仅能有效抑制SARS-CoV主蛋白酶, 而且对小鼠具有低毒性, 因此可以作为动物药效研究的候选化合物。
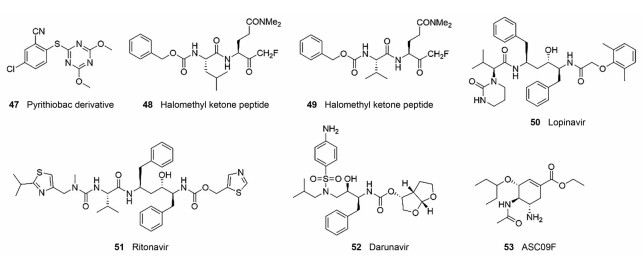
|
Figure 6 Representative compounds targeting coronavirus 3CLpro |
面对疫情暴发的紧迫性, “老药新用”的研发策略能大大缩短药物研发的时间成本, 在安全性上有一定的保障, 是目前对抗疫情的有效手段。化合物50~53 (lopinavir、ritonavir、darunavir和ASC09F, 图 6)都是FDA批准的HIV-1蛋白酶抑制剂, 有文献报道他们也能有效抑制SARS-CoV主蛋白酶[45, 46]。另有文献报道, lopinavir和ritonavir还能有效抑制MERS-CoV主蛋白酶, 其抑制活性EC50分别为11.6和24.9 μmol·L-1, CC50值均大于50 μmol·L-1 [47-49]。低剂量的lopinavir能抑制ritonavir的肝脏代谢, 因此药物联用能进一步提高药物的抗病毒治疗效果。目前已有多种药物联用策略对抗新冠病毒, 例如lopinavir与ritonavir等, 其中darunavir与科比司他(cobicistat)联用、ASC09F与奥司他韦(oseltamivir)联用已进入III期临床试验[46]。
2.5 木瓜样蛋白酶木瓜样蛋白酶(papain-like protease, PLpro)对于病毒复制至关重要, 也参与通过宿主细胞蛋白去泛素化或ISG修饰阻断宿主细胞对病毒感染的先天免疫应答[50]。多种天然产物表现出对木瓜样蛋白酶的抑制活性。从构树中提取的多酚衍生物54~55 (图 7)能够非竞争性抑制SARS-CoV木瓜样蛋白酶活性, 其IC50分别为9.2和3.7 μmol·L-1, 并且化合物55还有较好的抑制SARS-CoV木瓜样蛋白酶去泛素化作用。但对MERS-CoV木瓜样蛋白酶, 化合物54 (IC50 = 42.1 μmol·L-1)和化合物55 (IC50 = 112.5 μmol·L-1)表现较弱的抑制作用[51]。从赤杨中提取的二芳基庚烷类化合物56 (diarylheptanoid, IC50 = 4.1 ± 0.3 μmol·L-1, 图 7)[52], 从丹参中提取的化合物57 (tanshinone, IC50 = 0.8 ± 0.2 μmol·L-1, 图 7)[53]以及从毛泡桐树果实中提取的化合物58 (geranylated flavonoid, IC50 = 5.0 ± 0.06 μmol·L-1, 图 7)都具有SARS-CoV木瓜样蛋白酶的抑制活性[54]。

|
Figure 7 Representative compounds targeting coronavirus PLpro |
通过高通量筛选, Lee等[55]从25 000个化合物中得到木瓜样蛋白酶抑制剂59 (F2124-0890, 图 7), 它既是MERS-CoV的竞争性抑制剂(IC50 = 6.0 μmol·L-1), 也是SARS-CoV的变构抑制剂(IC50 = 11 μmol·L-1)。Chou等[56]从960个化学分子库中筛选出两种含硫羰基的SARS-CoV木瓜样蛋白酶抑制剂60 (6MP, IC50 = 21.6 μmol·L-1, 图 7)和化合物61 (6TG, IC50 = 5 μmol·L-1, 图 7)。另外化合物60~62对MERS-CoV木瓜样蛋白酶也有抑制作用(60, IC50 = 26.9 μmol·L-1; 61, IC50 = 24.4 μmol·L-1; 62, N-ethylmaleimdie, IC50 = 45 μmol·L-1, 图 7)[57]。有文献报道戒酒药物双硫仑(63, disulfiram, 图 7)能与化合物61和62发挥协同抑制作用, 同时也是MERS-CoV的变构抑制剂(IC50 = 22.7 μmol·L-1)和SARS-CoV的竞争抑制剂(IC50 = 14.2 μmol·L-1)[58]。Ratia等[59]从50 080个化合物中筛选出两个含萘甲胺基团的SARS-CoV木瓜样蛋白酶的抑制剂64 (IC50 = 20.1 ± 1.1 μmol·L-1, 图 7)和化合物65 (IC50 = 59 ± 7.8 μmol·L-1, 图 7)。对化合物64结构改造获得化合物66 (IC50 = 0.6 ± 0.1 μmol·L-1, EC50 = 14.5 ± 0.8 μmol·L-1, 图 7), 对化合物65结构改造获得化合物67~71均具有SARS-CoV抑制活性(67, IC50 = 0.32 ± 0.01 μmol·L-1, EC50 = 9.1 ± 0.5 μmol·L-1; 68, IC50 = 0.15 ± 0.01 μmol·L-1, EC50 = 5.4 ± 0.6 μmol·L-1; 69, IC50 = 0.49 ± 0.01 μmol·L-1, EC50 = 11.6 ± 0.8 μmol·L-1; 70, IC50 = 0.39 ± 0.01 μmol·L-1, EC50 = 8.3 ± 0.6 μmol·L-1; 71, IC50 = 0.35 ± 0.02 μmol·L-1, EC50 = 9.5 ± 1.3 μmol·L-1, 图 7)[60], 并且小鼠肝脏的微粒体实验表明化合物70和71具有明显的代谢稳定性, 是推进体内研究的可行候选化合物[61]。
2.6 RNA依赖的RNA多聚酶RNA依赖的RNA多聚酶(RNA-dependent RNA polymerase, RdRp)负责病毒RNA基因组的复制和转录, 是重要的抗病毒靶标之一[62]。RdRp与Nsp7、Nsp8蛋白形成三元复合物, 催化冠状病毒RNA的合成, 在SARS-CoV-2的复制和转录周期中起着核心作用[63]。文献报道化合物72 (法匹拉韦, 图 8)在对MERS-CoV感染6天的小鼠中治疗效果达到100%,并且对SARS-CoV-2的RNA多聚酶具有一定的抑制活性(EC50 = 61.88 μmol·L-1; CC50 > 400 μmol·L-1)[64, 65]。化合物73 (利巴韦林, 图 8)在体内被代谢成核苷类似物, 能阻断病毒RNA的合成, 曾与干扰素α2b、α2a、β1a联合用药治疗MERS-CoV感染患者, 对SARS-CoV-2也具有抑制活性(EC50 = 109.5 μmol·L-1; CC50 > 400 μmol·L-1)[46, 65]。化合物74 (图 8)对SARS-CoV RNA多聚酶的抑制活性EC90为6 μmol·L-1, 活性高且选择性强[66]。化合物75瑞德西韦(图 8)对SARS-CoV-2的抑制活性EC50达到0.77 μmol·L-1, 现已进入III期临床试验[65]。
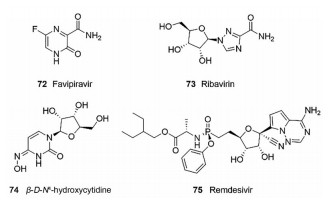
|
Figure 8 Representative compounds targeting coronavirus RdRp |
解旋酶(helicase)由3个主要结构域组成: N端金属结合域(MBD)、铰链结构域和NTPase/解旋酶结构域。解旋酶以依赖NTP的方式催化双链核苷酸解旋成单链结构, 参与病毒的复制过程[67, 68]。重要的是, 解旋酶由于其序列保守性和在所有冠状病毒中的必要性, 已被确定为开发抗病毒药物的理想靶标之一, 其上的NTPase/解旋酶结构域是设计解旋酶抑制剂的重要靶点[69, 70]。
文献报道化合物76 (图 9)能同时抑制SARS-CoV解旋酶ATPase活性(IC50 = 4 μmol·L-1)和helicase活性(IC50 = 11 μmol·L-1), 可用作开发多靶点或广谱抗病毒药物[71]。Tanner等[72]的SARS-CoV解旋酶抑制剂化合物77 (巴南宁, ATPase IC50 = 2.3 μmol·L-1; helicase IC50 = 3.0 μmol·L-1, 图 9)及其衍生物78~80都同时抑制解旋酶的ATPase和helicase活性(78, ATPase IC50 = 0.54 μmol·L-1, helicase IC50 = 7.0 μmol·L-1; 79, ATPase IC50 = 0.68 μmol·L-1, helicase IC50 = 2.7 μmol·L-1; 80, ATPase IC50 = 2.8 μmol·L-1, helicase IC50 = 5.4 μmol·L-1, 图 9)。
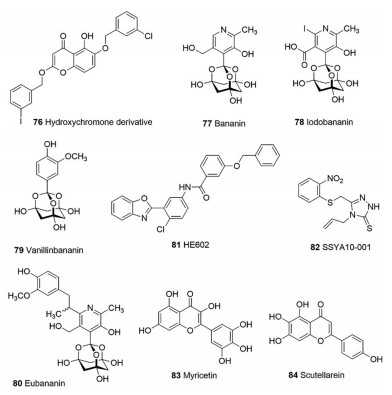
|
Figure 9 Representative compounds targeting coronavirus helicase |
基于高通量筛选, Kao等[29]从50 240个小分子中筛选出具有SARS-CoV解旋酶抑制活性的化合物81 (HE602, IC50 = 6.9 μmol·L-1; EC50 = 6 μmol·L-1, 图 9), 其能有效抑制解旋酶的ATPase活性。Adedeji等[67]从约3 000个化合物筛选得到具有抑制SARS-CoV解旋酶解旋活性的化合物82 (SSYA10-001, EC50 = 7 μmol·L-1; CC50 > 500 μmol·L-1, 图 9), 其酶动力学结果表明化合物82是解旋酶的非竞争性抑制剂, 不影响ATP水解或与核酸底物结合。随后, 他们发现化合物82对MERS-CoV解旋酶也有抑制解旋活性的作用(EC50 = 25 μmol·L-1), 并且作用位点与SARS-CoV相似(Y277、R507和R508)[69]。Yu等[70]筛选得到两种类黄酮天然产物, 化合物83 (杨梅素, IC50 = 2.71 ± 0.19 μmol·L-1, 图 9)和化合物84 (野黄芩素, IC50 = 0.86 ± 0.48 μmol·L-1, 图 9), 对SARS-CoV解旋酶具有ATPase抑制作用而不影响helicase活性。
2.8 鸟嘌呤-N7甲基转移酶鸟嘌呤-N7甲基转移酶(guanine-N7 methyltransferase, N7-MTase)是一种双功能酶, 其N端结构域具有核酸内切酶活性, 校正RNA致死性突变; 其C端具有鸟嘌呤-N7甲基转移酶活性, 对mRNA加帽[73]。Bouvet等[74]找到化合物85 (图 10)金精三羧酸在体外同时抑制SARS-CoV的鸟嘌呤-N7甲基转移酶和2'-O-甲基转移酶活性, IC50值分别为2.1和6.4 μmol·L-1。
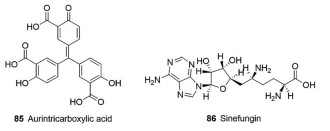
|
Figure 10 Representative compounds targeting coronavirus N7- MTase and 2'-O-Mtase |
2'-O-甲基转移酶(2'-O-methyl-transferase, 2'-O-Mtase)是S-腺苷高半胱氨酸(SAM)依赖的非结构蛋白, 在病毒非结构蛋白nsp10作为激动剂的条件下可以加强与RNA的亲和力, 并且逃脱宿主的免疫反应。Decroly等[75]发现化合物86 (西奈芬净, 图 10)能够与SAM结合口袋相结合, 从而抑制SARS-CoV的甲基转移酶活性。Aouadi等[76]在MERS-CoV的甲基转移酶抑制实验中, 测试了18个化合物, 发现SAM类似物西奈芬净显示出较强的抑制作用, IC50值为7.4 ± 0.9 μmol·L-1。
3 靶向宿主蛋白的活性化合物 3.1 血管紧张素转换酶2血管紧张素转换酶2 (angiotensin-converting enzyme 2, ACE2)是冠状病毒刺突糖蛋白的结合受体, 可有效结合冠状病毒刺突糖蛋白的S1结构域, 介导冠状病毒识别和入侵宿主细胞[77, 78]。通过虚拟筛选, Huentelman等[79]基于血管紧张素酶2闭合构象从约14 000个化合物中挑选出化合物87 (图 11), 实验证明其能有效抑制SARS-CoV刺突糖蛋白与血管紧张素转换酶2的结合(IC50 = 57 μmol·L-1, Ki = 459 μmol·L-1)。
3.2 组织蛋白酶L组织蛋白酶L (cathepsin L, CTSL)是冠状病毒感染过程中重要的活化蛋白酶, 可以激活刺突糖蛋白的膜融合[80], 因此可以作为阻断病毒侵入的靶标。化合物88 (K11777, 图 11)是一种半胱氨酸蛋白酶抑制剂, 作用靶标包括人组织蛋白酶和寄生虫的组织蛋白酶样蛋白酶。Zhou等[81]研究发现它可以通过靶向组织蛋白酶阻断病毒刺突蛋白酶激活途径, 因此对冠状病毒有广谱的抑制作用(SARS-CoV IC50 = 0.68 ± 0.09 nmol·L-1; HCoV-229E IC50 = 1.48 ± 0.13 nmol·L-1; HCoV-NL63 IC50 = 6.78 ± 0.24 nmol·L-1; MERS-CoV IC50 = 46.12 ± 6.63 nmol·L-1)。通过高通量筛选, Simmons等[80]获得组织蛋白酶L抑制剂89 (MDL28170, IC50 = 2.5 nmol·L-1, IC95 = 0.8 μmol·L-1, 图 11), 能够有效抑制SARS-CoV的复制, 同时获得组织蛋白酶抑制剂90~93, 能有效抑制HIV-luc (SARS-S) (90, IC95 = 1.8 μmol·L-1; 91, IC95 = 0.8 μmol·L-1; 92, IC95 = 1.3 μmol·L-1; 93, IC95 = 2.0 μmol·L-1, 图 11)。Adedeji等[6]从3 000个药理活性小分子库中筛选得到组织蛋白酶L抑制剂94 (SSAA09E1, 图 11)对SARS-CoV有抑制活性, 其IC50为5.33 μmol·L-1。Zhou等[82]筛选得到糖肽类抗生素化合物95 (teicoplanin, 图 11)能与宿主组织蛋白酶L结合, 对SARS-CoV (IC50 = 3.76 ± 1.09 μmol·L-1)和MERS-CoV (IC50 = 0.63 ± 0.34 μmol·L-1)均具有抑制作用。
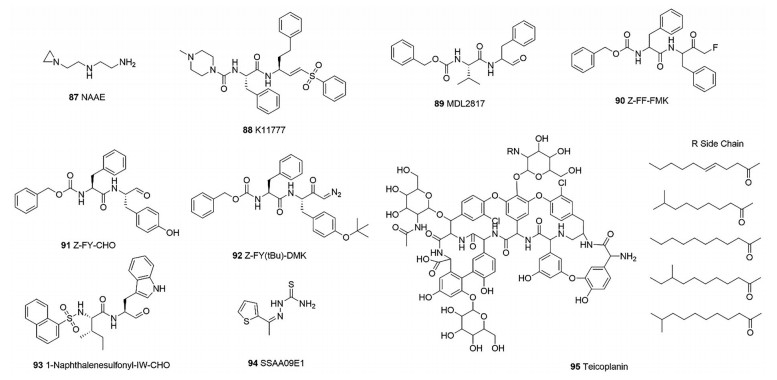
|
Figure 11 Representative compounds targeting host proteins ACE2 and CTSL |
弗林蛋白酶(furin)能水解刺突糖蛋白, 可以增强病毒与宿主细胞膜的融合[83, 84]。有文献报道弗林蛋白酶抑制剂96 (图 12)在75 μmol·L-1浓度时对MERS-CoV有一定的抑制作用, 导致MERS-CoV刺突糖蛋白裂解产物减少[85]。
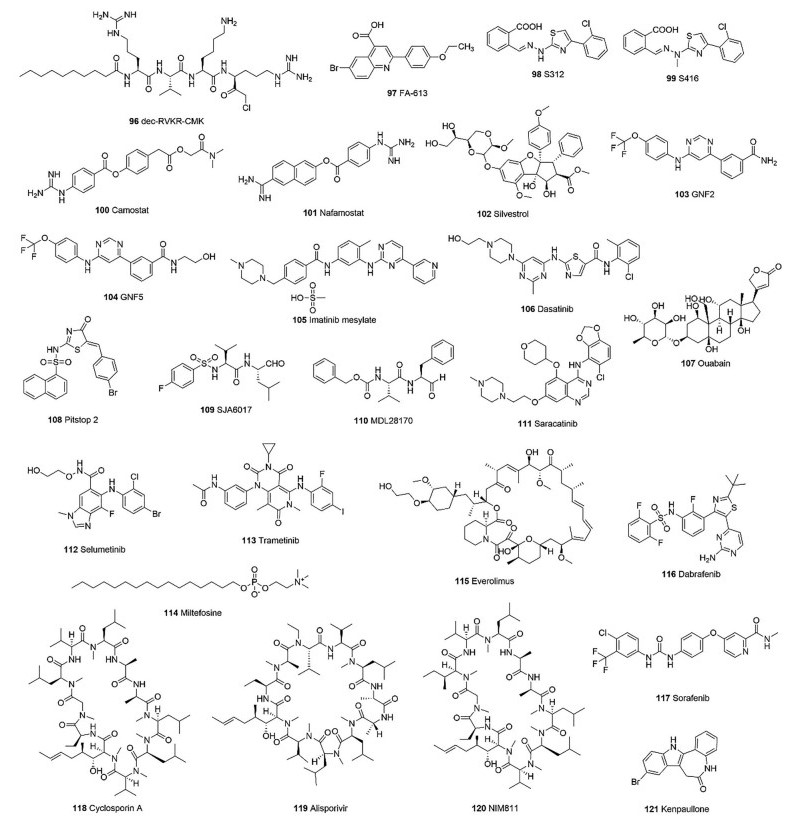
|
Figure 12 Representative compounds targeting host proteins |
二氢乳清酸脱氢酶(dihydroorotate dehydrogenase, DHODH)是嘧啶从头生物合成途径中的关键酶, 其抑制剂对RNA病毒具有广谱抗病毒作用, 包括冠状病毒SARS-CoV-2[86]。化合物97 (图 12)通过靶向二氢乳清酸脱氢酶, 对病毒产生抑制作用, 对许多流感病毒表现出较低摩尔的活性, 且没有明显的细胞毒性, 在SARS-CoV感染细胞中的EC50值为8.4 ± 1.2 μmol·L-1, 在MERS-CoV感染细胞中的EC50值为10.1 ± 0.2 μmol·L-1 [87]。Xiong等[86]通过虚拟筛选从280 000个化合物中找到两种二氢乳清酸脱氢酶抑制剂化合物98、99 (S312 SI > 64.62; S416 SI > 5882, 图 12), 实验证明其对SARS-CoV-2有一定抑制作用, 其EC50分别为1.55 μmol·L-1和17 nmol·L-1, 且都具抗RNA病毒广谱性。
3.5 跨膜丝氨酸蛋白酶2跨膜丝氨酸蛋白酶2 (transmembrane protease serine 2, TMPRSS2)水解并激活冠状病毒刺突糖蛋白, 在细胞表面上诱导病毒-细胞膜融合, 影响病毒的侵入过程[88-90]。化合物100 (卡莫司他, 图 12)能与跨膜丝氨酸蛋白酶2结合, 在SARS-CoV感染的致病小鼠模型中表现出抗病毒活性[81], 在1和10 μmol·L-1浓度时可有效抑制MERS-CoV膜融合, 在100 μmol·L-1浓度时完全抑制其膜融合[91]。化合物101 (奈莫司他, 图 12)作为抗凝药物, 能有效抑制SARS-CoV-2 (EC50 = 22.50 μmol·L-1, CC50 > 100 μmol·L-1)[65], Yamamoto等[92]认为化合物101最可能是通过结合跨膜丝氨酸蛋白酶2来抑制病毒刺突糖蛋白蛋白水解, 从而干扰刺突糖蛋白介导的膜融合。
3.6 真核起始因子4A-I真核起始因子4A-I (eukaryotic initiation factor 4A-I, eIF4A)具有ATP依赖性ATPase活性和RNA依赖性双链RNA解旋活性。它以类似于HCV NS3解旋酶结合RNA的方式结合RNA[93]。Müller等[94]报道了一种靶向真核起始因子4A-I的天然产物102 (图 12), 对MERS-CoV和HCoV-229E的抑制活性EC50分别为1.3和3 nmol·L-1。
3.7 酪氨酸蛋白激酶ABL1酪氨酸蛋白激酶ABL1 (tyrosine-protein kinase ABL1, ABL1)在许多病毒感染过程中都扮演着重要角色, 参与许多细胞功能, 提供细胞信号传导和肌动蛋白细胞骨架组织之间的通讯, 还参与T细胞信号传导, 胚胎发育以及癌症中的细胞迁移和侵袭[95]。因此被认为是开发广谱抗病毒药物的一个靶点。Sisk等[96]发现了两个ABL1抑制剂103 (GNF2, 图 12)和104 (GNF5, 图 12), 它们均能与ABL1的肉豆蔻酸结合位点结合而导致激酶失活, 从而使感染细胞的数量减少90%和80%。甲磺酸伊马替尼(105)和达沙替尼(106, 图 12)也是ABL1抑制剂, 能有效抑制SARS-CoV和MERS-CoV。其中, 达沙替尼对MERS-CoV的EC50为5.4 μmol·L-1, 对SARS-CoV的EC50为2.1 μmol·L-1, 由此可见, SARS-CoV对ABL1抑制剂更具敏感性[97]。
3.8 钠/钾转运ATPase钠/钾转运ATPase (Sodium/potassium-transporting ATPase subunit alpha-1, ATP1A1)具有钾离子和钠离子的主动转运功能[98], 也参与信号转导[99, 100]。化合物107 (乌本苷, 图 12)被报道能够靶向钠/钾转运ATPase, 抑制冠状病毒入侵的早期阶段, 50 nmol·L-1的低浓度乌本苷可以抑制MERS-CoV的感染[101]。
3.9 网格蛋白网格蛋白(clathrin)介导的内吞作用调节细胞表面重要质膜蛋白的内吞摄取。诸如细菌和病毒等病原体往往也利用网格蛋白的内吞机制以进入细胞内部[102]。网格蛋白介导的内吞作用是HCOV-NL63进入细胞的主要途径, 有文献报道萘磺酰胺衍生物108 (pitstop 2, 图 12)能与网格蛋白的末端结构域结合, 抑制HCOV-NL63入侵宿主细胞。在LLC-Mk2细胞中进行化合物108与DMSO的对照实验, 结果发现蛋白仅存在于细胞表面, 抑制剂阻断了蛋白的内吞作用[103]。
3.10 M-钙蛋白酶M-钙蛋白酶(M-calpain, Capn)是一种钙离子依赖性半胱氨酸蛋白酶, 通过酶解细胞内多种酶及细胞骨架蛋白系统, 参与细胞刺激应答、增殖、分化等诸多生物过程。M-钙蛋白酶在SARS-CoV感染的早期阶段有重要作用[104]。靶向M-钙蛋白酶的化合物109 (SJA6017, 图 12)和110 (MDL28170, 图 12), 能有效抑制SARS-CoV的感染。在SARS-CoV感染的Vero76细胞中, 化合物109的EC50值为2 μmol·L-1, 化合物110的EC50值为0.5 μmol·L-1[66]。
3.11 Src家族激酶SFKSrc蛋白酪氨酸激酶(SFK)家族在调节细胞表面受体的信号传导中起着关键作用[105]。抗癌药物塞卡替尼(111, 图 12)通过靶向Src家族激酶SFK, 能有效抑制MERS-CoV, 其抑制活性EC50为2.9 ± 0.6 μmol·L-1, CC50为57 ± 5.5 μmol·L-1 [106]。
3.12 ERK激酶/MAPK激酶ERK激酶/MAPK激酶参与的信号传导途径控制许多细胞类型的生长、增殖、分化和存活的分子信号网络, 因此是重要的药物靶标[107]。司美替尼(112)和曲美替尼(113, 图 12)通过靶向ERK/MAPK激酶对MERS-CoV感染过程起抑制作用, 在10 μmol·L-1浓度下, 抑制效果接近100%[108]。
3.13 AKT激酶AKT激酶参与的信号通路调节细胞的基本功能, 如转录、翻译、增殖、生长和存活[109]。抗肿瘤药物114 (米替福新, 图 12)通过靶向AKT激酶, 作用于PI3K/AKT/mTOR信号通路, 对MERS-CoV起抑制作用, 在10 μmol·L-1浓度下对MERS-CoV的抑制作用为28%[108]。
3.14 mTOR激酶mTOR是一种非典型丝氨酸/苏氨酸蛋白激酶, 可磷酸化下游核糖体激酶调控细胞的生长和增殖。依维莫司(115, 图 12)通过靶向mTOR激酶, 作用于PI3K/AKT/mTOR信号通路, 对MERS-CoV起预防和抑制作用, 在10 μmol·L-1浓度下有56%的抑制作用和59%的预防作用[108]。
3.15 Raf激酶Raf激酶属于蛋白丝氨酸/苏氨酸激酶家族, 参与Ras-Raf-MEK-ERK信号传导级联反应, 调节多种生物过程, 包括细胞凋亡、分化、增殖和癌变。达帕菲尼(116)和索拉非尼(117, 图 12)通过靶向Raf激酶, 作用于ERK/MAPK信号通路, 对MERS-CoV有抑制活性, 在10 μmol·L-1浓度下的抑制率为45%[108]。
3.16 亲环蛋白亲环蛋白(cyclophilin, Cyp)是一类结构高度保守的多功能蛋白质, 能够催化含脯氨酸的寡肽底物顺反异构作用, 对蛋白质折叠和组装起重要作用, 还具有分子伴侣作用, 在应激反应中参与调节信号转导途径, 并影响RNA的剪接过程。化合物118及其衍生物119~120 (图 12)通过靶向亲环蛋白, 阻碍HCoV-229E的核衣壳蛋白与亲环蛋白结合, 从而有效抑制HCoV-229E复制(118, EC50 = 0.97 μmol·L-1, CC50 = 185.6 μmol·L-1; 119, EC50 = 1.37 μmol·L-1, CC50 = 280.1 μmol·L-1; 120, EC50 = 1.19 μmol·L-1, CC50 = 207.9 μmol·L-1)[110]。
3.17 糖原合成酶激酶3β糖原合成酶激酶3β (glycogen synthase kinase-3 beta, GSK-3β)是一种高度保守的蛋白丝氨酸/苏氨酸激酶, 该激酶的失调会引发癌症、糖尿病、精神分裂症和躁郁症等多种疾病[111]。在感染的Vero E6细胞中, 化合物121 (图 12)通过靶向糖原合成酶激酶3β, 可降低SARS-CoV核衣壳蛋白的磷酸化, 使得病毒RNA水平降低到50%左右[112]。
4 靶标未知的活性化合物基于表型的药物筛选在新药研发中广泛应用, 但后期靶标鉴定是困扰活性化合物活性优化和作用机制研究的一大难题。Chan等[113]从具有临床药理活性的2 334个化合物进行筛选, 找到许多对MERS-CoV有抑制活性的化合物(表 3, 图 13)。抗精神类疾病的药物122和123, 能有效抑制MERS-CoV感染, 其抑制活性EC50分别为11.2和2.5 μmol·L-1, CC50分别为23.6和24.5 μmol·L-1。抗组胺药物124, 抗原生动物药物125, 抗癌药物126、127, 抗抑郁药物128也都具有MERS-CoV抑制活性。其中抗肿瘤药物129对HCoV-OC43、HCoV-NL63和MERS-CoV都有抑制作用, EC50分别为1.58、0.23和1.54 μmol·L-1。Ko等[114]从5 406种化合物中筛选得到具有抗MERS-CoV感染的潜在治疗药物130~136 (表 4, 图 13), 包括强心药物130~132, 抗虫剂133, 糖皮质激素药134, 抗肿瘤药135和抗病毒药136。Dyall等[97, 115]从290种临床批准和临床研究阶段化合物中筛选, 得到多种具有MERS-CoV和SARS-CoV抑制活性的化合物(表 5, 图 13), 包括多种神经递质受体阻滞剂137~144和雌激素受体拮抗剂145、146。
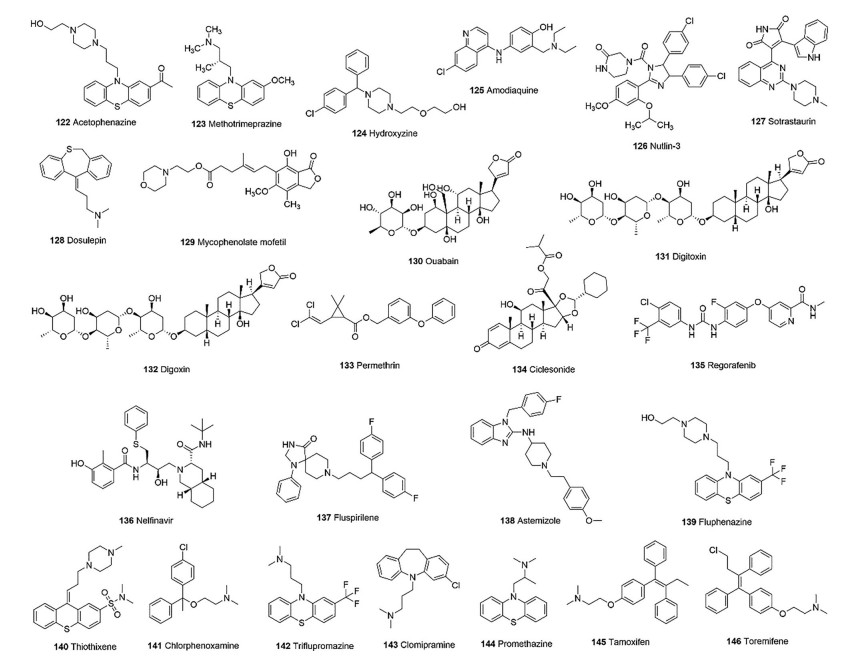
|
Figure 13 Representative compounds against MERS-CoV and SARS-CoV |
为进一步探索这些靶标未知的活性化合物的作用机制, 助力抗冠状病毒药物研发, 本课题组通过应用新报道的COVID-19靶标蛋白预测平台D3Docking[116], 对这些化合物的可能作用靶标开展预测。D3Docking包含了20种新冠病毒自身编码的蛋白质的三维结构以及22种与病毒侵入、复制和释放相关的人源蛋白的三维结构, 并运用多靶标、多构象、多位点的反向分子对接策略来预测药物作用靶标, 可望有效降低对接结果的假阴性率。并且, 为提高针对一些依赖金属离子的靶标蛋白的对接准确性, D3Docking在Smina对接软件[117]基础上添加了金属离子的相关作用项。例如, 在冠状病毒RdRp蛋白中, 镁离子对于RNA以及一些抑制剂(remdesivir等)的结合是不可或缺的[118]。最终预测结果如表 3~5所示, 为减少假阳性, 列出了预测的活性化合物的前3个可能作用靶标及其对接打分值, 以供靶标确证研究人员参考。特别需要指出的是, 基于分子对接的靶标预测挑战性极大, 既受制于方法本身的可靠性, 又决定于靶标蛋白库的完整性, 还与药物作用过程的复杂性密切相关, 需要综合考虑多种因素灵活运用。
| Table 3 The active compounds for MERS-CoV screened by Chan et al. with predicted targets of SARS-CoV-2. a: Antiviral activity against MERS-CoV (μmol·L-1); b: The top three possible targets predicted by D3docking (Docking score: kcal·mol-1). Target full name: cGMP-specific 3', 5'-cyclic phosphodiesterase (PDE5), RAC-alpha serine/threonine-protein kinase (AKT1), dihydroorotate dehydrogenase (DHODH), nonstructural protein 2 (Nsp2), tyrosine-protein kinase ABL2 (ABL2), transmembrane protease serine 2 (TMPRSS2), spike protein (S protein), RNA-dependent RNA polymerase (RdRp), caveolin-2 (CAV2), guanine-N7 methyltransferase (N7-MTase) |
| Table 4 The active compounds for MERS-CoV screened by Ko et al. with predicted targets of SARS-CoV-2. a: Antiviral activity against MERS-CoV (μmol·L-1); b: The top three possible targets predicted by D3docking (Docking score: kcal·mol-1). Target full name: cGMP-spe‐ cific 3', 5'-cyclic phosphodiesterase (PDE5), RAC-alpha serine/threonine-protein kinase (AKT1), dihydroorotate dehydrogenase (DHODH), non-structural protein 4 (Nsp4), spike protein (S protein), 3C-like proteinase (3CLpro), angiotensin converting enzyme 2 (ACE2), uridylatespecific endoribonuclease (NendoU), ADP ribose phosphatase (ADRP), c-Jun N-terminal kinase 2 (JNK2). c: Target multiple kinases such as VEGFR, EGFR and B-Raf |
| Table 5 The active compounds for MERS-CoV and SARS-CoV screened by Dyall et al. with predicted targets of SARS-CoV-2. a: Antiviral activity against MERS-CoV (μmol·L-1); b: Antiviral activity against SARS-CoV (μmol·L-1); c: The top three possible targets predicted by D3docking (Docking score: kcal·mol-1). Target full name: cGMP-specific 3', 5'-cyclic phosphodiesterase (PDE5), RAC-alpha serine/ threonine-protein kinase (AKT1), dihydroorotate dehydrogenase (DHODH), non-structural protein 2 (Nsp2), spike protein (S protein), c-Jun N-terminal kinase 1 (JNK1), guanine-N7 methyltransferase (N7-MTase) |
目前COVID-19疫情已席卷全球, 形势严峻, 抗SARS-CoV-2的药物研发刻不容缓。作为冠状病毒属, SARS-CoV-2与其他冠状病毒如SARS-CoV和MERS-CoV有较高的序列和结构相似性。因此, 面对SARS-CoV-2药物研发的紧迫性, 攻关科研者在研发创新药物时, 也需要借鉴其他抗冠状病毒药物的研发思路。而作为RNA病毒, SARS-CoV-2的变异性相比DNA病毒更强, 广谱抗RNA病毒药物的研发或许有助于应对SARS-CoV-2的高突变率问题。在当今信息化时代下, 攻关科研者需要全方面掌握关于SARS-CoV-2的多学科研究新动态, 为更快、更好地解决疫情提供理论支撑。本文根据已报道的冠状病毒相关靶标, 分类阐述了目前已知的冠状病毒活性化合物, 还对靶标不明确的化合物通过反向对接平台预测潜在靶标, 以期为相关科研人员提供帮助。新药研发往往需要很大的时间成本, 老药新用是一个很好的解决手段, 能有效缩短时间成本。但是一款可以真正应用于人体的药物, 无法省略药物研发的整个流程。然而, 目前还没有足够的针对SARS-CoV-2的体内药物活性测试平台。因此需要加快动物实验特别是与人类亲缘关系相近的动物实验测试平台建立, 以降低活性化合物进入临床实验后的失败率。
作者贡献:朱维良, 徐志建和李慧玉负责文章选题和课题设计; 王晓宇, 朱正诞和穆凯洁负责文献调研; 王晓宇, 彭诚, 杨延青, 石禹龙负责文章撰写; 王晓宇, 彭诚和朱维良负责文章修改
利益冲突:本文的所有作者不存在利益冲突
| [1] |
Pillaiyar T, Meenakshisundaram S, Manickam M. Recent discovery and development of inhibitors targeting coronaviruses[J]. Drug Discov Today, 2020, 25: 668-688. DOI:10.1016/j.drudis.2020.01.015 |
| [2] |
Liu QY, Wang XL. Strategies for the development of drugs targeting novel coronavirus 2019-nCoV[J]. Acta Pharm Sin (药学学报), 2020, 55: 181-188. |
| [3] |
Wang HR, Wang X. Advances in research of novel coronavirus related drugs and biological products[J]. Acta Pharm Sin (药学学报), 2020, 55: 349-354. |
| [4] |
Zhang C, Chen SB, Zhang J, et al. Analysis of chemical drugs applied for clinical trial for the treatment of COVID-19[J]. Acta Pharm Sin (药学学报), 2020, 55: 355-365. |
| [5] |
Walls AC, Park YJ, Tortorici MA, et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein[J]. Cell, 2020, 181: 281-292.e286. DOI:10.1016/j.cell.2020.02.058 |
| [6] |
Adedeji AO, Severson W, Jonsson C, et al. Novel inhibitors of severe acute respiratory syndrome coronavirus entry that act by three distinct mechanisms[J]. J Virol, 2013, 87: 8017-8028. DOI:10.1128/JVI.00998-13 |
| [7] |
Van Der Meer FJ, De Haan CA, Schuurman NM, et al. The carbohydrate-binding plant lectins and the non-peptidic antibiotic pradimicin A target the glycans of the coronavirus envelope glycoproteins[J]. J Antimicrob Chemother, 2007, 60: 741-749. DOI:10.1093/jac/dkm301 |
| [8] |
Kim JY, Kim YI, Park SJ, et al. Safe, high-throughput screening of natural compounds of MERS-CoV entry inhibitors using a pseudovirus expressing MERS-CoV spike protein[J]. Int J Antimicrob Agents, 2018, 52: 730-732. DOI:10.1016/j.ijantimicag.2018.05.003 |
| [9] |
Schelle B, Karl N, Ludewig B, et al. Selective replication of coronavirus genomes that express nucleocapsid protein[J]. J Virol, 2005, 79: 6620-6630. DOI:10.1128/JVI.79.11.6620-6630.2005 |
| [10] |
Mcbride R, Van Zyl M, Fielding BC. The coronavirus nucleocapsid is a multifunctional protein[J]. Viruses, 2014, 6: 2991-3018. DOI:10.3390/v6082991 |
| [11] |
Lin SY, Liu CL, Chang YM, et al. Structural basis for the identification of the N-terminal domain of coronavirus nucleocapsid protein as an antiviral target[J]. J Med Chem, 2014, 57: 2247-2257. DOI:10.1021/jm500089r |
| [12] |
Surya W, Li Y, Torres J. Structural model of the SARS coronavirus E channel in LMPG micelles[J]. Biochim Biophys Acta Biomembr, 2018, 1860: 1309-1317. DOI:10.1016/j.bbamem.2018.02.017 |
| [13] |
Dediego ML, Nieto-Torres JL, Jiménez-Guardeño JM, et al. Severe acute respiratory syndrome coronavirus envelope protein regulates cell stress response and apoptosis[J]. PLoS Pathog, 2011, 7. |
| [14] |
Verdiá-Báguena C, Nieto-Torres JL, Alcaraz A, et al. Coronavirus E protein forms ion channels with functionally and structurally-involved membrane lipids[J]. Virology, 2012, 432: 485-494. DOI:10.1016/j.virol.2012.07.005 |
| [15] |
Torres J, Parthasarathy K, Lin X, et al. Model of a putative pore:the pentameric alpha-helical bundle of SARS coronavirus E protein in lipid bilayers[J]. Biophys J, 2006, 91: 938-947. DOI:10.1529/biophysj.105.080119 |
| [16] |
Pervushin K, Tan E, Parthasarathy K, et al. Structure and inhibition of the SARS coronavirus envelope protein ion channel[J]. PLoS Pathog, 2009, 5. |
| [17] |
Jo S, Kim H, Kim S, et al. Characteristics of flavonoids as potent MERS-CoV 3C-like protease inhibitors MERS-CoV 3C-like protease inhibitors [J]. Chem Biol Drug
Des, 2019, 94: 2023-2030.[J]. Chem Biol Drug Des, 2019, 94: 2023-2030. DOI:10.1111/cbdd.13604 |
| [18] |
Anand K, Ziebuhr J, Wadhwani P, et al. Coronavirus main proteinase (3CLpro) structure:basis for design of anti-SARS drugs[J]. Science, 2003, 300: 1763-1767. DOI:10.1126/science.1085658 |
| [19] |
Muramatsu T, Takemoto C, Kim YT, et al. SARS-CoV 3CL protease cleaves its C-terminal autoprocessing site by novel subsite cooperativity[J]. Proc Natl Acad Sci U S A, 2016, 113: 12997-13002. DOI:10.1073/pnas.1601327113 |
| [20] |
Ryu YB, Park SJ, Kim YM, et al. SARS-CoV 3CLpro inhibitory effects of quinone-methide triterpenes from Tripterygium regelii[J]. Bioorg Med Chem Lett, 2010, 20: 1873-1876. DOI:10.1016/j.bmcl.2010.01.152 |
| [21] |
Wen CC, Kuo YH, Jan JT, et al. Specific plant terpenoids and lignoids possess potent antiviral activities against severe acute respiratory syndrome coronavirus[J]. J Med Chem, 2007, 50: 4087-4095. DOI:10.1021/jm070295s |
| [22] |
Ryu YB, Jeong HJ, Kim JH, et al. Biflavonoids from Torreya nucifera displaying SARS-CoV 3CL(pro) inhibition[J]. Bioorg Med Chem, 2010, 18: 7940-7947. DOI:10.1016/j.bmc.2010.09.035 |
| [23] |
Park JY, Kim JH, Kwon JM, et al. Dieckol, a SARS-CoV 3CL(pro) inhibitor, isolated from the edible brown algae Ecklonia cava[J]. Bioorg Med Chem, 2013, 21: 3730-3737. DOI:10.1016/j.bmc.2013.04.026 |
| [24] |
Yang S, Chen SJ, Hsu MF, et al. Synthesis, crystal structure, structure-activity relationships, and antiviral activity of a potent SARS coronavirus 3CL protease inhibitor[J]. J Med Chem, 2006, 49: 4971-4980. DOI:10.1021/jm0603926 |
| [25] |
Ghosh AK, Xi K, Ratia K, et al. Design and synthesis of peptidomimetic severe acute respiratory syndrome chymotrypsin-like protease inhibitors[J]. J Med Chem, 2005, 48: 6767-6771. DOI:10.1021/jm050548m |
| [26] |
Ghosh AK, Xi K, Grum-Tokars V, et al. Structure-based design, synthesis, and biological evaluation of peptidomimetic SARS-CoV 3CLpro inhibitors[J]. Bioorg Med Chem Lett, 2007, 17: 5876-5880. DOI:10.1016/j.bmcl.2007.08.031 |
| [27] |
Shie JJ, Fang JM, Kuo CJ, et al. Discovery of potent anilide inhibitors against the severe acute respiratory syndrome 3CL protease[J]. J Med Chem, 2005, 48: 4469-4473. DOI:10.1021/jm050184y |
| [28] |
Ghosh AK, Gong G, Grum-Tokars V, et al. Design, synthesis and antiviral efficacy of a series of potent chloropyridyl ester-derived SARS-CoV 3CLpro inhibitors[J]. Bioorg Med Chem Lett, 2008, 18: 5684-5688. DOI:10.1016/j.bmcl.2008.08.082 |
| [29] |
Kao RY, Tsui WH, Lee TS, et al. Identification of novel small-molecule inhibitors of severe acute respiratory syndrome-associated coronavirus by chemical genetics[J]. Chem Biol, 2004, 11: 1293-1299. DOI:10.1016/j.chembiol.2004.07.013 |
| [30] |
Konno H, Wakabayashi M, Takanuma D, et al. Design and synthesis of a series of serine derivatives as small molecule inhibitors of the SARS coronavirus 3CL protease[J]. Bioorg Med Chem, 2016, 24: 1241-1254. DOI:10.1016/j.bmc.2016.01.052 |
| [31] |
Konno H, Onuma T, Nitanai I, et al. Synthesis and evaluation of phenylisoserine derivatives for the SARS-CoV 3CL protease inhibitor[J]. Bioorg Med Chem Lett, 2017, 27: 2746-2751. DOI:10.1016/j.bmcl.2017.04.056 |
| [32] |
Ohnishi K, Hattori Y, Kobayashi K, et al. Evaluation of a non-prime site substituent and warheads combined with a decahydroisoquinolin scaffold as a SARS 3CL protease inhibitor[J]. Bioorg Med Chem, 2019, 27: 425-435. DOI:10.1016/j.bmc.2018.12.019 |
| [33] |
Lee TW, Cherney MM, Liu J, et al. Crystal structures reveal an induced-fit binding of a substrate-like aza-peptide epoxide to SARS coronavirus main peptidase[J]. J Mol Biol, 2007, 366: 916-932. DOI:10.1016/j.jmb.2006.11.078 |
| [34] |
Martina E, Stiefl N, Degel B, et al. Screening of electrophilic compounds yields an aziridinyl peptide as new active-site directed SARS-CoV main protease inhibitor[J]. Bioorg Med Chem Lett, 2005, 15: 5365-5369. DOI:10.1016/j.bmcl.2005.09.012 |
| [35] |
Chen L, Gui C, Luo X, et al. Cinanserin is an inhibitor of the 3C-like proteinase of severe acute respiratory syndrome coronavirus and strongly reduces virus replication in vitro[J]. J Virol, 2005, 79: 7095-7103. DOI:10.1128/JVI.79.11.7095-7103.2005 |
| [36] |
Shao YM, Yang WB, Peng HP, et al. Structure-based design and synthesis of highly potent SARS-CoV 3CL protease inhibitors[J]. Chembiochem, 2007, 8: 1654-1657. DOI:10.1002/cbic.200700254 |
| [37] |
Chen LR, Wang YC, Lin YW, et al. Synthesis and evaluation of isatin derivatives as effective SARS coronavirus 3CL protease inhibitors[J]. Bioorg Med Chem Lett, 2005, 15: 3058-3062. DOI:10.1016/j.bmcl.2005.04.027 |
| [38] |
Kumar V, Tan KP, Wang YM, et al. Identification, synthesis and evaluation of SARS-CoV and MERS-CoV 3C-like protease inhibitors[J]. Bioorg Med Chem, 2016, 24: 3035-3042. DOI:10.1016/j.bmc.2016.05.013 |
| [39] |
Kumar V, Shin JS, Shie JJ, et al. Identification and evaluation of potent Middle East respiratory syndrome coronavirus (MERS-CoV) 3CL(Pro) inhibitors[J]. Antiviral Res, 2017, 141: 101-106. DOI:10.1016/j.antiviral.2017.02.007 |
| [40] |
Kim Y, Liu H, Galasiti Kankanamalage AC, et al. Reversal of the progression of fatal coronavirus infection in cats by a broad-spectrum coronavirus protease inhibitor[J]. PLoS Pathog, 2016, 12. |
| [41] |
Galasiti Kankanamalage AC, Kim Y, Damalanka VC, et al. Structure-guided design of potent and permeable inhibitors of MERS coronavirus 3CL protease that utilize a piperidine moiety as a novel design element[J]. Eur J Med Chem, 2018, 150: 334-346. DOI:10.1016/j.ejmech.2018.03.004 |
| [42] |
Karypidou K, Ribone SR, Quevedo MA, et al. Synthesis, biological evaluation and molecular modeling of a novel series of fused 1, 2, 3-triazoles as potential anti-coronavirus agents[J]. Bioorg Med Chem Lett, 2018, 28: 3472-3476. DOI:10.1016/j.bmcl.2018.09.019 |
| [43] |
Wu RJ, Zhou KX, Yang H, et al. Chemical synthesis, crystal structure, versatile evaluation of their biological activities and molecular simulations of novel pyrithiobac derivatives[J]. Eur J Med Chem, 2019, 167: 472-484. DOI:10.1016/j.ejmech.2019.02.002 |
| [44] |
Zhang HZ, Zhang H, Kemnitzer W, et al. Design and synthesis of dipeptidyl glutaminyl fluoromethyl ketones as potent severe acute respiratory syndrome coronovirus (SARS-CoV) inhibitors[J]. J Med Chem, 2006, 49: 1198-1201. DOI:10.1021/jm0507678 |
| [45] |
Nukoolkarn V, Lee VS, Malaisree M, et al. Molecular dynamic simulations analysis of ritonavir and lopinavir as SARS-CoV 3CL(pro) inhibitors[J]. J Theor Biol, 2008, 254: 861-867. DOI:10.1016/j.jtbi.2008.07.030 |
| [46] |
Li G, De Clercq E. Therapeutic options for the 2019 novel coronavirus (2019-nCoV)[J]. Nat Rev Drug Discov, 2020, 19: 149-150. DOI:10.1038/d41573-020-00016-0 |
| [47] |
Sheahan TP, Sims AC, Leist SR, et al. Comparative therapeutic efficacy of remdesivir and combination lopinavir, ritonavir, and interferon beta against MERS-CoV[J]. Nat Commun, 2020, 11: 222. DOI:10.1038/s41467-019-13940-6 |
| [48] |
Kim UJ, Won EJ, Kee SJ, et al. Combination therapy with lopinavir/ritonavir, ribavirin and interferon-alpha for Middle East respiratory syndrome[J]. Antivir Ther, 2016, 21: 455-459. |
| [49] |
Arabi YM, Alothman A, Balkhy HH, et al. Treatment of Middle East Respiratory Syndrome with a combination of lopinavir-ritonavir and interferon-beta1b (MIRACLE trial):study protocol for a randomized controlled trial[J]. Trials, 2018, 19: 81. DOI:10.1186/s13063-017-2427-0 |
| [50] |
Frieman M, Ratia K, Johnston RE, et al. Severe acute respiratory syndrome coronavirus papain-like protease ubiquitin-like domain and catalytic domain regulate antagonism of IRF3 and NF-kappaB signaling[J]. J Virol, 2009, 83: 6689-6705. DOI:10.1128/JVI.02220-08 |
| [51] |
Park JY, Yuk HJ, Ryu HW, et al. Evaluation of polyphenols from Broussonetia papyrifera as coronavirus protease inhibitors[J]. J Enzyme Inhib Med Chem, 2017, 32: 504-515. DOI:10.1080/14756366.2016.1265519 |
| [52] |
Park JY, Jeong HJ, Kim JH, et al. Diarylheptanoids from Alnus japonica inhibit papain-like protease of severe acute respiratory syndrome coronavirus[J]. Biol Pharm Bull, 2012, 35: 2036-2042. DOI:10.1248/bpb.b12-00623 |
| [53] |
Park JY, Kim JH, Kim YM, et al. Tanshinones as selective and slow-binding inhibitors for SARS-CoV cysteine proteases[J]. Bioorg Med Chem, 2012, 20: 5928-5935. DOI:10.1016/j.bmc.2012.07.038 |
| [54] |
Cho JK, Curtis-Long MJ, Lee KH, et al. Geranylated flavonoids displaying SARS-CoV papain-like protease inhibition from the fruits of Paulownia tomentosa[J]. Bioorg Med Chem, 2013, 21: 3051-3057. DOI:10.1016/j.bmc.2013.03.027 |
| [55] |
Lee H, Lei H, Santarsiero BD, et al. Inhibitor recognition specificity of MERS-CoV papain-like protease may differ from that of SARS-CoV[J]. ACS Chem Biol, 2015, 10: 1456-1465. DOI:10.1021/cb500917m |
| [56] |
Chou CY, Chien CH, Han YS, et al. Thiopurine analogues inhibit papain-like protease of severe acute respiratory syndrome coronavirus[J]. Biochem Pharmacol, 2008, 75: 1601-1609. DOI:10.1016/j.bcp.2008.01.005 |
| [57] |
Cheng KW, Cheng SC, Chen WY, et al. Thiopurine analogs and mycophenolic acid synergistically inhibit the papain-like protease of Middle East respiratory syndrome coronavirus[J]. Antiviral Res, 2015, 115: 9-16. DOI:10.1016/j.antiviral.2014.12.011 |
| [58] |
Lin MH, Moses DC, Hsieh CH, et al. Disulfiram can inhibit MERS and SARS coronavirus papain-like proteases via different modes[J]. Antiviral Res, 2018, 150: 155-163. DOI:10.1016/j.antiviral.2017.12.015 |
| [59] |
Ratia K, Pegan S, Takayama J, et al. A noncovalent class of papain-like protease/deubiquitinase inhibitors blocks SARS virus replication[J]. Proc Natl Acad Sci U S A, 2008, 105: 16119-16124. DOI:10.1073/pnas.0805240105 |
| [60] |
Ghosh AK, Takayama J, Rao KV, et al. Severe acute respiratory syndrome coronavirus papain-like novel protease inhibitors:design, synthesis, protein-ligand X-ray structure and biological evaluation[J]. J Med Chem, 2010, 53: 4968-4979. DOI:10.1021/jm1004489 |
| [61] |
Baez-Santos YM, Barraza SJ, Wilson MW, et al. X-ray structural and biological evaluation of a series of potent and highly selective inhibitors of human coronavirus papain-like proteases[J]. J Med Chem, 2014, 57: 2393-2412. DOI:10.1021/jm401712t |
| [62] |
Ahn DG, Choi JK, Taylor DR. Biochemical characterization of a recombinant SARS coronavirus nsp12 RNA-dependent RNA polymerase capable of copying viral RNA templates[J]. Arch Virol, 2012, 157: 2095-2104. DOI:10.1007/s00705-012-1404-x |
| [63] |
Lung J, Lin YS, Yang YH, et al. The potential chemical structure of anti-SARS-CoV-2 RNA-dependent RNA polymerase[J]. J Med Virol, 2020. |
| [64] |
Oestereich L, Ludtke A, Wurr S. Successful treatment of advanced Ebola virus infection with T-705(favipiravir) in a small animal model[J]. Antiviral Res, 2014, 105: 17-21. DOI:10.1016/j.antiviral.2014.02.014 |
| [65] |
Wang M, Cao R, Zhang L, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro[J]. Cell Res, 2020, 30: 269-271. DOI:10.1038/s41422-020-0282-0 |
| [66] |
Barnard DL, Hubbard VD, Burton J, et al. Inhibition of severe acute respiratory syndrome-associated coronavirus (SARSCoV) by calpain inhibitors and beta-D-N4-hydroxycytidine[J]. Antivir Chem Chemother, 2004, 15: 15-22. DOI:10.1177/095632020401500102 |
| [67] |
Adedeji AO, Singh K, Calcaterra NE, et al. Severe acute respiratory syndrome coronavirus replication inhibitor that interferes with the nucleic acid unwinding of the viral helicase[J]. Antimicrob Agents Chemother, 2012, 56: 4718-4728. DOI:10.1128/AAC.00957-12 |
| [68] |
Jang KJ, Jeong S, Kang DY, et al. A high ATP concentration enhances the cooperative translocation of the SARS coronavirus helicase nsP13 in the unwinding of duplex RNA[J]. Sci Rep, 2020, 10: 4481. DOI:10.1038/s41598-020-61432-1 |
| [69] |
Adedeji AO, Singh K, Kassim A, et al. Evaluation of SSYA10-001 as a replication inhibitor of severe acute respiratory syndrome, mouse hepatitis, and Middle East respiratory syndrome coronaviruses[J]. Antimicrob Agents Chemother, 2014, 58: 4894-4898. DOI:10.1128/AAC.02994-14 |
| [70] |
Yu MS, Lee J, Lee JM, et al. Identification of myricetin and scutellarein as novel chemical inhibitors of the SARS coronavirus helicase, nsP13[J]. Bioorg Med Chem Lett, 2012, 22: 4049-4054. DOI:10.1016/j.bmcl.2012.04.081 |
| [71] |
Kim MK, Yu MS, Park HR, et al. 2, 6-Bis-arylmethyloxy-5-hydroxychromones with antiviral activity against both hepatitis C virus (HCV) and SARS-associated coronavirus (SCV)[J]. Eur J Med Chem, 2011, 46: 5698-5704. DOI:10.1016/j.ejmech.2011.09.005 |
| [72] |
Tanner JA, Zheng BJ, Zhou J, et al. The adamantane-derived bananins are potent inhibitors of the helicase activities and replication of SARS coronavirus[J]. Chem Biol, 2005, 12: 303-311. DOI:10.1016/j.chembiol.2005.01.006 |
| [73] |
Bouvet M, Imbert I, Subissi L, et al. RNA 3'-end mismatch excision by the severe acute respiratory syndrome coronavirus nonstructural protein nsp10/nsp14 exoribonuclease complex[J]. Proc Natl Acad Sci U S A, 2012, 109: 9372-9377. DOI:10.1073/pnas.1201130109 |
| [74] |
Bouvet M, Debarnot C, Imbert I, et al. In vitro reconstitution of SARS-coronavirus mRNA cap methylation[J]. PLoS Pathog, 2010, 6. |
| [75] |
Decroly E, Debarnot C, Ferron F, et al. Crystal structure and functional analysis of the SARS-coronavirus RNA cap 2'-O-methyltransferase nsp10/nsp16 complex[J]. PLoS Pathog, 2011, 7. |
| [76] |
Aouadi W, Blanjoie A, Vasseur JJ, et al. Binding of the methyl donor S-adenosyl-l-methionine to middle east respiratory syndrome coronavirus 2'-O-methyltransferase nsp16 promotes recruitment of the allosteric activator nsp10[J]. J Virol, 2017, 91: e02217-02216. |
| [77] |
Li W, Moore MJ, Vasilieva N, et al. Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus[J]. Nature, 2003, 426: 450-454. DOI:10.1038/nature02145 |
| [78] |
Li W, Sui J, Huang IC, et al. The S proteins of human coronavirus NL63 and severe acute respiratory syndrome coronavirus bind overlapping regions of ACE2[J]. Virology, 2007, 367: 367-374. DOI:10.1016/j.virol.2007.04.035 |
| [79] |
Huentelman MJ, Zubcevic J, Hernandez Prada JA, et al. Structure-based discovery of a novel angiotensin-converting enzyme 2 inhibitor[J]. Hypertension, 2004, 44: 903-906. DOI:10.1161/01.HYP.0000146120.29648.36 |
| [80] |
Simmons G, Gosalia DN, Rennekamp AJ, et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry[J]. Proc Natl Acad Sci U S A, 2005, 102: 11876-11881. DOI:10.1073/pnas.0505577102 |
| [81] |
Zhou Y, Vedantham P, Lu K, et al. Protease inhibitors targeting coronavirus and filovirus entry[J]. Antiviral Res, 2015, 116: 76-84. DOI:10.1016/j.antiviral.2015.01.011 |
| [82] |
Zhou N, Pan T, Zhang J, et al. Glycopeptide antibiotics potently inhibit cathepsin L in the late endosome/lysosome and block the entry of Ebola virus, Middle East respiratory syndrome coronavirus (MERS-CoV), and severe acute respiratory syndrome coronavirus (SARS-CoV)[J]. J Biol Chem, 2016, 291: 9218-9232. DOI:10.1074/jbc.M116.716100 |
| [83] |
Izaguirre G. The proteolytic regulation of virus cell entry by furin and other proprotein convertases[J]. Viruses, 2019, 11: 837. DOI:10.3390/v11090837 |
| [84] |
Follis KE, York J, Nunberg JH. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell-cell fusion but does not affect virion entry[J]. Virology, 2006, 350: 358-369. DOI:10.1016/j.virol.2006.02.003 |
| [85] |
Millet JK, Whittaker GR. Host cell entry of Middle East respiratory syndrome coronavirus after two-step, furin-mediated activation of the spike protein[J]. Proc Natl Acad Sci U S A, 2014, 111: 15214-15219. DOI:10.1073/pnas.1407087111 |
| [86] |
Xiong R, Zhang LK, Li SL, et al. Novel and potent inhibitors targeting DHODH, a rate-limiting enzyme in de novo pyrimidine biosynthesis, are broad-spectrum antiviral against RNA viruses including newly emerged coronavirus SARS-CoV-2[J]. bioRxiv, 2020. |
| [87] |
Cheung NN, Lai KK, Dai J, et al. Broad-spectrum inhibition of common respiratory RNA viruses by a pyrimidine synthesis inhibitor with involvement of the host antiviral response[J]. J Gen Virol, 2017, 98: 946-954. DOI:10.1099/jgv.0.000758 |
| [88] |
Hoffmann M, Kleine-Weber H, Schroeder S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor[J]. Cell, 2020, 181: 271-280. DOI:10.1016/j.cell.2020.02.052 |
| [89] |
Glowacka I, Bertram S, Muller MA. Evidence that TMPRSS2 activates the severe acute respiratory syndrome coronavirus spike protein for membrane fusion and reduces viral control by the humoral immune response[J]. J Virol, 2011, 85: 4122-4134. DOI:10.1128/JVI.02232-10 |
| [90] |
Matsuyama S, Nagata N, Shirato K, et al. Efficient activation of the severe acute respiratory syndrome coronavirus spike protein by the transmembrane protease TMPRSS2[J]. J Virol, 2010, 84: 12658-12664. DOI:10.1128/JVI.01542-10 |
| [91] |
Shirato K, Kawase M, Matsuyama S. Middle East respiratory syndrome coronavirus infection mediated by the transmembrane serine protease TMPRSS2[J]. J Virol, 2013, 87: 12552-12561. DOI:10.1128/JVI.01890-13 |
| [92] |
Yamamoto M, Matsuyama S, Li X, et al. Identification of nafamostat as a potent inhibitor of Middle East respiratory syndrome coronavirus S protein-mediated membrane fusion using the split-protein-based cell-cell fusion assay[J]. Antimicrob Agents Chemother, 2016, 60: 6532-6539. DOI:10.1128/AAC.01043-16 |
| [93] |
Gw Rogers Komar, Merrick WC. eIF4A:the godfather of the DEAD box helicases[J]. Prog Nucleic Acid Res Mol Biol, 2002, 72: 307-331. DOI:10.1016/S0079-6603(02)72073-4 |
| [94] |
Muller C, Schulte FW, Lange-Grunweller K, et al. Broad-spectrum antiviral activity of the eIF4A inhibitor silvestrol against corona and picornaviruses[J]. Antiviral Res, 2018, 150: 123-129. DOI:10.1016/j.antiviral.2017.12.010 |
| [95] |
Bradley WD, Koleske AJ. Regulation of cell migration and morphogenesis by Abl-family kinases:emerging mechanisms and physiological contexts[J]. J Cell Sci, 2009, 122: 3441-3454. DOI:10.1242/jcs.039859 |
| [96] |
Sisk JM, Frieman MB, Machamer CE. Coronavirus S protein-induced fusion is blocked prior to hemifusion by Abl kinase inhibitors[J]. J Gen Virol, 2018, 99: 619-630. DOI:10.1099/jgv.0.001047 |
| [97] |
Dyall J, Coleman CM, Hart BJ, et al. Repurposing of clinically developed drugs for treatment of Middle East respiratory syndrome coronavirus infection[J]. Antimicrob Agents Chemother, 2014, 58: 4885-4893. DOI:10.1128/AAC.03036-14 |
| [98] |
Skou JC. The identification of the sodium pump[J]. Biosci Rep, 1998, 18: 155-169. DOI:10.1023/A:1020196612909 |
| [99] |
Haas M, Askari A, Xie Z. Involvement of Src and epidermal growth factor receptor in the signal-transducing function of Na+/K+-ATPase.[J]. J Biol Chem, 2000, 275: 27832-27837. |
| [100] |
Mohammadi K, Kometiani P, Xie Z, et al. Role of protein kinase C in the signal pathways that link Na+/K+-ATPase to ERK1/2.[J]. J Biol Chem, 2001, 276: 42050-42056. DOI:10.1074/jbc.M107892200 |
| [101] |
Burkard C, Verheije MH, Haagmans BL, et al. ATP1A1-mediated Src signaling inhibits coronavirus entry into host cells[J]. J Virol, 2015, 89: 4434-4448. DOI:10.1128/JVI.03274-14 |
| [102] |
Von Kleist L, Stahlschmidt W, Bulut H. Role of the clathrin terminal domain in regulating coated pit dynamics revealed by small molecule inhibition[J]. Cell, 2011, 146: 471-484. DOI:10.1016/j.cell.2011.06.025 |
| [103] |
Milewska A, Nowak P, Owczarek K. Entry of human coronavirus NL63 into the cell[J]. J Virol, 2018, 92: e01933-01917. |
| [104] |
Schneider M, Ackermann K, Stuart M, et al. Severe acute respiratory syndrome coronavirus replication is severely impaired by MG132 due to proteasome-independent inhibition of M-calpain[J]. J Virol, 2012, 86: 10112-10122. DOI:10.1128/JVI.01001-12 |
| [105] |
Parsons SJ, Parsons JT. Src family kinases, key regulators of signal transduction[J]. Oncogene, 2004, 23: 7906-7909. DOI:10.1038/sj.onc.1208160 |
| [106] |
Shin JS, Jung E, Kim M. Saracatinib inhibits Middle East respiratory syndrome-coronavirus replication in vitro[J]. Viruses, 2018, 10: 283. DOI:10.3390/v10060283 |
| [107] |
Orton RJ, Sturm OE, Vyshemirsky V. Computational modelling of the receptor-tyrosine-kinase-activated MAPK pathway[J]. Biochem J, 2005, 392: 249-261. DOI:10.1042/BJ20050908 |
| [108] |
Kindrachuk J, Ork B, Hart BJ, et al. Antiviral potential of ERK/MAPK and PI3K/AKT/mTOR signaling modulation for Middle East respiratory syndrome coronavirus infection as identified by temporal kinome analysis[J]. Antimicrob Agents Chemother, 2015, 59: 1088-1099. DOI:10.1128/AAC.03659-14 |
| [109] |
Osaki M, Oshimura M, Ito H. PI3K-Akt pathway:its functions and alterations in human cancer[J]. Apoptosis, 2004, 9: 667-676. DOI:10.1023/B:APPT.0000045801.15585.dd |
| [110] |
Ma-Lauer Y, Zheng Y, Malesevic M, et al. Influences of cyclosporin A and non-immunosuppressive derivatives on cellular cyclophilins and viral nucleocapsid protein during human coronavirus 229E replication[J]. Antiviral Res, 2020, 173: 104620. DOI:10.1016/j.antiviral.2019.104620 |
| [111] |
Llorens-Martín M, Jurado J, Hernández F, et al. GSK-3β, a pivotal kinase in Alzheimer disease[J]. Front Mol Neurosci, 2014, 7: 46. |
| [112] |
Wu CH, Yeh SH, Tsay YG, et al. Glycogen synthase kinase-3 regulates the phosphorylation of severe acute respiratory syndrome coronavirus nucleocapsid protein and viral replication[J]. J Biol Chem, 2009, 284: 5229-5239. DOI:10.1074/jbc.M805747200 |
| [113] |
Chan JF, Chan KH, Kao RY, et al. Broad-spectrum antivirals for the emerging Middle East respiratory syndrome coronavirus[J]. J Infect, 2013, 67: 606-616. DOI:10.1016/j.jinf.2013.09.029 |
| [114] |
Ko M, Chang SY, Byun SY, et al. Screening of FDA-approved drugs using a MERS-CoV clinical isolate from South Korea identifies potential therapeutic options for COVID-19[J]. bioRxiv, 2020. |
| [115] |
Liu Q, Xia S, Sun ZW, et al. Testing of Middle East respiratory syndrome coronavirus replication inhibitors for the ability to block viral entry[J]. Antimicrob Agents Chemother, 2015, 59: 742-744. DOI:10.1128/AAC.03977-14 |
| [116] |
Shi YL, Zhang XB, Mu KJ, et al. D3Targets-2019-nCoV:a webserver for predicting drug targets and for multi-target and multi-site based virtual screening against COVID-19[J]. Acta Pharm Sin B, 2020. |
| [117] |
Koes DR, Baumgartner MP, Camacho CJ. Lessons learned in empirical scoring with smina from the CSAR 2011 benchmarking exercise[J]. J Chem Inf Model, 2013, 53: 1893-1904. DOI:10.1021/ci300604z |
| [118] |
Gordon CJ, Tchesnokov EP, Feng JY, et al. The antiviral compound remdesivir potently inhibits RNA-dependent RNA polymerase from Middle East respiratory syndrome coronavirus[J]. J Biol Chem, 2020, 295: 4773-4779. DOI:10.1074/jbc.AC120.013056 |
 2020, Vol. 55
2020, Vol. 55


