新型冠状病毒(severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)引起的传染性肺炎在世界范围内快速蔓延, 已成为全球性紧急卫生事件[1-3]。基因组测序表明, 这种病毒与蝙蝠冠状病毒有96.2%的同源性, 与严重急性呼吸综合症相关冠状病毒(severe acute respiratory syndrome coronavirus, SARS-CoV)的鉴定序列共有79.5%[4-6]。这种新的冠状病毒被国际病毒分类委员会命名为严重急性呼吸系统综合症冠状病毒2 (SARS-CoV-2)。目前尚无预防和治疗SARS-CoV-2感染的疫苗或抗病毒药。尽管α-干扰素和抗HIV药物洛匹那韦/利托那韦的联合治疗已在临床上开展了试验性治疗, 但疗效非常有限, 并且存在毒副作用[7]。初步的临床研究显示, 吉利德公司开发的广谱抗病毒药物雷姆昔韦具有一定的疗效, 但还需要进一步证明其临床应用价值[8, 9]。因此, 迫切需要发展疗效好、安全性高的特定抗SARS-CoV-2药物。
基因组序列的相似性表明该病毒属于Betacoronavirus属的Sarbecovirus亚属[6]。冠状病毒是有包膜的正义的单链RNA病毒。基因组RNA长度约为30 kb, 具有5'帽结构和3'-poly-A尾巴, 并包含至少6个开放阅读框(open reading frame, ORF)[10, 11]。第一个ORF (ORF 1a/b)大约占基因组长度的三分之二, 编码pp1a和pp1ab两种多蛋白。这些多蛋白主要由3CL蛋白酶(3C-like protease, 3CLpro; 也称main protease, Mpro)和木瓜蛋白酶(papain-like proteases, PLPs)加工成16种非结构蛋白(Nsps)。3CL蛋白酶属于半胱氨酸蛋白酶, 活性形式为同源二聚体。3CL蛋白酶在冠状病毒中较为保守, 并且不同冠状病毒3CL蛋白酶的底物之间具有共同特征, 例如切割位点的P1位基本都是Gln[12, 13]。
3CL蛋白酶在冠状病毒的生命周期中起着至关重要的作用, 其失活可抑制病毒的复制。同时, 由于人体内不存在与3CL蛋白酶同源的蛋白酶, 因此其成为理想的抗冠状病毒靶标[13, 14]。本研究利用生物发光共振能量转移(BRET)的技术建立了细胞水平SARS-CoV-2病毒3CL蛋白酶抑制剂筛选模型, 并初步应用和评价该筛选模型。结果表明, 该方法可以应用于3CL蛋白酶抑制剂的筛选与评价, 为新药研发提供了有力工具。
材料与方法质粒构建 pRLuc-N2质粒购自PerkinElmer公司; pEYFP-N1购自Clontech公司; 以pEYFP-N1质粒为模板, 将3CL蛋白酶识别序列Linker (ITSAVLQSGFRK)通过引物设计, PCR扩增得到C端融合了Linker的增强的黄色荧光蛋白(enhanced yellow fluorescent protein, EYFP)基因片段, 将此片段插入pRLuc-N2, 得到pEYFP-linker-Rluc质粒。PcDNA3.1-3CL-flag质粒中带有密码子优化的SARS-CoV-2 3CL蛋白酶编码序列。
细胞培养及转染 293T细胞(本室保存)培养于含100单位青霉素、链霉素和10%胎牛血清的DMEM培养基中。6孔板每孔接种细胞4×105个, 培养24 h后转染, pEYFP-linker-Rluc质粒转染量为每孔300 ng, 其他质粒根据实验比例而定, 转染试剂为Lipofectamine 2000, 根据使用说明书进行转染。
生物发光共振能量转移比值(BRET ratio)测定 转染48 h后, 弃培养基, 6孔板每孔加入冰上预冷的PBS 1 mL收取细胞, 调整细胞数为每毫升1×106个, 取100 μL放入白色不透明、平底的96孔板中, 加入海肾荧光素酶(Renilla luciferase, Rluc)底物coelenterazine h (Promega公司), 终浓度为5 μmol·L-1, 测定470 nm (±10 nm)和535 nm (±10 nm)两处发射光强度。计算BRET比值。
免疫印迹(Western blotting)检测 2 000 ×g离心5 min收集细胞, RIPA [50 mmol·L-1 Tris-HCl (pH 7.4), 150 mmol·L-1 NaCl, 1% NP-40, 0.1% SDS, Roche蛋白酶抑制剂混合物]裂解, 提取细胞总蛋白。SDS-PAGE分离蛋白样品。转膜后, 先后与一抗和二抗孵育, 然后ECL显色。Anti-GFP抗体购自Santa Cruz公司, anti-β-actin抗体购自Abcam公司, anti-flag抗体购自博奥龙公司, 羊抗鼠(二抗)、羊抗兔(二抗)均购自中杉金桥公司。
高通量筛选流程及模型评价 10 cm细胞培养皿接种2.4×106个细胞, 培养24 h后转染, 转染12 h后, 胰酶消化, 加入DMEM培养基30 mL, 轻轻吹打, 使细胞分散为单细胞悬液, 接种于96孔板, 每孔150 μL。培养12 h后, 加入待测药物。药物处理培养16 h后, 吸去培养基, 每孔加入含5 μmol·L-1 coelenterazine h的PBS溶液100 μL, 测定BRET比值。为评价此筛选模型的性质, 按Gao等[15]的方法计算Z'因子。Z' = 1 - 3(STD3CL+group+STD3CL-group)/(MEAN3CL+group-MEAN3CL-group)。
统计学分析 实验数据用
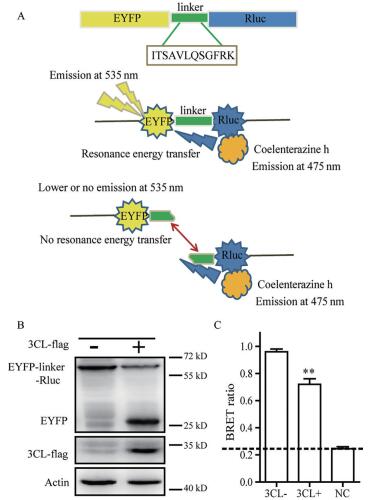
本研究依据BRET技术建立了SARS-CoV-2病毒3CL蛋白酶抑制剂筛选模型, 其工作原理如图1A所示。Linker序列是SARS-CoV-2多聚蛋白中相邻的12个氨基酸, 与SARS-CoV的序列保守一致, 为3CL蛋白酶特异识别。本文将Linker的编码序列插入到编码EYFP和RLuc之间, 构建了EYFP-linker-Rluc融合蛋白真核表达载体, 用于在细胞内表达3CL蛋白酶的底物。该模型的工作原理为, 在表达EYFP-linker-Rluc的细胞中, 加入Rluc底物coelenterazine h后, 将产生发射光(λem~475 nm), 在Rluc与EYFP距离小于100 Å时, Rluc催化产生的光波发生能量转移, 激发受体EYFP, 产生激发光(λem~535 nm)。当3CL蛋白酶酶切linker序列后, 使得EYFP和Rluc成为两个独立的蛋白。两者从融合状态改变成分离状态, 这将直接阻断能量转移, 导致BRET比值下降。当3CL蛋白酶活性被抑制时, BRET比值将上升(图1A)。
|
Figure 1 Overview of cell-based screening assay. A: Schematic representation of the bioluminescence resonance energy transfer (BRET) assay. EYFP-linker-Rluc fusion protein is cleaved efficiently by 3C-like protease (3CL); B: Western blotting analysis; C: Measurement of BRET ratio. n = 3, |
为了证明3CL蛋白酶能有效酶切EYFP-linker-Rluc序列, 并相应地引起BRET比值的改变, 采用EYFP-linker-Rluc和3CL表达质粒共转染293T细胞, 然后进行了Western blotting分析(图1B)和BRET比值测定(图1C)。如图1B所示, 当3CL蛋白酶存在时, 融合蛋白能够被有效切割。同时, 可以发现BRET比值下降(图1C), 与Western blotting结果相一致。以上结果表明, 3CL蛋白酶能够有效剪切EYFP-linker-Rluc融合蛋白, 并可导致BRET值的下降。同时, 利用pRLuc-N2与pEYFP-C1质粒共转293T细胞, 表达独立的EYFP和RLuc, 测定其BRET比值, 作为本底值进行差减, 后续的实验结果处理方法相同。
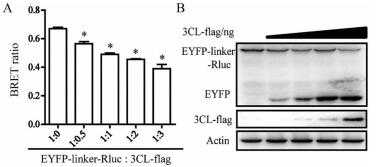
3 3CL蛋白酶活性的剂量关系为了明确细胞内3CL蛋白酶活性水平与其酶切融合蛋白EYFP-linker-Rluc效率的相关性, 本研究保持质粒pEYFP-linker-Rluc用量不变, 增加3CL表达质粒p3CL-flag的用量, 进行BRET比值测定(图 2A)和Western blotting检测(图 2B), 进一步分析底物剪切的情况。结果表明, 随着细胞内3CL蛋白酶表达量增高, BRET比值持续下降(图 2A)。同时, 伴随着融合蛋白EYFP-linker-Rluc的减少, 剪切产物EYFP逐渐增多(图 2B)。上述结果表明, 胞内3CL蛋白酶活性水平与底物剪切效率具有良好的量效关系。
|
Figure 2 Dose-dependence of 3CL activity. A: Measurement of BRET ratio; B: Western blotting analysis. n = 3, |
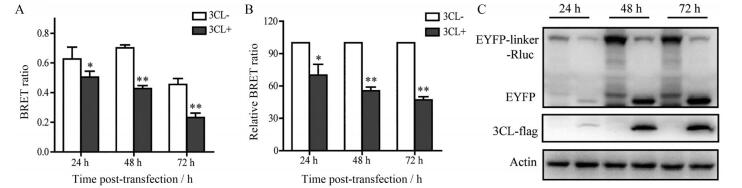
为了进一步明确筛选模型的实验条件, 本研究分析了不同测定时间对BRET结果的影响。利用EYFP-linker-Rluc和3CL表达质粒共转染293T细胞, 在不同时间点(24、48和72 h)收集细胞样品, 然后进行BRET比值测定(图 3A和B)和Western blotting分析(图 3C)。结果显示, 与转染后24 h相比, 在48与72 h进行检测时, 有无蛋白酶表达的BRET值相差较大。当BRET值的差值越大, 模型的灵敏度和重复性越高。为了节省检测时间, 本研究最终采用48 h为最佳检测时间。
|
Figure 3 Time-point of 3CL activity. A: Measurement of BRET ratio; B: Relative BRET ratio; C: Western blotting analysis. n = 3, |
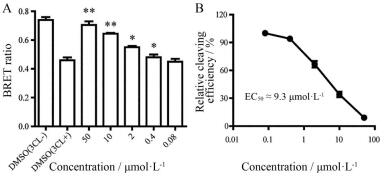
为了初步验证筛选的应用价值, 本研究测试了4个已报道的SARS-CoV-2 3CL蛋白酶小分子抑制剂ebselen、disulfiram、tideglusid与carmofur[16]。据报道, 上述化合物可以在体外抑制SARS-CoV-2 3CL蛋白酶, 但只有ebselen具有细胞水平抗SARS-CoV-2活性[16]。结果显示, 终浓度20 μmol·L-1化合物处理可以不同程度地提高BRET比值(图 4A), 其中ebselen的效果最为明显, 而tideglusid与carmofur处理未见明显变化。与之相应, 酶切效率分析发现, ebselen可以抑制55%左右的酶切能力, tideglusid与carmofur并不能抑制蛋白酶活性(图 4B)。

|
Figure 4 Influence of different drug treatments on intracellular 3CL activity. A: Measurement of BRET ratio; B: Relative cleaving efficiency. n = 3, |
进而本研究在该模型中测定了活性较好的ebselen的量效关系(图 5), 得到半数有效浓度(EC50)约为9.3 μmol·L-1。该结果与文献[16]报道的抗病毒活性EC50 4.67 μmol·L-1基本相当, 表明该筛选模型与细胞水平抗病毒活性分析具有较好的一致性。
|
Figure 5 Dose dependence of ebselen on intracellular 3CL activity. A: Measurement of BRET ratio; B: Relative cleaving efficiency. n = 3, |
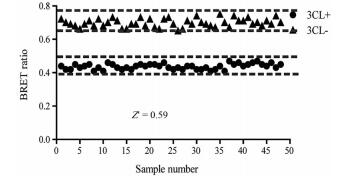
Z'因子是评价高通量筛选方法稳定性的一个重要参数。当Z'因子大于0.4, 则认为该方法较稳定, 适用于高通量药物筛选。计算得出本模型的Z'因子为0.59, 可以应用于高通量药物筛选(图 6)。
|
Figure 6 Determination of Z' factor in BRET-based high throughput screening assay (Z' = 0.59) |
由SARS-CoV-2引起的传染性肺炎在世界范围内广泛传播, 涉及到全球大部分国家和地区, 其传播速度快、涉及范围广、死亡率高, 对人民的生命造成了极大的威胁, 也给全球的经济带来了极大的冲击。因此, 急需开发有效的抗SARS-CoV-2的药物。SARS-CoV-2 3CL蛋白酶作为SARS-CoV-2病毒主要的蛋白水解酶, 可切割SARS-CoV-2的多聚蛋白, 在病毒新的生命周期中发挥了重要的作用, 因此该酶是抗病毒药物的理想靶标。目前SARS-CoV-2 3CL蛋白酶的晶体结构已经解析[16, 17], 酶的性质与抑制剂筛选等工作也取得了巨大进展[17]。
Jin等[16]通过计算机辅助药物设计鉴定了抑制剂N3, 并随后确定了3CL与该化合物的复合物的晶体结构, 并且通过基于结构的虚拟筛选和高通量筛选相结合的手段分析了10 000多种化合物; Dai等[18]设计并合成了针对Mpro的两种先导化合物(11a和11b)。两者的醛基共价键合到Mpro的Cys145, 均表现出优异的抑制活性和有效的抗SARS-CoV-2感染活性; Khan等[19]应用了计算药物设计方法从FDA批准的抗病毒药物以及合成来源的天然和类药物化合物的内部数据库中识别出几个胰凝乳蛋白酶样蛋白酶抑制剂为有前途的药物。
虽然针对此靶点的抑制剂筛选工作取得了巨大进展, 但是大部分工作均以虚拟筛选与体外酶水平筛选为技术手段。本研究首先建立了基于BRET技术的细胞水平筛选模型。与体外酶水平筛选方法相比, 该方法不仅可以尽可能排除由于非特异因素干扰酶活性而产生的假阳性结果, 同时还可以初步考量化合物的细胞毒性和进入细胞能力等因素。如上述结果所示, 该模型测定ebselen的EC50约为9.3 μmol·L-1, 与文献[16]报道的抗病毒活性EC50 4.67 μmol·L-1基本相当, 但与体外抑酶活性(IC50, 0.67 μmol·L-1)差别较大。同时, 前期研究发现tideglusid与carmofur尽管具有体外抑制3CL蛋白酶的活性, 但未发现抗SARS-CoV-2的能力。与抗病毒活性相一致, 本模型中未发现tideglusid与carmofur在细胞水平上具有抑制3CL蛋白酶的能力。这一结果表明, 该筛选模型与细胞水平抗病毒活性分析具有较好的一致性, 可以更好地反映或预测蛋白酶抑制剂的抗病毒能力, 为SARS-CoV-2蛋白酶抑制剂研发提供重要参考。
作者贡献: 岑山和马铃负责设计实验思路, 完成文章撰写; 马铃负责完成主要的实验部分; 赵建元、郭赛赛和谢永丽负责完成数据分析; 所有作者均对本文有所贡献。
利益冲突: 无利益冲突。
| [1] |
Zhu N, Zhang D, Wang W, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. N Engl J Med, 2020, 382: 727-733. DOI:10.1056/NEJMoa2001017 |
| [2] |
Li Q, Guan X, Wu P, et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia[J]. N Engl J Med, 2020, 382: 1199-1207. DOI:10.1056/NEJMoa2001316 |
| [3] |
Chan JFW, Yuan S, Kok KH, et al. A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission:a study of a family cluster[J]. Lancet, 2020, 395: 514-523. DOI:10.1016/S0140-6736(20)30154-9 |
| [4] |
Zhou P, Yang XL, Wang XG, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579: 270-273. DOI:10.1038/s41586-020-2012-7 |
| [5] |
Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratory disease in China[J]. Nature, 2020, 579: 265-269. DOI:10.1038/s41586-020-2008-3 |
| [6] |
Lu R, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding[J]. Lancet, 2020, 395: 565-574. DOI:10.1016/S0140-6736(20)30251-8 |
| [7] |
Cao B, Wang Y, Wen D, et al. A trial of lopinavir-ritonavir in adults hospitalized with severe covid-19[J]. N Engl J Med, 2020, 382: 1787-1799. DOI:10.1056/NEJMoa2001282 |
| [8] |
Holshue ML, DeBolt C, Lindquist S, et al. First case of 2019 novel coronavirus in the United States[J]. N Engl J Med, 2020, 382: 929-936. DOI:10.1056/NEJMoa2001191 |
| [9] |
Wang M, Cao R, Zhang L, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro[J]. Cell Res, 2020, 30: 269-271. DOI:10.1038/s41422-020-0282-0 |
| [10] |
Chen Y, Liu Q, Guo D. Emerging coronaviruses:genome structure, replication, and pathogenesis[J]. J Med Virol, 2020, 92: 418-423. DOI:10.1002/jmv.25681 |
| [11] |
Hussain S, Pan JA, Chen Y, et al. Identification of novel subgenomic RNAs and noncanonical transcription initiation signals of severe acute respiratory syndrome coronavirus[J]. J Virol, 2005, 79: 5288. DOI:10.1128/JVI.79.9.5288-5295.2005 |
| [12] |
Hayden FG, Turner RB, Gwaltney JM, et al. Phase II, randomized, double-blind, placebo-controlled studies of ruprintrivir nasal spray 2-percent suspension for prevention and treatment of experimentally induced rhinovirus colds in healthy volunteers[J]. Antimicrob Agents Chemother, 2003, 47: 3907. DOI:10.1128/AAC.47.12.3907-3916.2003 |
| [13] |
Kim Y, Liu H, Galasiti Kankanamalage AC, et al. Reversal of the progression of fatal coronavirus infection in cats by a broad-spectrum coronavirus protease inhibitor[J]. PLoS Pathog, 2016, 12: e1005531. DOI:10.1371/journal.ppat.1005531 |
| [14] |
Yang H, Xie W, Xue X, et al. Design of wide-spectrum inhibitors targeting coronavirus main proteases[J]. PLoS Biol, 2005, 3: e324. DOI:10.1371/journal.pbio.0030324 |
| [15] |
Gao Q, Wang Z, Liu Z, et al. A cell-based high-throughput approach to identify inhibitors of influenza A virus[J]. Acta Pharm Sin B, 2014, 4: 301-306. DOI:10.1016/j.apsb.2014.06.005 |
| [16] |
Jin Z, Du X, Xu Y, et al. Structure of M(pro) from SARS-CoV-2 and discovery of its inhibitors[J]. Nature, 2020, 582: 289-293. DOI:10.1038/s41586-020-2223-y |
| [17] |
Zhang L, Lin D, Sun X, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors[J]. Science, 2020, 368: 409-412. |
| [18] |
Dai W, Zhang B, Su H, et al. Structure-based design of antiviral drug candidates targeting the SARS-CoV-2 main protease[J]. Science, 2020, 368: 1331-1335. DOI:10.1126/science.abb4489 |
| [19] |
Khan SA, Zia K, Ashraf S, et al. Identification of chymotrypsin-like protease inhibitors of SARS-CoV-2via integrated computational approach[J]. J Biomol Struct Dyn, 2020. DOI:10.1080/07391102.2020.1751298 |
 2020, Vol. 55
2020, Vol. 55


