2. 西藏藏医药大学, 西藏 拉萨 850000;
3. 北京中医药大学, 北京 100029
2. Tibetan Traditional Medical College, Lhasa 850000, China;
3. Beijing University of Chinese Medicine, Beijing 100029, China
青藏高原复杂的地质历史、多样化的生境及气候条件, 蕴育出丰富的动植物资源, 是研究物种分化和物种形成机制的理想地区; 同时, 高山植物常具有独特的次生产物结构类型及生物活性特质, 为寻找安全、高效的临床药物提供了更为广阔的研究空间。
龙胆科龙胆属Gentiana秦艽组Sect. Cruciata植物多分布于我国青藏高原[1], 物种及遗传背景丰富多样。该组多个物种常作为中药秦艽及“解吉那保” (རྒྱ་རྒྱུད་ནག་པོ་)等藏药材的基原植物, 具有重要的药用价值[2, 3]。
藏医药学是祖国民族医药学的重要组成部分, 具有悠久的发展历史及丰富的科学与藏族文化内涵。深入开展藏药品种整理, 探讨高山植物物种遗传多样性及藏药材品质与具体产地的相关性等问题, 可为藏药材客观、准确的品种鉴定、活性物质基础研究、质量标准的完善、濒危藏药植物的就地保护及栽培学研究等工作打下坚实基础, 具有重要的科学意义。但由于种种原因, 藏药品种整理等工作尚不能适应藏医药学发展的需要, 当务之急是开展相关深入的基础性研究工作。藏药“解吉那保”, 花、根入药, 能消肿, 治白喉等[3, 4]。课题组前期与西藏藏医药大学合作开展了相关民族药物学考察、基原植物标本采集、物种分类学鉴定及遗传背景初步分析等工作[5-10]。
粗茎秦艽Gentiana crassicaulis Duthie ex Burk., “解吉那保”主流品种之一, 也是中药秦艽基原植物之一。我国特有物种, 自然分布区涉及西藏、云南、四川、贵州、青海及甘肃等省区, 多见于海拔2 100~4 500 m的高山草甸、林下及林缘[1]。
近年来, 野生资源状况不容乐观, 粗茎秦艽G. crassicaulis Duthie ex Burk.及其秦艽组近缘物种麻花艽G. straminea Maxim.、小秦艽G. dahurica Fisch.及大叶秦艽G. macrophylla Pall.均被列入《国家重点保护野生药材物种名录》[11]。药用植物种质资源是中药生产的源头, 也是中药可持续利用的物质基础。对粗茎秦艽及其近缘药用种类开展深入的遗传背景分析, 进而开展种质资源评价与就地保护工作, 紧迫性不言而喻。
扩增片段长度多态性(amplified fragment length polymorphism, AFLP)是一种新的分子标记技术, 通过对基因组DNA的限制性酶切片段进行选择性扩增而揭示其多态性, 具有灵敏、稳定、多态性丰富等优点[12]。近年来, 该技术在中药种质资源的遗传多样性研究等领域得到广泛应用, 如对枸杞属Lycium[13]、滇重楼Paris polyphylla var. yunnanensis[14]、短柄乌头Aconitum brachypodum[15]及三叶木通Akebia trifoliata[16]等药用植物的遗传多样性分析等。为此, 在品种整理、自然分布区广泛取样及分类学鉴定基础上, 采用AFLP技术, 对163份粗茎秦艽种质样本遗传背景进行深入分析, 构建DNA指纹谱; 以期为高山药用植物粗茎秦艽核心种质构建、深入开展藏药“解吉那保”及秦艽类品种的种质资源保护、药材生药学鉴定及道地性研究等提供基础资料。
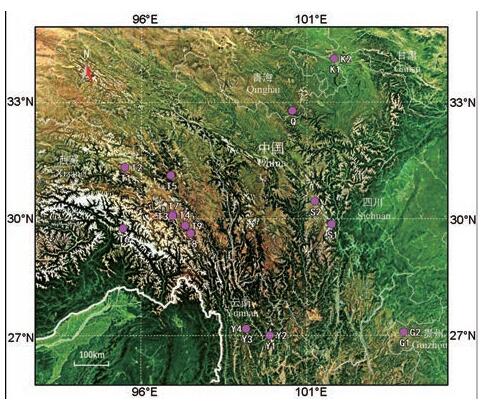
材料与方法民族植物学考察及分类学鉴定 自2010年起与西藏藏医药大学合作开展藏药“解吉那保”主流品种整理工作, 在西藏及云南等6省区粗茎秦艽G. crassicaulis自然分布区广泛采样, 共计20个居群, 163份种质样本(图 1和表 1); 同时, 取新鲜叶片硅胶快速干燥, 备用。采集外类群标本, 麻花艽G. straminea 2份、椭圆叶花锚Halenia elliptica 2份。去英国皇家植物园(Kew)植物标本馆查阅粗茎秦艽G. crassicaulis及其近缘物种的模式标本, 以确保物种各居群样本分类学鉴定的准确性。凭证标本存放于上海中医药大学中药学院药用植物标本室。
|
Figure 1 Map of the geographical distribution and sampled populations of G. crassicaulis |
| Table 1 Samples and voucher specimens. *cultivated; **outgroup. M: Gentiana straminea; H: Halenia elliptica |
总DNA提取 取适量干燥叶片, 加入液氮研磨成细粉状, 参考Doyle的CTAB法[17]并适当改良提取粗茎秦艽基因组DNA。
接头和引物 参考Vos等[18]方法合成AFLP接头和引物。EcoRⅠ接头1: 5′-CTCGTAGACTGCGCGTA CC-3′, 接头2: 5′-AATTGGTACGCAGTCTAC-3′; MseⅠ接头1: 5′-GACGATGAGTCCTGAG-3′, 接头2: 5′-TAC TCAGGACTCAT-3′。预扩增引物: EcoRI+A: 5′-GAC TGCGTACCAATTCA-3′; MseI+C: 5′-GATGAGTCCT GAGTAAC-3′。从64对引物中筛选出12对带型分布均匀、多态性高且分辨力强的引物进行AFLP分析。
酶切和连接 酶切、连接一步进行, 反应体系:模板DNA 500 ng, 10×Cut Smart Buffer 3 μL, EcoRⅠ-HF 0.5 μL, MseⅠ 0.5 μL, 10×T4 DNA Ligase Buffer 3 μL, T4 DNA Ligase (350 U·μL-1) 0.1 μL, 5 μmol·L-1EcoRI双链接头2 μL, 50 μmol·L-1 MseⅠ双链接头1 μL, ddH2O补充至30 μL, 混匀。反应程序: 37 ℃, 4 h; 65 ℃, 20 min。酶切-连接产物稀释10倍进行预扩增。
预扩增 体系:10×PCR Buffer 2 μL, MgCl2(25 mmol·L-1)1.2 μL, dNTP mixture (10 mmol·L-1)0.4 μL, 引物EA (10 μmol·L-1)1 μL, 引物MC (10 μmol·L-1)1 μL, Taq DNA聚合酶0.4 μL, 酶连产物10倍稀释液2 μL, ddH2O补充至20 μL, 离心混匀。循环参数:95℃, 3 min; 94℃, 30 s, 56℃, 45 s, 72℃, 80 s, 30 cycles; 72℃, 8 min。预扩增产物稀释20倍作为选择性扩增模板。
选择性扩增 体系:10×PCR Buffer 3 μL, MgCl2(25 mmol·L-1)1.8 μL, dNTP mixture (10 mmol·L-1)0.6 μL, 引物1(10 μmol·L-1)1 μL, 引物2(10 μmol·L-1)1 μL, Taq DNA聚合酶0.4 μL, 预扩产物20倍稀释液2 μL, ddH2O补充至30 μL, 离心混匀。循环参数:95℃, 5 min; 94℃, 30 s, 65℃(每循环降低0.7℃), 30 s, 72℃, 60 s, 12 cycles; 94℃, 30 s, 56℃, 45 s, 72℃, 80 s, 23 cycles; 72℃, 5 min。
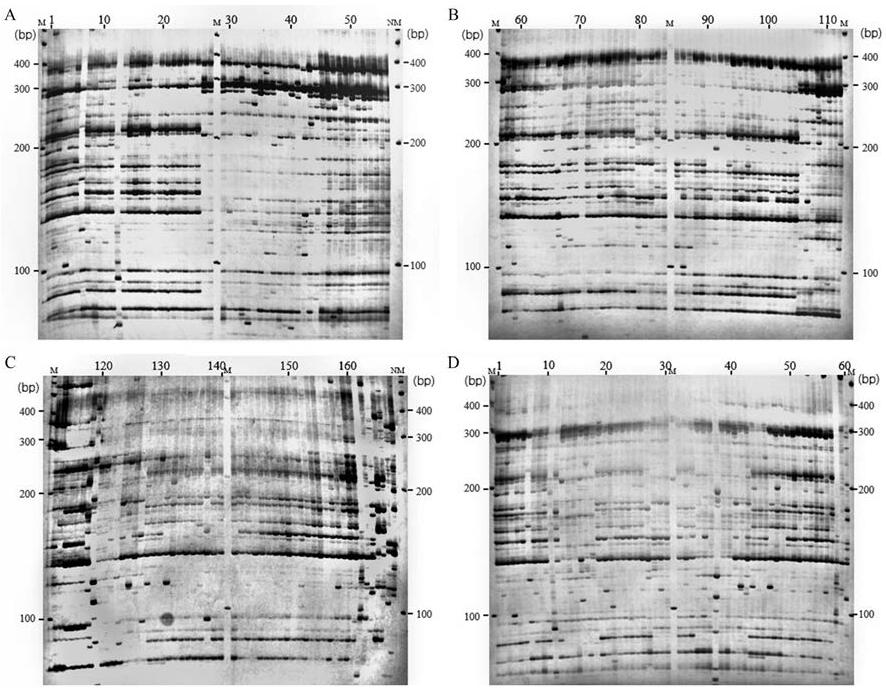
变性聚丙烯酰胺凝胶电泳 上样, 10%变性聚丙烯酰胺凝胶电泳。银染法染色, 拍照。样品1~167按顺序点样(三块胶); 同时为尽量减少由于样品处于不同胶板造成的读带误差, 粗茎秦艽每个居群均再取前3份样品平行点于另一块胶板中, 以做读带参照(图 2)。
|
Figure 2 AFLP fingerprints of genomic DNA of G. crassicaulis using primer combination MseⅠ+CAG/EcoRⅠ+ACG. M: SM0331 DNA Ladder; N: Negative; A-C: G. crassicaulis (lanes 1-163), G. straminea (lanes 164-165) and Halenia elliptica (lanes 166-167); D: Populations of G. crassicaulis. T1 (lanes 1-3), T2 (lanes 4-6), K1 (lanes 7-9), K2 (lanes 10-12), G1 (lanes 13-15), G2 (lanes 16-18), T3 (lanes 19-21), T4 (lanes 22-24), T5 (lanes 25-27), S1 (lanes 28-30), S2 (lanes 31-33), Y1 (lanes 34-36), Y2 (lanes 37-39), Y3 (lanes 40-42), Q (lanes 43-45), T6 (lanes 46-48), T7 (lanes 49-51), T8 (lanes 52-54), T9 (lanes 55-57) and Y4 (lanes 58-60) |
数据分析 在相同迁移率位置, 每个样品的扩增带按有(记为“1”)或无(记为“0”)记录, 只记录清晰易辨条带, 由此得到“0, 1”矩阵。使用POPGENE 32软件计算相关遗传参数; 同时使用NTSYS pc-V.2.10e进行UPGMA聚类分析, 以检验居群间和个体间的亲缘关系。利用GenAlEx 6.5软件进行PCA分析、分子方差分析(AMOVA)、Mantel检验以及空间自相关分析。
cpDNA SSR及SNP标记分析 为探讨四川康定县居群S1的分化问题, 测定cpDNA psbI-trnS (GCU)、trnS (GCU)-trnG (UCC)、rpoB-trnC (GCA)及ycf3 intron1序列, 以筛选SSR标记; 测定cpDNA ccsA、petA-psbJ、rpl16 intron及rpl33序列, 以筛选SNP标记。
结果 1 “解吉那保”基原植物形态特征及分类学位置多年生草本。茎基部被枯存的纤维状叶鞘包裹。基生叶莲座丛状; 茎生叶对生, 最上部叶密集呈苞叶状包被花序。花多数, 茎顶簇生呈头状; 花冠冠檐蓝紫色, 长2~2.2 cm。我国特有物种。分布于西藏、云南、四川、贵州、青海及甘肃等省区。生于山坡草地、高山草甸、灌丛等, 海拔2 100~4 500 m。形态特征及后选模式标本(Lectotype, Pratt 463; K!)见图 3。

|
Figure 3 Gentiana crassicaulis (Zhao ZL, et al 2017XZ009, alt. 3928 m, Zuogong, Tibet). A: Habit; B: Inflorescence; C: Lectotype (Pratt 463; K!) |
经查阅模式标本、参考相关分类学文献并进行分类学鉴定, 其基原为龙胆科龙胆属Gentiana秦艽组Sect. Cruciata植物粗茎秦艽Gentiana crassicaulis Duthie ex Burk.。据此, 对实验中所涉及的各居群标本逐一鉴定, 以确保物种遗传背景分析结果的科学性。
2 AFLP扩增结果从64对“3+3”引物组合中筛选出扩增带纹清晰, 多态性丰富且多态性位点百分率较高的引物组合12对, 对样品进行扩增。扩增片段多在50~500 bp之间, 共获得315个位点, 其中多态性位点(PL) 254个, 多态位点百分率(PPL)平均值为80.63%, 表明粗茎秦艽种质资源的遗传背景存在一定多样性。每对引物扩增情况见表 2, 其中MseⅠ+CAG/EcoRⅠ+ACG扩增图谱见图 2。
| Table 2 Selective amplification of the restriction fragments within G. crassicaulis using 12 primer combinations. PPL: Percentage of polymorphic loci |
对检测群体遗传多样性指标多态位点百分率(PPL)、观察等位基因数(Na)、有效等位基因数(Ne)、Nei's基因多样性(H)及Shannon信息指数(Im)进行统计, 结果见表 3。粗茎秦艽居群PPL变化区间为0.95%~19.37%, 平均百分比为6.85%; Na变化区间为1.009 5~1.193 7, 平均为1.068 7; Ne变化区间为1.005 4~1.126 2, 平均为1.039 9; H变化区间为0.0038~0.073 6, 平均为0.023 3; Im变化区间为0.005 2~0.109 2, 平均为0.035 0。物种水平的Na则为1.727 0, Ne为1.315 0, H为0.189 9, Im为0.292 7。显示粗茎秦艽物种不同居群间遗传多样性有差异, 其中Y4居群遗传多样性水平最高, Nei's基因多样性和Shannon信息指数最大, 分别为0.073 6和0.109 2。
| Table 3 Analysis of genetic diversity within populations. Na: Observed number of alleles; Ne: Effective number of alleles; H: Gene diversity; Im: Shannon's Information index |
POPGENE分析软件显示:粗茎秦艽居群总变异系数(Ht: total genetic diversity for species)为0.185 3, 群体内的遗传多样性(Hs: genetic diversity within populations)为0.023 3, 群体间的遗传多样性(Dst: inter-subpopulation gene diversity)为0.162 0, 遗传分化系数(Gst: coefficient of gene differentiation)为0.874 3, 基因流(Nm: gene flow)为0.071 9。居群间遗传分化远高于居群内, 遗传变异主要存在于居群间(87%), 居群内部的遗传变异较小(13%)。
GenAlEx 6.5软件AMOVA分析时, 居群间的遗传分化系数一般用分化指数Fst表示, 它通过等位基因频率计算得到, 但其需要居群处于Hardy-Weinberg平衡时才有意义[19, 20]。由于AFLP为显性标记, 不能满足此条件, 通常以AMOVA分析计算的Fst相似数Φst (即表 4中的PhiPT值)来代替, 而Gst是POPGENE1.32软件在假定Hardy-Weinberg平衡情形下估算的居群间的遗传分化值。因此, Gst与Φst都是Fst的近似值[21]。由此, Gst和Φst分别为0.874 3和0.866, 二者非常接近, AMOVA分析和POPGENE分析软件的相关推断相吻合, 见表 4。遗传分化指数Fst在0~0.05之间表示群体间无分化; 0.05~0.15间为中度分化; 0.15~0.25间为高度分化; 大于0.25表示极高的分化[22]。显然, 粗茎秦艽不同居群间遗传分化高, 遗传差异显著性极大。
| Table 4 AMOVA based on AFLP data set from G. crassicaulis. PhiPT = AP/(WP+AP) = AP/TOT, AP = Est. Var. Among Pops, WP = Est. Var. Within Pops. The significance test for PhiPT was based on 999 permutations across the full data set (P < 0.001) |
采用非加权组平均法(unweighted pair-group method with arithmetic means, UPGMA)进行聚类分析, 结果显示:首先外类群龙胆科花锚属椭圆叶花锚Halenia elliptica及龙胆属近缘种麻花艽G. straminea被依次分出; 而粗茎秦艽G. crassicaulis所有20个居群共聚为另一大支。粗茎秦艽物种内, 则分为I、II两支:支I由西藏、甘肃、青海及四川各居群组成, 支II由贵州及云南各居群组成; 同时, 除云南Y4居群(栽培)外, 相同省区内各居群均分别聚为一支, 显现出高度的地域相关性, 即相同居群个体或空间距离较近的居群多显示出更为接近的亲缘关系(图 4)。

|
Figure 4 UPGMA phenogram based on AFLP data set from 20 populations of G. crassicaulis. Outgroup: G. straminea and Halenia elliptica |
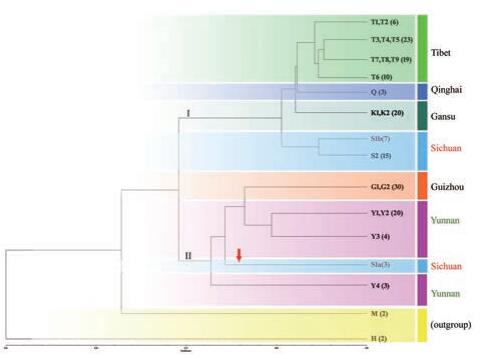
基于粗茎秦艽物种163份种质构建UPGMA聚类树。拓扑结构基本一致:首先, 外类群分出; 物种内分为两大支, 西藏、甘肃、青海及四川大部分个体(S2居群全部个体及由S1居群7株个体组成的S1b)构成支I; 值得注意的是, 支II除了包括贵州及云南各居群个体, 四川少数个体(S1居群3个组成的S1a)也在此聚类(图 5)。
|
Figure 5 UPGMA phenogram based on AFLP data set from 163 individuals of G. crassicaulis. Outgroup: G. straminea and H. elliptica. In brackets: Individual numbers |
主成分分析(PCA)可直观反映种质之间的遗传及分类学特性。GenAlEx 6.5软件[23-25]分析显示:前两个坐标分别占总遗传变异的40.46%和11.57%;总体结果与UPGMA聚类树拓扑结构基本一致, 均表现出明显的地理相关性。外类群椭圆叶花锚H. elliptica及麻花艽G. straminea (M, H)在第Ⅲ象限。四川居群S1共有10株个体, 除了其中3个外, 甘肃、青海及四川其余个体均位于第Ⅳ象限, 西藏样品均位于第Ⅰ象限。贵州居群集中在第Ⅱ象限。云南居群均位于第Ⅲ象限, 其中, 2个野生居群(Y1、Y2)个体位置较为集中, 而栽培个体(Y3、Y4)较为分散; 四川居群S1另3株个体亦在此象限(图 6)。

|
Figure 6 PCA based on AFLP data set from 163 individuals of G. crassicaulis. Outgroup: G. straminea and H. elliptica |
Mantel检验显示:粗茎秦艽居群间遗传距离与地理距离具有显著正相关关系, 相关系数Rxy=0.541, 且极其显著(P < 0.01)。随着地理距离增加, 物种内居群间遗传距离显著增大, 地理分布特性是影响粗茎秦艽种群遗传结构的重要因素之一。
空间自相关分析(spatial autocorrelation analysis), 将采样点地理距离划分为不同等级, 用空间自相关系数检验特定距离等级内各群体的遗传结构是否符合随机分布假设。若群体存在显著遗传差异, 自相关系数可以量化差异的程度[26]。根据粗茎秦艽各居群分布, 划分100、200、300、…、1 000 km共10个距离等级, 计算不同距离等级下的多位点相关系数r[27]。该系数值介于-1到1之间, 为0时表示不存在空间自相关[28]。结果显示:居群间距离在小于350 km时, r > 0, 呈现显著性正相关; 在350~1 000 km时, r < 0, 呈现显著性负相关。在一定范围内, 距离越小, r值越大。提示:居群间距离对于其遗传相似度有显著影响, 邻近群体具有较高的遗传相似度(图 7)。

|
Figure 7 Correlogram of the spatial autocorrelation analysis on 20 populations of G. crassicaulis |
对cpDNA psbI-trnS (GCU)、trnS (GCU)-trnG (UCC)、rpoB-trnC (GCA)及ycf3 intron1序列中SSR标记分析显示: S1a 3份种质内与S1b 7份种质内位点分别保持一致, 两者只有1个位点相同, 差异较为明显; 而S1b和居群S2有3个位点相同, 显示了更为接近的亲缘关系(表 5)。cpDNA ccsA、petA-psbJ、rpl16 intron及rpl33序列中SNP标记分析显示: S1a 3份种质内与S1b 7份种质内位点分别保持一致, 两者存在4个位点变异, 而S1b和居群S2则无差异; SSR与SNP标记均支持AFLP指纹谱分析结果(表 6)。
| Table 5 The polymorphic cpSSR loci of G. crassicaulis in Sichuan Province. *Location/sequence length (bp) |
| Table 6 The cpDNA SNPs of G. crassicaulis in Sichuan Province. *Location/sequence length (bp) |
国产特有物种粗茎秦艽G. crassicaulis, 自然分布区涉及范围广, 各居群个体生长小环境复杂多样; 植株高矮, 基生叶的较宽或较窄, 花色(蓝色、灰色及乳白色等)等亦有变化。如何把握种内个体形态特征的变异幅度, 准确鉴定不同产地实验样本, 是构建物种AFLP指纹谱关键的第一步。课题组在多年野外考察、分布区广泛取样(6省区20个居群163株个体)基础上, 结合模式标本查阅与形态学观察与比较, 进而开展物种鉴定; 同时, 以龙胆科花锚属椭圆叶花锚H. elliptica及龙胆属近缘种麻花艽G. straminea为外类群, 用于系统学分析等, 结果显示, 首先外类群被依次分出; 而粗茎秦艽物种所有20个居群共聚为另一大支, 鉴定工作达到预期目标。
道地药材是传统公认且来源于特定产地的名优正品药材, 为进一步揭示中药材道地性的科学内涵, 有许多工作要做[29]。内生菌与植物的互作关系及对宿主代谢的影响是近年来热点领域之一, 其在探讨药用植物道地性成因方面, 拓展了新的思路[30]。本工作基于筛选的12对引物组合, 构建的AFLP指纹谱, 在一定程度上反映出粗茎秦艽物种的遗传特性; 亦显示了AFLP分子标记技术在解析物种内不同个体遗传背景与产地空间地理相关性等方面的优势。因此, AFLP分子标记结合多学科新方法开展相关工作, 将深化对中药材道地性的认知。
藏药材传统生产方式多为就地采集野生资源加以利用。“解吉那保”主流品种之一粗茎秦艽分布于西藏、云南、四川、贵州、青海及甘肃等省区, 自然分布区内不同产地(居群) AFLP指纹谱各有特性, 其次生产物是否亦有一定差异, 对药材质量是否会产生影响, 值得进一步探讨。
有研究表明[31], 青藏高原特有种菊叶红景天Rhodiola chrysanthemifolia (Crassulaceae)遗传变异主要存在于居群间(77.3%); 造成居群间高度分化的主要因素可能是在恶劣的山地环境中种群的历史、地理和遗传隔离。本研究工作显示, 粗茎秦艽种内存在丰富的遗传多样性, 且遗传变异亦主要存在于居群间。遗传多样性丰富可能与物种生长环境复杂, 基因流受阻以及长期的自然选择有关。同时, Mantel检验表明粗茎秦艽居群间遗传距离与地理距离具有显著的正相关关系; 有关高山植物紫花针茅Stipa purpurea的研究[32]也发现了同样的规律。
在个体聚类与PCA等分析中注意到:来自四川康定县的居群S1, 其10个种质被分为S1a、S1b两类, 比例为3:7。S1a显示了与S1b及四川道孚县居群S2较远的亲缘关系。此状况, 在指纹谱中亦有反映(图 2, 泳道80~89)。为此, 尝试结合序列分子标记探究其原因。获得的叶绿体基因组SSR及SNP标记均支持AFLP指纹谱分析结果(表 5、6)。
聚类分析显示, 粗茎秦艽在自然分布区内种群分化为两大支, 一支由西藏、甘肃、青海及四川各居群组成, 另一支由贵州及云南各居群组成。这是否暗含物种起源、迁移路线与分化的规律性?值得应用植物群体遗传学、谱系地理学以及传粉生物学等多学科的证据来深入探究。同时, AFLP指纹技术显现了在种下居群甚至个体遗传特性间的分辨率, 相信结合居群次生产物指纹分析等, 将在药材道地性探究及系统的种质资源保护等方面有更为广阔的应用前景。
作者贡献:宗粉粉是本文的第一作者, 负责分子生物学实验及稿件撰写; 倪梁红负责样品采集工作; 嘎务负责民族植物学考察; 刘铜华参与研究工作总体设计; 赵志礼为本文的通讯作者, 负责样品分类学鉴定、研究工作总体设计及稿件修改等工作。
利益冲突:本文的研究内容无任何利益冲突。
| [1] |
Ho TN. Gentianaceae in Flora Reipublicae Popularis Sinicae:Vol 62(中国植物志:62卷)[M]. Beijing: Science Press, 1988: 59-75.
|
| [2] |
Chinese Pharmacopoeia Commission.Pharmacopoeia of the People's Republic of China: Vol I (中华人民共和国药典: 一部)[S]. Beijing: China Medical Science Press, 2015: 270-271.
|
| [3] |
Yang YC. Tibetan Medicine (藏药志)[M]. Xining: Qinghai People Press, 1991: 11-12.
|
| [4] |
Dimaer Danzeng Pengcuo. Jing Zhu Ben Cao (晶珠本草)[M]. Shanghai: Shanghai Science & Technology Press, 1986: 119.
|
| [5] |
Zhao ZL, Dorje Gaawe, Wang ZT. Identification of medicinal plants used as Tibetan Traditional Medicine Jie-Ji[J]. J Ethnopharmacol, 2010, 132: 122-126. DOI:10.1016/j.jep.2010.07.051 |
| [6] |
Ni LH, Zhao ZL. A morphometric comparison of three closely related species of Gentiana (Gentianaceae), endemic to the region of the Qinghai-Tibet Plateau[J]. Botany, 2018, 96: 209-215. DOI:10.1139/cjb-2017-0166 |
| [7] |
Ni LH, Zhao ZL, Xu HX, et al. Chloroplast genome structures in Gentiana (Gentianaceae), based on three medicinal alpine plants used in Tibetan herbal medicine[J]. Curr Genet, 2017, 63: 241-252. DOI:10.1007/s00294-016-0631-1 |
| [8] |
Ni LH, Zhao ZL, Xiong B, et al. A strategy for identifying six species of Sect. Cruciata (Gentiana) in Gansu using DNA barcode sequences[J]. Acta Pharm Sin (药学学报), 2016, 51: 821-827. |
| [9] |
Wei SJ, Zhao ZL, Ni LH, et al. Taxonomic identification of Tibetan herb Bawo Sebo and its chloroplast genome structure[J]. Acta Pharm Sin (药学学报), 2018, 53: 1009-1015. |
| [10] |
Lu JN, Zhao ZL, Ni LH, et al. The identification of Sect. Cruciata (Gentiana) species using mtDNA nad1/b-c and nad5/d-e fragments[J]. Acta Pharm Sin (药学学报), 2019, 54: 166-172. |
| [11] |
Zhou XJ, Xu HF, Shun QS. Resource Science of Chinese Medicinal Materials (中药资源学)[M]. Shanghai: Shanghai Science and Technology Literature Press, 2007: 370.
|
| [12] |
Arif IA, Bakir MA, Khan HA, et al. A brief review of molecular techniques to assess plant diversity[J]. Int J Mol Sci, 2010, 11: 2079-2096. DOI:10.3390/ijms11052079 |
| [13] |
Li YL, Fan YF, Dai GL, et al. Analysis of genetic diversity for wolfberry germplasms by AFLP technology[J]. Chin Tradit Herb Drugs (中草药), 2011, 42: 770-773. |
| [14] |
Li YB, Wang L, He ZJ, et al. AFLP analysis on genetic diversity of P. polyphylla var. yunnanensis[J]. Bot Res (植物学研究), 2018, 7: 45-53. |
| [15] |
Du CH, Pu CX, Liu XL, et al. AFLP analysis of genetic diversity of Aconitum brachypodum[J]. Chin Tradit Herb Drugs (中草药), 2018, 49: 439-443. |
| [16] |
Zhang Z, Jian JY, Wang Z. AFLP analysis on genetic diversity of Akebia trifoliata in Qinling Mountains of Shaanxi[J]. Chin Tradit Herb Drugs (中草药), 2016, 47: 3890-3895. |
| [17] |
Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf material[J]. Phyochem Bull, 1987, 19: 11-15. |
| [18] |
Vos P, Hogers R, Bleeker M, et al. AFLP:a new technique for DNA fingerprinting[J]. Nucleic Acids Res, 1995, 23: 4407-4414. DOI:10.1093/nar/23.21.4407 |
| [19] |
Lynch M, Milligan BG. Analysis of population genetic structure with RAPD markers[J]. Mol Ecol, 1994, 3: 91-99. DOI:10.1111/j.1365-294X.1994.tb00109.x |
| [20] |
Palacios C, González-Candelas F. Analysis of population genetic structure and variability using RAPD markers in the endemic and endangered Limonium duforii (Plumbaginaceae)[J]. Mol Ecol, 1997, 6: 1107-1121. DOI:10.1046/j.1365-294X.1997.00283.x |
| [21] |
Zhang DQ, Yang YP. A statistical and comparative analysis of genetic diversity detected by different molecular markers[J]. Acta Bot Yunnan (云南植物研究), 2008, 30: 159-167. |
| [22] |
Wright S. Evolution in mendelian populations[J]. Genetics, 1931, 16: 97-159. |
| [23] |
Peakall R, Smouse PE. GenAlEx 6.5:genetic analysis in Excel. Population genetic software for teaching and research-an update[J]. Bioinformatics, 2012, 28: 2537-2539. DOI:10.1093/bioinformatics/bts460 |
| [24] |
Peakall R, Smouse PE. GENALEX 6:genetic analysis in Excel. Population genetic software for teaching and research[J]. Mol Ecol Notes, 2010, 6: 288-295. |
| [25] |
Li X. Introduction to a population genetic software-GENALEX 6[J]. Chin Wind Plant Resour (中国野生植物资源), 2008, 27: 59-62. |
| [26] |
Wen B. Y Chromosome, mtDNA Polymorphisms and Genetic Structure of East Asian Population (Y染色体、mtDNA多态性与东亚人群的遗传结构)[D]. Shanghai: Fudan University, 2003.
|
| [27] |
Smouse PE, Peakall R. Spatial autocorrelation analysis of individual multiallele and multilocus genetic structure[J]. Heredity, 1999, 82: 561-573. DOI:10.1038/sj.hdy.6885180 |
| [28] |
Yang AH, Zhang JH, Tian H, et al. Microsatellite genetic diversity and fine-scale spatial genetic structure within a natural stand of Liriodendron chinense (Magnoliaceae) in Lanmushan, Duyun City, Guizhou Province[J]. Biodiv Sci (生物多样性), 2014, 22: 375-384. DOI:10.3724/SP.J.1003.2014.14013 |
| [29] |
Xiao XH, Chen SL, Huang LQ, et al. Survey of investigation on Daodi Chinese medicinal materials in China since 1980s[J]. China J Chin Mater Med (中国中药杂志), 2009, 34: 519-523. |
| [30] |
Cui JL, Guo SX, Xiao PG. Interaction between endophytes and host plant and the role of endophytes in genuineness analysis of medicinal plant[J]. Acta Pharm Sin (药学学报), 2017, 52: 214-221. |
| [31] |
Xia T, Chen SL, Chen SY, et al. ISSR analysis of genetic diversity of the Qinghai-Tibet Plateau endemic Rhodiola chrysanthemifolia (Crassulaceae)[J]. Biochem System Ecol, 2007, 35: 209-214. DOI:10.1016/j.bse.2006.09.016 |
| [32] |
Liu WS, Dong M, Song ZP, et al. Genetic diversity pattern of Stipa purpurea populations in the hinterland of Qinghai-Tibet Plateau[J]. Ann Appl Biol, 2009, 154: 57-65. DOI:10.1111/j.1744-7348.2008.00274.x |
 2020, Vol. 55
2020, Vol. 55


