2. 广州医大新药创制有限公司, 广东 广州 511436
2. GMU Medical Drug Development Co., Ltd., Guangzhou 511436, China
表观遗传修饰调控基因表达主要基于3种机制: DNA甲基化、组蛋白修饰和基于RNA的调控[1]。基因表达的改变是心力衰竭的显著生物学标志, 与DNA甲基化相关[2]。DNA甲基化是最显著的表观遗传特征之一, 由1个甲基基团与胞嘧啶5'碳位共价键结合而发生, 主要存在于哺乳动物基因组中的CpG二核苷酸序列中[3]。研究发现, DNA甲基化及去甲基化均参与心脏病的发病过程, DNA甲基化对各种因素引起的心脏功能保护均具有直接相关性[4]。鉴于DNA甲基化的表观遗传调控具有丰富的层次, 故可逆的RNA修饰通常被认为是“RNA表观遗传”形式的另一个生物调控领域[5]。在真核细胞mRNA中, N6-甲基腺苷(m6A)和5-甲基胞嘧啶(m5C)这两种RNA修饰被广泛研究, 它们出现在3'非翻译区(3' untranslated region, 3' UTR)和终止密码子位点, 提示其参与了基因的开关和/或促进介导了下游通路蛋白质的相互作用[6]。近年来, 针对心衰特异性甲基化改变的研究大多集中于基因和启动子特异性改变领域[7, 8], 然而全基因组DNA及总RNA甲基化的含量测定对于理解人类心脏疾病的分子病理学、衡量心脏疾病预防的潜在作用、以及监测人体临床试验中正在评估的药物治疗效应等方面, 仍然是一种有价值的技术手段, 是用于监测生物体内大规模表观遗传事件的良好的生物标记物。Kurdyukov等[9]详细介绍了几种不确定差异甲基化区域的全基因组甲基化分析的检测方法, 其中的高效液相色谱-紫外光谱技术目前仍被认为是测定水解DNA样品中脱氧胞苷和甲基化胞嘧啶含量的金标准, 但由于需要相对大量(3~10 μg)的待测样品, 该方法的应用受到很大限制。液相色谱-串联质谱联用作为高效液相色谱-紫外光谱法的替代方法, 快速、准确、简便、重现性高, 其所需样品量少, 且不受劣质DNA及RNA的不利影响, 已有研究利用该方法检测了孕妇外周血白细胞中m5C和5-羟甲基胞嘧啶[10]。本文采用了超高效液相色谱-串联质谱法同时检测了大鼠心肌组织及外周血淋巴细胞中DNA及RNA甲基化率的变化, 希望能够证实甲基化与心衰、外周血淋巴细胞的关系, 为临床的诊断提供理论依据。
材料与方法仪器设备 Agilent 1290 Infinity Ⅱ超高效液相色谱仪和G6465A Ultivo LC/TQ仪(美国安捷伦公司); RoVent™小动物呼吸机(美国Kent Scientific公司); Vevo 3100高分辨率小动物超声影像系统(加拿大VisualSonics公司); KZ-Ⅱ高效组织研磨仪(中国Servicebio公司); 5810R台式高速冷冻离心机(德国Eppendorf公司); SQP十万分之一电子分析天平和Arium Comfort超纯水机(德国Sartorius公司); NanoDrop One超微量分光光度计和Finnpipette® F3移液器(美国Thermo Fisher Scientific公司); HE33迷你型核酸潜水电泳和GE AI600超灵敏多功能生物分子成像仪(美国General Electric公司); MDF-U700VX超低温冰箱(日本Panasonic公司); SMZ1000体视显微镜(日本Nikon公司); Revolve FL正倒置一体显微镜(美国Echo公司); HD120-RT8电热恒温循环水浴锅(英国PRIMA公司)。
试剂 S1核酸酶和大肠杆菌C75碱性磷酸酶(日本TaKaRa公司); 磷酸二酯酶Ⅰ、HPLC级甲酸和2'-脱氧鸟苷水合物(2'-deoxyguanosine hydrate, dG) (中国阿拉丁公司); 离心柱型动物组织基因组DNA提取试剂盒(中国鼎国昌盛公司); 大鼠外周血淋巴细胞分离液(中国天津灏洋公司); 血液基因组DNA提取试剂盒(中国东胜公司); HPLC级甲醇(美国Thermo Fisher Scientific公司); 5-甲基脱氧胞苷(5-methyl-2'-deoxycytidine, 5mdC)、5-甲基胞苷(5-methylcytidine, 5mrC)、鸟苷(guanosine, rG)、N6-甲基腺苷(N6-methyladenosine, m6A)和腺苷(adenoside, rA) (中国麦克林公司)。
实验动物 SPF级SD雄性大鼠, 体重200~230 g, 购于济南朋悦实验动物繁育有限公司, 许可证号: SCXK (鲁) 2014 0007。大鼠分笼喂养, 每笼4只左右, 定期更换垫料, 保持环境清洁, 避免噪音等外界条件干扰。动物福利和实验过程均遵循广州医科大学动物伦理委员会的规定。
心肌梗死(myocardial infarction, MI)模型的制备 大鼠经10%水合氯醛(400 mg·kg-1)麻醉后, 背卧位固定于鼠板上, 将鼠板70度倾斜。将大鼠气管插管连接小动物呼吸机, 潮气量2~4 mL, 呼吸频率60~70次·min-1, 呼吸比1:1。进行气管插管, 观察呼吸机屏幕上显示的波形及大鼠胸部起伏状态, 若胸部规律性平稳起伏, 说明成功插入气管中。于左胸第3和4肋间打开约l.5 cm纵开口, 向上剪断第3肋, 钝性分离各肌层, 打开胸腔, 用扩胸器撑开胸腔, 轻轻撕开心包膜后, 用眼科弯镊轻拨左心耳, 在肺动脉圆锥与左心耳之间下约2 mm处用6/0手术线结扎冠状动脉左前降支, 结扎后可见左冠状动脉下左心室区域变白, 搏动减弱, 用4/0手术线逐层关胸, 密切观察大鼠的呼吸状况。假手术组(sham)的手术操作过程同模型组, 但只将6/0线从左冠状动脉下穿过不结扎。大鼠术后连续3天腹腔注射8万单位的青霉素预防感染, 常规饲养。分别于术后1、4及8周对大鼠进行超声心动图及相关指标检测。
超声心动图检测大鼠心脏结构和功能 大鼠经异氟烷麻醉后, 胸前脱毛备皮, 用医用胶带固定在操作台上, 涂适量耦合剂, 将仪器的探头放在大鼠左胸前, 调整探头的深度约为2.0~2.5 cm, 当获得左室长轴切面后, 将探头90度旋转, 获取短轴切面, 测量收缩末期左房前后径, 将M型取样线放于不同的位置即可获得相应的数据, 测量左室收缩末期内径(left ventricular end systolic diameter, LVIDs)和舒张末期内径(left ventricular end diastolic diameter, LVIDd)、左室射血分数(left ventricular ejection fraction, LVEF)及左室短轴缩短分数(left ventricular fraction shortening, LVFS), 测量4个心动周期取平均值。
样品处理 ①心肌组织标本采集:分别将sham组、术后1、4及8周大鼠实施安乐死, 开胸采血后取出心脏, 用无菌4 ℃预冷磷酸盐缓冲液(phosphate buffered saline, PBS)清洗心脏内积血, 滤纸吸干, 分离梗死区与非梗死区组织。实验组取大鼠心脏梗死边缘区域心肌组织, 对照组在心脏相同区域取材。液氮迅速冷冻后, 置于-80 ℃冻存。② HE (hematoxylin-eosin)染色及masson染色:将留取的心脏标本固定于多聚甲醛中, 制备蜡块, 切片机切片后进行HE染色。将以上石蜡包埋切片常规脱蜡至水溶液, 并置于masson复合染液进行染色。③心肌组织及血液样品RNA及DNA的提取:抽取大鼠心脏血于肝素钠抗凝管中并应用大鼠外周血淋巴细胞分离液分离淋巴细胞; 心肌组织样本从-80 ℃冰箱取出并应用Trizol法提取RNA; 应用动物组织基因组DNA提取试剂盒及血液基因组DNA提取试剂盒进行DNA提取。实验步骤均按照试剂盒说明书进行。RNA及DNA样本经NanoDrop One超微量分光光度计检测纯度和浓度。④ RNA及DNA的酶解:将500 ng基因组DNA和1 μg总RNA样品混入同一eppendorf (EP)管中, 加入无核酶水至10 μL, 95 ℃加热5 min, 迅速置于冰上冷却2 min, 短暂离心后加入S1核酸酶和10×S1核酸酶缓冲液各1 μL, 于37 ℃孵育2 h。在上述混合物中加入碱性磷酸酶(0.5 U) 1 μL、10×碱性磷酸酶缓冲液4 μL、磷酸二酯酶Ⅰ (0.002 U) 1 μL及超纯水22 μL, 于37 ℃继续孵育2 h。酶解后取2 μL样品用琼脂糖凝胶电泳检验酶解效果。随后加入超纯水160 μL, 于10 000×g、4 ℃离心10 min, 吸取上清液, 置于-80 ℃冻存或直接进样分析。⑤ DNA及RNA的鉴定:由于该液相色谱-串联质谱法测定结果的准确度源于DNA及RNA必须完全分解为单个核苷酸, 因此为验证心肌组织DNA及RNA的质量, 以及酶解DNA及RNA片段的效果, 对酶解前后的DNA及RNA样本进行1%的琼脂糖凝胶电泳以检验提取和酶解DNA及RNA的效果。
仪器设置 ①串联质谱条件:应用安捷伦喷射流离子聚焦电喷雾离子源(electrospray ionization with Agilent jet steam technology, AJS ESI)、正离子模式(positive ion mode, ESI+)和多反应监测模式(multiple reaction monitoring, MRM)进行定量; 毛细管电压3 000 V, 喷嘴电压500 V, 干燥气温度300 ℃, 干燥气流速8.0 L·min-1, 雾化气压力35 psi, 鞘气温度350 ℃, 鞘气流速11.0 L·min-1。②超高效液相色谱条件: EclipsePlusC18 RRHD (1.8 μm, 2.1 mm×50 mm); 流动相A为含0.1%甲酸的超纯水, 流动相B为含0.1%甲酸的甲醇; 流速为0.2 mL·min-1, 柱温35 ℃, 进样量5 μL; 梯度洗脱程序为, 0~1.35 min, 0.05 B; 1.35~2.60 min, 0.05~0.95 B; 2.6~3.5 min, 0.95 B; 3.5~3.51 min, 0.95~0.05 B; 3.51~5.5 min, 0.05 B。洗针液为1:1甲醇-水溶液, 泵头密封垫冲洗液为10%异丙醇-水溶液。
储液和工作液的配制 ①储液的配制:用电子天平分别称取5mdC、dG、5mrC、rG、m6A及rA各10 mg, 溶于10 mL超纯水中, 配制为1 mg·mL-1储液, 充分振荡溶解。②标准曲线工作液的配制:将不同浓度5mdC (2.5、5、10、25、50和100 ng·mL-1)加入到500 ng·mL-1 dG中, 将不同浓度5mrC (2、4、10、20、40和80 ng·mL-1)加入到2 000 ng·mL-1 rG中, 将不同浓度m6A (10、20、40、60、80和120 ng·mL-1)加入到2 000 ng·mL-1 rA中, 充分振荡混匀。以上溶液均保存于-20 ℃冰箱, 使用时提前室温溶解。③质控溶液的配制:将不同浓度5mdC (2.5、4、18和80 ng·mL-1)加入到500 ng·mL-1 dG中, 将不同浓度5mrC (2、3、15和60 ng·mL-1)加入到2 000 ng·mL-1 rG中, 将不同浓度m6A (10、15、50和100 ng·mL-1)加入到2 000 ng·mL-1 rA中, 分别制备4个不同浓度水平的质控溶液, 充分振荡混匀。以上溶液保存于-20 ℃冰箱, 使用时提前室温溶解。
方法学验证 称取两份标准品, 一份配制标准曲线样品, 另一份配制质控样品。利用“储液和工作液的配制”所述步骤配制的标准曲线工作液和质控溶液进行方法学验证, 按照生物样品测定方法验证指导原则[11], 考察残留效应、准确度与精密度、基质效应与回收率和短期及长期稳定性。
统计学分析 采用Agilent MassHunter Quantitative Analysis B.09.00软件绘制每一个分析批的标准曲线并检测未知样品的甲基化水平。采用GraphPad Prism 5统计软件进行统计分析, 使用One-way ANOVA比较不同组甲基化率的差异, P < 0.05认为差异具有统计学意义。
结果 1 超声心动图及组织切片染色手术1、4及8周后大鼠超声结果如图 1所示。心肌梗死组大鼠心腔扩大, 运动幅度减低, LVEF (图 1A)和LVFS (图 1B)下降, LVIDs (图 1C)和LVIDd (图 1D)增加, 与sham组相比均具有非常显著的统计学差异(P < 0.001)。同时, 与sham组相比, 模型组大鼠左心室腔呈现进行性扩张现象, 左心室前壁变薄, 几乎无波动(图 1E)。应用体视显微镜进行的HE染色心脏全景拍片结果显示, 模型组大鼠左心室壁显著变薄, 纤维化程度严重, 心室腔较sham组显著扩张(图 1F)。应用正倒置一体显微镜观察的masson染色结果显示, sham组大鼠心肌组织呈红色, 组织排列整齐, 视野中无胶原纤维出现。而模型组大鼠心肌组织梗死边缘区心肌纤维排列紊乱, 核溶解, 形成胶原纤维组织, 且残存心肌组织较少, 心肌细胞分布不均, 结构破坏严重(图 1G)。实验结果提示心梗模型构建成功。

|
Figure 1 Echocardiographic assessments of left ventricular (LV) function. A: Ejection fraction (EF); B: Fractional shortening (FS); C: Left ventricular end-systolic diameter (LVIDs); D: Left ventricular end-diastolic diameter (LVIDd). n = 5-10, |
对1 μg·mL-1标准溶液进行全扫描, 确认得到各化合物离子的质量数。应用选择离子监测模式(selected ion monitoring, SIM)优化毛细管出口电压(fragmentor), 以保证母离子(precursor ion)的传出效率。应用子离子扫描(product ion scan)和已优化好的毛细管出口电压选择定量离子, 以优化碰撞能量(collision energy, CE), 得到优化的子离子响应(表 1)。应用MRM模式定量已优化的毛细管出口电压和碰撞能量。当流动相流速为0.2 mL·min-1、梯度洗脱条件为“超高效液相色谱条件”项所述时, 各化合物保留时间不同, 可以完全分离(表 2)。
| Table 1 Mass spectrometry operating parameters. CE: Collision energy |
| Table 2 The retention time of 5mdC, 5mrC, m6A, dG, rG, and rA |
考察3批样品的准确度和精密度, 每批包括1条标准曲线, 4个不同浓度水平的质控样品, 分别为最低定量下限(lower limit of quantification, LLOQ)、低值质控(low level quantitation control, LQC)、中值质控(middle level quantitation control, MQC)和高值质控(high level quantitation control, HQC), 每个浓度平行5份。表 3为批内及批间的准确度与精密度结果, 其中Inter-assay CV/%数据体现批间精密度变异系数结果。测量值均在要求区间内(LLOQ CV% ≤ 20%, 其余浓度水平CV% ≤ 15%), 说明此方法的稳定性和重现性较好。
| Table 3 Validation of within-run and between-run accuracy and precision (n = 5). C: Concentration; Exp: Experiment; CV: Coefficient of variance; RE: Relative error |
用初始流动相稀释1 mg·mL-1的5mdC、dG、5mrC、rG、m6A及rA的储液至500 ng·mL-1, 图 2为连续进针6次500 ng·mL-1混合溶液后的1次空白样本, 从图中可以看出, 连续高浓度进样后, 残留响应值远远低于LLOQ的20%。
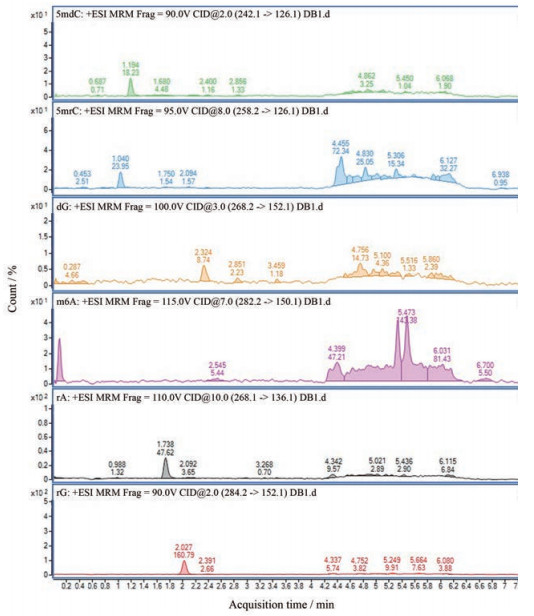
|
Figure 2 The liquid chromatographic of 5mdC (A), 5mrC (B), dG (C), m6A (D), rA (E), and rG (F) in blank sample |
将不同浓度水平的5mdC、dG、5mrC、rG、m6A及rA标准品溶液分别加到酶解缓冲液及超纯水中, 其中5mdC与dG, 5mrC与rG, m6A与rA标准品溶液各水平浓度保持一致, 按照“样品处理”项下RNA及DNA的酶解步骤离心后进样检测, 比较响应值差异, 考察基质效应。将空白基质按照“样品处理”项下RNA及DNA的酶解步骤离心后取上清, 加入不同浓度水平的5mdC、dG、5mrC、rG、m6A及rA标准品溶液, 比较相同基质条件下处理前后的响应值差异, 考察回收率。表 4为回收率及基质效应结果, 回收率结果无明显浓度依赖性, 各浓度水平的基质效应均在85%~115%内, 说明基质效应不会影响样品的实际检测分析。
|
Table 4 Validation of recovery and matrix effect (n = 5, |
短期稳定性考察3个不同浓度水平的质控溶液, 分别为LQC、MQC和HQC放置于室温2 h和自动进样器4 h稳定性。长期稳定性考察放置于4 ℃冰箱7天和-20 ℃冰箱30天稳定性。表 5为工作液短期及长期稳定性结果, 测量值均在要求区间内(LLOQ CV% ≤ 20%, 其余浓度水平CV% ≤ 15%), 说明工作液在此时间范围内保持稳定。
| Table 5 Validation of working solution stability (n = 5). LQC: Low level quantitation control; MQC: Middle level quantitation control; HQC: High level quantitation control; RT: Room temperature |
以混合标样中5mdC和dG、5mrC和rG、m6A和rA的物质的质量比值及其在质谱中的响应面积的比值构建回归曲线, 获得定量全基因组DNA及总RNA甲基化率的标准工作曲线。结果显示, 5mdC/dG、5mrC/rG及m6A/rA在0.5%~20%、0.1%~4%及0.5%~6%内具备良好的线性相关性, 可用于甲基化率的定量分析。
5 心肌梗死大鼠心肌组织及外周血淋巴细胞中甲基化水平变化定量测定心肌梗死大鼠心肌组织及外周血淋巴细胞中全基因组DNA及总RNA的甲基化水平, 并将其与sham组进行比较, 结果显示, 心梗组DNA及RNA甲基化率均有增加趋势, 其中8w组与sham组比较, 具有显著性差异(P < 0.01), 且外周血淋巴细胞与心肌组织中的甲基化率变化相一致(图 3)。
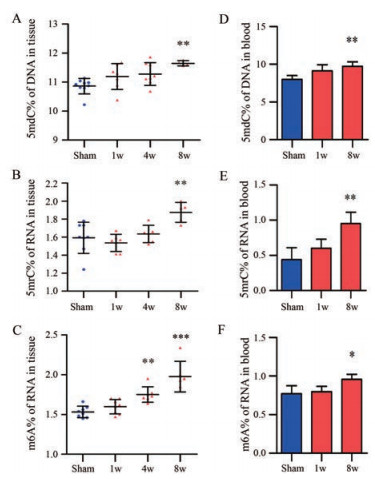
|
Figure 3 Increased 5mdC in genomic DNA, 5mrC, and m6A in total RNA in failing rat (post-myocardial infarction ischemic) hearts and blood. Quantification of 5mdC in genomic DNA in LV (A), 5mrC in total RNA in LV (B), and m6A in total RNA in LV (C). n = 4-9, |
表观遗传调控在衰竭心脏的表型反应中起着关键作用[12]。了解心衰过程中的表观遗传学变化可为早期诊断、有效的治疗、降低医疗成本和改善患者的生活质量奠定基础。已有文献证实冠心病患者的全基因组DNA甲基化显著高于对照组[13], Jiang等[14]也发现老年冠心病患者外周血单核细胞DNA甲基化和羟甲基化水平明显升高, 并与冠状动脉粥样硬化程度呈正相关。动态mRNA的转录后修饰对其稳定性、核输出、细胞分裂、翻译为蛋白质、降解和干细胞多能性至关重要[15]。最近发表了两项研究, 通过调节肥胖基因(fat mass and obesity associated, FTO)或m6A甲基转移酶3 (N6-adenosine-methyltransferase subunit, METTL3)研究m6A在应激后心脏重塑和功能中的作用[16, 17]。在衰竭的哺乳动物心脏和低氧的心肌细胞中, m6A是最普遍和功能相关的RNA修饰之一, m6A的失调是哺乳动物心力衰竭的重要标志[16]。RNA甲基化m6A水平的增加会导致代偿性心肌肥大, 而m6A减少则会导致偏心性心肌细胞重塑和功能障碍, METTL3介导的m6A的mRNA甲基化响应机制同样也用于维持正常的心脏功能[17]。
基因特异性分析对于整合DNA甲基化模式与基因表达、染色质修饰和基因启动子转录因子组装的信息至关重要, 而全基因组DNA及总RNA甲基化检测则体现了甲基化水平的总体情况, 对于理解基因特异性甲基化模式和基因组稳定性之间的关系同样至关重要。组织是研究表观遗传学最直观的模型, 但由于临床上较难获取, 所以外周血逐渐成为一种替代检测途径。从冠状动脉旁路手术患者采集的外周血白细胞(peripheral blood leucocyte, PBL)和心房活检的一项研究也证实了组织间DNA甲基化的高相关性(R2 = 0.83)[18]。本研究采用了UPLC-ESI-MS/MS法同时对大鼠心肌组织及外周血淋巴细胞中全基因组DNA及总RNA的甲基化水平进行了检测, 初步证明了心肌梗死大鼠心脏组织及外周血中的甲基化发生改变, 且甲基化率与心肌梗死的严重程度呈正相关, 提示心力衰竭的发生和发展与DNA及RNA的甲基化存在密切联系。而组织间的变化呈现一致性, 提示外周血淋巴细胞可能成为心脏组织的良好替代物。
虽然该研究从整体甲基化水平上证实了心衰过程中伴随着甲基化程度的增加, 但性别和种属的潜在影响, 以及心衰的发病机制中疾病与甲基化的因果关系都有待研究。本研究从心肌组织及外周血两个方面揭示了全基因组DNA和总RNA甲基化水平与心衰的发生和发展具有紧密联系, 初步表明了DNA和RNA甲基化可能在心力衰竭的发生发展中具有一定的潜在作用, 为心脏疾病的进一步研究提供了依据。
作者贡献:林忠晓负责部分液质方法学验证实验和文章作图; 张燕芬参与了样品制备内容的撰写和文献整理; 胡渊参与了动物饲养及动物实验样品的制备; 常继硕是文章框架的构思者及第一作者; 余细勇指导论文写作并对论文进行了修改和检查。
利益冲突:所有作者均不存在利益冲突。
| [1] |
Sabia C, Picascia A, Grimaldi V, et al. The epigenetic promise to improve prognosis of heart failure and heart transplantation[J]. Transplant Rev (Orlando), 2017, 31: 249-256. DOI:10.1016/j.trre.2017.08.004 |
| [2] |
Movassagh M, Choy MK, Knowles DA, et al. Distinct epigenomic features in end-stage failing human hearts[J]. Circulation, 2011, 124: 2411-2422. DOI:10.1161/CIRCULATIONAHA.111.040071 |
| [3] |
Feinberg AP. Phenotypic plasticity and the epigenetics of human disease[J]. Nature, 2007, 447: 433-440. DOI:10.1038/nature05919 |
| [4] |
Li G, Xing XY, Zhang MS, et al. Research progress in epigenetic regulation of myocardial ischemia/reperfusion injury by traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2016, 51: 1047-1053. |
| [5] |
He C. Grand challenge commentary:RNA epigenetics?[J]. Nat Chem Biol, 2010, 6: 863-865. DOI:10.1038/nchembio.482 |
| [6] |
Chandola U, Das R, Panda B. Role of the N6-methyladenosine RNA mark in gene regulation and its implications on development and disease[J]. Brief Funct Genomics, 2015, 14: 169-179. DOI:10.1093/bfgp/elu039 |
| [7] |
Pepin ME, Ha CM, Crossman DK, et al. Genome-wide DNA methylation encodes cardiac transcriptional reprogramming in human ischemic heart failure[J]. Lab Invest, 2019, 99: 371-386. DOI:10.1038/s41374-018-0104-x |
| [8] |
Li B, Feng ZH, Sun H, et al. The blood genome-wide DNA methylation analysis reveals novel epigenetic changes in human heart failure[J]. Eur Rev Med Pharmacol Sci, 2017, 21: 1828-1836. |
| [9] |
Kurdyukov S, Bullock M. DNA methylation analysis:choosing the right method[J]. Biology (Basel), 2016, 5: 3. |
| [10] |
Pauwels S, Duca RC, Devlieger R, et al. Maternal methyl-group donor intake and global DNA (hydroxy) methylation before and during pregnancy[J]. Nutrients, 2016, 8: 474. DOI:10.3390/nu8080474 |
| [11] |
Booth B, Arnold ME, DeSilva B, et al. Workshop report:crystal city V——quantitative bioanalytical method validation and implementation:the 2013 revised FDA guidance[J]. AAPS J, 2015, 17: 277-288. DOI:10.1208/s12248-014-9696-2 |
| [12] |
Cao DJ. Epigenetic regulation and heart failure[J]. Expert Rev Cardiovasc Ther, 2014, 12: 1087-1098. DOI:10.1586/14779072.2014.942285 |
| [13] |
Sharma P, Kumar J, Garg G, et al. Detection of altered global DNA methylation in coronary artery disease patients[J]. DNA Cell Biol, 2008, 27: 357-365. DOI:10.1089/dna.2007.0694 |
| [14] |
Jiang D, Sun M, You L, et al. DNA methylation and hydroxymethylation are associated with the degree of coronary atherosclerosis in elderly patients with coronary heart disease[J]. Life Sci, 2019, 224: 241-248. DOI:10.1016/j.lfs.2019.03.021 |
| [15] |
Nachtergaele S, He C. Chemical modifications in the life of an mRNA transcript[J]. Annu Rev Genet, 2018, 52: 349-372. DOI:10.1146/annurev-genet-120417-031522 |
| [16] |
Mathiyalagan P, Adamiak M, Mayourian J, et al. FTO-dependent N(6)-methyladenosine regulates cardiac function during remodeling and repair[J]. Circulation, 2019, 139: 518-532. DOI:10.1161/CIRCULATIONAHA.118.033794 |
| [17] |
Dorn LE, Lasman L, Chen J, et al. The N6-methyladenosine mRNA methylase METTL3 controls cardiac homeostasis and hypertrophy[J]. Circulation, 2019, 139: 533-545. DOI:10.1161/CIRCULATIONAHA.118.036146 |
| [18] |
Ma B, Wilker EH, Willis-Owen SA, et al. Predicting DNA methylation level across human tissues[J]. Nucleic Acids Res, 2014, 42: 3515-3528. DOI:10.1093/nar/gkt1380 |
 2020, Vol. 55
2020, Vol. 55


