作者贡献: 所有作者都参与了文献调研、手稿设计与撰写。杨万镇、涂杰开展文献调研和手稿撰写, 刘娜、盛春泉获取经费资助并指导整个研究。所有作者都认可手稿的最终版本。
利益冲突:本文的发表不存在任何利益冲突。
2. 中国人民解放军海军军医大学, 上海 200433
2. The Second Military Medical University, Shanghai 200433, China
真核生物内物质的分解代谢主要有两条途径:泛素蛋白酶降解途径和自噬降解途径。细胞内短寿命蛋白主要依赖泛素化降解途径, 长寿命蛋白的降解则主要依赖于自噬降解途径, 更重要的是, 生物大分子和完整的细胞器的降解只能通过自噬途径来完成[1, 2]。
自噬是一种在进化中高度保守的过程, 是真核细胞内广泛存在且特有的主要降解系统[3]。当细胞处于营养不足、生长因子缺乏或缺氧等各种应激状态时, 会将不需要的蛋白质或受损的细胞器运送到溶酶体(动物)或液泡(真菌和植物)中进行降解和再循环[4]。自噬不仅是细胞应对外界刺激的一种自我保护机制, 而且在维持细胞内的正常生命活动、物质循环利用和内环境的稳定等方面发挥着重要的作用[5, 6]。自噬可分为选择性自噬及非选择性自噬。选择性自噬主要用于保护细胞结构, 清除受损的细胞器和胞质物质[7], 如线粒体自噬、过氧化物酶体自噬、内质网自噬及核糖体自噬等[8]; 非选择性自噬则在细胞饥饿时发挥重要作用, 可为细胞提供氨基酸等营养物质, 如巨自噬和微自噬。
研究表明, 在哺乳动物细胞中, 自噬与胚胎发育、细胞分化、免疫和炎症有关[9, 10]; 在植物细胞中, 自噬对于幼苗发育、植物生长、衰老、抗逆性、新陈代谢和繁殖至关重要[11, 12]; 在真菌细胞中, 自噬影响其生长、形态、发育、脂质周转和繁殖[13-15]。目前, 真菌自噬在模式生物酵母菌中已经得到较为深入的研究, 常用的模式菌种主要为酿酒酵母(Saccharomyces cerevisiae)、巴斯德毕赤酵母(Komagataella pastoris)和法夫酵母(K.phaffii)[16]。
近几十年来, 大量研究表明自噬在感染、癌症、心脏病和神经退化等各种疾病的致病过程中起着重要作用[3]。虽然自噬在真菌感染和抗真菌药物研发等方面的相关研究尚处于起步阶段, 但已有研究表明自噬与真菌细胞的分化、发育和毒力等密切相关。因此, 深入了解真菌细胞的自噬过程, 并基于此开展相关药物研发工作, 将为真菌感染的治疗提供一种新的思路。本文综述了自噬和真菌细胞自噬的最新研究进展, 重点讨论自噬与真菌致病性之间的联系。
2 自噬的发生途径酵母的选择性自噬是由细胞质到液泡靶向(cytoplasm-to-vacuole targeting, Cvt)途径介导的, 胞质内的物质被运送到液泡或溶酶体后进行降解, 酵母的Cvt途径可作为一种模型便于开展其他生物体自噬的相关研究[5, 17]。根据包裹底物分子及运送至降解细胞器方式的不同, 可以将自噬的发生途径分为3类:巨自噬、微自噬和分子伴侣介导的自噬[3, 18, 19]。①巨自噬(macroautophagy):在巨自噬过程中, 部分受损的细胞质和细胞器被具有双层膜的分隔膜(也称为吞噬泡)包裹形成中间细胞器—自噬体, 这是巨自噬发生途径的典型特征。自噬体可与溶酶体(一种降解细胞器)融合成为“自噬溶酶体”, 被吞噬的物质在自噬溶酶体中降解, 降解产物被释放到细胞质中重新用于物质和能量的合成, 以满足机体营养需要, 维持细胞稳态; ②微自噬(microautophagy):在微自噬过程中, 溶酶体自身通过内陷溶酶体膜来吞噬部分受损的细胞质和细胞器, 被吞噬的物质直接在溶酶体中降解为小分子, 这一过程在高等真核生物中的功能尚不清楚, 而在真菌中参与了细胞器的选择性降解; ③分子伴侣介导的自噬(chaperone-mediated autophagy, CMA):此类自噬过程不涉及膜重组, 底物蛋白在分子伴侣介导下, 可直接跨溶酶体膜进行转运。伴侣蛋白如热休克蛋白Hsc70 (heat shock cognate70)和辅伴侣蛋白可特异性地识别含有五肽Lys-Phe-Glu-Arg-Gln (KFERQ)样基序的可溶性胞质蛋白底物, 使其与溶酶体上的受体跨膜蛋白Lamp-2A (Lamp-2的一个异构体)相结合, 通过多聚体蛋白复合物被运送进入溶酶体内进行降解。与巨自噬和微自噬相比, 分子伴侣介导的自噬降解途径在清除蛋白时具有选择性, 其只能介导可溶性蛋白的降解。在自噬的3种发生途径中, 巨自噬是一个非选择性的过程, 是主要的自噬类型, 相关的研究也最为广泛。因此, 下文简称巨自噬为“自噬”, 其发生过程如图 1所示。
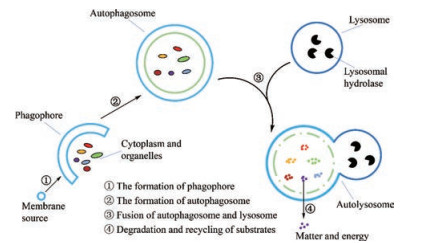
|
Figure 1 The process of autophagy |
自噬在真菌细胞的生长、发育和致病过程中起着重要的作用。在过去十多年中, 细胞生物学家以酿酒酵母(Saccharomyces cerevisiae)为模式生物展开自噬相关基因(autophagy-related genes, ATG)的筛选和研究, 用Atg表示其对应的自噬相关蛋白。ATG的发现为探索自噬体形成提供了关键信息, 也给自噬的研究带来了极大的便利。
自噬是一种动态的、高度诱导的降解系统, 能对环境和生理变化做出反应, 涉及各种ATG[6], 研究其保守和进化对于了解自噬的形成、进化和调控具有重要意义。自1992年第一个自噬相关基因ATG1被发现以来, 已在酿酒酵母中鉴定出42个驱动自噬过程的自噬相关基因, 目前已经证实其中18个核心基因对于自噬关键步骤是必不可少的, 其他24个ATG基因参与诱导特定的自噬途径或选择性自噬[6, 20]。Wang等[21]系统地鉴定了331个物种中已知的41个ATG基因, 并分析了它们在真菌细胞基因中的分布。经基因组学分析, 可将ATG基因分为3组:第一组包括20个高度保守的ATG基因, 分别是ATG1~9、ATG11~13、ATG15、ATG18、ATG20~22、ATG24和ATG26~27, 它们的同源基因出现在超过80%的已测序的真菌物种中。虽然大多数(20个中的14个)是酵母核心自噬基因, 但其中6个是选择性自噬的特异性基因; 第二组包括7个ATG基因, 分别是ATG10、ATG16、ATG17、ATG28、ATG29、ATG33和ATG37, 这些基因仅在子囊菌门中存在, 在担子菌门或基础真菌组中不存在; 第三组包括14个ATG基因, 分别是ATG14、ATG19、ATG23、ATG25、ATG30~32、ATG34~36和ATG38~41, 它们只存在于酵母菌物种中, 是酿酒酵母中的核心自噬基因。Parzych等[20]在酿酒酵母中鉴定出YBR139W并将其命名为ATG42。Atg42 (Ybr139w)是一种液泡丝氨酸羧肽酶, 在液泡蛋白水解过程和自噬末端步骤中起重要作用。在酵母和病原真菌中, 缺乏Atg42 (Ybr139w)的细胞在蛋白质合成、液泡酶原激活和自噬体形成等方面存在缺陷。
虽然自噬过程通常被认为是保守的, 事实上许多真菌物种缺乏非保守组分, 却仍然具有自噬过程。并且, 参与自噬体形成的重要功能基因包含保守和非保守的ATG组分[22]。因此, 自噬相关基因在整个基因范围内的保守和进化有待进一步探究。研究表明, 非保守的ATG基因在特定物种中具有可替代性, 例如人类病原体新生隐球菌中不存在ATG17, 需要在感染过程中进行自噬, 而哺乳动物细胞缺乏ATG17同源基因, 但有一个功能对等的基因[23, 24]。
4 真菌自噬调控机制自噬调控因子包括:自噬诱导剂雷帕霉素(rapamycin)的靶标雷帕霉素靶蛋白(target of rapamycin, TOR), 是自噬的关键调控因子之一, 能在饥饿、缺氧、伤害或活性氧(reactive oxygen species, ROS)积累等条件下诱导自噬; 5'-腺苷酸活化蛋白激酶(adenosine 5'-monophosphate activated protein kinase, AMPK)能监测机体能量, 可对缺氧和低能量等应激状态做出反应; 真核翻译起始因子2α (eukaryotic initiation factor 2α, eIF2α)可响应营养饥饿、双链RNA和内质网(endoplasmic reticulum, ER)应激; BH3-only蛋白含有Bcl-2同源-3结构域并干扰Bcl-2/Bcl-XL对Beclin 1/PI3K复合物的抑制; 其他自噬调控因子还包括肿瘤抑制蛋白p53 (tumor suppressor protein 53, p53)、死亡相关蛋白激酶(death-associated protein kinase, DAPK)、肌醇需求激酶1 (inositol requiring enzyme 1, IRE-1)、应激激活激酶c-Jun-N端激酶、三磷酸肌醇受体(inositol 1, 4, 5-triphate receptor, IP3R)、鸟苷三磷酸酶(guanosine triphosphatase, GTPases)、细胞外调节蛋白激酶1和2 (extracellular regulated protein kinases 1/2, Erk1/2)、神经酰胺和钙等[25-27]。
自噬过程所必需的蛋白质可分为以下几个核心功能基团:响应上游信号以启动自噬体形成的丝氨酸/苏氨酸蛋白激酶复合物: Atg1、Atg13和Atg17;介导囊泡成核的Ⅲ类磷脂酰肌醇3激酶(phosphatidylinositol 3-kinase, PI3K)复合物: Atg6、Atg14、液泡分拣蛋白(vacuolar protein sorting, Vps) 15和Vps34;介导自噬体扩增和成熟的两个泛素样蛋白结合系统: Atg8泛素样蛋白结合系统(Atg3、Atg4、Atg7和Atg8)和Atg12泛素样蛋白结合系统(Atg5、Atg7、Atg10、Atg12和Atg16);通过3-磷酸磷脂酰肌醇依赖的方式与Atg2-Atg18复合体相互作用来调节自噬体形成的整合膜蛋白Atg9;以及介导Atg蛋白从成熟自噬体分解的循环系统: Atg2、Atg9、Atg18和Atg41[28-30]。
目前, 大量真菌细胞发生自噬所需的Atg蛋白已被发现, 但是对真菌自噬调控的分子机制的研究还处于初级阶段。真菌自噬信号转导途径受TOR调控, TOR是丝氨酸/苏氨酸蛋白激酶, 属于PI3K蛋白激酶家族, 是PI3K/蛋白激酶B信号通路下游效应蛋白。在多种生物中, TOR是自噬的负调节因子, 能够用于感知营养, 调节蛋白质的转录、翻译及降解[31, 32]。真菌在营养饥饿条件下, 通过雷帕霉素处理后, TOR激酶的活性受到抑制, 最终诱导真菌自噬。ATG1是第一个被发现的ATG基因, 编码丝氨酸/苏氨酸激酶[33], 由Atg1、Atg13和Atg17蛋白结合所形成的Atg1-Atg13-Atg17复合物是诱导自噬所必需的。真菌在正常的生长条件下, Atg1、Atg13和Atg17之间的这种联系被高度磷酸化所抑制, 而当真菌受外界环境刺激(如饥饿、缺氧、伤害或ROS积累)或经雷帕霉素处理后, 自噬上游调控蛋白TOR的活性降低, 导致Atg13迅速去磷酸化, 随后与Atg17结合并激活Atg1, 形成Atg1-Atg13-Atg17复合物, 并定位于自噬起始位点(又称自噬前体结构, pre-autophagosomal, PAS), 从而诱导自噬发生(图 2)。
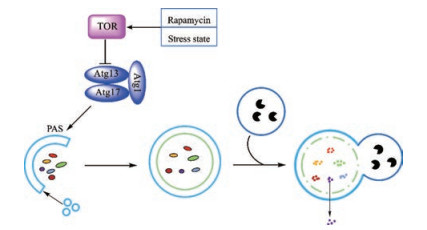
|
Figure 2 A simplified model of autophagy regulation. TOR: Target of rapamycin; Atg: Autophagy-related protein; PAS: Pre-autophagosomal |
自噬主要是一种分解代谢过程, 用于应对营养缺乏和各种应激条件。自噬作为病原真菌致病机制的一个重要组成部分, 可在营养缺乏时被诱导[5]。事实上, 如果长时间处于营养饥饿状态, 将会导致真菌的一种程序性细胞死亡, 这种死亡依赖于特定的自噬蛋白和蛋白酶的表达[34]。
新生隐球菌是免疫受损宿主的主要真菌病原体, 人体环境可对新生隐球菌施加各种应激性压力。因此, 新生隐球菌对这些压力的自噬反应往往决定其在宿主中的生存。新生隐球菌感染通常是由肺部吸入酵母态细胞或孢子, 引起肺部感染, 然后通过血管传播, 导致隐球菌脑膜炎[35]。最近的研究表明, ATG基因可通过调节自噬功能对新生隐球菌的毒力产生一定影响, 例如, Hu等[23]发现在37 ℃营养条件下, Vps34虽然对于新生隐球菌荚膜、黑素和尿素酶等毒力因子的表达不是必需的, 但是对于新生隐球菌的生长是必不可少的。在小鼠新生隐球菌感染模型中, Vps34缺失会引起Atg8标记的囊泡的形成缺陷, 最终抑制自噬, 导致新生隐球菌的毒力显著降低; 为了从分子角度分析自噬在新生隐球菌感染期间对VPS34表型的影响, 构建了一株通过RNA干扰(RNA interference, RNAi)抑制Atg8表达的新生隐球菌菌株, 发现Atg8的RNAi可使自噬能力降低, 导致真菌对饥饿应激的敏感性增加、毒力减弱[23]; 而ATG7的缺失会导致感染小鼠的肺部细胞体积增加、存活率下降[36]。最近发表的一项研究发现, 在氮饥饿条件下, 新生隐球菌的ATG1、ATG7、ATG8和ATG9基因中的任何一个基因的缺失都会破坏其氨基酸动态平衡。虽然这4个基因的突变体(atgΔ)在体外没有对新生隐球菌的毒力因子产生明显的影响, 但它们在小鼠新生隐球菌感染模型中对新生隐球菌的毒力都有贡献[37]。
Zhao等[38]构建了22株ATG缺陷菌株, 研究其在新生隐球菌毒力、致病机制、自噬通量、应激反应和耐饥性等方面的作用。结果表明, 新生隐球菌的自噬通量取决于Atg11、Atg1-Atg13复合物、Atg5-Atg12-Atg16复合物和Atg2-Atg18复合物, 在37 ℃时新生隐球菌的生长和漆酶产量会受到Atg6和Atg14-03 (Atg14的同源蛋白, CNAG_03608)影响。另外, atg2∆ (ATG2基因缺失突变体)和atg6∆ (ATG6基因缺失突变体)菌株会对过氧化氢引起的氧化胁迫敏感。并且, 约有一半的atg∆菌株对1.5 mol·L-1 NaCl表现出较高的敏感性, 而在梅隆氏菌(Galleria mellonella)模型中的毒力明显低于野生型。上述研究证明, 新生隐球菌的自噬和其毒力之间关系密切, 自噬相关基因的缺失会增加真菌对应激反应的敏感性, 通过干扰应激反应通路、抑制真菌细胞的自噬而减弱其毒力。自噬在真菌病原体的感染过程中是一个与毒力相关的生存机制, 对真菌病原体自噬进行分子层面的解析可能为抗真菌药物的研究找到新的途径。
Atg10是Atg12-Atg5结合所需的一种重要而特异的泛素结合酶(E2)[39], 它在酿酒酵母自噬体的形成过程中是不可或缺的, 目前还未在新生隐球菌中检测到酿酒酵母Atg10的同源物。虽然已有报道在新生隐球菌发现了Atg10类似物, 但它与已知的Atg10同源物的相似性极低[38]。Atg17、19、21、23、29和31的直系同源基因在新生隐球菌中也未发现, 推测可能是因为新生隐球菌的相关蛋白相似性很低, 或者没有这些蛋白。与酿酒酵母相比, 这些ATG基因对新生隐球菌的碳氮饥饿耐受性有不同的贡献。值得注意的是, 在新生隐球菌中发现了哺乳动物Atg101 (CNAG_07646)的同源蛋白, 这是酿酒酵母和高等真核生物之间的主要区别之一。酿酒酵母中的Atg29和Atg31在哺乳动物中已被Atg101取代[40, 41], 这表明新生隐球菌更接近哺乳动物, 而不是酿酒酵母。新生隐球菌作为一种人类病原体, 发现其与哺乳动物Atg101的同源性具有重要的意义。
6 自噬的检测策略目前已有多个通过调节自噬达到治疗目的的候选药物进入临床试验。自噬作为抗真菌药物研发的潜在靶点, 其重要性日益凸显, 研究人员正在努力筛选作用于自噬过程的新型抗真菌药物。为了确定这些药物是否确实影响了自噬, 以及自噬过程中的哪些步骤受到了影响, 需要选择适当的方法对自噬进行检测, 下面将介绍几种主要的自噬检测方法。
6.1 透射电子显微镜早在20世纪50年代, 研究人员通过透射电子显微镜(transmission electron microscopy, TEM)观察到了溶酶体引起的细胞质区域的局灶性降解[42], 这是首次检测到自噬, 这一现象仍然是自噬过程发生的标志。后来的分析表明, 局灶性降解现象始于一种特殊的双膜结构(现称为吞噬体)对部分细胞质的隔离, 该结构成熟后形成自噬体, 其边界仍然是双膜, 随后自噬体与溶酶体融合形成自噬溶酶体, 并对被吞噬的物质进行降解。
TEM在自噬研究中十分重要, 此类方法有以下几大特点:首先, TEM是目前唯一一种可在纳米分辨率下揭示自噬结构形态的工具, 能够准确识别并清晰地显示这些结构在其自然状态下的位置; 其次, 如果遵循适当的采样规则, 可进行定量研究; 从自噬体到自噬溶酶体的形成是一个动态、连续的过程, TEM可以用于监测自噬过程中的形态变化; 此外, 该方法可以用来分析自噬诱导或缺陷菌株中的空泡化和自噬体的存在, 并且还可以用来监测选择性自噬和非选择性自噬。但这种方法也存在一些弊端, 例如操作极其繁琐, 很难用来评估不同类型细胞中的自噬活性。
6.2 自噬体标记蛋白Atg8/LC3的检测和定量分析微管蛋白1轻链3 (Atg8/LC3)是一种泛素样蛋白, 其作为自噬体膜上的标记蛋白, 是检测自噬最常用的自噬相关蛋白, 在真菌细胞中常称之为Atg8。在酵母和其他几种生物中, Atg8通常与磷脂酰乙醇胺(phosphatidyl ethanolamine, PE)结合形成Atg8-PE, 特定情况下, 也可以与磷脂酰丝氨酸结合[43]。在哺乳动物细胞中, Atg8的同源物分为两个蛋白亚家族:微管蛋白1轻链3 (LC3)和GABAA受体结合蛋白(GABARAP)[44]。这两个蛋白亚家族均有两种存在形式:非脂化和脂化形式, 分别称为LC3-Ⅰ和LC3-Ⅱ及GABARAP和GABARAP-PE。LC3蛋白参与了吞噬泡的形成, GABARAP亚家族参与了自噬体的形成[45]。LC3蛋白合成后在其C端即被具有蛋白内切酶活性的Atg4剪切生成LC3-Ⅰ, LC3-Ⅰ散在分布于细胞浆内。在自噬体形成过程中, LC3-Ⅰ和PE偶联形成自噬体的结构蛋白LC3-Ⅱ, 并定位于自噬体内膜和外膜[46]。Atg蛋白定位于自噬膜上后, 通常仅在自噬过程中的某一阶段发挥作用, 而LC3-Ⅱ则能始终稳定地保留在自噬体膜上, 直到与液泡/溶酶体融合。因此LC3-Ⅱ可被用来作为自噬体的标志物, 且LC3-Ⅱ的水平在某种程度上反映了自噬体的数量, 通过蛋白质免疫印迹法(Western blot)可以对两种类型不同大小的蛋白进行检测。
虽然Atg8/LC3是自噬检测手段中使用最为广泛的一种标记蛋白, 但要注意的一点是, 哺乳动物的LC3存在不同亚型, 必须考虑其亲和力和特异性。除了自噬体外, 在一些非自噬体膜上也发现了LC3-Ⅱ, 故LC3-Ⅱ水平并不能量化实际的自噬活性。研究者要意识到Atg8/LC3的稳态水平和自噬通量之间的区别, 目前还没有任何证据表明LC3-Ⅱ (或自噬体)的增加能代表自噬通量的增加。因此, 在进行自噬通量分析时, 应当结合其他方法来分析自噬的变化。
6.3 应用苯甲基磺酰氟检测自噬苯甲基磺酰氟(phenylmethylsulfonyl fluoride, PMSF)是一种液泡蛋白酶抑制剂, 可以防止液泡内自噬体的降解, 可用于观察液泡内自噬体的存在[47]。液泡中自噬体的积累可以被认为是自噬的一个标志, 但液泡内自噬体降解发生得相对较快, 因此必须使用PMSF来观察自噬体。在饥饿条件下, 用液泡蛋白酶抑制剂PMSF处理酿酒酵母(BJ926、BJ2407和BJ3505等细胞)野生型菌株和突变型菌株(缺乏液泡蛋白酶A、B及羧肽酶Y)的培养液, 所有野生型菌株均观察到液泡化现象, 但突变体菌株在其液泡内没有表现出自噬体的积累[48]。
6.4 利用荧光探针检测自噬单丹磺酰尸胺(monodansylcadaverine, MDC)是一种嗜酸性自体荧光染料, 在荧光或共聚焦激光扫描显微镜下检测的激发/发射波长为355/512 nm, 能对酸性的溶酶体进行特异性标记染色, 常被用作指示剂来检测哺乳动物细胞和真菌细胞中自噬晚期阶段[49, 50]。在真菌自噬检测实验中, 自噬诱导突变体在PMSF处理后的空泡中有MDC的积累, 而自噬缺陷突变体则没有MDC的积累[51]。
自噬过程在细胞内是随时间不断变化的, 仅凭观察到的自噬体出现无法说明自噬活性的改变, 要想准确反映这种变化必须实现体内自噬活性的实时监测。Liu等[52]报道了一种新型近红外荧光探针分子HQO, 它是一种pH敏感探针, 在不同pH条件下具有不同的荧光性质: HQO碱性形式(酮结构)在可见光区域表现出吸收(λmax = 475和515 nm), 而酸性形式(烯醇结构, HQOH+)在近红外区域表现出吸收(λmax = 710 nm)。HQO在生理pH条件下, 会选择性地聚集在线粒体中, 激发/发射波长为530/650 nm。当受损的线粒体与溶酶体融合形成自噬溶酶体时, 自噬溶酶体作为一种酸性细胞器, 随着pH的降低, 可使HQO向质子化的HQOH+形式转化, 导致其波长红移到710/750 nm。由于HQO和HQOH+在低极性环境(如胶束中)表现出不同的吸收和发射, 它们可以同时探测活细胞中的线粒体和自噬溶酶体, 因此可以用于监测活细胞中的线粒体自噬水平。
6.5 通过流式和多光谱成像细胞术进行中高通量自噬分析将荧光显微镜与新型自噬探针相结合虽然可以实现对自噬的单细胞分析, 但实现自动化的中、高通量分析仍具有一定的挑战性。目前, 研究人员已经建立了多种新方法, 例如快速监测自噬的流式商业试剂盒的出现及多光谱成像技术的优化, 使得利用流式细胞技术来确定自噬通量成为可能, 并且在具有异质LC3的细胞或微生物感染等非正常生理条件的情况下, 也可以准确地定量分析自噬通量[53, 54]。EGFP-LC3是自噬降解的底物, EGFP-LC3的总荧光强度可以用来指示活细胞中的自噬水平。当诱导自噬时, 细胞总荧光的变化情况可以精确地量化[55]。
使用流式和多光谱成像细胞技术进行中、高通量的自噬分析, 不仅可以分析更多数量细胞的自噬通量, 并且可以进行表面标记的检测, 提供更稳定的数据。然而, 该方法也存在一定的缺点, 流式细胞技术仅测量GFP-LC3总水平的变化, 而GFP-LC3总水平会伴随转录、翻译或pH值的改变而发生变化。因此, 在没有足够的细胞通透性的情况下, 这种方法不能准确地评估自噬体的定位或脂化LC3-Ⅱ的产生。
6.6 通过测量真菌菌丝强度检测自噬菌丝是真菌细胞的一种重要毒力因子, 菌丝强度对真菌细胞的生物学行为具有重要意义。据Kim等[56]报道, 雷帕霉素诱导的自噬在即使有充足外源性碳源的情况下, 也会引起细胞壁成分的降解, 由于细胞壁强度的降低, 真菌菌丝会发生一系列变化, 包括膨胀性和均匀性的丧失、菌丝抗张强度的降低以及菌丝形态明显的破坏[57]。因此, 雷帕霉素诱导的自噬效应能够降低菌丝强度。
Quintanilla等[58]开发了一种快速、高效的新方法来表征菌丝强度的大小, 该研究通过对比构巢曲霉(Aspergillus nidulans, 野生型)和构巢曲霉突变体(Anatg8∆, 缺失自噬核心基因ATG8)在经雷帕霉素诱导和空白对照两种条件下的菌丝强度, 结果发现在对照条件下, 突变菌株的菌丝强度比野生型的菌丝强度弱; 在雷帕霉素诱导的条件下, 突变菌株的菌丝强度与亲本菌株的菌丝强度相比也明显变弱。结果表明, 真菌菌丝强度与自噬之间存在一定的联系, 构巢曲霉突变体中自噬核心基因ATG8缺失导致自噬受到抑制, 菌丝强度相应减弱。因此, 测量真菌菌丝强度的大小可作为检测真菌细胞自噬水平的一种有效手段。
7 真菌自噬药靶的研究目前, 国内外关于自噬药靶的研究多集中在肿瘤细胞自噬现象。大量的临床前研究证明, 抑制自噬可以改善癌症的治疗效果, 在特定癌基因驱动的动物肿瘤模型中, 肿瘤在使用自噬抑制剂的情况下会逐渐消退。氯喹(chloroquine, CQ)和羟基氯喹(hydroxychloroquine, HCQ)是美国食品和药物管理局(FDA)最初批准的4-氨基喹啉类抗疟药物, 主要用于治疗疟疾、类风湿性关节炎和系统性红斑狼疮等疾病。此外, 氯喹和羟基氯喹可以使溶酶体脱酸, 阻止自噬体与溶酶体的融合, 从而抑制自噬[59], 因此它们还作为自噬抑制剂被广泛用于癌症临床试验研究。Vps34-IN-1 (抑制Vps34)、Mdivi-1 (抑制线粒体自噬)和NSC185058 (抑制ATG4B)等自噬抑制剂正处于临床前研究, 基于自噬相关靶点进行抗肿瘤药物研发是一种很有前途的新策略。
目前, 国内外对于病原真菌细胞自噬的研究处于起步阶段, 并且尚未有真菌中自噬药靶的相关研究报道。虽然, 酿酒酵母中的自噬相关蛋白Atg在病原真菌中大多存在同源蛋白, 但是, 目前还未发现针对Atg蛋白的特异性抑制剂。在研究人员对真菌自噬调控机制和自噬相关基因、自噬相关蛋白具有一定了解的基础上, 如果设计一种能够选择性作用于真菌自噬某个环节的荧光探针, 既能调控真菌细胞自噬又能监测活细胞内的自噬水平, 或将发现一种以自噬为靶点的新型抗真菌荧光探针。
Benhamou等[60]报道了两种基于唑类抗真菌药物的荧光探针的设计、合成和表征, 并评价了它们在唑类抗真菌药物研究中的适用性。尽管唑类抗真菌药物已在临床广泛应用, 但关于其在真菌细胞内摄取和定位的基本问题仍未得到解答。目前, 已知氟康唑、酮康唑和伊曲康唑等唑类抗真菌药物都含有两个保守核心结构:一个间二卤化苯环和一个三唑或咪唑环, 文献[60]报道的这两种唑类荧光探针则是将荧光染料片段丹磺酰染料和Cyanine5 (Cy5)染料与唑类保守核心结构连接合成得到的。这两种唑类荧光探针均能够定位到白色念珠菌SC5314细胞的线粒体且均保持了一定的抗真菌活性。此外, Liu等[52]发现了能够监测活细胞线粒体自噬的新型近红外荧光探针HQO, 以上研究结果为真菌自噬药靶的研究提供了新的思路。以自噬相关蛋白Atg作为全新的潜在药物研发靶点, 设计并合成能够选择性定位到真菌细胞并调控自噬、动态监测真菌细胞自噬的全新分子探针, 对于抗真菌药物的研究将具有深远的意义。
8 总结和展望细胞通过自噬的精确调控, 可维持内环境的稳态, 从而免受外界应激及不利因素的损伤。自噬在真菌感染的发生、发展和治疗过程中的作用是非常复杂的, 既可能是抑制真菌生长的机制, 也可能是真菌细胞存活的机制; 既可能提高抗真菌药物的疗效, 也可能以相反的作用方式使真菌细胞产生耐药性及放疗抵抗效应, 影响抗真菌药物对真菌感染的治疗效果。因此, 监测自噬在细胞中的发生过程是非常重要。自噬是一个动态的、连续的过程, 不是单一的、静态的, 应结合自噬发生时细胞所处的环境状态去选择合适的检测方法, 对自噬进行实时监测。
目前, 自噬过程已经得到初步阐明, 自噬检测手段也在不断发展, 但基于自噬进行抗真菌药物的研发仍存在很大的挑战, 其关键在于如何精准调控病原真菌在不同阶段的自噬。研究人员正利用代谢组学和蛋白质组学的方法来研究自噬途径和代谢产物, 希望发现新的途径或新的代谢产物, 这将有助于阐明自噬在病原真菌中的过程和功能, 将为研发以自噬为靶点的抗真菌候选药物奠定基础。
| [1] |
Mizushima N. Collaboration of proteolytic systems[J]. Auto-phagy, 2007, 3: 179-180. |
| [2] |
Qu X, Yu J, Bhagat G, et al. Promotion of tumorigenesis by heterozygous disruption of the beclin 1 autophagy gene[J]. J Clin Invest, 2003, 112: 1809-1820. DOI:10.1172/JCI20039 |
| [3] |
Mizushima N, Levine B, Cuervo AM, et al. Autophagy fights disease through cellular self-digestion[J]. Nature, 2008, 451: 1069-1075. DOI:10.1038/nature06639 |
| [4] |
Zhu XM, Li L, Wu M, et al. Current opinions on autophagy in pathogenicity of fungi[J]. Virulence, 2019, 10: 481-489. DOI:10.1080/21505594.2018.1551011 |
| [5] |
Levine B, Kroemer G. Autophagy in the pathogenesis of disease[J]. Cell, 2008, 132: 27-42. DOI:10.1016/j.cell.2007.12.018 |
| [6] |
Farré JC, Subramani S. Mechanistic insights into selective autophagy pathways: lessons from yeast[J]. Nat Rev Mol Cell Biol, 2016, 17: 537-552. |
| [7] |
Klionsky DJ, Emr SD. Autophagy as a regulated pathway of cellular degradation[J]. Science, 2000, 290: 1717-1721. DOI:10.1126/science.290.5497.1717 |
| [8] |
Lemasters JJ. Selective mitochondrial autophagy, or mitophagy, as a targeted defense against oxidative stress, mitochondrial dysfunction, and aging[J]. Rejuvenation Res, 2005, 8: 3-5. DOI:10.1089/rej.2005.8.3 |
| [9] |
Mizushima N, Levine B. Autophagy in mammalian development and differentiation[J]. Nat Cell Biol, 2010, 12: 823-830. DOI:10.1038/ncb0910-823 |
| [10] |
Kanayama M, Shinohara ML. Roles of autophagy and autophagy-related proteins in antifungal immunity[J]. Front Immunol, 2016, 7: 47. |
| [11] |
Michaeli S, Galili G, Genschik P, et al. Autophagy in plants--what's new on the menu?[J]. Trends Plant Sci, 2016, 21: 134-144. DOI:10.1016/j.tplants.2015.10.008 |
| [12] |
Olmedilla A, Sandalio LM. Selective autophagy of peroxisomes in plants: from housekeeping to development and stress responses[J]. Front Plant Sci, 2019, 10: 1021. DOI:10.3389/fpls.2019.01021 |
| [13] |
Kershaw MJ, Talbot NJ. Genome-wide functional analysis reveals that infection-associated fungal autophagy is necessary for rice blast disease[J]. Proc Natl Acad Sci U S A, 2009, 106: 15967-15972. DOI:10.1073/pnas.0901477106 |
| [14] |
Voigt O, Pöggeler S. Autophagy genes Smatg8 and Smatg4 are required for fruiting-body development, vegetative growth and ascospore germination in the filamentous ascomycete Sordaria macrospora[J]. Autophagy, 2013, 9: 33-49. DOI:10.4161/auto.22398 |
| [15] |
Lv W, Wang C, Yang N, et al. Genome-wide functional analysis reveals that autophagy is necessary for growth, sporulation, deoxynivalenol production and virulence in Fusarium graminearum[J]. Sci Rep, 2017, 7: 11062. DOI:10.1038/s41598-017-11640-z |
| [16] |
Ying SH, Feng MG. Insight into vital role of autophagy in sustaining biological control potential of fungal pathogens against pest insects and nematodes[J]. Virulence, 2019, 10: 429-437. DOI:10.1080/21505594.2018.1518089 |
| [17] |
Lynch-Day MA, Klionsky DJ. The Cvt pathway as a model for selective autophagy[J]. FEBS Lett, 2010, 584: 1359-1366. DOI:10.1016/j.febslet.2010.02.013 |
| [18] |
Klionsky DJ. The molecular machinery of autophagy: unanswered questions[J]. J Cell Sci, 2005, 118: 7-18. DOI:10.1242/jcs.01620 |
| [19] |
Stolz A, Ernst A, Dikic I. Cargo recognition and trafficking in selective autophagy[J]. Nat Cell Biol, 2014, 16: 495-501. DOI:10.1038/ncb2979 |
| [20] |
Parzych KR, Ariosa A, Mari M, et al. A newly characterized vacuolar serine carboxypeptidase, Atg42/Ybr139w, is required for normal vacuole function and the terminal steps of autophagy in the yeast Saccharomyces cerevisiae[J]. Mol Biol Cell, 2018, 29: 1089-1099. DOI:10.1091/mbc.E17-08-0516 |
| [21] |
Wang Q, Liu H, Xu H, et al. Independent losses and duplications of autophagy-related genes in fungal tree of life[J]. Environ Microbiol, 2019, 21: 226-243. DOI:10.1111/1462-2920.14451 |
| [22] |
Suzuki H, Osawa T, Fujioka Y, et al. Structural biology of the core autophagy machinery[J]. Curr Opin Struct Biol, 2017, 43: 10-17. DOI:10.1016/j.sbi.2016.09.010 |
| [23] |
Hu G, Hacham M, Waterman SR, et al. PI3K signaling of autophagy is required for starvation tolerance and virulenceof Cryptococcus neoformans[J]. J Clin Invest, 2008, 118: 1186-1197. DOI:10.1172/JCI32053 |
| [24] |
Hara T, Mizushima N. Role of ULK-FIP200 complex in mammalian autophagy: FIP200, a counterpart of yeast Atg17?[J]. Autophagy, 2009, 5: 85-87. DOI:10.4161/auto.5.1.7180 |
| [25] |
Martinez-Vicente M, Cuervo AM. Autophagy and neurodegene-ration: when the cleaning crew goes on strike[J]. Lancet Neurol, 2007, 6: 352-361. DOI:10.1016/S1474-4422(07)70076-5 |
| [26] |
Meijer AJ, Codogno P. Signalling and autophagy regulation in health, aging and disease[J]. Mol Aspects Med, 2006, 27: 411-425. DOI:10.1016/j.mam.2006.08.002 |
| [27] |
Maiuri MC, Le Toumelin G, Criollo A, et al. Functional and physical interaction between Bcl-X(L) and a BH3-like domain in Beclin-1[J]. EMBO J, 2007, 26: 2527-2539. DOI:10.1038/sj.emboj.7601689 |
| [28] |
Noda T, Kim J, Huang WP, et al. Apg9p/Cvt7p is an integral membrane protein required for transport vesicle formation in the Cvt and autophagy pathways[J]. J Cell Biol, 2000, 148: 465-480. DOI:10.1083/jcb.148.3.465 |
| [29] |
Noda NN, Fujioka Y. Atg1 family kinases in autophagy initiation[J]. Cell Mol Life Sci, 2015, 72: 3083-3096. DOI:10.1007/s00018-015-1917-z |
| [30] |
Cebollero E, van der Vaart A, Zhao M, et al. Phosphatidylinositol-3-phosphate clearance plays a key role in autophagosome completion[J]. Curr Biol, 2012, 22: 1545-1553. DOI:10.1016/j.cub.2012.06.029 |
| [31] |
Noda T, Ohsumi Y. Tor, a phosphatidylinositol kinase homologue, controls autophagy in yeast[J]. J Biol Chem, 1998, 273: 3963-3966. DOI:10.1074/jbc.273.7.3963 |
| [32] |
Liu Y, Bassham DC. TOR is a negative regulator of autophagy in Arabidopsis thaliana[J]. PLoS One, 2010, 5: e11883. DOI:10.1371/journal.pone.0011883 |
| [33] |
Tsukada M, Ohsumi Y. Isolation and characterization of autophagy-defective mutants of Saccharomyces cerevisiae[J]. FEBS Lett, 1993, 333: 169-174. DOI:10.1016/0014-5793(93)80398-E |
| [34] |
Khan IA, Lu JP, Liu XH, et al. Multifunction of autophagy-related genes in filamentous fungi[J]. Microbiol Res, 2012, 167: 339-345. DOI:10.1016/j.micres.2012.01.004 |
| [35] |
Rajasingham R, Smith RM, Park BJ, et al. Global burden of disease of HIV-associated cryptococcal meningitis: an updated analysis[J]. Lancet Infect Dis, 2017, 17: 873-881. DOI:10.1016/S1473-3099(17)30243-8 |
| [36] |
Oliveira DL, Fonseca FL, Zamith-Miranda D, et al. The putative autophagy regulator Atg7 affects the physiology and pathogenic mechanisms of Cryptococcus neoformans[J]. Future Microbiol, 2016, 11: 1405-1419. DOI:10.2217/fmb-2016-0090 |
| [37] |
Ding H, Caza M, Dong Y, et al. ATG genes influence the virulence of Cryptococcus neoformans through contributions beyond core autophagy functions[J]. Infect Immun, 2018, 86: e00069-18. |
| [38] |
Zhao X, Feng W, Zhu X, et al. Conserved autophagy pathway contributes to stress tolerance and virulence and differentially controls autophagic flux upon nutrient starvation in Cryptococcus neoformans[J]. Front Microbiol, 2019, 10: 2690. DOI:10.3389/fmicb.2019.02690 |
| [39] |
Shintani T, Mizushima N, Ogawa Y, et al. Apg10p, a novel protein-conjugating enzyme essential for autophagy in yeast[J]. EMBO J, 1999, 18: 5234-5241. DOI:10.1093/emboj/18.19.5234 |
| [40] |
Hegedus K, Nagy P, Gaspari Z, et al. The putative HORMA domain protein Atg101 dimerizes and is required for starvation-induced and selective autophagy in Drosophila[J]. Biomed Res Int, 2014, 2014: 470482. |
| [41] |
Nanji T, Liu X, Chew LH, et al. Conserved and unique features of the fission yeast core Atg1 complex[J]. Autophagy, 2017, 13: 2018-2027. DOI:10.1080/15548627.2017.1382782 |
| [42] |
Eskelinen EL, Reggiori F, Baba M, et al. Seeing is believing: the impact of electron microscopy on autophagy research[J]. Autophagy, 2011, 7: 935-956. DOI:10.4161/auto.7.9.15760 |
| [43] |
Sou YS, Tanida I, Komatsu M, et al. Phosphatidylserine in addition to phosphatidylethanolamine is an in vitro target of the mammalian Atg8 modifiers, LC3, GABARAP, and GATE-16[J]. J Biol Chem, 2006, 281: 3017-3024. DOI:10.1074/jbc.M505888200 |
| [44] |
Le Grand JN, Chakrama FZ, Seguin-Py S, et al. GABARAPL1 (GEC1): original or copycat?[J]. Autophagy, 2011, 7: 1098-1107. DOI:10.4161/auto.7.10.15904 |
| [45] |
Weidberg H, Shvets E, Shpilka T, et al. LC3 and GATE-16/GABARAP subfamilies are both essential yet act differently in autophagosome biogenesis[J]. EMBO J, 2010, 29: 1792-1802. DOI:10.1038/emboj.2010.74 |
| [46] |
Kimura S, Fujita N, Noda T, et al. Monitoring autophagy in mammalian cultured cells through the dynamics of LC3[J]. Methods Enzymol, 2009, 452: 1-12. DOI:10.1016/S0076-6879(08)03601-X |
| [47] |
Pinan-Lucarre B, Clave C. Monitoring autophagy in the filamentous fungus Podospora anserina[J]. Methods Enzymol, 2008, 451: 251-270. DOI:10.1016/S0076-6879(08)03218-7 |
| [48] |
Takeshige K, Baba M, Tsuboi S, et al. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction[J]. J Cell Biol, 1992, 119: 301-311. DOI:10.1083/jcb.119.2.301 |
| [49] |
Perry CN, Kyoi S, Hariharan N, et al. Novel methods for measuring cardiac autophagy in vivo[J]. Methods Enzymol, 2009, 453: 325-342. DOI:10.1016/S0076-6879(08)04016-0 |
| [50] |
Biederbick A, Kern HF, Elsasser HP. Monodansylcadaverine (MDC) is a specific in vivo marker for autophagic vacuoles[J]. Eur J Cell Biol, 1995, 66: 3-14. |
| [51] |
Veneault-Fourrey C, Barooah M, Egan M, et al. Autophagic fungal cell death is necessary for infection by the rice blast fungus[J]. Science, 2006, 312: 580-583. DOI:10.1126/science.1124550 |
| [52] |
Liu Y, Zhou J, Wang L, et al. A cyanine dye to probe mitophagy: simultaneous detection of mitochondria and autolysosomes in live cells[J]. J Am Chem Soc, 2016, 138: 12368-12374. DOI:10.1021/jacs.6b04048 |
| [53] |
de la Calle C, Joubert PE, Law HK, et al. Simultaneous assessment of autophagy and apoptosis using multispectral imaging cytometry[J]. Autophagy, 2011, 7: 1045-1051. DOI:10.4161/auto.7.9.16252 |
| [54] |
Degtyarev M, Reichelt M, Lin K. Novel quantitative autophagy analysis by organelle flow cytometry after cell sonication[J]. PLoS One, 2014, 9: e87707. DOI:10.1371/journal.pone.0087707 |
| [55] |
Shvets E, Fass E, Elazar Z. Utilizing flow cytometry to monitor autophagy in living mammalian cells[J]. Autophagy, 2008, 4: 621-628. |
| [56] |
Kim Y, Islam N, Moss BJ, et al. Autophagy induced by rapamycin and carbon-starvation have distinct proteome profiles in Aspergillus nidulans[J]. Biotechnol Bioeng, 2011, 108: 2705-2715. DOI:10.1002/bit.23223 |
| [57] |
Otang WM, Grierson DS, Ndip RN. The Effect of the acetone extract of Arctotis arctotoides (Asteraceae) on the growth and ultrastructure of some opportunistic fungi associated with HIV/AIDS[J]. Int J Mol Sci, 2011, 12: 9226-9235. DOI:10.3390/ijms12129226 |
| [58] |
Quintanilla D, Chelius C, Iambamrung S, et al. A fast and simple method to estimate relative, hyphal tensile-strength of filamentous fungi used to assess the effect of autophagy[J]. Biotechnol Bioeng, 2018, 115: 597-605. DOI:10.1002/bit.26490 |
| [59] |
Yang YP, Hu LF, Zheng HF, et al. Application and interpretation of current autophagy inhibitors and activators[J]. Acta Pharmacol Sin, 2013, 34: 625-635. DOI:10.1038/aps.2013.5 |
| [60] |
Benhamou RI, Bibi M, Steinbuch KB, et al. Real-time imaging of the azole class of antifungal drugs in live candida cells[J]. ACS Chem Biol, 2017, 12: 1769-1777. DOI:10.1021/acschembio.7b00339 |
 2020, Vol. 55
2020, Vol. 55


