2. 云南省高校外用给药系统与制剂技术研究重点实验室, 云南 昆明 650500
2. Key Laboratory of Applied Drug Delivery System and Preparation Technology, Kunming 650500, China
β-内酰胺类抗生素包含青霉素类、头孢霉素类、碳青霉烯类以及单酰胺菌素类, 占整个世界抗生素市场55%的份额[1]。然而药物的滥用加速了细菌耐药性的发展。其中, β-内酰胺酶(BLs)的产生是最重要的耐药性机制, 对β-内酰胺类抗生素构成最大威胁, 能够破坏其药效团β-内酰胺环。目前, 由于BLs的快速扩散和进化, 已从临床分离菌株中鉴定出超过1 300种BLs[2]。Ambler等[3]根据其氨基酸序列和作用机制不同将BLs分为4类: A类、B类、C类和D类(路线图 1)。被称为丝氨酸β-内酰胺酶(SBLs)的A、C和D类酶使用活性位点丝氨酸(Ser)对β-内酰胺环的羰基进行亲核进攻。B类酶, 亦称为金属-β-内酰胺酶(MBLs), 使用Zn结合的羟基对β-内酰胺的羰基进行亲核进攻[4]。
|
Scheme 1 Classifications and mechanisms of β-lactamases (BLs) |
SBLs抑制剂与β-内酰胺抗生素联合用药是临床上治疗由携带各种SBLs的细菌引发炎症感染的主流疗法。目前, 已获FDA批准的SBLs抑制剂包括克拉维酸、舒巴坦、他唑巴坦以及近年刚上市的阿维巴坦与vaborbactam等。与SBLs不同, MBLs是锌离子依赖性酶, 其不能被SBLs抑制剂抑制[5]。所有MBLs共有αβ/βα夹心结构, 但根据氨基酸序列和锌离子结合基序的不同, MBLs可进一步分为3个亚类, B1类、B2类和B3类[6]。Bush等[2]总结了所有MBLs的信息, 如表 1所示。
| Table 1 Classifications and characteristics of metallo-β-lactamases (MBLs) |
三种类型MBLs典型代表的晶体结构都已获得解析[7-9]。在B1类MBLs中, Zn1由3个His残基和水分子进行形似四面体构象配位, Zn2与Asp、His、Cys残基和水分子进行三角双锥构象的螯合。而对于B3类MBLs, 与B1类明显的区别在于His残基替代Cys残基参与Zn2螯合。B1与B3类都能够水解除单环内酰胺外所有β-内酰胺抗生素。值得注意的是B2类MBLs只有一个与B1类相同残基配位的Zn2参与β-内酰胺的水解[10]。上述所有MBLs的金属结合氨基酸序列都是高度保守的, 任一残基的突变都将导致水解活性的丧失[11]。
MBLs水解机制中存在两个不同于SBLs方面, 包括Zn结合水分子作为亲核试剂以及没有形成共价键。这使得MBLs抑制剂的研发显得毫无进展, 以至于携带MBLs的细菌被称为超级细菌。目前临床上尚无药物能够治疗携带MBLs的细菌引发的感染, 原因如下所述。首先, 克拉维酸钾等SBLs抑制剂与SBLs上Ser共价结合形成稳定的酰基-酶中间体, 不可逆地使SBLs失活。这是基于SBLs抑制剂与β-内酰胺抗生素的结构相似性。而MBLs通过锌离子络合稳定的羟基阴离子用于水解β-内酰胺环。在水解过程中并没有形成共价键结合的中间体, 这表明它是可逆反应, 且SBLs抑制剂对MBLs无效。其次, MBLs的Zn结合氨基酸序列同样存在于哺乳动物体内酶中, 如乙二醛酶Ⅱ、芳基硫酸酯酶、黄素蛋白和环化酶/脱水酶等[12]。因此, MBLs抑制剂也有可能抑制哺乳动物中的同源酶, 导致毒性的产生。例如, EDTA是众所周知的具有高毒性的NDM-1抑制剂。第三, 与SBLs不同, MBLs的氨基酸序列千变万化, 活性位点氨基酸序列微妙的不同将会导致显著的变化。因此, 很难找到抑制所有MBLs的通用抑制剂[13]。
新德里金属-β-内酰胺酶(NDM-1)属于B1类MBLs, 2008年首次从一个离开印度前往瑞典的病人体内分离得到的肺炎克雷伯菌和大肠杆菌中检出[14]。它可以水解几乎所有的β-内酰胺类抗生素, 包括号称抗生素最后防线的碳青霉烯类。它可通过质粒等基因转移在世界范围内进行快速传播[15]。美国疾病控制和预防中心(CDC)宣布抗生素的黄金时代已经结束, 经典的β-内酰胺类抗生素对携带blaNDM-1的“超级细菌”引起的感染毫无治疗作用[16]。超级细菌仅对多粘菌素、替加环素等敏感, 但它们都具有较高毒性[17]。NDM-1位于细胞外膜与内膜之间的细胞周质中, 并固定在外膜上。它可以通过外膜囊泡(OMVs)分泌到细胞外, 赋予附近细菌耐药性[18]。蛋白数据库(Protein Data Bank)显示NDM-1相关晶体已达81种, 其中晶体结构中配体包含各种β-内酰胺类抗生素水解型与潜在的NDM-1抑制剂。晶体结构解析表明, 负责β-内酰胺环水解的两个锌离子位于NDM-1活性位点中, 形成宽而浅的底物结合囊腔, 因此可以较容易地容纳不同类型的β-内酰胺类抗生素。其中, 1个锌离子与3个相同的氨基酸His120、His122和His189连接; 另一种锌离子被3种不同的氨基酸Asp124、Cys208和His250包围[9]。NDM-1和美罗培南(MEM)的共结晶体结构揭示了β-内酰胺类抗生素如何被识别与水解(图 1)[19]。

|
Figure 1 Crystal structure and hydrolytic mechanism of NDM-1 |
同时, NDM-1中那些维持活性位点构象并参与底物识别的氨基酸残基已在表 2中列出。所有这些氨基酸残基在β-内酰胺抗生素的水解中起重要作用, 并可以为NDM-1抑制剂的研发奠定基础[20]。其中基于Lys211与Cys208的共价键结合NDM-1抑制剂已见报道[21-24]。
| Table 2 Summary of essential amino acids in the active site of NDM-1 |
目前已报道的具有潜在NDM-1抑制作用的小分子化合物多达500多个, 但临床上尚没有批准的NDM-1抑制剂[25]。最近, VenatoRx制药公司宣布头孢吡肟(cefepime)与taniborbactam (VNRX-5133)联合用药, 治疗携带SBLs与MBLs的耐碳青霉烯类肠杆菌(CRE)及耐碳青霉烯铜绿假单胞菌(CRPA)等耐药菌导致的复杂尿路感染进入Ⅲ期临床研究[26]。本文将对已发表NDM-1抑制剂进行活性结构探讨, 以期归纳出药效团为后续NDM-1抑制剂的开发奠定基础。根据机制不同, NDM-1抑制剂可分为共价键结合抑制剂与非共价键结合抑制剂。
2 非共价键结合抑制剂Yang等[27]通过研究NDM-1异二金属类似物包括CoCo型和ZnCo型发现NDM-1的锌离子在水解β-内酰胺抗生素中至关重要。NDM-1酶中亲电子的锌离子通过离子-偶极相互作用与NDM-1抑制剂的富电子取代基配位从而产生NDM-1抑制活性。
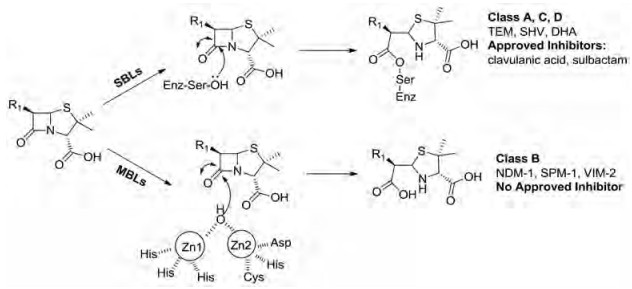
2.1 巯基化合物Klingler等[28]评估了captopril (化合物1)等11种FDA已批准的含巯基药物的NDM-1抑制活性(图 2)。但并非所有的化合物都显示出优异的抑制活性, 结果表明抑制NDM-1不仅需要络合锌离子的巯基, 还需要化合物能够与活性位点附近氨基酸产生结合作用。Captopril作为血管紧张素转化酶(ACE)抑制剂, 其NDM-1酶的半数最大抑制浓度值(IC50值)为6.4 μmol·L-1。Guo等[29]通过手性拆分发现D-captopril (化合物2)与L-captopril显示出不同的抑制NDM-1的IC50值, 分别为7.9和202.0 μmol·L-1。通过对captopril的结构修饰和构效关系(SAR)研究, Li等[30]确定抑制NDM-1的药效基团是巯基羧酸。Skagseth等[31]利用生物电子等排体理论, 将羧基取代为磷酸酯、磷酸或者四氮唑等, 或者将巯基乙酰基保护, 抑制NDM-1活性降低, 进一步论证了巯基羧酸基团的重要性。分子对接研究揭露NDM-1的两个锌离子与巯基官能团作用, Asn220残基与膦酸酯基作用。Thiorphan (3)作为脑啡肽酶抑制剂, 在NDM-1酶上显示出1.8 μmol·L-1的IC50值。Dimercaprol (化合物4)是一种用于治疗有毒金属中毒的药物, 其NDM-1酶的IC50值为1.3 μmol·L-1。治疗威尔逊氏病的药物penicillamine, 同样显示出NDM-1酶抑制活性。
|
Figure 2 Structures and activity of thiol-containing NDM-1 inhibitors |
González等[32]基于NDM-1识别并水解β-内酰胺抗生素机制, 设计并合成了双噻唑烷衍生物, 其中化合物10的IC50值可达23 ± 2 μmol·L-1。NDM-1与化合物10的配合物的晶体结构(PDB:4U4L)进一步揭示了化合物10通过其巯基与两个锌离子相互作用, 其羧基通过两个水分子与NDM-1的Lys211产生相互作用。如前所述, Lys211被证明参与底物识别并调节底物朝向便于进行水解[9, 33]。
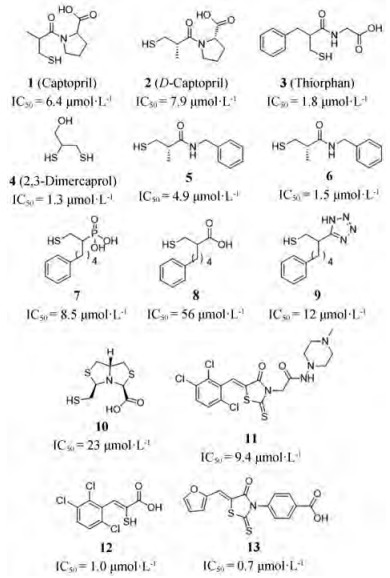
Brem等[34]研究发现硫代烯酸酯化合物12抑制NDM-1的IC50值为1.0 μmol·L-1。化合物12是罗丹宁衍生物11的代谢产物。19F NMR谱中蛋白底物复合物的化学位移变化能够证实化合物12与NDM-1酶的结合。这种19F蛋白化学位移分析方法由Rydzik及其同事发现。通过将三氟丙酮基团引入到接近活性位点的氨基酸残基碳原子中, 建立了一种标记的NDM-1酶。化学环境的微小变化都能引起这种标记酶的19F NMR谱化学位移改变[35]。Xiang等[36]基于化合物11设计并合成了26个罗丹宁衍生物, 分别含有苄基、杂环、萘基、脂肪族羧基和芳香族羧基等取代基。大部分的衍生物显示出了对NDM-1较好的抑制作用, IC50值从0.69到47 μmol·L-1。化合物14与NDM-1分子对接模拟显示硝基作为锌离子配体能够与Zn和Asp124产生作用, 苯环能与Phe70形成π-π堆积相互作用(图 3)。
|
Figure 3 Mechanism of compound 14 |
Chen等[37]基于分子片段的药物发现方法筛选出2, 6-二吡啶甲酸15 (DPA, 图 4)为NDM-1抑制剂, 其IC50为0.52 μmol·L-1。故以其为核心骨架进行进一步的化学修饰, 能够抑制临床上最重要的MBLs如NDM-1、IMP-1和VIM-2的化合物20被优选出来, 并且它没有显著抑制其他锌离子依赖性金属酶的活性。机制研究显示, 尽管DPA显示了螯合NDM-1中锌离子的倾向, 而衍生物20则可以进一步与NDM-1形成稳定的NDM-1/Zn/抑制剂三元复合体。Hinchliffe等[38]依据生物电子等排理论, 利用磷酸酯基团替代一个DPA羧酸基团设计合成了一系列6-膦酰基甲基吡啶-2-羧酸酯(PMPCs)衍生物(化合物19)。其竞争性的缓慢的与NDM-1结合, IC50值为0.3~7.2 μmol·L-1。SAR研究表明双羧酸基团或膦酸基团是抑制NDM-1的药效团, 剔除一个羧酸基团则抑制活性大大降低。机制研究表明PMPCs通过与活性位点结合从而抑制NDM-1, 而不是仅仅螯合锌离子。
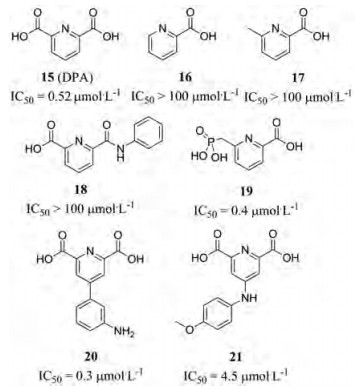
|
Figure 4 Structures and activity of pyridinedicarboxylic acid derivatives |
Brem等[39]报道了第一种双重BLs抑制剂环状硼酸衍生物。通过模仿常见的内酰胺抗生素水解过程中产生的高能四面体中间体, 它能够抑制SBLs与MBLs。其中化合物23 (图 5)能够降低所有NDM-1高表达的菌株中MEM的MIC值, 最高可达64倍。晶体结构证明C-3位羧酸根可同时络合Zn2和Lys211。其次, 在MBL活性位点中, 苯基硼酸酯双环与头孢菌素二氢噻嗪环非常相似, 两者都可以与Trp87和Phe61残基形成疏水作用。最后, 两个“环外”硼结合羟基与Zn1络合, 与Asn220的乙酰氨基侧链形成氢键。Santucci等[40]第一次报道了非环状硼酸衍生物26~28具有抑制MBLs和SBLs活性。根据分子对接结果, 衍生物通过硼酸基团与Zn1络合, 羧基与Lys211残基产生氢键作用。环状硼酸与非环状硼酸都显示出良好的NDM-1抑制活性, 但前者活性明显强于后者, 这得益于环状硼酸的双环结构。
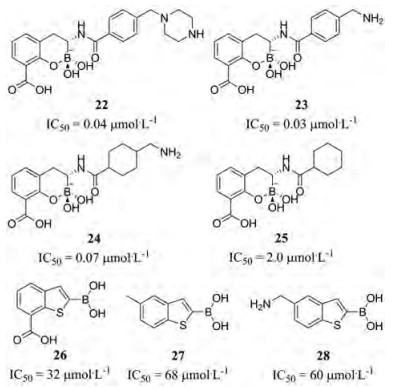
|
Figure 5 Structures and activity of cyclic and acyclic boric acid derivatives |
其中, 双环结构的化合物29 (taniborbactam, VNRX-5133)已被FDA批准进入Ⅲ期临床实验[26]。它同样是一种双重BLs抑制剂, 能够抑制SBLs和一些临床上重要的MBLs, 包括NDM-1, 其IC50为0.01 μmol·L-1。晶体结构分析VNRX-5133硼原子可能以sp2杂化方式, 类似于β-内酰胺抗生素的羰基, 与NDM-1上的Zn络合的羟基反应, 产生紧密结合的sp3配合物, 模仿水解过程中形成的四面体中间体。需要注意的是, 理论上硼酸盐的sp3形式可以与NDM-1活性位点结合, 直接置换锌离子络合的羟基, 但目前只能发现与NDM-1结合模式为硼酸sp2形式[41] (路线图 2)。

|
Scheme 2 Mechanism of taniborbactam |
锌离子缺失是锌离子依赖性酶失活的经典范例[42]。故而金属离子络合剂显示出NDM-1酶抑制活性已被广泛报道。EDTA (化合物30, 图 6)能够与锌离子结合产生NDM-1抑制作用, 其IC50为1.6 μmol·L-1 [43]。其他金属离子螯合剂包括NOTA (化合物31)、DOTA (化合物32)和ME1071 (化合物33)也被证明为NDM-1的抑制剂, 能够恢复抗生素耐药菌株对MEM的敏感性[44, 45]。但是, 这些能够螯合Zn、Fe和Cu等各种金属离子的非选择性螯合剂会产生严重的毒副作用[42]。而且Zn是许多酶的关键元素, 金属螯合剂对体内Zn的过度消耗可能导致疾病[46]。TPEN, 一种具有不同于上述螯合剂的软碱性配体, 通过甲基吡啶胺基团表现出Zn选择性的螯合作用[47]。Schnaars等[48]设计合成了一系列三甲基吡啶胺(TPA)多肽连接衍生物。这些衍生物能够恢复NDM-1高表达铜绿假单胞菌和肺炎克雷伯菌临床菌株对MEM敏感性。衍生物50 μmol·L-1浓度时, MEM的MIC降低32~256倍。此类衍生物对SBLs高表达的临床耐药菌却无活性, 间接论证了TPA的Zn螯合作用。备受关注的是具有高选择性NDM-1抑制剂曲霉明AMA的发现, 一种从Aspegillus versicolor真菌中分离的天然产物[49]。AMA之前被认为是一种抗高血压药, 具有抑制血管紧张素转化酶(ACE)的作用[50]。AMA可以高度选择性地抑制NDM-1, IC50为4.0 ± 1.0 μmol·L-1, 并完全恢复MEM对NDM-1高表达的耐药菌的抗菌活性。与上述金属络合剂不同, 在体内也可以保持AMA的体外功效。动物存活研究表明, 仅AMA和MEM单独给药都无法治愈由NDM-1高表达的肺炎克雷伯菌引起的致命感染。但是MEM (10 mg·kg-1)和AMA (10 mg·kg-1)的联合给药却可使生存率大大提高到95%。令人惊讶的是, 与EDTA的半致死率(LD50值)为29 mg·kg-1相比, AMA的LD50值为160 mg·kg-1。ICP-MS研究结果表明, AMA可以从NDM-1酶中提取单个Zn2+离子。AMA的缺点是亲水性高, CLogP值为-5, 这可能限制了其在临床中的进一步的开发与应用。2016年, Lei和Wright分别完成了AMA分子的立体选择性全合成, 并且纠正了King首次提出的(R, R, S)构型, AMA活性分子实际为(S, S, S)构型[51, 52]。Zhang等[53]进一步设计合成了7个新的AMA衍生物, SAR研究表明在C1和C4以及C7和C10位的羧基对活性至关重要, 而用脯氨酸代替天冬氨酸残基则可以基本保持活性。
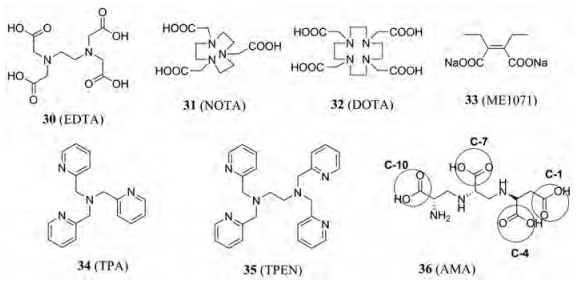
|
Figure 6 Structures of metal chelating agents |
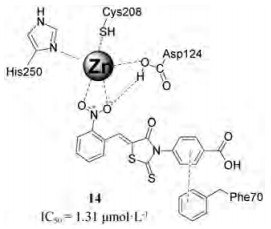
NDM-1仅存在一个Cys208残基, 且作为Zn2配体氨基酸以维持保守的活性位点结构。故此Cys残基可为NDM-1抑制剂设计提供潜在的靶标。Thomas等[54]通过高通量筛选得到两种不可逆的巯基修饰剂对氯汞苯甲酸37 (pCBM)和硝普钠38 (sodium nitroprusside) (图 7), 它们分别能够与NDM-1上Cys不可逆的共价结合从而表现出NDM-1抑制活性, 其IC50分别为2.3和9.0 μmol·L-1。前课题组已论证依布硒啉39 (Ebs)是一种用于脑缺血和中风的药物, 已被论证为有效的NDM-1抑制剂[21]。ESI-MS结果表明它通过与NDM-1酶活性位点上的Cys208残基形成S-Se键产生共价结合, 致使NDM-1酶丢失Zn2从而丧失活性。体外研究表明, MEM和Ebs联合用药能降低MEM在NDM-1高表达的大肠杆菌中的MIC值, 最高可达512倍。本文作者进一步对Ebs进行SAR研究, 筛选出能够恢复MEM对NDM-1高表达的临床耐药菌的杀菌活性的衍生物40[22]。Su等[55]进一步探讨了化合物41 (ebsulfur)衍生物对NDM-1的抑制活性, 其IC50为0.16~9 μmol·L-1, 且能够逆转NDM-1高表达大肠杆菌对头孢唑林的耐药性。荧光显色实验进一步证明了ebsulfur与NDM-1形成S-S键的抑制作用机制。
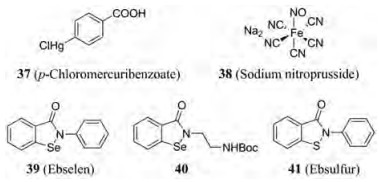
|
Figure 7 Structures of covalent inhibitors of NDM-1 |
Thomas等[23]研究发现Lys211也可作为NDM-1共价抑制剂的靶点。头孢克洛42能够与Lys211形成共价键, 而Lys211被认为是高度保守的, 并且与NDM-1的金属簇相邻。初步的活性机制如路线图 3。
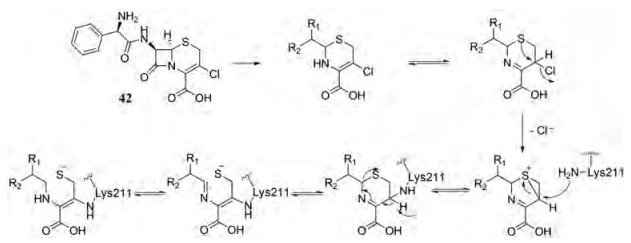
|
Scheme 3 Mechanism of cefaclor |
Thomas等[24]同样报道一种能够与NDM-1活性位点Lys211共价键结合的邻芳氧羰基异羟肟酸酯化合物43。虽然它不够稳定, 但是也为NDM-1抑制剂的研发开阔了思路。其机制如路线图 4所示。

|
Scheme 4 Mechanism of compound 43 |
NDM-1赋予的抗生素耐药性已经成为人类治疗感染的头号威胁, 亟需开发出能够抑制NDM-1恢复细菌敏感性的抑制剂。尽管虚拟化合物库的高通量筛选和酶-配体复合物的结晶可能为NDM-1抑制剂的研发提供帮助, 但所有NDM-1抑制剂包括上述带有各种Zn结合官能团的化合物均未获得批准进入临床应用[56]。这是由于活性位点囊腔大而浅, 能够与几乎所有类型的β-内酰胺抗生素结合以及与配体结合后的水分子重组, 使得计算机辅助药物筛选以及合理的药物设计策略显得不那么可靠。且NDM-1一直在进化, 多达14个亚种已经被鉴别[57]。更值得注意的是, 人体中含有许多金属蛋白酶。靶向细菌NDM-1而非人体金属蛋白酶的化合物可能是未来NDM-1抑制剂研发的先决条件。尽管Zn在人类蛋白质中起着重要作用, 但具有Zn结合功能(ZBF)的分子如上述含巯基化合物、含羧基化合物、吡啶二甲酸衍生物以及硼酸衍生物等仍显示出不错的前景, 特别是广谱抑制剂硼酸衍生物VNRX-5133已进入Ⅲ期临床研究。只要将合理药物设计策略与对NDM-1酶作用机制更为深入的理解相结合, 就能寻找出恢复细菌对抗生素敏感性的先导化合物。
| [1] |
Van Boeckel TP, Gandra S, Ashok A, et al. Global antibiotic consumption 2000 to 2010:an analysis of national pharmaceutical sales data[J]. Lancet Infect Dis, 2014, 14: 742-750. DOI:10.1016/S1473-3099(14)70780-7 |
| [2] |
Bush K. Proliferation and significance of clinically relevant β-lactamases[J]. Ann N Y Acad Sci, 2013, 1277: 84-90. DOI:10.1111/nyas.12023 |
| [3] |
Ambler R. The structure of beta-lactamases[J]. Philos Trans R Soc London B Biol Sci, 1980, 289: 321-331. DOI:10.1098/rstb.1980.0049 |
| [4] |
Chen J, Shang XH, Hu F, et al. β-Lactamase inhibitors:an update[J]. Mini-Rev Med Chem, 2013, 13: 1846-1861. DOI:10.2174/13895575113139990074 |
| [5] |
Sabath L, Abraham E. Zinc as a cofactor for cephalosporinase from Bacillus cereus 569[J]. Biochem J, 1966, 98: 11C. DOI:10.1042/bj0980011C |
| [6] |
Galleni M, Lamotte-Brasseur J, Rossolini GM, et al. Standard numbering scheme for class B β-lactamases[J]. Antimicrob Agents Chemother, 2001, 45: 660-663. DOI:10.1128/AAC.45.3.660-663.2001 |
| [7] |
Fabiane SM, Sohi MK, Wan T, et al. Crystal structure of the zinc-dependent β-lactamase from Bacillus cereus at 1.9 Å resolution:binuclear active site with features of a mononuclear enzyme[J]. Biochemistry, 1998, 37: 12404-12411. DOI:10.1021/bi980506i |
| [8] |
Concha NO, Janson CA, Rowling P, et al. Crystal structure of the IMP-1 metallo β-lactamase from pseudomonas aeruginosa and its complex with a mercaptocarboxylate inhibitor: binding determinants of a potent, broad-spectrum inhibitor[J]. Biochemistry, 2000, 39: 4288-4298. DOI:10.1021/bi992569m |
| [9] |
Zhang H, Hao Q. Crystal structure of NDM-1 reveals a common β-lactam hydrolysis mechanism[J]. FASEB J, 2011, 25: 2574-2582. DOI:10.1096/fj.11-184036 |
| [10] |
Hernandez Valladares M, Felici A, Weber G, et al. Zn (Ⅱ) dependence of the Aeromonas hydrophila AE036 metallo-β-lactamase activity and stability[J]. Biochemistry, 1997, 36: 11534-11541. DOI:10.1021/bi971056h |
| [11] |
Prosperi-Meys C, Bebrone C, Rossolini GM, et al. Mutational analysis of the two zinc-binding sites of the Bacillus cereus 569/H/9 metallo-β-lactamase[J]. Biochem J, 2002, 363: 687-696. DOI:10.1042/bj3630687 |
| [12] |
Daiyasu H, Osaka K, Ishino Y, et al. Expansion of the zinc metallo-hydrolase family of the β-lactamase fold[J]. FEBS Lett, 2001, 503: 1-6. DOI:10.1016/S0014-5793(01)02686-2 |
| [13] |
Li T, Wang Q, Chen F, et al. Biochemical characteristics of New Delhi metallo-β-lactamase-1 show unexpected difference to other MBLs[J]. PLoS One, 2013, 8: e61914. DOI:10.1371/journal.pone.0061914 |
| [14] |
Yong D, Toleman MA, Giske CG, et al. Characterization of a new metallo-β-lactamase gene, bla(NDM-1), and a novel erythromycin esterase gene carried on a unique genetic structure in klebsiella pneumoniae sequence type 14 from India[J]. Antimicrob Agents Chemother, 2009, 53: 5046-5054. DOI:10.1128/AAC.00774-09 |
| [15] |
Hudson CM, Bent ZW, Meagher RJ, et al. Resistance determinants and mobile genetic elements of an NDM-1-encoding Klebsiella pneumoniae strain[J]. PLoS One, 2014, 9: e99209. DOI:10.1371/journal.pone.0099209 |
| [16] |
Bush K, Fisher JF. Epidemiological expansion, structural studies, and clinical challenges of new β-lactamases from Gram-negative bacteria[J]. Annu Rev Microbiol, 2011, 65: 455-478. DOI:10.1146/annurev-micro-090110-102911 |
| [17] |
Rogers BA, Sidjabat HE, Silvey A, et al. Treatment options for New Delhi metallo-beta-lactamase-harboring Enterobacteriaceae[J]. Microb Drug Resist, 2013, 19: 100-103. DOI:10.1089/mdr.2012.0063 |
| [18] |
González LJ, Bahr G, Nakashige TG, et al. Membrane anchoring stabilizes and favors secretion of New Delhi metallo-β-lactamase[J]. Nat Chem Biol, 2016, 12: 516-522. DOI:10.1038/nchembio.2083 |
| [19] |
King DT, Worrall LJ, Gruninger R, et al. New Delhi metallo-β-lactamase:structural insights into β-lactam recognition and inhibition[J]. J Am Chem Soc, 2012, 134: 11362-11365. DOI:10.1021/ja303579d |
| [20] |
Groundwater PW, Xu S, Lai F, et al. New Delhi metallo-β-lactamase-1:structure, inhibitors and detection of producers[J]. Future Med Chem, 2016, 8: 993-1012. DOI:10.4155/fmc-2016-0015 |
| [21] |
Chiou J, Wan S, Chan KF, et al. Ebselen as a potent covalent inhibitor of New Delhi metallo-β-lactamase (NDM-1)[J]. Chem Commun (Camb), 2015, 51: 9543-9546. DOI:10.1039/C5CC02594J |
| [22] |
Jin WB, Xu C, Cheng Q, et al. Investigation of synergistic antimicrobial effects of the drug combinations of meropenem and 1, 2-benzisoselenazol-3(2H)-one derivatives on carbapenem-resistant Enterobacteriaceae producing NDM-1[J]. Eur J Med Chem, 2018, 155: 285. DOI:10.1016/j.ejmech.2018.06.007 |
| [23] |
Thomas PW, Cammarata M, Brodbelt JS, et al. Covalent inhibition of New Delhi metallo-β-lactamase-1(NDM-1) by cefaclor[J]. ChemBioChem, 2014, 15: 2541-2548. DOI:10.1002/cbic.201402268 |
| [24] |
Thomas PW, Cammarata M, Brodbelt JS, et al. A lysine-targeted affinity label for serine-β-lactamase also covalently modifies New Delhi metallo-β-lactamase-1(NDM-1)[J]. Biochemistry, 2019, 58: 2834-2843. DOI:10.1021/acs.biochem.9b00393 |
| [25] |
Linciano P, Cendron L, Gianquinto E, et al. Ten years with New Delhi metallo-β-lactamase-1(NDM-1):from structural insights to inhibitor design[J]. ACS Infect Dis, 2019, 5: 9-34. DOI:10.1021/acsinfecdis.8b00247 |
| [26] |
Krajnc A, Brem J, Hinchliffe P, et al. Bicyclic boronate VNRX-5133 inhibits metallo- and serine-β-lactamases[J]. J Med Chem, 2019, 62: 8544-8556. DOI:10.1021/acs.jmedchem.9b00911 |
| [27] |
Yang H, Aitha M, Marts AR, et al. Spectroscopic and mechanistic studies of heterodimetallic forms of metallo-β-lactamase NDM-1[J]. J Am Chem Soc, 2014, 136: 7273-7285. DOI:10.1021/ja410376s |
| [28] |
Klingler FM, Wichelhaus TA, Frank D, et al. Approved drugs containing thiols as inhibitors of metallo-β-lactamases:strategy to combat multidrug-resistant bacteria[J]. J Med Chem, 2015, 58: 3626-3630. DOI:10.1021/jm501844d |
| [29] |
Guo Y, Wang J, Niu G, et al. A structural view of the antibiotic degradation enzyme NDM-1 from a superbug[J]. Protein Cell, 2011, 2: 384-394. DOI:10.1007/s13238-011-1055-9 |
| [30] |
Li N, Xu Y, Xia Q, et al. Simplified captopril analogues as NDM-1 inhibitors[J]. Bioorg Med Chem Lett, 2014, 24: 386-389. DOI:10.1016/j.bmcl.2013.10.068 |
| [31] |
Skagseth S, Akhter S, Paulsen MH, et al. Metallo-β-lactamase inhibitors by bioisosteric replacement:preparation, activity and binding[J]. Eur J Med Chem, 2017, 135: 159-173. DOI:10.1016/j.ejmech.2017.04.035 |
| [32] |
González MM, Kosmopoulou M, Mojica MF, et al. Bisthiazolidines:a substrate-mimicking scaffold as an inhibitor of the NDM-1 carbapenemase[J]. ACS infect dis, 2015, 1: 544-554. DOI:10.1021/acsinfecdis.5b00046 |
| [33] |
Chiou J, Leung TYC, Chen S. Molecular mechanisms of substrate recognition and specificity of New Delhi metallo-β-lactamase[J]. Antimicrob Agents Chemother, 2014, 58: 5372-5378. DOI:10.1128/AAC.01977-13 |
| [34] |
Brem J, van Berkel SS, Aik W, et al. Rhodanine hydrolysis leads to potent thioenolate mediated metallo-β-lactamase inhibition[J]. Nat Chem, 2014, 6: 1084-1090. DOI:10.1038/nchem.2110 |
| [35] |
Rydzik AM, Brem J, van Berkel SS, et al. Monitoring conformational changes in the NDM-1 metallo-β-lactamase by 19F NMR spectroscopy[J]. Angew Chem Int Ed Engl, 2014, 53: 3129-3133. DOI:10.1002/anie.201310866 |
| [36] |
Xiang Y, Chen C, Wang WM, et al. Rhodanine as a potent scaffold for the development of broad-spectrum metallo-β-lactamase inhibitors[J]. ACS Med Chem Lett, 2018, 9: 359-364. DOI:10.1021/acsmedchemlett.7b00548 |
| [37] |
Chen AY, Thomas PW, Stewart AC, et al. Dipicolinic acid derivatives as inhibitors of new delhi metallo-β-lactamase-1[J]. J Med Chem, 2017, 60: 7267-7283. DOI:10.1021/acs.jmedchem.7b00407 |
| [38] |
Hinchliffe P, Tanner CA, Krismanich AP, et al. Structural and kinetic studies of the potent inhibition of metallo-β-lactamases by 6-phosphonomethylpyridine-2-carboxylates[J]. Biochemistry, 2018, 57: 1880-1892. DOI:10.1021/acs.biochem.7b01299 |
| [39] |
Brem J, Cain R, Cahill S, et al. Structural basis of metallo-β-lactamase, serine-β-lactamase and penicillin-binding protein inhibition by cyclic boronates[J]. Nat Commun, 2016, 7: 12406. DOI:10.1038/ncomms12406 |
| [40] |
Santucci M, Spyrakis F, Cross S, et al. Computational and biological profile of boronic acids for the detection of bacterial serine- and metallo-β-lactamases[J]. Sci Rep, 2017, 7: 17716. DOI:10.1038/s41598-017-17399-7 |
| [41] |
Krajnc A, Lang PA, Panduwawala TD, et al. Will morphing boron-based inhibitors beat the β-lactamases?[J]. Curr Opin Chem Biol, 2019, 50: 101-110. DOI:10.1016/j.cbpa.2019.03.001 |
| [42] |
Aaseth J, Skaug MA, Cao Y, et al. Chelation in metal intoxication-principles and paradigms[J]. J Trace Elem Med Biol, 2015, 31: 260-266. DOI:10.1016/j.jtemb.2014.10.001 |
| [43] |
Ma J, McLeod S, MacCormack K, et al. Real-time monitoring of New Delhi metallo-β-lactamase activity in living bacterial cells by 1H NMR spectroscopy[J]. Angew Chem Int Ed Engl, 2014, 53: 2130-2133. DOI:10.1002/anie.201308636 |
| [44] |
Livermore DM, Mushtaq S, Morinaka A, et al. Activity of carbapenems with ME1071(disodium 2, 3-diethylmaleate) against Enterobacteriaceae and Acinetobacter spp. with carbapenemases, including NDM enzymes[J]. J Antimicrob Chemother, 2013, 68: 153-158. DOI:10.1093/jac/dks350 |
| [45] |
Somboro AM, Tiwari D, Bester LA, et al. NOTA:a potent metallo-β-lactamase inhibitor[J]. J Antimicrob Chemother, 2015, 70: 1594-1596. DOI:10.1093/jac/dku538 |
| [46] |
Andreini C, Bertini I. A bioinformatics view of zinc enzymes[J]. J Inorg Biochem, 2012, 111: 150-156. DOI:10.1016/j.jinorgbio.2011.11.020 |
| [47] |
Donadelli M, Dalla Pozza E, Costanzo C, et al. Zinc depletion efficiently inhibits pancreatic cancer cell growth by increasing the ratio of antiproliferative/proliferative genes[J]. J Cell Biochem, 2008, 104: 202-212. DOI:10.1002/jcb.21613 |
| [48] |
Schnaars C, Kildahl-Andersen G, Prandina A, et al. Synthesis and preclinical evaluation of TPA-based zinc chelators as metallo-β-lactamase inhibitors[J]. ACS Infect Dis, 2018, 4: 1407-1422. DOI:10.1021/acsinfecdis.8b00137 |
| [49] |
King AM, Reid-Yu SA, Wang W, et al. Aspergillomarasmine A overcomes metallo-β-lactamase antibiotic resistance[J]. Nature, 2014, 510: 503-506. DOI:10.1038/nature13445 |
| [50] |
Mikami Y, Suzuki T. Novel microbial inhibitors of angiotensin-converting enzyme, aspergillomarasmines A and B[J]. Agric Biol Chem, 1983, 47: 2693-2695. |
| [51] |
Liao D, Yang S, Wang J, et al. Total synthesis and structural reassignment of aspergillomarasmine A[J]. Angew Chem Int Ed Engl, 2016, 128: 4363-4367. DOI:10.1002/ange.201509960 |
| [52] |
von Nussbaum F, Schiffer G. Aspergillomarasmine A, an inhibitor of bacterial metallo-β-lactamases conferring blaNDM and blaVIM resistance[J]. Angew Chem Int Ed Engl, 2014, 53: 11696-11698. DOI:10.1002/anie.201407921 |
| [53] |
Zhang J, Wang S, Wei Q, et al. Synthesis and biological evaluation of aspergillomarasmine A derivatives as novel NDM-1 inhibitor to overcome antibiotics resistance[J]. Bioorg Med Chem, 2017, 25: 5133-5141. DOI:10.1016/j.bmc.2017.07.025 |
| [54] |
Thomas PW, Spicer T, Cammarata M, et al. An altered zinc-binding site confers resistance to a covalent inactivator of New Delhi metallo-beta-lactamase-1(NDM-1) discovered by high-throughput screening[J]. Bioorg Med Chem, 2013, 21: 3138-3146. DOI:10.1016/j.bmc.2013.03.031 |
| [55] |
Su J, Liu J, Chen C, et al. Ebsulfur as a potent scaffold for inhibition and labelling of New Delhi metallo-β-lactamase-1in vitro and in vivo[J]. Bioinorg Chem, 2019, 84: 192-201. DOI:10.1016/j.bioorg.2018.11.035 |
| [56] |
Ma YR, Zhao HX, Gao QZ. Advances in the study of New Delhi metallo-β-lactamase 1[J]. Acta Pharm Sin (药学学报), 2018, 53: 28-36. |
| [57] |
Mojica MF, Bonomo RA, Fast W. B1-metallo-beta-lactamases:where do we stand?[J]. Curr Drug Targets, 2016, 17: 1029. DOI:10.2174/1389450116666151001105622 |
 2020, Vol. 55
2020, Vol. 55


