2. 中国中药有限公司, 北京 102600;
3. 中药资源教育部工程研究中心, 北京 100193
2. China National Traditional Chinese Medicine Co., Ltd., Beijing 102600, China;
3. Engineering Research Center of Chinese Medicine Resources, Ministry of Education, Beijing 100193, China
本草基因组学是利用组学技术研究中药基原物种的遗传信息及其调控网络, 阐明中药防治人类疾病分子机制的学科[1]。陈士林等[2]提出本草基因组计划, 是针对具有重要经济价值的药用植物和代表不同次生代谢途径的模式药用植物开展的基因组层面的系列研究计划, 主要包括中草药结构基因组、转录组、功能基因组、蛋白组、代谢组、表观基因组、宏基因组等, 其中叶绿体基因组作为中草药结构基因组的重要组成部分, 可以为物种的鉴定、系统发育以及叶绿体基因工程研究提供数据基础[3]。叶绿体是绿色植物进行光合作用的细胞器, 普遍存在于真核自养生物中。叶绿体基因组非常保守[4], 主要表现在基因组结构、基因排列顺序和基因种类等方面。被子植物的大部分叶绿体基因组以共价、闭合、环形双链DNA的多拷贝形式存在, 为环状四分体结构, 由2个序列相同方向相反的重复区(inverted repeats, IRs)、1个大单拷贝区(large single copy, LSC)和1个小单拷贝区(small single copy, SSC)组成[5], 基因组长度通常介于120~160 kb之间, 具有母系遗传、小分子量、多拷贝、分子进化速率慢等特点[6-8]。自1986年烟草(Nicotiana tabacum)和地钱(Marchantia polymorpha)叶绿体全基因组被首次报道以来[9, 10], 目前已经完成了多种植物叶绿体基因组的序列测定, 并研究了其结构、功能与表达。此外, 叶绿体全基因组序列的测定不仅加速了物种的进化、迁徙等方面的研究, 还对物种的鉴定、分子标记、系统发育和转基因研究也存在着巨大的推动作用, 这在揭示物种进化及其种间亲缘关系等方面具有重要价值, 将推动药用植物的叶绿体基因组的深入研究和相关产业的发展, 进一步促进本草基因组学科的发展[11-14]。
石斛属(Dendrobium)是兰科(Orchidaceae)的第二大属, 全球共有1 500余种, 中国有76种, 主要分布在西南及华南地区, 其中可作药用的有40余种[15]。细茎石斛(Dendrobium moniliforme (L.) Sw.)为兰科石斛属多年生草本植物, 是石斛属分布最广泛的种类之一, 主要分布于中国、日本、韩国、印度和尼泊尔等地, 在东亚跨海间断分布, 具有重要的药用以及观赏价值, 其干燥茎是药用石斛的重要来源[16], 也是正品枫斗的基原之一[17], 通常被称为“铜皮枫斗”[18]; 其味甘, 性微寒, 具有益胃生津, 滋阴清热等功效, 用于热病津伤, 口干烦渴, 阴虚火旺等症[19]。细茎石斛含有较高的多糖、生物碱及微量元素等[20-22]。现代药理研究表明其对咽喉疾病、肠胃疾病、白内障、心血管疾病、糖尿病和抑制肿瘤生长具有显著疗效[23]。细茎石斛分子水平的研究如群体遗传学、谱系地理学等也有相关报道[24-26]。分布于中国的细茎石斛的复合种的界定一直有争议, 最新研究把复合种定义为具有细长茎干, 叶尖稍微两裂, 花朵着生于老的无叶片茎干上端, 白色苞片, 有棕色斑块, 蕊柱基部无毛或有时有毛, 并基于形态学以及分子证据的石斛属系统分类的研究, 认为该复合种是一个单系, 分布于中国的细茎石斛的复合种包含有细茎石斛(D. moniliforme)、梵净山石斛(D. fanjingshanense)、霍山石斛(D. huoshanense)、西畴石斛(D. xichouense)、河南石斛(D. henanense)和琉球石斛(D. okinawense) 6个种[27]。近年来, 叶绿体基因组已经应用于石斛属药用植物的条形码鉴定、分子标记和系统发育等研究[28-30], 推动了石斛属物种的鉴定和系统进化研究的进程。本研究通过对细茎石斛叶绿体基因组测序和分析, 为细茎石斛及其复合种的鉴定和系统发育关系的研究提供依据, 对于细茎石斛药材的质量控制、临床用药以及合理开发利用具有重要意义, 同时也为细茎石斛本草基因组学的研究奠定基础。
材料与方法实验材料 本实验用细茎石斛的组培样品进行DNA提取, 产地为安徽霍山, 样品经中国医学科学院药用植物研究所林余霖研究员鉴定, 凭证标本保存于中国医学科学院药用植物研究所。
总DNA提取和测序 总DNA采用DNease Plant Mini Kit试剂盒(Qiagen, 德国)法提取, 利用微量分光光度计(Nanodrop 2000, 美国)检测总DNA的浓度并用1%的琼脂糖凝胶电泳检测总DNA的质量。利用Illumina HiseqX测序平台构建插入片段为500 bp的文库, 并进行双末端测序, 双端各测150 bp。
叶绿体基因组序列的拼接 用Trimmomatic软件去除原始数据中的低质量区, 以美国国立生物信息中心(NCBI)上已公布的叶绿体基因组序列构建局部序列比对检索(BLASTn)数据库, 然后用高质量读序和此数据库进行比对, 根据覆盖度和相似度提取叶绿体相关读长。用软件SOAPdenovo 2将筛选出的叶绿体读长拼接成若干个contigs, 然后用SSPACE软件将contigs连接成完整的叶绿体基因组序列, 用GapCloser软件填补空缺位点。
叶绿体基因组注释和结构分析 用软件Dual Organellar GenoMe Annotator (DOGMA, http://dogma.ccbb.utexas.edu/)和CPGAVAS对序列进行初步注释并手工校正, 用tRNAscan-SE软件注释tRNA, 基因、内含子以及编码区的边界均与参考序列进行比对。将细茎石斛的GenBank格式文件用Organellar Genome DRAW (OGDRAW) v1.2在线绘图工具(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制叶绿体全基因组物理图谱。使用软件MEGA 6.0 (molecular evolutionary genetics analysis 6.0)分析GC含量。
密码子使用分析 本研究采用软件CodonW (http://codonw.sourceforge.net/)分析细茎石斛的密码子使用情况。
长重复序列和简单序列重复 (simple sequence repeats, SSR)结构分析 利用REPuter软件(https://bibiserv.cebitec.uni-bielefeld.de/reputer)识别细茎石斛叶绿体基因组中的长重复序列。利用MISA软件(http://pgrc.ipk-gatersleben.de/misa/)测定SSR位点类型与数目, 其参数设置与李西文等[31]研究一致。本文中主要搜索完全重复型SSR位点, 循环排列或成反向互补的SSR被当作同一种类型。
序列变异及系统发育关系分析 本研究应用在线基因组比对工具mVISTA对6条石斛属叶绿体基因组进行全基因组比对, 并用DnaSP v5.10测定叶绿体基因组间的核酸变异值。利用MAFFT version 5软件对33条石斛属植物叶绿体基因组序列(表 1)进行全基因组比对, 利用IQTREE软件进行建树模型选择以及构建Maximum Likelihood (ML)系统发育树。
| Table 1 Information of the 33 Dendrobium species. *Sequence from this study |
细茎石斛(D. moniliforme)的叶绿体全基因组为常见的四分体结构, 由两个反向重复区IRs、一个大单拷贝区LSC和一个小单拷贝区SSC组成(图 1)。拼接后的细茎石斛叶绿体基因组(GenBank登录号MN617018)全长为150 754 bp, GC含量为37.50%。各区段长度分别为: 84 818 bp (LSC)、14 124 bp (SSC)、25 906 bp (IRs), 4个区段中GC含量最高的是IR区(43.47%), 其次是LSC区(35.08%)和SSC区(30.25%)。
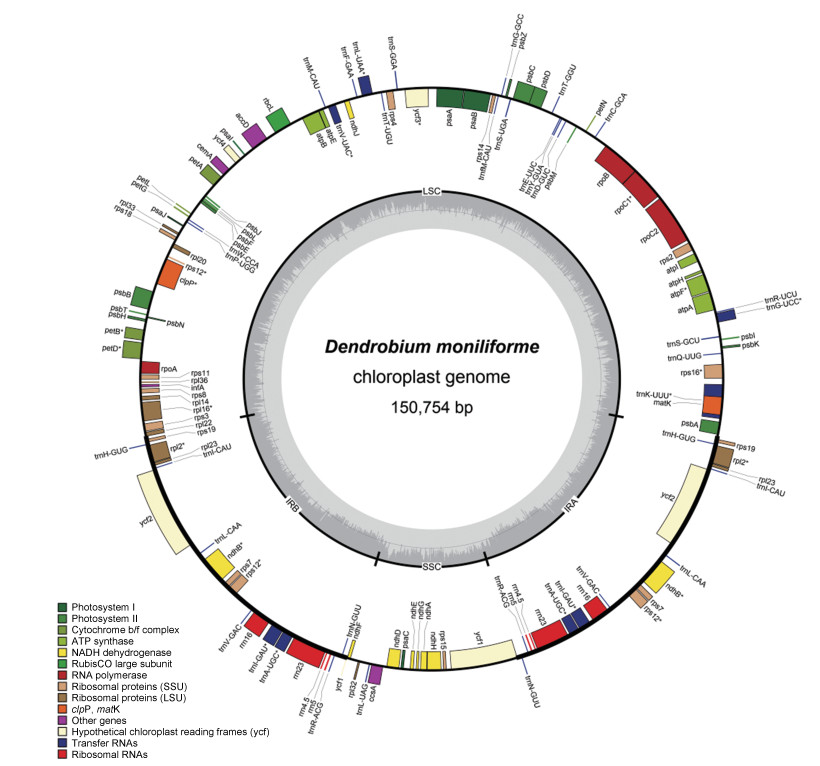
|
Figure 1 Gene maps of the complete chloroplast genome of D. moniliforme. Genes on the inside of the circle are transcribed clockwise, while those on the outside are transcribed counter clockwise. The same gene type is represented by the same color |
细茎石斛叶绿体基因组结构和基因组成可以分为以下4类:与光合作用有关的基因, 与自我复制有关的基因, 未知功能的蛋白基因和成熟酶基因(matK)、囊膜蛋白基因(cemA)等其他基因。共注释到77个蛋白编码基因、38个tRNA基因和8个rRNA基因。其中7个蛋白编码基因(rps19、rpl2、rpl23、ycf2、ndhB、rps7和rps12)、8个tRNA (trnH-GUG、trnI-CAU、trnL-CAA、trnV-GAC、trnI-GAU、trnA-UGC、trnR-ACG和trnN-GUU)和4个rRNA (rrn16、rrn23、rrn4.5和rrn5)位于反向重复区。有17个基因含有内含子, 其中3个基因(ycf3、clpP和rps12)含有两个内含子, 其余14个基因只有一个内含子(表 2)。rps12基因为反式剪切基因, 该基因的5′端位于LSC区, 3′端位于IR区。
| Table 2 Location and length of 17 chloroplast genes containing introns |
根据所有蛋白编码基因, 以相对同义密码子使用值(relative synonymous codon usage, RSCU)作为参考。RSCU值是特定密码子的使用频率与预期频率之间的比率, 用于检测编码序列内的同义密码子使用不均匀的方法, 密码子没有使用偏好性的值设置为1.00, RSCU值大于1.00的密码子实际使用率高于预期, 小于1.00的密码子实际使用率低于预期。结果显示, 在所有的密码子中, 编码亮氨酸(Leu)的密码子最多, 为2 385个。密码子总数不超过1 000的氨基酸有甲硫氨酸(Met)、脯氨酸(Pro)、酪氨酸(Tyr)、组氨酸(His)、天冬氨酸(Asp)、谷氨酰胺(Gln)、半胱氨酸(Cys)、色氨酸(Trp)、丝氨酸(Ser)、终止子(TER)。半胱氨酸(Cys)密码子最少, 为275个。图 2中显示了细茎石斛叶绿体基因组所有蛋白质编码基因的20个氨基酸和终止密码子的密码子含量。
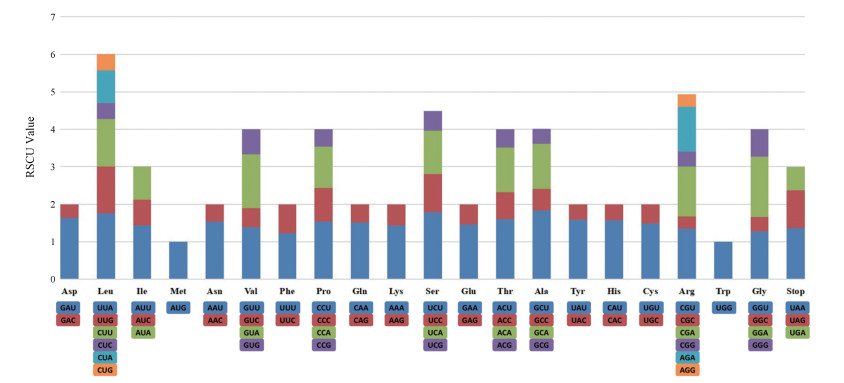
|
Figure 2 Codon content of 20 amino acid and stop codons in all protein-coding genes of the chloroplast genome of D. moniliforme |
长重复序列包括正向重复(forward, F)、回文重复(palindrome, P)、反向重复(reverse, R)和互补重复(complement, C), 对于所有重复类型, 重复长度大于等于30 bp, 序列相似度大于等于90%。细茎石斛长重复序列的数量分布结果显示, 其叶绿体基因组中有8个正向重复(其中30~39 bp的6个, 40~49 bp的2个)、27个回文重复(其中30~39 bp的20个, 40~49 bp的5个, 50~59 bp的2个)、5个反向重复(全部为30~39 bp)和2个互补重复(全部为30~39 bp)。这些重复中重复长度主要在30~39 bp范围内, 其中反向重复和互补重复没有超过40 bp的序列, 只有回文重复有超过50 bp的序列。
叶绿体简单重复序列具有丰富的多态性, 不仅具有标记数量丰富、共显性遗传、重复性高等优点, 同时兼顾叶绿体基因组结构简单、相对保守、单亲遗传等特点, 现已成为一种高效的分子标记, 在物种鉴定和系统进化分析等方面广泛应用[32, 33]。细茎石斛叶绿体全基因组共有53个符合条件的SSR位点。其中单碱基重复有30个、二碱基重复有13个、三碱基重复有2个、四碱基重复有6个、五碱基重复有2个。重复最多的序列是单碱基重复, 且全部是A/T的重复, 共有13处连续A及17处连续T的单碱基重复。然后依次是AT/AT (11次)、AAAG/CTTT (3次)、AG/CT (2次)、AAT/ATT (2次)、AATT/AATT (1次)、ACAG/CTGT (1次)、AGAT/ATCT (1次)、AATAG/ATTCT (1次)、ATATC/ATATG (1次)。另外, 单核苷酸至三核苷酸重复基序的碱基组成存在一定的碱基偏好性, 以富含A-T的重复基序为主。这与细茎石斛叶绿体全基因组富含A-T (AT含量为62.49%)一致, 这种偏好性可能与A-T比G-C更易变动有关[31]。
4 细茎石斛复合种叶绿体基因组比较本研究以细茎石斛为参照基因组利用在线基因组比对工具mVISTA[34]对已发表的复合种物种叶绿体全基因组序列进行全局比对(图 3)。结果显示, 6条叶绿体基因组序列中非编码区变异高于保守的蛋白编码区域, LSC区、SSC区变异明显大于IR区, rRNA基因高度保守, 几乎没有变异。由图看出, 变异较大的基因有trnK、trnL、ycf1和ycf2, 其他基因的保守程度非常高, 绝大多数的基因相似度都在90%以上。基因间区的变异大于基因区, 如trnS-trnG、atpA-atpF、trnE-trnT、trnF-trnV、psbB-psbT、rps12-trnV、ndhF-rpl32、trnV-rps12、rps19-psbA。
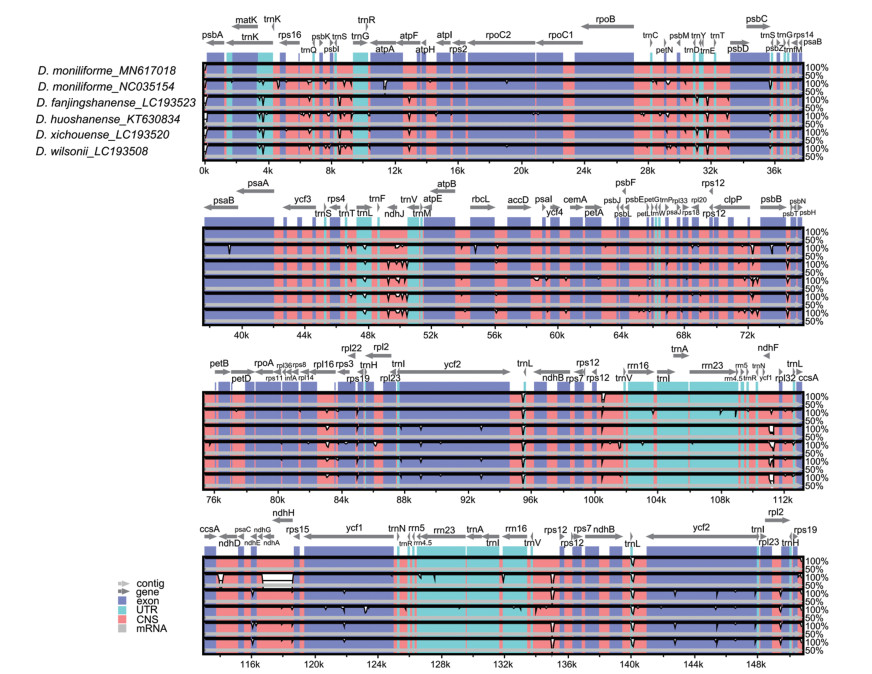
|
Figure 3 Global alignment of chloroplast genomes of D. moniliforme complex. Gray arrows and thick black lines above the alignment indicate genes with their orientation and the position of the IRs, respectively. A cutoff of 70% identity was used for the plots, and the Y-scale represents the percent identity ranging from 50% to 100% |
此外, 使用软件DnaSP分析检测细茎石斛复合种叶绿体基因组中的高变异区, 通过两两比对计算核苷酸变异程度(K值)以显示在序列水平上的变异程度(图 4)。结果显示复合种叶绿体基因组IR区的变异程度明显小于LSC区和SSC区, K值普遍在0.005以下; SSC区表现出较高的变异性, 其中有1处两两比对的峰都特别突出, K值均在0.01以上, 结合mVISTA图并根据具体位点, 此处为ndhF-rpl32; LSC区的变异程度居于IR区和SSC区之间, 其中也有1处两两比对的峰都特别突出, K值均在0.005以上, 结合mVISTA图并根据具体位点, 此处为trnF-trnV。叶绿体基因组两两比对结果显示, 细茎石斛与梵净山石斛之间K的平均值为0.000 35;细茎石斛与霍山石斛之间K的平均值为0.003 39;细茎石斛与西畴石斛之间K的平均值为0.001 45;霍山石斛与梵净山石斛之间K的平均值为0.003 40;霍山石斛与西畴石斛之间K的平均值为0.003 52;梵净山石斛与西畴石斛之间K的平均值为0.001 42, 说明霍山石斛与西畴石斛之间核酸变异程度最大, 而细茎石斛与梵净山石斛之间的变异程度最小。
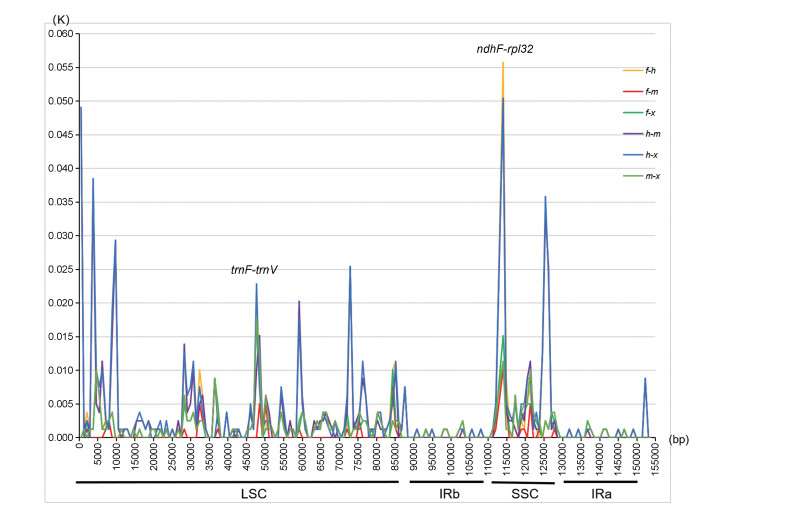
|
Figure 4 Nucleic acid variation information of chloroplast genomes of D. moniliforme complex. m: D. moniliforme; f: D. fanjingshanense; h: D. huoshanense; x: D. xichouense |
叶绿体基因组对系统发育研究具有重要意义[35, 36]。本研究利用从GenBank下载的32条石斛属植物叶绿体基因组序列和测得的细茎石斛的叶绿体基因组序列构建ML进化树, 其中Phalaenopsis aphrodite (蝴蝶兰)和P. equestris (小兰屿蝴蝶兰)为外类群。从ML进化树(图 5)可以看出细茎石斛复合种中的物种聚在同一个大支:本研究测得的细茎石斛与梵净山石斛聚为一支, 支持率为100%, 再与西畴石斛聚为一支, 支持率为72%, 再与GenBank数据库中的另外一条细茎石斛序列聚为一支, 支持率为100%。此外, 广东石斛(D. wilsonii)与细茎石斛、梵净山石斛、西畴石斛、霍山石斛聚在同一个大支, 说明广东石斛与细茎石斛复合种各物种的亲缘关系较近。

|
Figure 5 Phylogenetic tree constructed using ML method based on chloroplast genome sequences of 33 Dendrobium species |
叶绿体基因组序列可以分为蛋白编码区序列和非编码区序列。编码区序列进化速率较慢, 适用于目、科等较高的进化阶元水平的系统进化分析[37]; 非编码区不能编码蛋白质, 序列进化较快, 存在更多的变异信息, 又可以细分为内含子和基因间区, 更加适合于科、属甚至种等较低分类阶元水平的系统进化分析和分子鉴定[38]。石斛作为珍稀药材, 药用价值很大, 但不同物种间或同一物种在不同地区间药用价值也存在一定的差异。长期以来, 植物分类与鉴定主要依据形态学证据, 但形态性状又易受到环境影响, 普遍存在趋同和平行进化, 而遗传物质DNA的研究则能为物种系统进化和属内相似种的区分鉴定提供可靠的分子学证据, 针对石斛属物种系统进化的分析更有益于了解石斛属植物种间的系统进化位置关系[26]。Jin等[39]认为细茎石斛复合种包含广东石斛, Niu等[40]研究也发现广东石斛与细茎石斛复合种物种有较近的亲缘关系, 本研究利用从GenBank下载的32条石斛属植物叶绿体基因组序列和测得的细茎石斛叶绿体基因组序列构建ML进化树, 以探讨石斛属植物之间的系统进化位置关系, 结果显示细茎石斛、梵净山石斛、西畴石斛、霍山石斛聚在同一个大支, 与这些物种聚在同一大支的还有广东石斛, 说明广东石斛与细茎石斛复合种中的物种亲缘关系较近, 与前面的研究结论一致。
DNA条形码(DNA barcoding)是应用一段标准的、相对较短的、具有足够变异的、易扩增的DNA片段进行物种鉴定的一种分子鉴定技术[41]。自2003年提出以来, 该技术普遍用于动物物种的鉴定, 一段650 bp的线粒体COI基因片段已被公认为动物的核心DNA条形码序列。但在植物中, 由于种间杂交和基因渗透的存在, 很难找到像动物中COI一样通用的单个片段。叶绿体基因组长度是普通条形码序列的几百倍, 在种间或种内物种中存在足够的变异位点, 会进一步提高鉴定效率, 有报道提出将叶绿体基因组作为物种鉴定的超级条形码[42, 43], 超级条形码克服了传统条形码的许多局限。首先, 超级条形码的测序可以省略PCR扩增过程, PCR步骤的省略意味着即使材料中的DNA发生降解也仍然可以进行测序, 降低了对测序材料的要求, 扩大了条形码使用范围[44]。其次, 超级条形码包含了更多的信息, 对于物种鉴定更加敏感、高效[45, 46]。此外, 超级条形码克服了因基因缺失、测序质量低等缺点对条形码的使用限制。Zhu等[28]通过实验证明了DNA条形码或者叶绿体基因组高变异区都不能鉴定铁皮石斛及其近缘物种, 而完整的叶绿体基因组序列具有更高的分辨率, 可以用于鉴定铁皮石斛及其近缘物种。超级条形码目前已成功用于种群、种及个体的鉴定[47-51]。本研究中的系统进化树显示, 叶绿体全基因组序列也可作为细茎石斛复合种鉴定的超级条形码, 对细茎石斛复合种物种的鉴定和系统发育具有重要意义。随着测序技术和分析方法的迅速发展, 测序组装叶绿体基因组作为超级条形码将得到广泛的运用。
对于大多数被子植物来说, 叶绿体基因组编码区包含100~120类不同的基因, 大约70~88类基因能编码蛋白质、参与光合作用、基因表达, 30~32类基因编码tRNAs, 4类基因编码rRNAs[52, 53]。本研究中测得细茎石斛叶绿体基因组共注释到123个基因, 包括77个蛋白编码基因、38个tRNA基因和8个rRNA基因, 其中17个蛋白编码基因含有内含子。与核基因组相比, 叶绿体基因组进化速度慢且通常不发生遗传重组[54, 55]。除此之外, 叶绿体基因组较小, 不足其大小的万分之一; 具有多拷贝性, 在每个细胞的拷贝数介于1 000~10 000, 可使目的基因高水平表达; 对外源基因可稳定遗传, 通过外源基因的定点整合, 可避免位置效应和基因沉默现象[56]。由于叶绿体基因组有以上特点, 使其广泛运用于植物多样性、植物适应性和遗传工程研究中[57], 细茎石斛叶绿体基因组的获得为研究其叶绿体转化技术提供了基础。
植物细胞具有核基因组、叶绿体基因组和线粒体基因组3个基因组, 并且在一些种子植物中, 核基因组和叶绿体基因组是可转化的[58]。叶绿体转化是对植物叶绿体基因组进行的遗传转化, 基本原理是通过目的基因两端连接的叶绿体同源片段与叶绿体基因组发生同源重组双交换, 将目的基因整合进入叶绿体基因组, 并经转录、翻译、折叠和修饰等获得功能产物[59]。与核转化相比, 叶绿体的转化不会出现转基因沉默的现象, 外源蛋白的表达量更高; 可同时进行多种外源基因表达, 类似于原核体系中的多顺反子结构; 由于它具有母性遗传的特性, 在后代中不出现性状分离, 故转基因可在子代中稳定地遗传和表达, 可有效地避免外源DNA在田间扩散污染[60]。叶绿体转化作为一项安全高效的体系, 越来越受到重视, 成功进行叶绿体转化的物种也在不断增加[61], 细茎石斛叶绿体基因组测序, 可以根据其叶绿体基因组设计特异的叶绿体同源载体, 提高同源重组效率, 同时为细茎石斛叶绿体基因组在物种问题的解决方面奠定了基础。最近的研究逐步地扩展了人们对叶绿体基因工程的应用, 许多实例表明, 叶绿体转化在植物遗传改良上有着巨大的潜力, 同样在植物生物反应器的发展上也有巨大作用, 可持续且高效益地生产生物医药、生物酶以及化工原材料, 也被应用到基础研究中, 如叶绿体基因组的转录、mRNA编辑、光合作用和进化等[62]。叶绿体转化技术作为分子水平上的一种技术手段, 为植物基因工程的研究开辟了一个新的方向。
本草基因组学利用基因组注释信息, 发掘优良基因, 运用基因工程的手段打破生殖隔离, 培育药效成分含量高的具有优良农艺性状的新品种, 为药效成分的大量提取和广泛临床应用奠定了基础, 为占领中药基础研究领域的科技制高点提供了难得的机遇, 将推动功能基因组学、蛋白组学和代谢组学等现代生命科学技术在药用植物研究领域中的应用, 将是中药现代化的重要组成部分[2]。本草基因组学的研究成果将为阐明药用植物有效成分的合成和调控奠定基础, 进而促进植物类药物的筛选和生物合成研究, 同时还将加速药用植物优良品种的选育, 控制药用植物重要农艺性状的基因, 尤其是与生长发育、抗逆抗病、重要遗传性状及种质性状控制相关的基因, 是药用植物中一类重要的功能基因[63]。本草基因组学将会极大地推动前沿生命科学技术在药用植物和中药领域的应用, 在较短时间内实现药用植物和中药学研究的跨越式发展, 对药用植物物种的鉴定、系统发育及叶绿体基因工程研究有着重要意义。
叶绿体基因组是本草基因组学的重要组成部分, 随着生物信息学及高通量测序技术的不断发展, 人们对叶绿体基因组的研究将日益深入, 研究叶绿体基因组对于揭示叶绿体DNA的结构与起源、植物分子标记、物种亲缘关系等具有重要意义[52]。更重要的是, 叶绿体基因组研究还可以推动植物在分子育种、遗传转化等叶绿体基因工程方面的研究进程[50]。细茎石斛叶绿体基因组测序及系统发育分析不仅可以丰富叶绿体基因组序列的数量, 还可以为研究细茎石斛及其复合种的鉴定、系统发育、培育优良品种、实现该药用植物资源的可持续利用奠定基础, 也为细茎石斛本草基因组学的研究奠定基础。
| [1] |
Chen SL, Song JY. Herbgenomics[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 3881-3889. |
| [2] |
Chen SL, Sun YZ, Xu J, et al. Strategies of the study on Herb Genome Program[J]. Acta Pharm Sin (药学学报), 2010, 45: 807-812. |
| [3] |
Lin XH, Liu ZH, Li Q, et al. Chloroplast genome of medicinal plants[J]. Mod Tradit Chin Med Mater Med-World Sci Technol (世界科学技术-中医药现代化), 2010, 12: 442-446. |
| [4] |
Jansen RK, Ruhlman TA. Plastid Genomes of Seed Plants[M]. New York: Springer, 2012: 103-126.
|
| [5] |
Wang L, Dong WP, Zhou SL. Structural mutations and reorganizations in chloroplast genomes of flowering plants[J]. Acta Bot Boreal-Occid Sin (西北植物学报), 2012, 32: 1282-1288. |
| [6] |
Wakasugi T, Tsudzuki T, Sugiura M. The genomics of land plant chloroplasts:gene content and alteration of genomic information by RNA editing[J]. Photosyn Res, 2001, 70: 107-118. DOI:10.1023/A:1013892009589 |
| [7] |
Kahlau S, Aspinall S, Gray JC, et al. Sequence of the tomato chloroplast DNA and evolutionary comparison of solanaceous plastid genomes[J]. J Mol Evol, 2006, 63: 194-207. DOI:10.1007/s00239-005-0254-5 |
| [8] |
Shimada H, Sugiura M. Fine structural features of the chloroplast genome:comparison of the sequenced chloroplast genome[J]. Nucleic Acids Res, 1991, 19: 983-995. DOI:10.1093/nar/19.5.983 |
| [9] |
Sugiura M, Shinozaki K, Zaita N, et al. Clone bank of the tobacco (Nicotiana tabacum) chloroplast genome as a set of overlapping restriction endonuclease fragments:mapping of eleven ribosomal protein genes[J]. Plant Sci, 1986, 44: 211-217. DOI:10.1016/0168-9452(86)90093-2 |
| [10] |
Ohyama K, Fukuzawa H, Kohchi T, et al. Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha, chloroplast DNA[J]. Nature, 1986, 322: 572-574. DOI:10.1038/322572a0 |
| [11] |
Wu FH, Chan MT, Liao DC, et al. Complete chloroplast genome of Oncidium Gower Ramsey and evaluation of molecular markers for identification and breeding in Oncidiinae[J]. BMC Plant Biol, 2010, 10: 68-79. DOI:10.1186/1471-2229-10-68 |
| [12] |
Kuang DY, Wu H, Wang YL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae):implication for DNA barcoding and population genetics[J]. Genome, 2011, 54: 663-673. DOI:10.1139/g11-026 |
| [13] |
Nock CJ, Waters DLE, Edwards MA, et al. Chloroplast genome sequences from total DNA for plant identification[J]. Plant Biotechnol J, 2011, 9: 328-333. DOI:10.1111/j.1467-7652.2010.00558.x |
| [14] |
Takano A, Okada H. Phylogenetic relationships among subgenera, species, and varieties of Japanese Salvia L. (Lamiaceae)[J]. J Plant Res, 2011, 124: 245-252. DOI:10.1007/s10265-010-0367-9 |
| [15] |
Song JY, Guo SX, Xiao PG. Research of Dendrobium in the past 10 years[J]. Chin Pharm J (中国药学杂志), 2004, 39: 9-11. |
| [16] |
Zhang HY, Chen YJ, Pan LJ. Study on the pharmacognosy of Dendrobium moniliforme[J]. Guid J Tradit Chin Med Pharm (中医药导报), 2008, 14: 85-86. |
| [17] |
Bao XS, Shun QS. History and current status of Dendrobium medicinal herbs[J]. J Chin Med Mater (中药材), 1999, 22: 440-441. |
| [18] |
Qiu S, Zheng WJ, Xia K, et al. Volatile components in flowers of Dendrobium moniliforme[J]. Guihaia (广西植物), 2019, 39: 1482-1495. |
| [19] |
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. 2015 ed. Beijing: China Medical Science Press, 2015: 92.
|
| [20] |
Pauwels M, Vekemans X, Godé C, et al. Nuclear and chloroplast DNA phylogeography reveals vicariance among European populations of the model species for the study of metal tolerance, Arabidopsis halleri (Brassicaceae)[J]. New Phytol, 2012, 193: 916-928. DOI:10.1111/j.1469-8137.2011.04003.x |
| [21] |
Chen YL, Zhang M, Hua YF, et al. Studies on polysaccharide alkaloids and minerals from Dendrobium moniliforme[J]. China J Chin Mater Med (中国中药杂志), 2001, 26: 709-710. |
| [22] |
Hu GH, Xie CJ, Li HC. Detailed study of trace elements in wild Dendrobium moniliforme[J]. J Wenshan Univ (文山学院学报), 2010, 23: 123-125. |
| [23] |
Muhammad NA. Optimal extraction conditions and antioxidant activity of polysaccharide from Dendrobium monliforme[J]. J Anhui Agric Sci (安徽农业科学), 2018, 46: 1-5. |
| [24] |
Luo J. Comparative Genomic and Pedigree Geography of Chloroplasts of Dendrobium monliforme (Species with Discontinuous Distribution Across the Sea in East Asia) (东亚跨海间断分布物种-细茎石斛的叶绿体比较基因组及谱系地理学研究)[D]. Nanjing: Nanjing Normal University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10319-1017283204.htm
|
| [25] |
Ye MR. Affinity geography and conservation genetics of Dendrobium monliforme (东亚珍稀濒危植物细茎石斛的亲缘地理学和保护遗传学研究)[D]. Nanjing: Nanjing Normal University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10319-1015423738.htm
|
| [26] |
Xing WR, Hou BW, Guan JJ, et al. Sequence analysis of LEAFY homologous gene from Dendrobium moniliforme and application for identification of medicinal Dendrobium[J]. Acta Pharm Sin (药学学报), 2013, 48: 597-603. |
| [27] |
Xiang XG, Schuiteman A, Li DZ, et al. Molecular systematics of Dendrobium (Orchidaceae, Dendrobieae) from mainland Asia based on plastid and nuclear sequences[J]. Mol Phylogen Evol, 2013, 69: 950-960. DOI:10.1016/j.ympev.2013.06.009 |
| [28] |
Zhu SY, Niu ZT, Xue QY. Accurate authentication of Dendrobium officinale and its closely related species by comparative analysis of complete plastomes[J]. Acta Pharm Sin B, 2018, 8: 969-980. DOI:10.1016/j.apsb.2018.05.009 |
| [29] |
Niu ZT, Pan JJ, Xue QY. Plastome-wide comparison reveals new SNV resources for the authentication of Dendrobium huoshanense and its corresponding medicinal slice (HuoshanFengdou)[J]. Acta Pharm Sin B, 2018, 8: 466-477. DOI:10.1016/j.apsb.2017.12.004 |
| [30] |
Yan WJ, Niu ZT, Zhu SY. The complete chloroplast genome sequence of Dendrobium nobile[J]. Mitochondr DNA, 2016, 27: 4090-4092. DOI:10.3109/19401736.2014.1003858 |
| [31] |
Li XW, Gao HH, Wang YT, et al. Complete chloroplast genome sequence of Magnolia grandiflora and comparative analysis with related species[J]. Sci Sin Vit (中国科学:生命科学), 2012, 42: 947-956. |
| [32] |
Yang L, Zhao HS, Peng ZH, et al. Development and application of SSR molecular markers from the chloroplast genome of bamboo[J]. J Trop Subtrop Bot (热带亚热带植物学报), 2014, 22: 263-269. |
| [33] |
Tang DQ, Lu JJ, Fang W, et al. Development, characterization and utilization of GenBank microsatellite markers in Phyllostachys pubescens and related species[J]. Mol Breeding, 2010, 25: 299-311. DOI:10.1007/s11032-009-9333-4 |
| [34] |
Frazer KA, Pachter L, Poliakov A, et al. Vista:computational tools for comparative genomics[J]. Nucleic Acids Res, 2004, 32: W273-W279. DOI:10.1093/nar/gkh458 |
| [35] |
Hu Y, Woeste KE, Zhao P. Completion of the chloroplast genomes of five Chinese Juglans and their contribution to chloroplast phylogeny[J]. Front Plant Sci, 2016, 7: 1955-1970. |
| [36] |
Zhang YJ, Ma PF, Li DZ. High-throughput sequencing of six bamboo chloroplast genomes:phylogenetic implications for temperate woody bamboos (Poaceae:Bambusoideae)[J]. PLoS One, 2011, 6: e20596. DOI:10.1371/journal.pone.0020596 |
| [37] |
Li XW, Hu ZG, Lin XH, et al. High-throughput pyrosequencing of the complete chloroplast genome of Magnolia officinalis and its application in species identification[J]. Acta Pharm Sin (药学学报), 2012, 47: 124-130. |
| [38] |
Shaw J, Lickey EB, Schilling EE, et al. Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms:the tortoise and the hare Ⅲ[J]. Am J Bot, 2007, 94: 275-288. DOI:10.3732/ajb.94.3.275 |
| [39] |
Jin XH, Chen SH, Luo YB. Taxonomic revision of Dendrobium moniliforme complex (Orchidaceae)[J]. Sci Hort, 2009, 120: 143-145. DOI:10.1016/j.scienta.2008.10.002 |
| [40] |
Niu Z, Zhu S, Pan J, et al. Comparative analysis of Dendrobium plastomes and utility of plastomic mutational hotspots[J]. Sci Rep, 2017, 7: 2073. DOI:10.1038/s41598-017-02252-8 |
| [41] |
Hebert PDN, Cywinska A, Ball SL. Biological identifications through DNA barcodes[J]. Proc R Soc London B:Biol Sci, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218 |
| [42] |
Kane NC, Cronk Q. Botany without borders:barcoding in focus[J]. Mol Ecol, 2008, 17: 5175-5176. DOI:10.1111/j.1365-294X.2008.03972.x |
| [43] |
Li XW, Yang Y, Henry RJ, et al. Plant DNA barcoding:from gene to genome[J]. Biol Rev, 2015, 90: 157-166. DOI:10.1111/brv.12104 |
| [44] |
Coissac E, Hollingsworth PM, Lavergne S, et al. From barcodes to genomes:extending the concept of DNA barcoding[J]. Mol Ecol, 2016, 25: 1423-1428. DOI:10.1111/mec.13549 |
| [45] |
Parks M, Cronn R, Liston A. Increasing phylogenetic resolution at low taxonomic levels using massively parallel sequencing of chloroplast genomes[J]. BMC Biol, 2009, 7: 84. DOI:10.1186/1741-7007-7-84 |
| [46] |
Steele PR, Pires JC. Biodiversity assessment:state of the art techniques in phylogenomics and species identification[J]. Am J Bot, 2011, 98: 415-425. DOI:10.3732/ajb.1000296 |
| [47] |
Doorduin L, Gravendeel B, Lammers Y, et al. The complete chloroplast genome of 17 individuals of pest species Jacobaea vulgaris:SNPs, microsatellites and barcoding markers for population and phylogenetic studies[J]. DNA Res, 2011, 18: 93-105. DOI:10.1093/dnares/dsr002 |
| [48] |
Nock CJ, Waters DLE, Edwards MA, et al. Chloroplast genome sequences from total DNA for plant identification[J]. Plant Biotechnol J, 2011, 9: 328-333. DOI:10.1111/j.1467-7652.2010.00558.x |
| [49] |
Kane N, Sveinsson S, Dempewolf H, et al. Ultra-barcoding in cacao (Theobroma spp.; Malvaceae) using whole chloroplast genomes and nuclear ribosomal DNA[J]. Am J Bot, 2012, 99: 320-329. DOI:10.3732/ajb.1100570 |
| [50] |
Ma SJ, Zhou JG, Li Y, et al. Complete chloroplast genomes of Dioscorea opposite and D. collettii and screening specific DNA barcodes[J]. Sci Sin Vit (中国科学:生命科学), 2018, 48: 571-582. DOI:10.1360/N052017-00160 |
| [51] |
Chen XL, Zhou JG, Cui YX, et al. Identification of Ligularia herbs using the complete chloroplast genome as a super-barcode[J]. Front Pharmacol, 2018, 9: 695. DOI:10.3389/fphar.2018.00695 |
| [52] |
Tang P, Ruan QY, Peng C. Phylogeny in structure alterations of Poaceae cpDNA[J]. Chin Agric Sci Bull (中国农学通报), 2011, 27: 171-176. |
| [53] |
Zhang T, Fang Y, Wang X, et al. The complete chloroplast and mitochondrial genome sequences of Boea hygrometrice:insight into the evolution of plant organellar genomes[J]. PLoS One, 2012, 7(e30531). |
| [54] |
Clegg MT. Chloroplast gene sequences and the study of plant evolution[J]. Proc Natl Acad Sci U S A, 1993, 90: 363-367. DOI:10.1073/pnas.90.2.363 |
| [55] |
Clegg MT, Gaut BS, Learn GH, et al. Rates and patterns of chloroplast DNA evolution[J]. Proc Natl Acad Sci U S A, 1994, 91: 6795-6801. DOI:10.1073/pnas.91.15.6795 |
| [56] |
Verma D, Daniell H. Chloroplast vector systems for biotechnology applications[J]. Plant Physiol, 2007, 145: 1129-1143. DOI:10.1104/pp.107.106690 |
| [57] |
Sajjad A, Muhammad W, Abdul LK, et al. The complete chloroplast genome of wild rice (Oryza minuta) and its comparison to related species[J]. Front Plant Sci, 2017, 8: 304. |
| [58] |
Mu LS, He Y, Tian ZH. Research of plant chloroplast genetic transformation technology and its application[J]. J Yangtze Univ (Nat Sci Ed (长江大学学报(自科版)), 2017, 14: 52-57. |
| [59] |
Gong ZG, Xu F, Zhou HP, et al. Chloroplast transformation and the latest progress of its application in vaccine expression[J]. Gen Appl Biol (基因组学与应用生物学), 2012, 31: 310-319. |
| [60] |
Li D, Xie LL, Cui LL, et al. Research of chloroplast genetic transformation technology and its preliminary application in mechanized seed production of hybrid rice[J]. Hybrid Rice (杂交水稻), 2010, 25(S1): 332-337. |
| [61] |
Wang JH, Li YN, Ni PC, et al. Research of chloroplast transformation system[J]. Biotechnol Bull (生物技术通报), 2012(01): 1-6. |
| [62] |
Maliga P. Plastid transformation in higher plants[J]. Ann Rev Plant Biol, 2004, 55: 289-313. DOI:10.1146/annurev.arplant.55.031903.141633 |
| [63] |
Chen SL, Zhu XX, Li CF, et al. Genomics and synthetic biology of traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2012, 47: 1070-1078. |
 2020, Vol. 55
2020, Vol. 55


