2. 河北大学中医学院, 河北 保定 071002;
3. 江苏理工学院电气信息工程学院, 生物信息与医药工程研究所, 江苏 常州 213001
2. College of Traditional Chinese Medicine, Hebei University, Baoding 071002, China;
3. Institute of Bioinformatics and Medical Engineering, School of Electrical and Information Engineering, Jiangsu University of Technology, Changzhou 213001, China
密码子是遗传信息传递的基本单位, 每一种氨基酸至少对应一种密码子(如甲硫氨酸和色氨酸都只有一个密码子, 其他的氨基酸都对应2~6种密码子)。编码同一种氨基酸的密码子称为同义密码子。在早期的研究中显示, 蛋白质翻译过程中, 密码子的使用并不是随机发生的, 并且发现特定物种甚至特定基因通常会偏向于使用一种或几种固定的密码子, 这种现象被称为密码子使用偏好性[1, 2]。在密码子偏好性的形成过程中受到多种因素的影响, 例如GC含量、基因表达量、tRNA丰度及突变压力等[3, 4]。不同物种之间存在着密码子使用偏好性的差异, 即使在同种生物中, 不同基因的密码子使用偏好性也不相同。通过分析物种的密码子使用偏好性, 揭示物种密码子使用偏好性的特征, 对于生物的分子进化机制及基因工程等研究等都具有重要的意义。例如研究植物的叶绿体基因组的序列特征密码子碱基的组成规律, 不仅叶绿体遗传转化效率的提高, 也有利于目的基因的高效稳定表达[5]。
猪苓(Polyporus umbellatus)是我国传统珍贵药用真菌, 其药用记载最早出现在《神农本草经》上, 被列为中品[6]。猪苓性味归经及功效[7]与现代药理学证明, 猪苓的主要活性成分是猪苓多糖、麦角甾醇等, 对膀胱癌、急性肾炎和水肿都具有良好的疗效[8]。目前, 猪苓的研究主要集中在其有效药用活性成分、药理作用及其栽培技术等方面[9, 10]。课题组在长期研究猪苓栽培、资源等基础上, 利用二代高通量测序技术研究猪苓转录组, 揭示了猪苓防御蜜环菌入侵的分子机制[11]。为了进一步深入认识猪苓分子系统学及功能基因组学, 本研究基于本课题组完成的猪苓转录组数据, 对猪苓的密码子使用偏向性进行了系统研究, 并分析影响猪苓基因密码子使用偏好性的因素, 进一步对筛选出的猪苓基因密码子使用模式及最优密码子进行了分析, 为下一步开展猪苓系统进化的分子机制、基于密码子改造的基因功能验证研究提供理论依据。
材料与方法猪苓转录组 在NCBI序列片段存档(sequence read archive, SRA)数据库选取之前本课题组发表的猪苓转录组数据(SRP058382)[11]。利用Trinity 2.2.0软件拼接组装转录谱[12], 应用BLASTx和ESTScan软件分析猪苓转录组中的编码序列(coding sequences, CDS)。由于短序列无法正确计算有效密码子数, 为了减少样本误差, 本研究选择以ATG为起始密码子的非重复且长度大于300 bp的序列进行分析, 最终得到332条用于之后密码子偏好性分析的猪苓CDS序列。
密码子偏好性分析 利用CodonW 1.4.2(http://codonw.sourceforge.net/)软件计算密码子组成和使用偏好性分析。密码子组成的度量指标包括G+C含量(鸟嘌呤和胞嘧啶含量); A3s、T3s、G3s、C3s (同义密码子在第3位上腺嘌呤、胸腺嘧啶、鸟嘌呤和胞嘧啶的出现频率); GC3s (同义密码子第3位的G+C含量)等。密码子使用偏好性的度量指标包括:同义密码子相对使用度(relative synonymous codon usage, RSCU)、有效密码子数(effective number of codons, ENC)、密码子适应指数(codon adaption index, CAI)等。
ENC可用于检测单个基因CDS区密码子偏好程度, 它的取值范围在20到61之间, 这个值越接近20表示该基因所有氨基酸越偏向使用其同义密码子的某一密码子, 这个值越接近61表示该基因所有氨基酸的各同义密码子越被平均使用, 所以ENC的值越低, 表明该基因的密码子使用偏好性越强[13]。
CAI可以检测猪苓基因编码区同义密码子与密码子最佳使用相符合程度, 范围在0到1之间。CAI值较高, 基因具有较高的表达量, CAI值较低, 基因具有较低的表达量[14]。
RSCU是对59个同义密码子(不包括3个终止密码子TAG、TGG、TGA和仅由一个密码子编码的甲硫氨酸ATG及色氨酸TGG密码子)的使用偏好性评估。RSCU值是用同义密码子的实际观测值和同义密码子平均使用期望值之间的比值获得的[15]。
中性绘图分析 在生物体编码氨基酸的过程中, 除了编码精氨酸、亮氨酸和丝氨酸的密码子外, 在密码子第三位的碱基经常会出现同义突变, 中性绘图是以GC12 (密码子第一、二位GC含量的平均值)为纵坐标, 以GC3 (密码子第三位GC含量)为横坐标, 每个散点代表一个基因, 分析密码子3个位点碱基组成之间的相关性, 确定影响密码子偏好性的因素。
ENC-plot绘图 采用ENC绘图分析ENC值和GC3分布的关系, 主要用于检测碱基组成对密码子偏好性的影响。本文利用GC3为横坐标, ENC为纵坐标, 创建二维散点图, 并在图中附加ENC值的标准曲线。如果基因的点分布在标准曲线附近, 这表明密码子的使用偏好性主要受到了突变作用的影响, 如果基因点主要分布在期望曲线下方, 表明密码子使用偏好性主要受选择压力的影响。
PR2 (parity rule 2)奇偶偏好分析 奇偶规则表明, 在DNA的两条互补链之间如果不存在任何突变或者选择压力上的偏倚, 那么碱基含量上应该存在A=T和G=C关系。这种方法主要针对4个密码子编码的氨基酸, 包括Ser、Leu、Pro、Arg、Thr、Val、Ala和Gly。分别以基因中上述氨基酸对应密码子A3/(A3+T3)为纵坐标, 以G3/(G3+C3)为横坐标作图, 以(0.5, 0.5)坐标为中心, 从这个中心出发的矢量表示了PR2偏倚的程度和方向[16]。
最优密码子分析 对于最优密码子的确定, 以ENC值进行排序然后作为偏性标准, 选取ENC最大值和ENC最小值各10%的基因, 分别构建高低偏性库, 之后以△RSCU值为标准确定最优密码子, 取△RSCU > 0.08的密码子作为高表达优越密码子。
对应性分析 对应性分析是指采用多元统计方法研究不同基因密码子变异趋势。利用CodonW软件的RSCU对应分析功能, 将所有基因分布到59维向量空间, 从而得到密码子使用偏好性的特征。通过分析基因位置探究基因向量及基因间值的变异程度, 推测导致密码子偏好发生的原因。
结果 1 密码子组成分析利用CodonW软件分析猪苓转录组中所有候选基因的编码区序列GC含量。将猪苓转录组候选基因的CDS序列作为一个整体进行分析, 结果如图 1所示, GC含量范围在44.51%~64.6%, 主要分布在50%~60%之间, 平均含量为53.57%, GC1、GC2、GC3平均含量分别为56.28%、45.48%和57.98%。总体来看, GC2的值最小, GC1和GC3的值相近, 说明猪苓的CDS的密码子更倾向于使用鸟嘌呤(G)和胞嘧啶(C)这两种碱基。

|
Figure 1 Distribution of the GC content of coding sequences (CDS) in Polyporus umbellatus |
猪苓转录组CDS中性绘图分析结果(图 2)显示, GC12的取值范围为0.407~0.705, GC3的范围为0.356~0.771。GC12与GC3的回归系数为0.075, 说明GC12和GC3的相关性微弱。密码子第一、二位碱基和第三位碱基受突变的影响不同, 表明猪苓的CDS在进化过程中受到一定的突变压力, 影响了CDS本身的碱基组成。
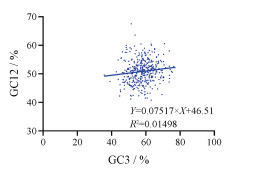
|
Figure 2 Neutrality plot (GC12 vs GC3) |
本研究显示猪苓CDS的ENC取值范围为38.46~61, 因此认为猪苓CDS的密码子偏向性较弱。以GC3为横坐标, ENC为纵坐标作图(图 3), 图 3中各点为基因分布情况。代表基因的大部分点都分布在远离期望曲线的地方, 只有小部分的基因点分布在期望曲线周围, 说明除了突变压力在猪苓密码子偏向性的形成中起到重要作用外, 其他因素比如遗传选择作用也在猪苓密码子偏向性的形成中起到重要作用。

|
Figure 3 Neutrality plot (ENC vs GC3). ENC: Effective number of codons |
为了准确的区分期望ENC与观测ENC之间的区别, 本研究进行了(ENCexp-ENCobs)/ENCexp计算[17], 如图 4所示, (ENCexp-ENCobs)/ENCexp的比值主要分布在0.00~0.10之间, 说明期望ENC与观测ENC之间存在很小差异。因此, 这个结果说明突变不是影响猪苓密码子偏向性形成的唯一因素, 但确实是一个很重要的因素。

|
Figure 4 Frequency distribution of the ENC ratio |
对应性分析结果表明, 第一向量轴的差异占11.31%, 第二向量轴占9.64%的差异, 说明第一轴对猪苓转录组密码子偏向性造成的影响最大。为了进一步说明GC含量对猪苓密码子偏向性的影响, GC含量不同的基因被着上不同的颜色, GC含量高于60%的基因被标记为红色, GC含量在45%~60%之间的基因被标记为蓝色, GC含量低于45%的基因被标记为绿色。如图 5所示, GC含量高于60%的基因在坐标系中分布的比较分散, 而GC含量小于60%的基因则分布的比较集中。
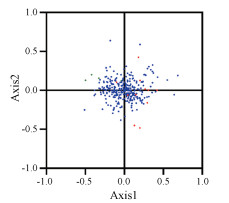
|
Figure 5 Correspondence analysis: genes with GC content higher than 60%, within 45%-60% and lower than 45% were plotted as red, blue and green dots, respectively |
另外, 本研究做了6个参数的相关性分析, 包括GC12、GC3、GCall、ENC、CAI以及Axis 1。如表 1所示, Axis 1与3个重要的参数存在显著相关性, 包括GCall、ENC及Axis 1 (r = 0.679, r = 0.466, r = 0.437, r = 0.218; P < 0.01), 说明突变压力及遗传选择作用在猪苓密码子偏向性形成过程中起到重要作用。
| Table 1 Correlation analysis of Polyporus umbellatus gene-related parameters. *P < 0.05; **P < 0.01. CAI: Codon adaption index |
采用PR2-plot分析了猪苓转录组332条CDS嘌呤(A和G)与嘧啶(T和C)之间的关系, 结果如图 6所示, 大部分基因位于y轴0.5以下, 矢量向下及左右两侧偏倚, 表明猪苓转录组基因的密码子第三位中T、G和C含量较高, 在密码子第三位A、T、C和G使用频率并不相等, 表明猪苓密码子偏向性不仅仅由突变引起, 还有其他因素影响, 比如说遗传选择因素。

|
Figure 6 Parity rule 2 (PR2)-bias plot [A3/(A3+T3) vs G3/(C3+G3)] |
猪苓CDS的CAI取值范围为0.13~0.372, 说明猪苓基因表达水平不高。同时, 对CAI和其他几个重要参数(ENC、GC3、GC)进行了相关性分析(图 7, 表 1), 结果显示CAI值与ENC之间存在显著的负相关性(r = -0.529, P < 0.01), CAI值与GC含量之间存在显著的正相关性(r =0.106, P < 0.01)。因此, 猪苓密码子偏好性的形成过程中受到基因表达水平的影响, GC含量高且表达水平高, 基因的密码子偏好性的程度越高。
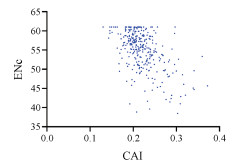
|
Figure 7 Neutrality plot (ENC vs CAI) |
以ENC值为偏性标准, 建立偏性库, 确定了猪苓CDS密码子最优密码子为23个(表 2)。在这些最优密码子中, 以G (7个)或C (14个)结尾的较多, 表明G或C结尾的密码子为猪苓转录组CDS的偏好使用。
| Table 2 Optimal codons of Polyporus umbellatus genes based on the RSCU values. RSCU: Relative synonymous codon usage; *Optimal codons |
研究物种的密码子使用偏好性对于指导基因工程相关的研究, 如重要基因在异源生物中的表达起到重要的作用, 但是由于缺少多数高等生物的基因组数据, 使得我们不能基于基因组数据对生物密码子使用偏好性进行研究。在2015年郭顺星等公布了药用真菌猪苓的转录组数据[11], 为猪苓的密码子使用偏好性奠定了基础。本研究为了了解猪苓转录组CDS密码子使用的偏好性特点, 首先对预测猪苓CDS序列进行筛选, 去除编码区小于300 bp的序列, 最后得到332条CDS密码子使用偏好性分析。
密码子使用偏好性与选择压相关, 通过影响RNA转录与翻译等多种过程以决定整体编译的合理性[18]。编码序列GC含量、转录终止子、转录因子识别序列、隐藏的终止密码、重复序列、CpG二核苷酸偏向性和隐藏的剪接信号等基因序列特征, 都与密码子使用偏好性相关[19]。刘思思等[20]最新研究指出, GC含量和突变压力的平衡调节在基因组的碱基组成中起到重要的作用, 这种调节是影响密码子使用偏好性的最普遍的作用之一。ENC⁃plot绘图分析结果表明, 猪苓部分CDS分布于标准曲线附近, 基因的实际ENC值与理论ENC值基本保持一致, 表明这些CDS的密码子使用偏向性受到GC含量(突变)的影响比较大; 而大多数CDS分布的离标准曲线较远, 表明实际ENC值与理论ENC值存在较大的差异, 受遗传选择的影响较大。因此, 猪苓转录组CDS密码子使用偏好性受到突变的影响, 更多的是受到了选择的作用。
本研究重点分析了猪苓转录组中CDS密码子使用特点, 发现猪苓CDS的最优密码子有23个。在这些最优密码子中, 以G或C结尾的较多, 表明G或C结尾的密码子为猪苓密码子CDS的偏好使用。GC含量被认为是衡量基因组结构进化的重要因素, 基因组不同简并密码子变化的方向和幅度与密码子自身的GC含量相关, GC含量会影响基因的密码子使用偏向性[21, 22]。从猪苓CDS中的GC含量可以看出, 其偏好使用G或C碱基结尾的密码子。猪苓转录组的CDS在密码子使用上的这一特征与赤芝基因[23]、大豆[24]、石榴[25]基因密码子使用规律相同。由此推断猪苓CDS的密码子偏好性的产生与猪苓CDS进化过程中GC到AT突变压力要比AT到GC突变压力高有关。
本研究对猪苓转录组数据中的CDS进行了密码子偏好性及其偏好程度研究, 结果显示猪苓的CDS密码子偏好性较弱, 突变压力和遗传选择都对其密码子使用偏好性产生影响, 这些结果可为猪苓的分子进化研究提供理论基础; 另外, 通过最优密码子分析共计得到21个偏好以G或C结尾的最优密码子, 这个结果可为猪苓基因在进行转基因研究时宿主的选择提供依据。
| [1] |
Grantham R, Gautier C, Gouy M. Codon frequencies in 119 individual genes confirm consistent choices of degenerate bases according to genome type[J]. Nucleic Acids Res, 1980, 8: 1893-1912. DOI:10.1093/nar/8.9.1893 |
| [2] |
Grantham R, Gautier C, Gouy M, et al. Codon catalog usage is a genome strategy modulated for gene expressivity[J]. Nucleic Acids Res, 1981, 9: r43-74. |
| [3] |
Holmquist GP, Filipski J. Organization of mutations along the genome:a prime determinant of genome evolution[J]. Trends Ecol Evol, 1994, 9: 65-69. DOI:10.1016/0169-5347(94)90277-1 |
| [4] |
Bernardi G. The isochore organization of the human genome and its evolutionary history-a review[J]. Gene, 1993, 135: 57-66. DOI:10.1016/0378-1119(93)90049-9 |
| [5] |
Chao YE, Chang Y, Wang MF, et al. Codon usage bias and cluster analysis on chloroplastic genes form seven crop species[J]. Acta Agric Boreali-Sin (华北农学报), 2012, 27: 60-64. |
| [6] |
Xing XK, Ma XT, Hart MM, et al. Genetic diversity and evolution of Chinese traditional medicinal fungus Polyporus umbellatus (Polyporales, Basidiomycota)[J]. PLoS One, 2013, 8: e58807. DOI:10.1371/journal.pone.0058807 |
| [7] |
Xing YM, Chen J, Lv YL, et al. Determination of optimal carbon source and pH value for sclerotial formation of Polyporus umbellatus under artificial conditions[J]. Mycol Prog, 10: 121-125. DOI:10.1007/s11557-010-0725-y |
| [8] |
Xing YM, Zhang LC, Liang HQ, et al. Sclerotial formation of Polyporus umbellatus by low temperature treatment under artificial conditions[J]. PLoS One, 2013, 8: e56190. DOI:10.1371/journal.pone.0056190 |
| [9] |
Liu YY, Guo SX. Nutritional factors determining sclerotial formation of Polyporus umbellatus[J]. Lett Appl Microbiol, 2009, 49: 283-288. DOI:10.1111/j.1472-765X.2009.02656.x |
| [10] |
Sun Y, Yasukawa K. New anti-inflammatory ergostane-type ecdysteroids from the sclerotium of Polyporus umbellatus[J]. Bioorg Med Chem Lett, 18: 3417-3420. DOI:10.1016/j.bmcl.2008.04.008 |
| [11] |
Liu MM, Xing YM, Zhang DW, et al. Transcriptome analysis of genes involved in defence response in Polyporus umbellatus with Armillaria mellea infection[J]. Sci Rep, 2015, 5: 16075. DOI:10.1038/srep16075 |
| [12] |
Haas BJ, Papanicolaou A, Yassour M, et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nat Protoc, 2013, 8: 1494-1512. DOI:10.1038/nprot.2013.084 |
| [13] |
Fuglsang A. Impact of bias discrepancy and amino acid usage on estimates of the effective number of codons used in a gene, and a test for selection on codon usage[J]. Gene, 2008, 410: 82-88. DOI:10.1016/j.gene.2007.12.001 |
| [14] |
Gupta SK, Bhattacharyya TK, Ghosh TC. Synonymous codon usage in Lactococcus lactis:mutational bias versus translational selection[J]. J Biomol Struct Dyn, 2004, 21: 527-536. DOI:10.1080/07391102.2004.10506946 |
| [15] |
Zalucki YM, Power PM, Jennings MP. Selection for efficient translation initiation biases codon usage at second amino acid position in secretory proteins[J]. Nucleic Acids Res, 2007, 35: 5748-5754. DOI:10.1093/nar/gkm577 |
| [16] |
Sinclair G, Choy FY. Synonymous codon usage bias and the expression of human glucocerebrosidase in the methylotrophic yeast, Pichia pastoris[J]. Protein Exp Purif, 2002, 26: 96-105. DOI:10.1016/S1046-5928(02)00526-0 |
| [17] |
Wright F. The 'effective number of codons' used in a gene[J]. Gene, 1990, 87: 23-29. DOI:10.1016/0378-1119(90)90491-9 |
| [18] |
Kumar N, Bera BC, Greenbaum BD, et al. Revelation of influencing factors in overall codon usage bias of equine influenza viruses[J]. PLoS One, 2016, 11: e0154376. DOI:10.1371/journal.pone.0154376 |
| [19] |
Quax TEF, Claassens NJ, Soell D, et al. Codon bias as a means to fine-tune gene expression[J]. Mol Cell, 2015, 59: 149-161. DOI:10.1016/j.molcel.2015.05.035 |
| [20] |
Liu S, Qiao Z, Wang X, et al. Analysis of codon usage patterns in "Lonicerae Flos" (Lonicera macranthoides Hand. -Mazz.) based on transcriptome data[J]. Gene, 2019, 705: 127-132. DOI:10.1016/j.gene.2019.04.065 |
| [21] |
Shackelton LA, Parrish CR, Holmes EC. Evolutionary basis of codon usage and nucleotide composition bias in vertebrate DNA viruses[J]. J Mol Evol, 2006, 62: 551-563. DOI:10.1007/s00239-005-0221-1 |
| [22] |
Van Hemert F, Berkhout B. Nucleotide composition of the Zika virus RNA genome and its codon usage[J]. Virol J, 2016, 13: 95. DOI:10.1186/s12985-016-0551-1 |
| [23] |
Zhu XX, Zhu YJ, Song JY, et al. Comparative studies on codon usage bias of Ganoderma lucidum based on analysis of genomic and transcriptomic data[J]. Acta Pharm Sin (药学学报), 2014, 49: 1340-1345. |
| [24] |
Zhang L, Jin LG, Luo L, et al. Analysis of nuclear gene codon bias on soybean genome and transcriptome[J]. Acta Agron Sin (作物学报), 2011, 37: 965-974. |
| [25] |
Zhang TK, Qi GH, Ye HL, et al. Codon usage bias in pomegranate transcriptome[J]. Acta Hort Sin (园艺学报), 2017, 44: 675-690. |
 2020, Vol. 55
2020, Vol. 55


