旋覆花属(Inula L.)属于菊科(Composite), 包含约100种植物, 分布于欧洲、非洲和亚洲等地区, 部分为广布种, 部分为特有种。我国有20余种和多个变种, 进一步分为5组11系[1, 2]。《中国药典》2015年版收载品种为菊科植物旋覆花(Inula japonica Thunb)和欧亚旋覆花(Inula britannica L.)的干燥头状花序, 具有降气、消痰、行水、止呕的功效, 用于风寒咳嗽、痰饮蓄结、胸膈痞闷、喘咳痰多、呕吐噫气、心下痞硬[3]。旋覆花富含甾醇、倍半萜及黄酮类化合物, 其中, 倍半萜类化学成分被认为是菊科旋覆花属植物的特征性成分, 且不同种旋覆花属植物的倍半萜含量有显著差异[4]。药理研究表明, 旋覆花属植物的提取物及单体成分在抗肿瘤、抗炎、抗血脂和护肝及治疗糖尿病等方面具有良好的生物活性[5]。
该属植物地方习用品和伪混品较多, 且药用功效有一定差异[6, 7]。如部分地区使用线叶旋覆花(Inula linaariifolia Turcz.), 云南、贵州使用同属植物水朝阳旋覆花(Inula helianthus-aquatuca C.Y.Wu ex Ling), 湖北、四川使用湖北旋覆花(Inula hupehensis), 甘肃使用沙旋覆花(Znula salsoloides (Turca.) Ostenf.)。伪混品有柳叶旋覆花(Inula salicina L.)和山白菊(Aster ageraloides Turcz.)。药用功效方面, 水煎剂旋覆花有镇咳、祛痰和抗炎作用, 水朝阳旋覆花有镇咳和抗炎作用, 湖北旋覆花有抗炎和祛痰作用。目前, 市售品混淆现象严重, 有报道显示, 线叶旋覆花煎剂刺激性较大, 会导致呕吐现象[8]; 此外, 近年来也有旋覆花引起皮肤过敏的报道[9]。因此, 早在上个世纪90年代, 王建华等[7]就对中药旋覆花的7种原植物进行过形态组织学研究, 使从药材来源上保证使用安全。但不能很好的区分干药材。刘义梅等[10]采用提取植物DNA, 通过比较ITS序列对旋覆花属8种药用植物(旋覆花、欧亚旋覆花、总状土木香、土木香、羊耳菊、泽兰羊耳菊、赤茎羊耳菊、显脉旋覆花)进行了鉴定, 发现能很好的区分。施之琪等[11]采用HPLC通过鉴定特征成分峰能区分旋覆花、水朝阳旋覆花、湖北旋覆花和山黄菊2种药材制成的配方颗粒, 为临床安全用药提供了参考。但从基因组水平, 对旋覆花属植物或产品进行区分的文章尚未见报道。
叶绿体基因组序列是植物物种鉴定和系统发育学研究的有效工具。与核基因组相比, 叶绿体基因组具有结构简单、分子量小和拷贝多的特点。同时, 叶绿体基因组具有高度保守性。在不同物种之间, 乃至同一物种不同个体间又存在着一定的局部区域的变异。叶绿体基因组的整体高度保守性和局部区域的变异性为其应用于植物系统发育学研究提供了基础与条件[12]。近年来随着基因组测序技术的发展, 越来越多植物的叶绿体全基因组序列得到解析。截止2019年9月, GenBank的RefSeq数据库已经收录了包括陆地植物、海洋植物在内的3 490种植物的叶绿体基因组信息[13]。然而, 旋覆花属植物叶绿体基因组序列尚未见发布。本文通过比较分析旋覆花属3个物种(旋覆花、湖北旋覆花和线叶旋覆花)的叶绿体基因组结构信息, 为该属物种精准鉴定和系统发育研究提供参考, 进一步为旋覆花属植物种质资源保护与开发提供科学依据。关于旋覆花族的起源, 《中国植物志》上有2种不同的观点, 一种观点认为其起源于千里光族, 另一种观点认为其起源于菜蓟族[2]。本研究旨在通过构建基因组系统发育树, 为阐明该族起源提供有力的证据。
材料和方法植物材料、DNA提取与测序 旋覆花采自河南南阳桐柏山脉, 线叶旋覆花采自河北鸽子窝公园, 由中国医学科学院药用植物研究所黄林芳研究员鉴定; 湖北旋覆花叶片采自华中药用植物园(湖北恩施), 由湖北省农科院中药材研究所尤金文教授鉴定。新鲜叶片摘取后放入液氮迅速冷冻, 随后放于-80 ℃冰箱待用。总DNA提取采用植物基因组DNA提取试剂盒(天根生物, 北京), 利用琼脂糖凝胶, NanoDrop2000微量分光光度计(Thermo Scientific, 美国)和Qubit3.0检测总DNA的浓度和质量, 取500 ng DNA构建文库, 采用Illumina Hiseq 2500平台测序, 旋覆花、湖北旋覆花、线叶旋覆花测序数据大小分别为5.2G、5.1 G、5.7G。其原始数据已经提交到GenBank, 相应的Bioproject ID为PRJNA604096。
叶绿体基因组组装和注释 选取拟南芥的rbcL基因作为种子序列, 采用NOVOPlasty (v.2.7.2)从原始测序数据中组装叶绿体基因组[14]。为验证组装的正确性, 采用Bowtie2 (V.2.0.1)[15]将所有原始reads映射(mapping)到旋覆花属物种的叶绿体基因组组装序列上, 通过reads映射的覆盖度及深度检测叶绿体基因组组装的正确性, 尤其是各contig连接处的正确性。叶绿体基因组的注释采用CPGAVAS2[16], 注释信息的编辑、校正采用Apollo[17]。基于CPGAVAS2软件绘制叶绿体基因组环状示意图[18]。利用sequin软件提交旋覆花、湖北旋覆花和线叶旋覆花叶绿体基因组序列及注释至GenBank, 登录号分别为MN974527、MN418395、MN954653。
重复序列分析 重复序列分析和密码子使用情况均使用CPGAVAS2分析获得。其中, 使用MISA发现微卫星序列[19], 参数设置阈值为:单、双、三、四、五、六核苷酸的单位数分别为10、6、5、5、5和5。使用TRF软件发现串联重复序列[20], 设定重复单元的大小≥7。采用VMATCH软件发现散布重复序列[21]。GC含量和密码子使用偏好均采用EMBOSS (v6.3.1)[15]中的Cusp程序计算获得。
系统发育分析 从PlasDB数据库(http://www.herbalgenomics.org/plasdb)下载来自管状花亚科11个族49个属和2个外类群物种的所有叶绿体编码蛋白质序列, 其中59个为共有蛋白质: atpA、atpE、atpF、atpH、rps2、rps3、rps4、rps7、rps8、rps11、rps12、rps15、rps18、rps19、rpl2、rpl14、rpl16、rpl20、rpl22、rpl23、rpl32、rpl33、rpl36、rpoA、rpoB、rpoC2、petA、petB、petD、petG、petL、psaA、psaB、psaC、psaI、psaJ、psbA、psbD、psbE、psbF、psbH、psbI、psbM、psbN、psbT、ndhB、ndhC、ndhE、ndhF、ndhG、ndhH、ndhI、ndhJ、ndhK、rbcL、matK、cemA、ccsA和ycf4。使用Clustal W进行多重序列比对。以拟南芥(Arabidopsis_thaliana)和烟草(Nicotiana_ tabacum)作为外类群, 利用RAxML (Randomized Axelerated Maximum Likelihood)软件实现的最大似然法构建系统发育树。具体参数设置为: “RAxMLHPC-PTHREADS-SSE3 -f -N 1000 -m PROTGAMMACPREV -x 551314260 -p 551314260 -o A_thaliana, N_tabacum T 20”。进化树分枝的可信度用Bootstrap1000次的平均值表示。
结果 1 旋覆花属3个物种叶绿体基因组结构特征比较旋覆花、湖北旋覆花和线叶旋覆花的叶绿体基因组均为典型环状DNA分子, 总长度分别为150 754、150 909和150 812 bp。三者均具有保守的四分状结构, 包括一个大单拷贝区(large single copy region, LSC), 长度分别为82 427、82 552、82 474 bp; 一个小单拷贝区(small single copy region, SSC), 长度分别为18 431、18 453、18 434 bp; 和一对反向重复区(inverted repeat, IR), 长度分别为24 948、24 952、24 952 bp (图 1)。三者叶绿体基因组的整体G/C含量分别为37.72%、37.71%、37.71%, 均低于IR区的G/C含量(43.01%、43.01%、43.00%), 但均高于SSC区(31.29%、31.25%、31.25%)和LSC区(36.05%、35.94%、35.96%)的G/C含量(表 1)。有研究者认为这是因为IR区存在4个高GC含量的rRNA基因[22], 也有研究者认为这表明LSC、SSC和IR区来源不同或具有不同选择压力[23]。
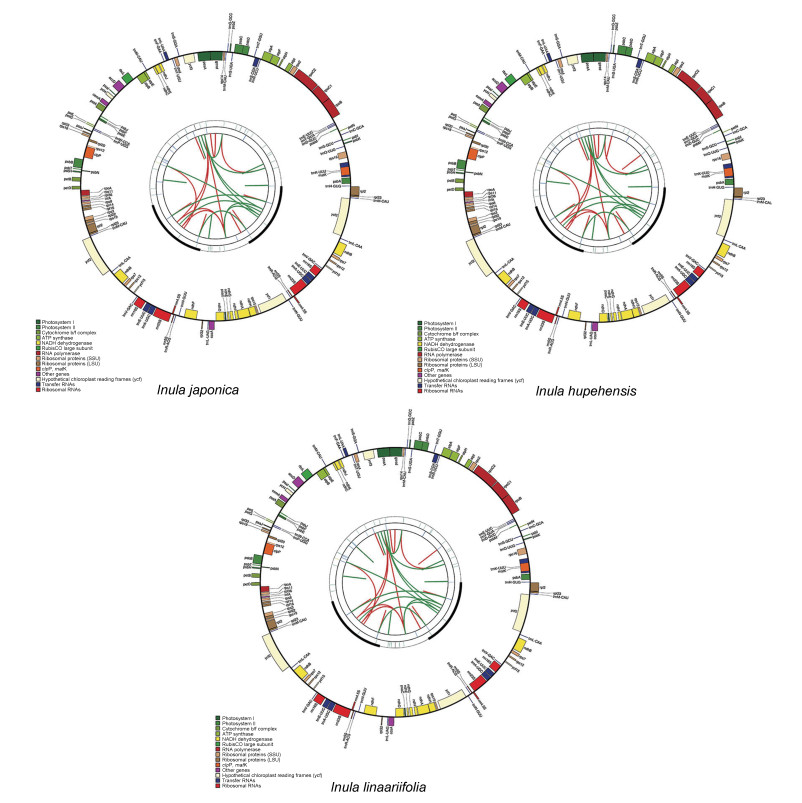
|
Figure 1 Graphic representation of features identified in three Inula L. species plastomes by using CPGAVAS2. The map contains four rings. From the center going outward, the first circle shows the forward and reverse repeats connected with red and green arcs, respectively. The next circle shows the tandem repeats marked with short bars. The third circle shows the microsatellite sequences identified using MISA. The fourth circle is drawn using draw gene map and shows the gene structure on the plastome. The genes were colored based on their functional categories, which are shown at the left corner. |
| Table 1 Summary of the plastome features for the three Inula L. species. LSC: Large single-copy region; SSC: Small single-copy region; IR: Inverted repeat |
在旋覆花属3个物种的叶绿体基因组中均预测到131个编码基因, 包括111个不同的基因, 其中蛋白质编码基因80个、转运RNA (transfer RNA)基因28个和核糖体RNA (ribosome RNA)基因4个(表 2)。三者均检测到9个基因(rps16、rpoC1、atpF、rpl2、ndhB、ndhA、petB、rpoA、ycf2)含1个内含子(intron), 2个基因(ycf3、clpP)含2个内含子。旋覆花、湖北旋覆花和线叶旋覆花叶绿体全基因组中蛋白质编码区(coding sequence, CDS)的长度分别为78 702、78 705、78 702 bp, 各自占其基因组总长度的52.21%、52.15%、52.19%; rRNA基因的长度均为9 394 bp, 分别占各自基因组总长度的6.23%、6.22%、6.23%; tRNA基因的长度均为2 726 bp, 均占各自基因组总长度的1.81%。此外, 旋覆花、湖北旋覆花和线叶旋覆花的叶绿体基因组非编码区均主要包括内含子和基因间隔区, 分别占各自整个基因组长度的39.75%、39.81%、39.78%。
| Table 2 Gene contents of the three Inula L. plastomes |
在旋覆花属3个物种中, 共发现86个蛋白编码基因, 编码26 235个密码子。其中包括编码20个氨基酸的61个唯一密码子和3个终止密码子。在这些密码子中, 编码频次最高的氨基酸为异亮氨酸, 编码2 793次, 占比10.65%, 编码频次最低的氨基酸为半胱氨酸, 只编码了294次, 占比1.12%。3个物种中相同密码子的相对同义密码子用法(relative synonymous codon usage, RSCU)值相似。此外, 3个物种质体蛋白质编码区(CDS)内密码子第三位的A/T含量(69.80%、69.80%、69.81%), 分别高于第一位(54.35%、54.34%、54.35%)和第二位的A/T含量(61.88%、61.88%、61.88%)。与其他大多数陆地植物中观察到的趋势一致, 密码子第三位的A/T含量较第一、二位的高[24]。
2 重复序列分析重复序列在基因组进化中起着重要的作用, 如大片DNA的插入、删除、重排等, 并且可以影响基因组的长度和基因的顺序[25]。本文主要分析叶绿体基因组中的3种重复序列, 即简单重复序列(simple sequence repeat, SSR), 又称微卫星重复序列(microsatellite sequence)、串联重复序列(tandem repeats sequence)和散在重复序列(scattered repetitive sequence)。在旋覆花属3个物种的叶绿体基因组中, 共检测到42个SSR (表 3)。这些SSR主要分布在IGS和内含子序列中, 共检测到38次。大多数单核苷酸重复序列由A/T重复序列组成, AT/AT二核苷酸重复序列是最常见的类型。这与Wang等[26]的报道一致, 即来自质体的SSR通常由短polyA或polyT重复序列组成, 在许多植物中很少含有串联G或C重复序列。在这3个物种的4个基因的CDS中, 共鉴定出4个SSRs, 包括rpoB、rpoC2、rpoA和ycf1。结果表明, 这3个质体在SSRs的数目和GC含量上具有高度的保守性。
| Table 3 Details for the simple sequence repeats in the three Inula L. species |
在旋覆花、湖北旋覆花和线叶旋覆花叶绿体基因组中分别发现有18、22、18个串联重复序列, 这些序列重复单元之间的相似性均大于90%。大多数重复只有2个重复单元。重复单元的长度从10 bp到32 bp不等。
散在重复序列的重复单元是以散在方式分布于基因组内, 主要有回文重复和正向回复2种类型。以e-value小于10-4为阈值, 旋覆花叶绿体基因组散在重复序列包括回文重复序列15条、正向重复序列20条; 湖北旋覆花为17、20条; 线叶旋覆花为18、20条。最长的散布重复单元为48 bp, 分布在2个ycf2基因外显子中。这些重复序列将为分子标记开发和系统发育分化研究提供有意义的线索。
3 基因组系统发育分析对来自管状花亚科49个属的51个物种和2个外类群物种(拟南芥, 烟草)的59个共有蛋白质序列用最大似然法构建进化树。总体上各属物种均与本族物种聚在一起, 与传统分类结果一致(图 2)。如图所示, 管状花亚科可分为4支, 鼠麴草族(Gnaphalieae)、春黄菊族(Anthemideae)聚为一支; 紫苑族(Astereae)单独为一支; 千里光族(Senecioneae)、旋覆花族(Inuleae)、向日葵族(Heliantheae)、泽兰族(Eupatorieae)等聚为一支; 菜蓟族(Cardueae)单独为一支。旋覆花族内, 旋覆花属3个物种与阔苞菊属阔苞菊(Pluchea indica)聚为一类, bootstrap值高(100%); 旋覆花属3个物种中, 旋覆花与湖北旋覆花亲缘关系较近, 二者与线叶旋覆花相对较远。本结果显示旋覆花族与千里光族亲缘关系较近, 与菜蓟族关系较远。这与《中国植物志》上认为的旋覆花族起源于千里光族的观点一致[2]。
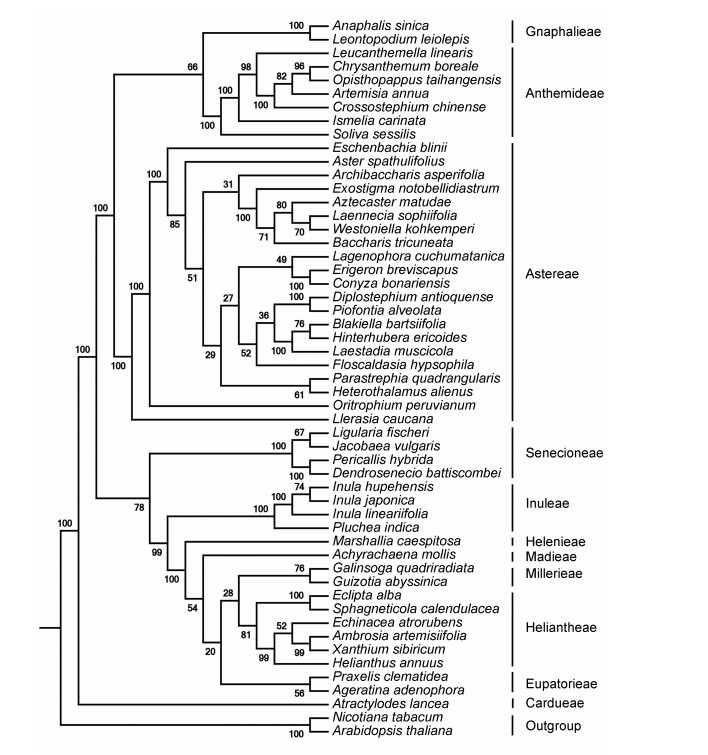
|
Figure 2 Molecular phylogenetic analyses. Plastome sequences of 59 common proteins present from 51 species were used to construct the phylogenetic tree with the maximum likelihood method implemented in the RaxML. Two taxa, namely, N. tabacum and A. thaliana, were used as outgroups. Tribes to which each species belongs are shown on the right side of the tree. Bootstrap supports were calculated from 1000 replicates |
本文首次报道了旋覆花属3个植物旋覆花、湖北旋覆花和线叶旋覆花的叶绿体基因组序列及结构组成。该属3个物种均为典型环状DNA分子, 且具有保守的四分状结构, 与已报道的同族阔苞菊叶绿体基因组大小相似[27]。共预测了130个蛋白质编码基因, 包括111个不同的基因, 其中蛋白质编码基因80个。
本研究从旋覆花、湖北旋覆花和线叶旋覆花叶绿体基因组中分别共检测到32、33、34个SSR; 共发现18、22、18个串联重复序列; 散在重复序列33、37、38条。这些重复序列将为接下来旋覆花属物种分子标记开发, 物种或产品鉴定提供理论依据。本课题组下一步研究将针对旋覆花属3个物种的差异序列进行分析筛选, 寻找合适的区域进行引物设计, 并采集实体样本进行PCR验证, 从分子水平鉴定旋覆花属物种。
关于旋覆花族的起源, 本研究通过对菊科管状花亚科11族48属植物进行系统进化分析发现, 旋覆花族与千里光族亲缘关系较近, 与菜蓟族亲缘关系较远, 首次为旋覆花族起源提供了直接分子证据。
| [1] |
Ling Y, Tseng YQ. Miscellaneous notes on Chinese Compositae[J]. J Univ Chin Acad Sci (中国科学院大学学报), 1978, 16: 82-86. |
| [2] |
Editorial Board of Flora of China, Chinese Academy of Sciences. Flora of China Vol. 75[J]. Beijing:Science Press, 1979, 75: 248-281. |
| [3] |
Chinese Pharmacopoeia Committee.Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Part 1. Beijing: China Medical Science Press, 2015: 325.
|
| [4] |
Yang X, Liu H, He YJ, et al. Simultaneous determination of the contents of five compounds in Inula plants[J]. J Shenyang Pharm Univ (沈阳药科大学学报), 2012, 29: 116-120. |
| [5] |
Fan LL, Chen JN, Zhang T, et al. Chemical constituents in Xuanfuhua (Inula L.) and their pharmacological activities[J]. Guid J Tradit Chin Med Pharm (中医药导报), 2017, 23: 40-43. |
| [6] |
Wang JH, Qi Z, Jia GS, et al. Study on the pharmacological effect of the Traditional Chinese Medicine Xuanfuhua (Inula L.) and its local customs[J]. Beijing J Tradit Chin Med (北京中医), 1997, 1: 42-44. |
| [7] |
Wang JH, Lou ZC. Herbalogical, morphological and histological studies of the Chinese drug XUAN-FU-HUA, Flos Inulae[J]. Acta Pharm Sin (药学学报), 1983, 18: 950-964. |
| [8] |
Li ZA. Adverse reactions caused by Inula linariaefolia[J]. J New Chin Med (新中医), 1985, 8: 55. |
| [9] |
Gao PF, Wang YF, Zhang XJ. A case of skin hypersensitivity caused by XUN-FU-HUA decocting[J]. Clin J Tradit Chin Med (中医药临床杂志), 2004, 13: 473. |
| [10] |
Liu YM, Yu S, Luo K, et al. ITS2 sequences analysis of eight medicinal plants in Inula L.[J]. Chin Tradit Herb Drug (中草药), 2014, 45: 410-414. |
| [11] |
Shi ZQ, Fu JW, Cai CH, et al. Study on the characteristic HPLC chromatograms of Inula japonica formula granules[J]. Tradit Chin Drug Res Clin Pharm (中药新药与临床药理), 2017, 28: 765-770. |
| [12] |
Olejniczak SA, Lojewska E, Kowalczyk T, et al. Chloroplasts:state of research and practical applications of plastome sequencing[J]. Planta, 2016, 244: 517-527. DOI:10.1007/s00425-016-2551-1 |
| [13] |
NIH, U.S. National Library of Medicine. NCBI, Genome.[2019-09-24]. https: //www.ncbi.nlm.nih.gov/genome/browse#!/organelles/[DB/OL].
|
| [14] |
Dierckxsens N, Mardulyn P, Smits G. NOVO Plasty:de novo assembly of organelle genomes from whole genome data[J]. Nucleic Acids Res, 2017, 45: e18. DOI:10.1093/nar/gkw1060 |
| [15] |
Langmead B, Trapnell C, Pop M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome[J]. Genome Biol, 2009, 10: R25. DOI:10.1186/gb-2009-10-3-r25 |
| [16] |
Shi L, Chen H, Jiang M, et al. CPGAVAS2, an integrated plastome sequence annotator and analyzer[J]. Nucleic Acids Res, 2019, 47: W65-W73. DOI:10.1093/nar/gkz345 |
| [17] |
Lewis SE, Searle SM, Harris N, et al. Apollo:a sequence annotation editor[J]. Genome Biol, 2002, 3: 82. |
| [18] |
Lohse M, Drechsel O, Bock R. Organellar Genome DRAW (OG-DRAW):a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J]. Curr Gen, 2007, 52: 267-274. DOI:10.1007/s00294-007-0161-y |
| [19] |
Beier S, Thiel T, Munch T, et al. MISA-web:a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33: 2583-2585. DOI:10.1093/bioinformatics/btx198 |
| [20] |
Benson G. Tandem repeats finder:a program to analyze DNA sequences[J]. Nucleic Acids Res, 1999, 27: 573-580. DOI:10.1093/nar/27.2.573 |
| [21] |
Kurtz S, Choudhuri JV, Ohlebusch E, et al. REPuter:the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Res, 2001, 29: 4633-4642. DOI:10.1093/nar/29.22.4633 |
| [22] |
Qian J. Study on the Chloroplast and Mitochondrial Genome of Salvia miltiorrhiza (丹参的叶绿体和线粒体基因组研究)[D]. Beijing: Chinese Academy of Medical Sciences, Peking Union Medical College, Tsinghua University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10023-1014345717.htm
|
| [23] |
Yang QQ, Jiang M, Wang LQ, et al. Complete chloroplast genome of Allium chinense:comparative genomic and phylogenetic analysis[J]. Acta Pharm Sin (药学学报), 2019, 54: 173-181. |
| [24] |
Nie XJ, Deng PC, Feng K, et al. Comparative analysis of codon usage patterns in chloroplast genomes of the Asteraceae family[J]. Plant Mol Biol Reporter, 2014, 32: 828-840. DOI:10.1007/s11105-013-0691-z |
| [25] |
Tangphatsornruang S, Sangsrakru D, Chanprasert J, et al. The chloroplast genome sequence of mungbean (Vigna radiata) determined by high-throughput pyrosequencing:structural organization and phylogenetic relationships[J]. DNA Res, 2009, 17: 11-22. |
| [26] |
Wang S, Shi C, Gao LZ. Plastid genome sequence of a wild woody oil species, Prinsepia utilis, provides insights into evolutionary and mutational patterns of Rosaceae chloroplast genomes[J]. PLoS One, 2013, 8: e73946. DOI:10.1371/journal.pone.0073946 |
| [27] |
Zhang Y, Zhang J, Yang Y, et al. Complete chloroplast genome of Pluchea indica (L.) Less. (Asteraceae) and its phylogenetic analysis[J]. Mitochondrial DNA B Resour, 2018, 2: 918-919. |
 2020, Vol. 55
2020, Vol. 55


