阿尔茨海默症(Alzheimer's disease, AD)是一种以学习能力衰退为主要临床表现的中枢神经系统退行性疾病[1, 2]。随着全球老龄化的日趋严重, 据估计到本世纪中叶, 每33秒就会出现一例新发AD患者, 全球AD患者数预计高达1.3亿, 其造成的死亡率也逐步升高[3]。然而, 目前仍然不存在有效预防或治疗AD发病的措施, 因此对有效药物干预的需求越来越明显。
抗AD的药物设计主要基于AD发病机制, 研究显示, AD主要的病理特征表现为β-淀粉样蛋白(amyloid-β peptide, Aβ)在细胞外聚合形成斑块和Tau蛋白异常磷酸化引起的细胞内神经原纤维缠结[4-7]。目前仅有4种FDA批准的药物用于AD治疗, 其中3种是胆碱脂酶抑制剂, 另外1种是非竞争性N-甲基-D-天冬氨酸(N-methyl-D-aspartate, NMDA)受体拮抗剂[8-10]。但以上药物仅能缓解临床症状, 不能有效阻止疾病的发病进程。究其原因, 最核心的问题是对AD的致病机制研究深度不够和缺失药物设计有效的靶点, 因此深入研究AD致病机制, 寻求有效的潜在靶点对于治疗AD的药物设计和研发具有重要意义。
蛋白质的翻译后修饰(post-translational modification, PTM)作为一种有效调节蛋白质功能的方式, 参与了多种生理及病理生物学过程, 其中对AD相关生物标志物的发现具有重要作用[11]。大多数与AD相关的蛋白质都需要PTM, 包括Tau蛋白、淀粉样前体蛋白(amyloid precursor protein, APP)、Aβ等。目前研究发现, Aβ42存在着不同类型的PTM, 包括磷酸化、糖基化、脱酰胺化、甲基化和焦谷氨酸化等[12-16]。这些修饰可以改变Aβ42的侧链结构, 而进一步影响其构象变化和聚合过程。Kumar等[17-19]通过动物模型验证了Aβ42的S8磷酸化修饰可加速Aβ42聚合物的形成, 并增加聚合物毒性; Jawhar等[14, 20-22]通过电子显微镜和动物模型研究发现, 氨基端截短型的Aβ42中E3和E11发生的焦谷氨酸化修饰会阻碍脑内Aβ清除机制, 加速聚合物的形成, 并且导致聚合物毒性增强。而与前两种修饰不同, Osaki等[15]应用荧光、圆二色光谱、红外吸收光谱技术发现Aβ42的N27发生脱酰胺化修饰会有效减慢聚合物形成的速率, 但具体机制尚不清楚。因此, 阐述脱酰胺化修饰抑制Aβ42聚合的机制有重要的研究价值, 不仅能为药物靶点的确定提供依据, 而且能为搜寻有效的小分子提供研究方向。
Aβ42单体是形成聚合物结构的基础, Aβ42二聚体是形成寡聚物的最小单位。研究表明, 富含无规卷曲或α螺旋结构的单体不易发生聚合, 而富含β折叠结构的单体易发生聚合, 两种结构的转变是聚合发生与否最重要的环节[23-25]。为此, 本文将通过分子动力学(molecular dynamics, MD)模拟方法, 从结构水平探索第27位天冬酰胺发生脱酰胺化修饰对于Aβ42单体结构改变的影响, 并研究N27脱酰胺化修饰抑制Aβ42聚合的分子机制, 以发现潜在的药物靶点, 为今后基于Aβ42为靶点的药物设计提供重要参考信息。
材料与方法构建体系 Aβ42单体结构(PDB ID: 5oqv[26])从蛋白质结构数据库(protein data bank, PDB)获得。该结构中的A链为未被修饰的Aβ42单体(Aβ42N27)。应用Vienna-PTM网站(http://vienna-ptm.univie.ac.at, 用于构建翻译后修饰的蛋白质结构平台, 并提供相对应分子力场[27, 28]), 将Aβ42单体中N27进行脱酰胺化修饰, 得到N27发生脱酰胺化修饰为D27的Aβ42单体结构(Aβ42D27), 并从该网站下载相应的分子力场。
分子动力学模拟 本研究中所有模拟均采用Gromacs软件进行(版本号: 5.0.1), 对于未修饰的Aβ42单体的模拟应用GROMOS96 54A7力场, 对于已修饰的Aβ42单体的模拟应用翻译后修饰GROMOS96 54A8力场[29]。模拟的详细设置如下:在初始结构中添加立方体盒子, Aβ42单体距离盒子边缘的最小距离为1.0 nm; 向盒子中添加TIP3P水模型和Na+离子或者Cl-离子, 使系统保持电中性; 然后对体系使用最大值为10 kJ·mol-1的下降法进行能量最小化, 得到的构象用于平衡模拟; 模拟温度设定为300 K, 使用LINCS算法进行位置约束, 首先进行500 ps的NVT平衡模拟, 使体系达到稳定的温度; 然后设置在一个标准大气压下进行1 000 ps的NPT平衡模拟, 最后进行100 ns的MD模拟。模拟过程中采用原子无位置约束, 所有时间步长均设定为2 fs。使用Particle-mesh Ewald (PME)方法计算长程静电相互作用, 格点宽度设为0.16 nm; 使用Lenard-Jones函数计算范德华力和短程静电相互作用, 截断距离设定为1.0 nm。为减少计算量, 使用LINCS算法对体系内所有成键原子之间的相对距离进行固定。
采用GROMACS内置模块程序和Python脚本对MD轨迹进行分析。使用GROMACS软件中的gmx_rmsd命令计算均方根偏差(root mean square deviation, RMSD); 使用gmx_rmsf命令计算均方根波动(root mean square fluctuations, RMSF); 使用GROMACS软件中的gmx do_dssp命令计算模拟过程中的二级结构变化; 使用GROMACS软件中的gmx_mdmat命令计算残基接触图, 截断距离设定为0.5 nm; 使用GROMACS软件中的gmx_cluster进行聚类计算, 阈值设定为0.5 nm。
结合能计算 采用MM-PBSA方法计算Aβ42结构中关键区域之间的结合能。结合能的计算表达式为: △Gbinding=EMM+Gsolvation-(GCTR+GNTR), 其中GCTR和GNTR分别为指定区域CTR和NTR中残基在溶剂中的总结合能, EMM是真空中的分子构象能, Gsolvation是溶剂化的自由能。分子构象能包括键合能和非键合能, 其中非键合能主要包括静电相互作用和范德华作用。溶剂化的自由能包括极性能和非极性能, 其中通过溶剂可及表面积计算极性化能, 通过用溶剂表面张力系数(γ)与溶剂可及表面积的积计算非极性化能, γ在Aβ42和水中的值分别设为1和80。
结果 1 结构稳定性分析为了研究27位天冬酰胺发生脱酰胺化修饰对Aβ42单体结构的稳定性影响, 本研究计算了两个模拟体系在整个模拟时间内Aβ42单体骨架原子的RMSD值, 结果如图 1a所示。从图中可以看出, 两个模拟体系的RMSD值在模拟过程的前10 ns内急剧上升, 20 ns之后呈现缓慢波动, 50~100 ns时波动趋于稳定, 表明两个体系的结构变化趋于收敛, 因此本研究中所有的分析均在50~100 ns的模拟轨迹中进行。另外, 尽管两个体系的骨架原子在模拟前具有相同结构, 但在模拟过程中发生修饰的Aβ42单体结构的RMSD值始终大于未发生修饰的Aβ42单体结构, 说明N27发生脱酰胺化修饰对于Aβ42结构的稳定性产生了一定影响。
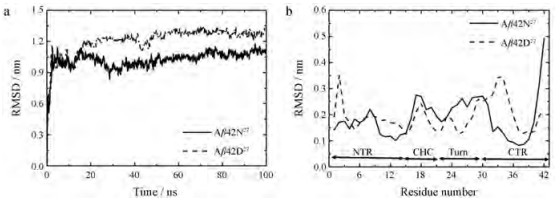
|
Figure 1 Comparison of structural flexibility between Aβ42N27 and Aβ42D27. a: The root mean square deviation (RMSD) of backbone atoms during 100 ns; b: The root mean square fluctuations (RMSF) of each residue from 50 to 100 ns. NTR: N-terminal region; CHC: Central hydrophobic cluster; CTR: C-terminal region |
为了进一步分析两个Aβ42单体结构中各个残基在模拟过程中的变化, 分别计算了50~100 ns模拟时间内Aβ42单体中每个残基骨架原子的RMSF值。RMSF表示残基构象的变化程度, 值越大说明残基在模拟过程中的构象变化幅度越大。两个体系的残基骨架原子的RMSF结果如图 1b所示, 从图中可以看出, 二者残基整体波动趋势大致相同, 但在个别区域内RMSF呈现了不同的改变。比如在Aβ42单体的Turn区域(E22~G29), N27发生脱酰胺化修饰可以明显降低该区域残基的RMSF波动, 这可能与脱酰胺化修饰后可以抑制Turn区自身结构的转变, 进而影响该区域的残基波动有关; 由于中央疏水(central hydrophobic cluster, CHC)区域与Turn区域相邻, 与Turn区域类似, 脱酰胺化修饰也降低了CHC区域(K16~A21)的RMSF值。与以上两个区域不同, NTR区域中残基(D1~F4和S8~Q15)的RMSF值明显高于未修饰的Aβ42单体中相应的残基, 而残基R5~D7波动又低于未发生修饰的Aβ42单体中相应的残基, 这可能与NTR区域中的残基结构灵活、无序有关。另外CTR区域(I31~V39)残基的RMSF值也明显高于未修饰的Aβ42单体中相应的残基, 表明该区域内的残基波动性增强, 可能会导致该区域内二级结构发生改变。
综上所述, Aβ42第27位天冬酰胺进行脱酰胺化修饰会引起不同位置残基RMSF值的波动性发生改变, 其中, CHC区和Turn区的残基RMSF值波动性降低, CTR区(I31~V39)和NTR区(S8~Q15)残基RMSF值波动性增强, 结构变得更为柔性。
2 盐桥分析基于Aβ42原纤维结构分析发现, Turn区域中的D23和K28易形成盐桥, 而形成的盐桥能够进一步稳定两个β折叠的回旋结构[30]。因此D23和K28形成盐桥的概率可以用来评估Aβ42单体形成聚合物的趋势。研究表明, 两个带电残基形成盐桥的条件是参与形成盐桥的原子间距离小于0.46 nm[31]。因此本研究计算了在平衡时间内两个体系中D23骨架C原子与K28骨架N原子之间的距离概率, 以判断盐桥的形成情况。结果如图 2a所示, 未发生修饰的Aβ42单体结构中的D23和K28距离小于0.46 nm的概率在25%左右, 而发生脱酰胺化修饰后的D23和K28距离小于0.46 nm的概率为零。结果表明, N27发生脱酰胺化修饰后, D23和K28之间距离增加, 无法形成盐桥。
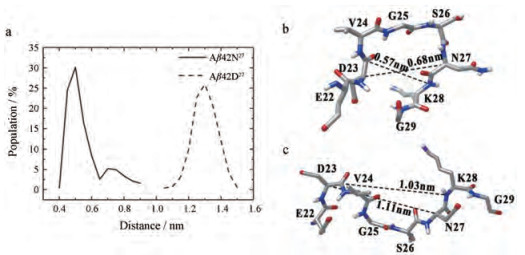
|
Figure 2 Comparison of the salt bridge formed between D23 and K28 in Aβ42N27 and Aβ42D27. a: The distance distributions between D23 and K28; b, c: The position of residues in turn region for Aβ42N27 and Aβ42D27, respectively |
Aβ42单体的K28侧链带有正电荷, D23侧链带有负电荷, N27侧链不带电荷, 但当N27侧链发生脱酰胺化修饰转变成天冬氨酸后, 则侧链带有负电荷, 与D23位带有相同的电荷, 将会发生排斥作用, 阻止K28与D23之间的盐桥形成。
为了进一步比较N27侧链发生脱酰胺化修饰之后对D23和K28周围残基结构的影响, 进行了两个模拟体系的Turn区的平均构象研究。通过图 2b、c可以发现, 未修饰时的D23骨架C原子和K28骨架N原子的平均距离是0.57 nm, 并且它们之间会形成转角结构; 当N27侧链发生脱酰胺化修饰时, D23和K28的平均距离增至1.03 nm, 原有的转角结构转变成比较舒展的无规卷曲结构, 并且D23与D27的平均距离也增至1.11 nm。另外, 转角结构的改变可能会进一步引起Aβ42整体二级结构、三级结构以及各个区域间的相互作用强度随之发生变化。
3 二级结构分析已有研究表明Aβ42的二级结构由无规卷曲或α螺旋转变成β折叠对Aβ42进行自组装后, 进一步形成各种聚合物至关重要[32, 33]。为了研究N27发生脱酰胺化修饰对于Aβ42单体形成二级结构的影响, 本文比较了两个体系在100 ns模拟时间内的二级结构的动态变化。结果如图 3所示, 在整个模拟时间内, 未发生修饰的Aβ42单体的CTR区域残基(L34~V36)和NTR区域残基(V12~H14)在10 ns左右发生了由无规卷曲转变成β折叠的结构转变, 并且存在时间高达75%以上。脱酰胺化之后的Aβ42单体的NTR区域形成β折叠结构的存在时间小于1%。CTR区域残基(I31~I32)和(M35~V36)虽然形成β折叠结构, 但存在时间只有40%左右。对比两个体系可发现, 修饰后的Aβ42在CTR与NTR区域出现β折叠结构的时间较晚, 存在时间较短, 并且参与形成β折叠的残基数量减少。在CHC区域(F19~A21), 未发生修饰的Aβ42单体在三种不同结构形式之间转变, 其中β折叠存在的时间为15%, 形成无规卷曲的时间为40%, 形成α螺旋的时间为20%。而当发生修饰之后, 该区域形成β折叠结构的时间小于0.05%, 大部分时间以形成转角结构为主(70%)。这与之前通过RMSF分析得到的修饰后CHC区域残基波动幅度变小的结果相符合。
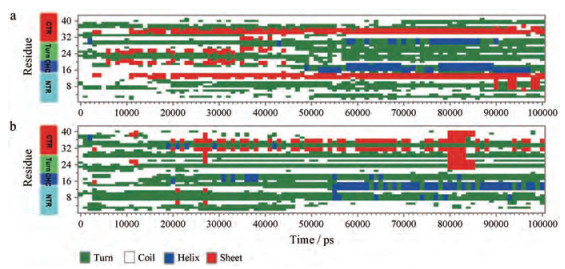
|
Figure 3 The evolution of secondary structure elements for Aβ42N27 (a) and Aβ42D27 (b) |
由于Aβ42第27位天冬酰胺位于Turn区域, 因此分析该区域的结构变化对于理解Aβ42的整体构象改变至关重要。Turn区域残基形成的二级结构形式如图 4所示, 未发生N27修饰的Aβ42单体的二级结构主要包括无规卷曲(39%)和β转角(47%), 其邻近的残基也都主要参与形成β转角(40%以上)。当发生N27脱酰胺化修饰后, Aβ42单体的D27、G25形成的二级结构均主要是无规卷曲(92%), 而其他残基还是以形成转角结构为主。可以发现, 修饰后的Aβ42的Turn区残基可以形成稳定的结构, 在不同二级结构之间相互转变的概率变小, 这也与脱酰胺修饰之后Turn区域残基RMSF波动较小相符。
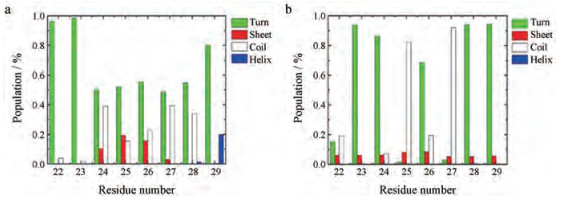
|
Figure 4 Probabilities of four secondary structures formed in residues 22–29 for Aβ42N27 (a) and Aβ42D27 (b) |
综上所述, 当27位天冬酰胺进行脱酰胺化修饰后, Aβ42单体形成的二级结构主要以无规卷曲和转角结构为主, NTR区域和CTR区域形成β折叠结构的趋势降低。
4 三级结构分析残基间的空间距离会影响其相互作用, 从而进一步影响蛋白质构象。影响相互作用的因素主要来自非邻近(序列上间隔大于3)残基之间的接触, 为此, 本文计算了在50~100 ns时间内Aβ42链内非邻近残基之间的接触。如图 5a左上三角部分显示, 没有修饰的Aβ42单体中残基A30~A42侧链与残基D1-K16侧链之间的接触比较密切, 该接触区域对应于Aβ42的CTR区域(L34~V36)与NTR区域(V12~H14)之间形成反向平行β折叠结构。而在图 5b左上三角部分显示, 在发生修饰后的Aβ42单体以上两个区域中的残基侧链之间几乎没有接触, 残基A21~I32与残基V36~A42侧链之间接触比较紧密。这与发生修饰后Aβ42单体的Turn区域形成了比较舒展延伸的结构、CTR区域中残基A30~L34形成转角结构和残基V36~A42形成与Turn区域结构相平行的结构相对应。
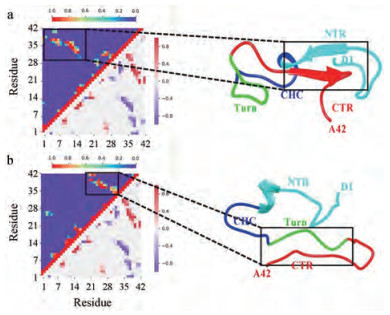
|
Figure 5 Intra chain residue contact probability matrix and the average structures of Aβ42 from the last 50 ns. The upper triangular portion in the matrix represents the normalized contacts probability for Aβ42N27 (a) and Aβ42D27 (b) and the lower triangular portion in the matrix is the probability differences between Aβ42N27 and Aβ42D27 |
通过分析Aβ42单体三级结构发现, 两个体系最主要的差异体现在CTR区域与NTR区域之间的相互接触。由于链内β折叠之间的相互作用对于Aβ42单体聚合发挥着关键作用, 因此本研究从50~100 ns模拟轨迹中选取5 000帧构象, 分别计算了两个体系CTR区域与NTR区域残基的结合能。通过计算得到未发生修饰的Aβ42单体的CTR区和NTR区之间的结合能为(-99.19 ± 8.89) kJ·mol-1; 而发生脱酰胺化修饰之后的Aβ42单体中两个区域之间的结合能下降为(-49.56 ± 3.79) kJ·mol-1。表明修饰之后Aβ42的CTR和NTR区域残基相互作用减弱, 这也与以上两个区域残基之间距离变远的结果相符合。
为了分析在以上两个区域内残基之间相互作用是以哪种作用方式为主, 本研究进一步把结合能进行了分解, 结果如表 1所示, 促进两个区域结合主要通过静电作用、范德华作用和非极性作用, 其中范德华作用起主要促进作用。而范德华作用也是引起这两个体系的两个区域结合能差异的最主要原因。
| Table 1 The calculated interaction free energy between residues at CTR and NTR regions and its individual components for Aβ42N27 and Aβ42D27. ΔEvdw: The van der Waals energy; ΔEelec: The electrostatic energy; ΔGpolar: The polar salvation energy; ΔGnonpolar: The nonpolar salvation energy; ΔGinteraction.energy: The binding free energy |
为了确定在结合过程中起重要作用的氨基酸, 本研究把结合能分解到每个残基上, 结果如图 6所示, 在未发生修饰的Aβ42单体中起主要作用的残基是V12、H13、H14、I32、L34、M35、V36、G37、V39;而修饰之后起主要作用的残基是I32和V40。可见修饰之后, NTR区域中各个残基对于与CTR区域相结合的贡献变小, 这与CTR区域之间的距离变大有关, 从而无法与CTR区域形成稳定的反向平行β折叠结构。而CTR区域中的I32和V40在与NTR区域结合中贡献较大, 与它们形成无规卷曲后, 与NTR区域中的残基距离接近有关。
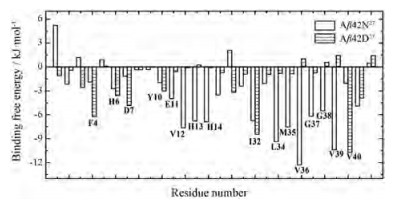
|
Figure 6 Contribution of individual residues to the interaction energy between residues at CTR and NTR regions. Key residues with great contribution are indicated by one letter code |
为了进一步分析Aβ42单体在模拟过程中的主要构象, 本文对50~100 ns轨迹中的结构进行了聚类分析。结果表明, 未发生修饰的Aβ42单体结构聚类的总数是23类, 其中前五类构象的数目占总数的95.78%;而发生修饰之后的Aβ42单体结构的类别为30类, 其中前五类构象数目占总数的91.54%。对比聚类结果可以发现, 27位天冬酰胺进行脱酰胺化修饰可以使Aβ42单体的构象发生较大变化, 这与之前RMSD结果相符。
分别提取的两个体系前五类构象在图 7中显示。通过对比未发生修饰的Aβ42单体结构, 可以得到以下结构特征: ①整体结构比较稳定, 除了第三类构象外, 其他四类构象(83.41%)中, NTR区域残基V12~H14与CTR区域残基L34~V36可以形成反向平行的β折叠结构, 并且β折叠结构区域之间易形成发夹结构; ②后四类构象中羧基末端和氨基末端残基呈现相同方向的舒展趋势, 并且它们之间的距离比较接近。
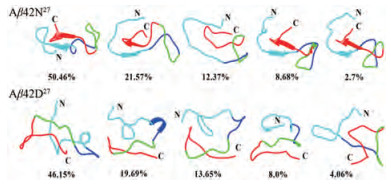
|
Figure 7 The representative structures of five most-populated conformational clusters for Aβ42N27 and Aβ42D27 are shown in cartoon representation and the corresponding percent is shown at the bottom of the structures. Different regions are represented by different color as follow: cyan, NTR region; blue, CHC region; green, Turn region; red, CTR region |
对于发生修饰的Aβ42单体结构可以得到以下特征: ①所有的构象都比较松散灵活, 含有丰富的无规卷曲结构; ②在前两类构象(65.84%)中均出现α螺旋结构, 其中在第一类构象(46.15%)中, NTR区域中残基E11~Q15形成α螺旋结构; 在第二类构象(19.69%)中, CHC区域中残基K16~F19形成α螺旋结构; ③所有构象中均未形成β折叠结构。另外, 羧基末端与氨基末端残基构象并未沿相同方向舒展, 它们之间的距离相对比较远。
讨论基础实验证实Aβ42的N27发生脱酰胺化修饰可以减慢聚合物的形成速率[15]。评估Aβ42单体形成聚合物能力的指标是其β折叠的含量, 本文通过MD模拟的方法证实了发生脱酰胺化修饰的Aβ42单体参与形成β折叠的残基数量减少, 并且在模拟过程中其结构从无规卷曲或α螺旋到β折叠的转变需要的时间较未发生修饰的Aβ42单体变长。这表明了本研究的模拟是与实验结果相符合的。
通过分析脱酰胺化修饰和非修饰Aβ42单体的结构, 发现N27脱酰胺化修饰破坏了Turn区D23和K28形成盐桥的条件, 同时阻止Turn区残基形成β转角结构。导致发生脱酰胺化修饰的Aβ42单体大部分区域形成无规卷曲, 残基RMSF值波动幅度增大, 形成的β折叠结构减少, 尤其在CTR区域和NTR区域, 两者之间无法形成稳定的反向平行β折叠结构, 显著影响了Aβ42单体之间的聚合作用。
之前研究已证明盐桥对于Aβ42聚合至关重要[34-36], 因此, 如果破坏Aβ42单体中D23与K28之间的盐桥形成条件, 就可有效抑制Aβ42单体中CTR与NTR区域形成的β折叠结构, 减弱Aβ42单体聚合能力。本研究结果显示, 当N27发生脱酰胺化修饰, 会阻碍D23与K28形成盐桥, 同时提示, 如果能发现可优先与Aβ42的Turn区残基相互作用的小分子, 将有可能达到治疗AD的作用。
| [1] |
Amariglio RE, Donohue MC, Marshall GA, et al. Tracking early decline in cognitive function in older individuals at risk for Alzheimer disease dementia:the Alzheimer's disease cooperative study cognitive function instrument[J]. JAMA Neurol, 2015, 72: 446-454. DOI:10.1001/jamaneurol.2014.3375 |
| [2] |
Dziedzic T, Pera J, Klimkowicz-Mrowiec A, et al. Biochemical and radiological markers of Alzheimer's disease progression[J]. J Alzheimers Dis, 2016, 50: 623-644. DOI:10.3233/IFS-150578 |
| [3] |
Association Alzheimer's. 2016 Alzheimer's disease facts and figures[J]. Alzheimers Dement, 2016, 12: 459-509. DOI:10.1016/j.jalz.2016.03.001 |
| [4] |
Masters CL, Simms G, Weinman NA, et al. Amyloid plaque core protein in Alzheimer disease and Down syndrome[J]. Proc Natl Acad Sci U S A, 1985, 82: 4245-4249. DOI:10.1073/pnas.82.12.4245 |
| [5] |
Hyman BT, Phelps CH, Beach TG, et al. National Institute on Aging-Alzheimer's association guidelines for the neuropathologic assessment of Alzheimer's disease[J]. Alzheimers Dement, 2012, 8: 1-13. DOI:10.1016/j.jalz.2011.10.007 |
| [6] |
Peng Y, Li YY, Li L, et al. Progress of clinical trials in Alzheimer's disease drugs[J]. Acta Pharm Sin (药学学报), 2016, 51: 1185-1195. DOI:10.16438/j.0513-4870.2015-1100 |
| [7] |
Zou Y, Qian Z, Chen Y, et al. Norepinephrine inhibits Alzheimer's amyloid-beta peptide aggregation and destabilizes amyloid-beta protofibrils:a molecular dynamics simulation study[J]. ACS Chem Neurosci, 2019, 10: 1585-1594. DOI:10.1021/acschemneuro.8b00537 |
| [8] |
Berk C, Paul G, Sabbagh M. Investigational drugs in Alzheimer's disease:current progress[J]. Expert Opin Investig Drugs, 2014, 23: 837-846. DOI:10.1517/13543784.2014.905542 |
| [9] |
Long JM, Holtzman DM. Alzheimer disease:an update on pathobiology and treatment strategies[J]. Cell, 2019, 179: 312-339. DOI:10.1016/j.cell.2019.09.001 |
| [10] |
Zhang BY, Pang XC, Jia H, et al. Repositioning drug discovery for Alzheimer's disease based on global marketed drug data[J]. Acta Pharm Sin (药学学报), 2019, 54: 1214-1224. |
| [11] |
Marcelli S, Corbo M, Iannuzzi F, et al. The involvement of post-translational modifications in Alzheimer's disease[J]. Curr Alzheimer Res, 2018, 15: 313-335. DOI:10.2174/1567205014666170505095109 |
| [12] |
Kumar S, Rezaei-Ghaleh N, Terwel D, et al. Extracellular phosphorylation of the amyloid beta-peptide promotes formation of toxic aggregates during the pathogenesis of Alzheimer's disease[J]. EMBO J, 2011, 30: 2255-2265. DOI:10.1038/emboj.2011.138 |
| [13] |
Halim A, Brinkmalm G, Rüetschi U, et al. Site-specific characterization of threonine, serine, and tyrosine glycosylations of amyloid precursor protein/amyloid beta-peptides in human cerebrospinal fluid[J]. Proc Natl Acad Sci U S A, 2011, 108: 11848-11853. DOI:10.1073/pnas.1102664108 |
| [14] |
Jawhar S, Wirths O, Bayer TA. Pyroglutamate amyloid-beta (A beta):a hatchet man in Alzheimer disease[J]. J Biol Chem, 2011, 286: 38825-38832. DOI:10.1074/jbc.R111.288308 |
| [15] |
Osaki D, Hiramatsu H. Citrullination and deamidation affect aggregation properties of amyloid beta-proteins[J]. Amyloid, 2016, 23: 234-241. DOI:10.1080/13506129.2016.1240076 |
| [16] |
Song L, Xue R, Ge P, et al. Identification of post-translational modifications of A beta peptide in platelet membranes from patients with cerebral amyloid angiopathy[J]. J Neurol Sci, 2017, 383: 11-17. DOI:10.1016/j.jns.2017.08.3269 |
| [17] |
Kumar S, Walter J. Phosphorylation of amyloid beta (A beta) peptides-a trigger for formation of toxic aggregates in Alzheimer's disease[J]. Aging (Albany NY), 2011, 3: 803-812. |
| [18] |
Kumar S, Singh S, Hinze D, et al. Phosphorylation of amyloid-beta peptide at serine 8 attenuates its clearance via insulin-degrading and angiotensin-converting enzymes[J]. J Biol Chem, 2012, 287: 8641-8651. DOI:10.1074/jbc.M111.279133 |
| [19] |
Moloney A, Sattelle DB, Lomas DA, et al. Alzheimer's disease:insights from drosophila melanogaster models[J]. Trends Biochem Sci, 2010, 35: 228-235. DOI:10.1016/j.tibs.2009.11.004 |
| [20] |
Cynis H, Frost JL, Crehan H, et al. Immunotherapy targeting pyroglutamate-3 A beta:prospects and challenges[J]. Mol Neurodegener, 2016, 11: 48. DOI:10.1186/s13024-016-0115-2 |
| [21] |
Schlenzig D, Manhart S, Cinar Y, et al. Pyroglutamate formation influences solubility and amyloidogenicity of amyloid peptides[J]. Biochemistry, 2009, 48: 7072-7078. DOI:10.1021/bi900818a |
| [22] |
Alexandru A, Jagla W, Graubner S, et al. Selective hippocampal neurodegeneration in transgenic mice expressing small amounts of truncated Abeta is induced by pyroglutamate-Abeta formation[J]. J Neurosci, 2011, 31: 12790-12801. DOI:10.1523/JNEUROSCI.1794-11.2011 |
| [23] |
Ahmed M, Davis J, Aucoin D, et al. Structural conversion of neurotoxic amyloid-beta(1-42) oligomers to fibrils[J]. Nat Struct Mol Biol, 2010, 17: 561-567. DOI:10.1038/nsmb.1799 |
| [24] |
Nguyent P, Derreumaux P. Understanding amyloid fibril nucleation and A beta oligomer/drug interactions from computer simulations[J]. Acc Chem Res, 2014, 47: 603-611. DOI:10.1021/ar4002075 |
| [25] |
Nelson R, Eisenberg D. Recent atomic models of amyloid fibril structure[J]. Curr Opin Struct Biol, 2006, 16: 260-265. DOI:10.1016/j.sbi.2006.03.007 |
| [26] |
Gremer L, Scholzel D, Schenk C, et al. Fibril structure of amyloid-beta(1-42) by cryo-electron microscopy[J]. Science, 2017, 358: 116-119. DOI:10.1126/science.aao2825 |
| [27] |
Margreitter C, Petrov D, Zagrovic B. Vienna-PTM web server:a toolkit for MD simulations of protein post-translational modifications[J]. Nucleic Acids Res, 2013, 41: 422-426. DOI:10.1093/nar/gkt416 |
| [28] |
Petrov D, Margreitter C, Grandits M, et al. A systematic framework for molecular dynamics simulations of protein post-translational modifications[J]. PLoS Comput Biol, 2013, 9: e1003154. DOI:10.1371/journal.pcbi.1003154 |
| [29] |
Reif MM, Winger M, Oostenbrink C. Testing of the GROMOS force-field parameter set 54A8:structural properties of electrolyte solutions, lipid bilayers, and proteins[J]. J Chem Theory Comput, 2013, 9: 1247-1264. DOI:10.1021/ct300874c |
| [30] |
Yang C, Zhu XL, Li JY, et al. Exploration of the mechanism for LPFFD inhibiting the formation of beta-sheet conformation of A beta(1-42) in water[J]. J Mol Model, 2010, 16: 813-821. DOI:10.1007/s00894-009-0594-y |
| [31] |
Truong PM, Viet MH, Nguyen PH, et al. Effect of Taiwan mutation (D7H) on structures of amyloid-beta peptides:replica exchange molecular dynamics study[J]. J Phys Chem B, 2014, 118: 8972-8981. DOI:10.1021/jp503652s |
| [32] |
Steckmann T, Bhandari YR, Chapagain PP, et al. Cooperative structural transitions in amyloid-like aggregation[J]. J Chem Phys, 2017, 146: 135103. DOI:10.1063/1.4979516 |
| [33] |
Chiti F, Dobson C M. Protein misfolding, amyloid formation, and human disease:a summary of progress over the last decade[J]. Annu Rev Biochem, 2017, 86: 27-68. DOI:10.1146/annurev-biochem-061516-045115 |
| [34] |
Mithu VS, Sarkar B, Bhowmik D, et al. Curcumin alters the salt bridge-containing turn region in amyloid beta(1-42) aggregates[J]. J Biol Chem, 2014, 289: 11122-11131. DOI:10.1074/jbc.M113.519447 |
| [35] |
Chandra B, Mithu VS, Bhowmik D, et al. Curcumin dictates divergent fates for the central salt bridges in amyloid-beta40 and amyloid-beta42[J]. Biophy J, 2017, 112: 1597-1608. DOI:10.1016/j.bpj.2017.02.043 |
| [36] |
Boopathi S, Kolandaivel P. Study on the inter- and intra-peptide salt-bridge mechanism of Abeta23-28 oligomer interaction with small molecules:QM/MM method[J]. Mol BioSyst, 2015, 11: 2031-2041. DOI:10.1039/C5MB00066A |
 2020, Vol. 55
2020, Vol. 55


