2. 山西大学化学化工学院, 山西 太原 030006;
3. 中国科学院过程工程研究所, 北京 100190
2. College of Chemistry and Chemical Engineering, Shanxi University, Taiyuan 030006, China;
3. Institute of Process Engineering, Chinese Academy of Sciences, Beijing 100190, China
寡糖为2~10个单糖通过α-或β-糖苷键连接组成的直链或支链天然碳水化合物, 广泛存在于动植物体内, 游离寡糖在自然界中含量相对较少, 多以混合物和糖缀合物的形式存在。糖类化合物体内合成是由多个单糖在相关酶作用下通过糖苷键脱水缩合而成, 没有系统的生物合成途径, 生成的糖链具有各种不规则分支和空间排列方式, 此外在糖基转移酶作用下还可能在蛋白质、脂质表面加上不同类型的糖分子, 形成结构各异的寡糖链, 因此糖类化合物结构复杂多样。游离寡糖、寡糖混合物和寡糖缀合物在研究中显示出多种生物活性, 如抗菌、抗病毒、抗凝血、抗氧化和抑制癌细胞转移等[1]。大量研究表明糖类化合物结构和生物活性密切相关, 由异构或手性引起的细微差异可能导致寡糖活性巨大差异[2]。目前糖类化合物构效关系研究发展缓慢, 对其结构进行快速有效解析成为当前糖生物学研究的重难点。
自然界中已发现的寡糖大部分为中性寡糖, 这类分子不带电荷呈中性, 如中性人乳寡糖、低聚果糖、低聚木糖等; 少数为酸性寡糖, 因糖链中含有酸性基团(硫酸基团或羧基等)而带大量负电荷, 如硫酸半乳聚糖、低聚半乳糖醛酸、肝素等, 中性和酸性寡糖具有较好的生物活性, 如中性人乳寡糖具有良好的抗病毒、免疫调节功效[3], Li等[4]研究发现低聚半乳糖醛酸具有抑菌、抗肿瘤及清除自由基活性; 自然界唯一的碱性寡糖为壳寡糖, 结构中因含有氨基而带正电, 可以与带负电荷有害物质结合清除毒质, 调节机体内环境; 寡糖链与蛋白质、脂质等生物大分子以共价键相连形成糖缀合物, 其中寡糖链作为撮合这些大分子的辅基, 其复杂结构可随着环境变化而变化, 并表现出强大的调节功能, 在细胞识别、黏附、传导信息等生物学功能中起到重要作用[5], 常见的糖缀合物有N-连接寡糖、O-连接寡糖和糖脂, 这些糖缀合物因寡糖链的存在而表现出广泛的生物活性, 如特定细胞表面糖肽具有的特异性结构可作为导向治疗药物的定向载体, 糖肽还具有抗感染及抗肿瘤转移活性。为了进一步揭示寡糖及糖缀合物的生物学作用规律, 了解寡糖结构以及和活性位点至关重要, 需要通过合适的方法分离纯化解析这些寡糖结构, 随着先进分析技术的发展, 快速有效分离鉴定某一寡糖组分成为可能。
1 寡糖结构寡糖的基本构成单元有葡萄糖(Glc)、阿拉伯糖(Ara)、半乳糖(Gal)、鼠李糖(Rha)、木糖(Xly)、甘露糖(Man)、岩藻糖(Fuc)和半乳糖醛酸(GalA)等, 寡糖结构中含有较多的羟基, 为亲水性物质, 常有多个同分异构体。结构多样性使寡糖有多种生物活性, 这些活性的产生可能是单一成分的寡糖的功效, 也可能是多种寡糖混合物的协同功效, 因此确定活性寡糖成分与结构成为寡糖药效研究中不可或缺的一环。寡糖结构经典表示方式为树形的分枝结构, 如图 1所示, 聚糖从还原端向非还原端延伸, 其中一种糖的异头碳与另一种糖的多个羟基之一连接形成不同分支结构。寡糖链中单糖组成、糖基构型、连接顺序和连接位点的不同, 导致树形分支结构的复杂性和寡糖结构多样[6]。
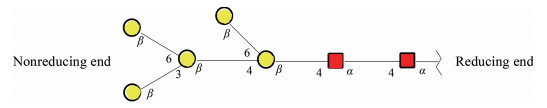
|
图 1 Oligosaccharide structure |
要完全解析清楚寡糖结构, 需要依次测定:寡糖纯度, 寡糖分子量, 组成单糖数目、种类, 单糖间连接位点与顺序, 糖苷键构型和空间构象。化学分析结合光谱学检测方法成为目前解析寡糖结构的主要手段, 但因寡糖自身微观不均一性, 存在多种异构体和衍生化产物, 导致结构异常复杂, 且糖分子缺乏生色团或荧光团, 检测方法有限, 难以快速准确解析寡糖结构。
2 寡糖提取分离技术目前获取寡糖的方法主要有3种, 第一种为从动植物中直接提取, 实验中常用寡糖提取方法为溶剂提取法, 利用相似相溶原理, 选择一定比例甲醇或乙醇溶剂反复抽提, 为保证寡糖提取率与寡糖纯度, 提取时间一般较长, 该方法适用范围广, 寡糖溶出完全, 如Zhou等[7]采用95%乙醇为溶剂提取桂花寡糖, 寡糖提取率达90.29%;此外还有超声波提取法和微波提取法, 超声波法提取寡糖利用空化作用破坏细胞, 无需加热, 寡糖提取速度快, 但需定期维护, 如Liu等[8]使用超声波辅助提取山药低聚糖得率比传统方法提高了3.91%;微波提取法利用低温穿透性加热, 获得的寡糖收率高, 提取速度快, 但成本较高, 如Wang等[9]通过微波提取大豆寡糖显著提高了寡糖含量; 第二种为先从植物中提取多糖, 后降解为目标寡糖, 这种提取方法操作简单, 适用范围广, 提取效率高, 为目前获取寡糖的主要手段, 但降解过程随机性较强, 降解产物组成及结构复杂多样, 无法精准控制降解工艺[10], 常用降解条件有酸降解法和酶降解法, 二者都能获得含量较高的目标寡糖, 本课题组用三氟乙酸将黄芪多糖降解后得到聚合度为3~11的寡糖组分[11], 模拟人工胃酸液降解黄芪多糖, 获得双糖至八糖[12], Xie等[13]研究确定了酸降解琼胶多糖和琼胶酶降解多糖制备琼胶寡糖的最佳工艺条件, 降解度均达到90%左右; 第三种为寡糖化学合成, 有机合成可精准制备不同结构寡糖, 但难度较大, 合成过程中单糖分子上的多个羟基可以与另一个单糖分子的羟基形成糖苷键, 因此寡糖合成形成过程中需要频繁地保护与脱保护, 此外合成具有生物活性的寡糖往往还得保持一定的空间结构, 这些因素大大增加了寡糖合成的难度, 目前化学合成法大致分为两类:一类是用未被保护的糖基受体与被保护的糖基供体进行反应, 另一类是用被保护的糖基受体与被保护的糖基供体进行反应, 不同的保护与脱保护策略则构成了不同合成方法, 如模块化合成和“一锅法”合成使制备结构确定且多样化的肝素寡糖成为可能[14]。
寡糖结构中含有较多的羟基, 极性较大, 且由于携带不同取代基而显示出不同的化学性质, 寡糖通常由提取到的多糖降解后得到。而多糖的获得可根据多糖性质不同选择提取方法, 中性糖提取用条件温和的水提法, 可保持中性糖的结构特征不被破坏; 酸性糖提取用酸提法, 该法不适用于中性糖, 中性糖的支链糖苷键在酸性环境中易发生断裂; 弱碱可用于提取结构稳定的糖, 碱性较高易使糖分子发生β-消除反应而结构改变, 适宜的碱性环境可以去除糖分子中碱不溶性杂质[15]。多糖降解得到多种寡糖混合物, 常用方法为通过用不同浓度的三氟乙酸(TFA)或盐酸(HCl)对多糖水解, 通过控制酸浓度、反应温度和反应时间进行可控降解, 或者通过特异性酶在一定pH、酶解温度、底物浓度下可控降解。根据寡糖性质选择合适的分离方法, 可分离纯化得到纯度较高的寡糖单体。寡糖混合物分离主要存在以下问题: ①寡糖分支多、异构体多, 结构相似的寡糖难以分离; ②寡糖结构缺乏生色团或荧光团, 难以被检测, 未衍生寡糖只能用蒸发光检测器或灵敏度较低的示差检测器检测; ③从生物中提取的寡糖缀合物含量一般较少, 分离从这些糖缀合物中释放的寡糖链, 往往需要较高灵敏度检测, 通常需要结合柱前衍生化方法以提高检测灵敏度[16]。目前常用寡糖分离纯化方法有两种, 一种为使用合适填料柱色谱分离, 另一种为将寡糖衍生化后通过液相色谱分离制备。
2.1 柱色谱分离色谱法是分离纯化寡糖传统方法, 常见有凝胶柱色谱、纤维素柱色谱等。凝胶柱色谱根据寡糖分子量大小分离寡糖, 洗脱液通常为水, 操作简单。凝胶填料有葡聚糖凝胶和聚丙烯酰胺凝胶, 葡聚糖凝胶有不同粒度, 超细级葡聚糖凝胶用于高分辨分离, 粗级和中级凝胶用于制备型分离, 但葡聚糖凝胶易被污染, 分辨率高的细颗粒流速较慢, 难以进行快速高效分离; 聚丙烯酰胺凝胶是丙烯酰胺与甲叉双丙烯酰胺交联而成, 聚丙烯酰胺凝胶亲水性好, 基本不带电荷, 吸附效应较小, 洗脱液pH范围在1~10, 选择不同孔径聚丙烯酰胺凝胶, 可较好分离聚合度25以内的各种寡糖, 如Peng等[17]利用聚丙烯酰胺凝胶柱分离制备纤维低聚糖得到纯度为98.45%、98.65%和98.12%的纤维五糖、四糖和三糖组分。纤维素柱色谱可用于分离酸性寡糖, 中性寡糖, 甲基化寡糖, 洗脱液易通过, 洗脱速度较快, 聚合度小的寡糖先流出, 聚合度大的后流出, 应用范围较广, 如用水、乙醇-水等分离中性寡糖, 正丁醇-乙酸-水等分离酸性寡糖[18]。Akpinar等[19]用纤维素柱色谱经乙醇-水洗脱, 成功分离纤维二糖到纤维五糖混合物, 在分离过程中聚合度高纤维寡糖溶解度较低且保留时间较长。
2.2 高效液相色谱分离(HPLC)高效液相色谱法常用于不同寡糖组分的分离分析, 常用的色谱柱为:亲水作用色谱柱(HILIC)、反相C18柱、高效离子交换色谱柱、凝胶柱等。分离纯化时根据纯化条件选择相应填料可保证寡糖分离效果。未衍生化寡糖混合物的分离分析可使用HPLC-RID法, 其优点为稳定性好, 操作方便, 但只能进行等度洗脱, 对寡糖混合物分离效果较差; HILIC-HPLC-ELSD法广泛用于糖类化合物分离检测, 其灵敏度比示差检测器高, 基线稳定, 适合梯度洗脱, 但要求流动相及缓冲盐必须具有挥发性, 且样品在检测器中无法保留, 操作复杂, 价格昂贵; 通过柱前衍生化方法, 使糖链带上紫外或荧光基团, 可通过HPLC-UV法分离分析寡糖, 大大提高检测灵敏度和分离效率, 但分离纯化后的衍生物通常需经过还原处理获得寡糖单体, 样品损耗较大, 常见柱前衍生化方法有: ①糖链还原端衍生化, 通过还原胺化法在糖链还原端引入发色基团, 常用衍生化试剂有: 1-苯基-3-甲基-5-吡唑酮(PMP), 对氨基苯甲酸乙酯(ABEE), 2-氨基吡啶(AP), 3-氨基-9-乙基咔唑(AEC)等, 这种衍生化方法仅作用于糖链还原端, 适用于大部分糖链, 应用广泛; ②糖链非还原端衍生化, 氨基糖可利用其具有胺的性质进行氨基衍生化, 寡糖上的羟基也可衍生化带上苯甲酰基, 酸性糖中羧基通过与对氨基苯磺酸(SA)或7-氨基萘-1, 3-二磺酸(ANDSA)中的氨基形成酰胺键, 上述方法均可在不同程度上提高寡糖检测分离效率[16], Wang等[20]用PMP为衍生化试剂对寡糖链进行标记, 聚合度为1~16的葡聚糖得到很好分离, 并用MALDI-TOF-MS质谱分析了它们的结构。
2.3 高效液相阴离子交换色谱分离(HPAEC)HPAEC利用糖链上的羟基在碱性条件下可生成氧负离子, 不同糖分子中羟基的电离常数有细微的差别, 这种差别使得氧负离子与阴离子交换树脂相互作用产生差异并表现在不同的洗脱位置, 从而达到分离目的, 通常洗脱液为强碱(NaOH), 洗脱体积较大, 这使HPAEC应用受到一定限制, 强碱性洗脱液要求分离与检测系统具有耐碱性, 脉冲电流检测技术(PAD)所用金电极是糖类分析的理想电极, 检测灵敏度可达1×10-12 mol·L-1, 且HPAEC-PAD分离检测寡糖不需要衍生化, 目前应用最为广泛[21]。强碱容易使糖发生异构化和降解, 可通过缩短分离时间减少影响, 如能在足够短时间内分离还原性单糖和寡糖, 异构化和降解可以忽略不计。Ding等[22]使用HPAEC-PAD分离巴戟天系列寡糖得到不同寡糖专属性较强的特有峰, 且在短时间内使各个寡糖达到较好的分离度。但HPAEC分离得到的样品需经过脱盐分离步骤, 才能进行质谱分析。
2.4 多孔石墨化碳色谱分离(PGC)石墨化碳柱分离原理为根据分子空间结构和分子极性分离, 分子结构越平和石墨化碳结合越紧密, 保留更强, 带有羟基、羧基、氨基的大极性寡糖分子可引起石墨化碳表面极化, 寡糖分子和石墨化碳之间作用加强, 这种作用可以很好保留寡糖分子, 提高分离效率, 还可以很好地分离复杂寡糖异构体。PGC柱容量大, 适合于寡糖的制备性操作, PGC柱分离不需要对寡糖分子进行柱前衍生化, 可以很好地分离非衍生化的寡糖和带有少量氨基酸残基的糖肽[23]。Lang等[24]采用PGC联合离线ESI-MSn技术对唾液酸化人乳寡糖混合物进行了分离分析, 得到不同分子量的单体寡糖, 且较好地分离了两个单唾液酸化二糖异构体和两个单唾液酸化四糖异构体。
2.5 高效毛细管电泳(HPCE)毛细管电泳利用电介质中带电粒子在电场中的迁移作用分离纯化化学组分, 高效毛细管电泳分离能力强、灵敏度高、快速简便、样品需量少。大多数寡糖为中性糖, 不带电荷, 极性很大而且没有发色基团或荧光基团, HPCE分离分析这些寡糖时常需衍生化或形成络合物使之带上发色基团、荧光基团或电荷。常用紫外衍生试剂有: α-萘胺、对肼基苯磺酸、1-苯基-3-甲基-5-吡唑酮(PMP)等, 既有紫外吸收又是荧光衍生剂: 2-氨基吡啶、6-氨基喹啉、8-氨基萘基-1, 3, 6-三磺酸(ANTS)等, Sun等[25]用ANTS还原氨化κ-卡拉胶寡糖, 使其带上紫外吸收基团, 高效分离了不同聚合度的κ-卡拉胶寡糖。
3 寡糖结构分析寡糖结构鉴定的传统方法为, 通过亲水色谱柱等方法分离后的寡糖单体经示差折光检测、蒸发光检测达到一定纯度后, 采用质谱法获得分子离子峰确定寡糖分子量, 然后通过制备部分甲基化乙酰化样品(PMAA), 进行GC-MS分析确定单糖连接位点, 推测出糖链中各单糖的数目、分支位点和末端糖种类, 最后通过13C NMR归属碳信号, 确定产生苷化位移的碳, 13H NMR结合2D NMR确定糖苷键构型、糖链构象、连接顺序、结构特征等, 综合以上分析结果得到寡糖结构。核磁共振对仪器精密度和图谱解析能力要求较高, 传统方法通常较耗时且需要样品量较大。质谱分析也是分析寡糖结构的一种手段, 具有高灵敏度, 耐受混合物的特点, 质谱图中结构信息直观、需要的样品量较少, 且对样品纯度要求低, 此外还可大大减少分析时间及工作量, 质谱分析法是目前分析糖类结构的有效手段。质谱法表征糖类结构依赖于从质谱中获得全部结构细节, 随着分析技术不断发展和改进, 分离分析寡糖时选择合适的寡糖分离方法、电离方法、离子激活方法、串联质谱和数据收集处理软件, 可对寡糖结构进行精确解析。
寡糖通过串联质谱解析方法大致流程为:首先进行寡糖样品分离制备纯化, 然后选择合适的电离方式, 电离产生形成母离子, 选择合适的离子碎裂方式将寡糖母离子碎裂成碎片离子, 收集并统计碎片离子对应的质荷比信息和数量信息, 得到质谱图, 最后通过对质谱数据的分析, 得到寡糖结构详细信息。
3.1 寡糖质谱电离方式寡糖电离方法有基质辅助激光解吸电离(MALDI)、电喷雾电离(ESI)和快速原子轰击电离(FAB)。其中FAB常用于酸性寡糖的电离[26], 但因FAB测量中性与碱性寡糖灵敏度较低, 样品常需要衍生化提高灵敏度, 也不能区分质量相同的单糖和连接方式, 且测定寡糖混合物时存在压制信号等缺点, 在测定寡糖结构方面逐渐被更好的电离方式所代替[27]。目前最广泛使用的寡糖电离方法是基质辅助激光解吸电离(MALDI)和电喷雾电离(ESI), 与快速原子轰击电离(FAB)相比, 它们电离能量小, 电离过程中产生较少的碎裂, 电离后主要产生分子离子, 可提供分子量信息。不同电离方式都有正负离子模式两种, 含有酸性基团(磺酸基或磷酸基)的寡糖用负离子模式分析效果较好; 含有碱性基团(氨基)的寡糖用正离子模式分析效果好; 正负离子两种电离模式都适用于天然中性寡糖电离。寡糖电离前大多数情况下需要全甲基化或全乙酰化以降低其极性来改善电离效果, 且衍生化寡糖与未衍生化寡糖的裂解模式不同。
3.1.1 电喷雾电离(ESI)ESI是目前分析寡糖结构常用方法之一, 是一种将样品溶液以喷雾形式在高电场中电离产生气相离子的电离方法, 电离后的质谱图通常会出现[M+nH]n+或[M-nH]n-峰, 这使得ESI-MS可测定的分子量范围大大增加, 目前可测定聚合度达25的聚糖, 但由于电离能量低, 灵敏度随糖分子量增加而降低。酸性寡糖常用负离子模式电离产生[M-nH]n-峰, 中性寡糖一般产生[M+nH]n+或[M+Na]+峰, ESI-MS可得到寡糖的单糖组成、连接方式及分支和聚合度等信息[28]。研究表明ESI电离灵敏度与样品溶液的表面活性有关, 寡糖溶液亲水性限制了喷雾液滴表面活性, 可通过减小液滴大小或衍生化降低寡糖溶液亲水性来提高液滴表面活性, 提高电离效率[29], 如传统ESI接口处液滴通常较大, 对未衍生化寡糖的结构分析效果较差, 采用静态或流动纳米电喷雾可减小液滴尺寸, 灵敏度显著增加。样品的酸碱度和盐的存在对分子离子的形成有重要影响, 如ESI-MS分析前于样品中加入NaAc或NH4Ac, 可获得满意的正离子图谱, 待测样品中含有高浓度盐时, ESI-MS灵敏度将显著降低[30]。ESI-MS可分析含有羧基或硫酸根的寡糖链, 全甲基化的酸性寡糖能在正离子模式下形成丰度较高的金属加合离子, 用以准确判断寡糖分子量。ESI电离不同异构体寡糖产生的母离子进一步裂解后产生不同的碎片离子, 可以此来区分寡糖异构体[27]。ESI-MS可检测区分非衍生化的寡糖O-聚糖和N-聚糖, 并确定其分子量、单糖组成及聚合度及组成[31]。ESI可与在线分离技术, 如高效液相色谱(HPLC)或毛细管电泳(CE)结合分析寡糖混合物, 中性和酸性寡糖的衍生化后有利于液相分离并可提高检测灵敏度, 如Walker等[32]用疏水性试剂将N-聚糖衍生化形成腙, 提高了N-聚糖混合物的色谱分离效率, 增加了ESI电离检测灵敏度, Guo等[33]用3-氨基-9-乙基咔唑(AEC)衍生壳寡糖混合物, 可以检测到聚合度为1-8的寡糖, 未衍生化寡糖混合物只能检测到聚合度2~6的寡糖。
3.1.2 基质辅助激光解吸电离(MALDI)MALDI是指用脉冲式激光照射样品与基质形成的共晶体薄膜, 基质吸收激光能量及解吸样品, 气化并电离得到质谱图。MALDI电离对糖类化合物有更高的灵敏度, 电离质量范围广(1×102~1×106 Da), 杂质干扰小。MALDI质谱图比ESI质谱图简单, 因为在正负两种电离模式下, 大多数离子可通过质子化或去质子化形成带正电或负电的单电荷离子, 与碱金属或碱土金属形成加合物也可以形成单电荷离子。MALDI电离对寡糖结构分析主要是进行多级质谱, 可根据碎片离子峰的类型和强度, 精确测定混合物中寡糖的详细结构, 区分同分异构糖苷键。与ESI不同, MALDI电离效率随着分子量的增加而保持恒定, MALDI电离产生的分子离子稳定不易裂解, 电离糖缀合物主要产生[M+H]+峰及少量多电荷离子峰, 电离中性寡糖只产生[M+Na]+离子峰, 因此对中性寡糖分析优于ESI, 特别是对于从糖蛋白释放的中性寡糖混合物[34]。MALDI比ESI电离能量高, 可导致不稳定基团(如磺酸基、磷酸基、硅酸基)碎裂产生相应碎片离子, 使图谱分析变得复杂, 完全甲基化能使寡糖和糖肽中的不稳定基团变得稳定, 减少不稳定基团碎片离子干扰。MALDI与在线分离技术结合较为困难, 通常将寡糖离线分离后通过MALDI分析, 寡糖样品加入含有阴离子的盐(如NO3-和Cl-)形成配合物, 可以改善中性寡糖在负离子模式中的电离, 在串联质谱产生具有结构特征碎片离子[35, 36]。基质的选择对MALDI产生的离子类型有较大影响, 如阴离子掺杂的液体基质可提高N-聚糖结构解析时的灵敏度[37]。
3.2 质谱中寡糖裂解方式寡糖电离后的分子离子进一步裂解, 可得到与寡糖精细结构相关的碎片离子, 碎裂方式及命名规则[38]如图 2所示, 含有非还原末端的片段离子记为Ai, Bi, Ci, 含有糖苷配基(苷元)或还原糖单元的还原末端离子记为Xj, Yj, Zj, 下标表示相对于末端的位置。A和X表示跨环断裂离子, 非还原端第一个糖环跨环断裂后, 非还原端到断裂部位的碎片离子为A1, 依次为A2到Ai, 还原端第一个糖环跨环断裂后, 还原端到断裂部位的碎片离子为X0, 依次为X1到Xj; 同理, 糖环糖苷键的O左键断裂, 非还原端到断裂部位的碎片离子依次记为B1, B2至Bi, 还原端到断裂部位的碎片离子记为Y0, Y1至Yj; 糖环糖苷键的O右键断裂, 非还原端到断裂部位的碎片离子依次记为C1, C2至Ci, 还原端到断裂部位的碎片离子记为Z0, Z1至Zj; 糖环中O和糖环C1位相连的键记为0, 顺时针依次为1到5, 例如跨环断裂位置为第一个糖环的0键和2键, 记为0, 2A1; 支链碎片记为α、β、λ, α为最大支链, 如B1α表示糖链中最大支链从非还原端开始第一个糖苷键O左键断裂后所形成的非还原端寡糖链离子碎片。其中Bi、Yj和Ci、Zj糖苷键断裂碎片可提供寡糖序列和分支信息, Ai、Xj跨环断裂碎片可提供糖苷键类型和连接方式信息, 遵循上述裂解规律, 可大大简便寡糖序列分析过程。
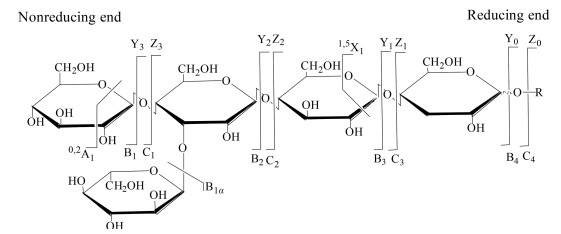
|
图 2 Nomenclature of oligosaccharides fragments generated by tandem MS |
普通一级质谱只能得到的分子量等少量结构信息, 一级质谱根据组分质量分离不同混合物, 选择分子离子在串联质谱中进一步激活裂解, 形成离子碎片质谱图, 根据碎片离子可推断寡糖精确的结构信息, 目前ESI-MSn和MALDI-MSn是分析糖类结构主要方法。影响质谱图因素主要有寡糖衍生物类型和母离子的类型, 分析结构时可选择寡糖天然结构、全甲基化寡糖和寡糖还原端衍生物, 电离产生母离子有质子化离子[M+nH]n+、去质子化离子[M-nH]n-和金属加合离子[M+Na]+ (电荷由质子携带表示为[M(X)+H]+, 电子由金属阳离子携带表示为[M+mX]m+)[39]。串联质量分析器可选择特定母离子进行分析, 因此可直接对未分离纯化的寡糖混合物进行分析。母离子类型有多种, 天然寡糖结构可电离形成母离子[M+nH]n+、[M-nH]n-和[M+Na]+; 全甲基化寡糖和寡糖乙酰化衍生物电离形成的母离子有, MALDI电离产生的[M+H]+、[M+Na]+, 或ESI电离产生的多电荷离子[40]。根据寡糖分子离子活化裂解的子离子碎片, 可获得寡糖序列、单糖组成、连接方式及结构修饰等信息, 而且串联质谱可以区分结构相近的同分异构体[41]。
已应用于寡糖结构分析的两大类裂解分子离子方法[2]:第一种是能量激活法, 主要有低能碰撞诱导解离(CID)和红外多光子解离(IRMPD), 它们将母离子裂解后主要产生糖苷键断裂碎片离子, 其中低能CID可提供较多碎片离子, 目前应用最广泛, CID和IRMPD在解离时可破坏糖链中的不稳定键, 对糖肽和含不稳定修饰基团的寡糖分析时, 得到的谱图复杂, 难以解析, 通过改进ESI喷雾溶液、甲基化、乙酰化、衍生化以及将不稳定基团与金属结合等方法提高寡糖稳定性可改善这一状况。此外还有高能CID和真空紫外光解离(VUVPD), 它们裂解母离子主要发生跨环裂解, 但目前应用尚不广泛。金属加合物母离子经CID裂解不会重排, 可产生较多糖苷键断裂离子和少量跨环断裂离子, 能够为结构鉴定提供更详细的信息[42]; 未衍生化的中性寡糖及糖脂类化合物(神经节苷脂)电离后, 带正电和带负电的母离子经CID裂解后都能得到大量的结构信息碎片, 并互相补充[43]。负离子模式下电离的酸性寡糖母离子经CID裂解后也可产生大量结构信息碎片, 如糖胺聚糖和含唾液酸的酸性寡糖[44]; 带正电母离子经CID裂解, 糖苷键容易断裂并引起残基重排导致结构序列错乱, 如天然N-聚糖电离产生带正电分子离子经CID裂解后, 发生重排导致子离子碎片错乱。
第二种是通过电子转移, 将电子转入多电荷分子离子或从多电荷分子离子转出电子, 产生自由基离子, 然后裂解成碎片离子, 这种方法不仅可以得到寡糖的详细结构信息, 还可产生保留不稳定修饰基团的寡糖骨架碎片。目前主要有电子捕获解离(ECD)、电子转移解离(ETD)、电子分离解离(EDD)、负电子转移解离(NETD)和负电子捕获解离(niECD)。ECD和ETD适用于解离多电荷正离子, 跨环裂解是ECD中的主要裂解途径, 常用于中性寡糖结构表征, 并且可根据糖肽产生的碎片来判断糖基化位点, 并保留糖链主链[45]; EDD和NETD适用于解离多电荷负离子, 如酸性寡糖和糖肽[46]; niECD适用于解离带负电寡糖离子, 解离过程中可产生骨架断裂, 如糖胺聚糖[47]。
3.3.1 质子化母离子裂解寡糖缀合物或中性寡糖分子经电离后加合一个或多个质子形成质子化母离子。大小和支链不同寡糖的质子化母离子经CID裂解后, 产生的碎片离子不同, 可用于区分寡糖链中细微的结构差异, 例如唾液酸糖缀合物(N-聚糖)的母离子[M+H]+, 经CID活化后产生丰富的Bi和Yj离子片段, 这些信息可确定寡糖序列[48], 中性糖缀合物(神经节苷脂)的母离子[M+H]+, 经CID活化后产生丰富的Bi和Yj离子片段, 且得到离子丰度较低的跨环碎裂离子峰[49]。CID活化小分子量寡糖母离子时, 糖苷键断裂和跨环断裂均发生, 可以获得更精细的结构信息, 而大分子量寡糖质子化母离子活化后大部分为糖苷键断裂, 所得到的支化结构信息较少。衍生化寡糖乙酰己糖胺(HexNAc)经ESI电离产生质子化母离子, 经CID裂解后还原端断裂产生一系列子离子, 可用于区分N-连接寡糖的类别, 如对α-2, 3-和α-2, 6-连接的唾液酸寡糖异构体的区分[50]。对于中性寡糖, 可通过改变分离条件来控制不同前体离子的产生, 如在低浓度乙酸钠存在下电离寡糖产生加钠前体离子, 在乙酸铵存在下电离寡糖产生容易裂解为质子化离子的氨化前体离子, 相比而言, 在高能CID裂解条件下, 质子化母离子能比加钠母离子产生更多的糖苷键碎片离子, 提供更丰富的结构信息[51]。
3.3.2 去质子化母离子碎裂在ESI-MS负离子模式下电离寡糖生成去质子化母离子, 经CID裂解产生丰富的糖苷键裂解离子和跨环裂解离子, 已被广泛应用于中性或酸性寡糖结构分析。天然寡糖母离子[M-H]-经CID活化后, 游离羟基上的活泼氢存在, 有利于糖苷键裂解, 抑制跨环裂解, 因此全甲基化寡糖质谱图中观察到的某些跨环裂解离子信息在天然寡糖质谱图中不存在[52]。对比去质子化和质子化母离子的子离子, 可以发现质子转移在寡糖碎裂模式中具有重要作用, 如中性寡糖麦芽七糖(一种线性多糖)母离子[M+H]-经CID裂解后, 主要产生糖苷键碎裂离子, 而其去质子化母离子[M-H]-裂解后仅产生跨环碎裂2, 4Am离子[53]; 一种有支链的中性壳寡糖母离子[M+H]-经裂解后, 主要产生糖苷键碎裂离子, 而其去质子化母离子[M-H]-主要产生2, 4A3离子碎片, 和少量糖苷键碎裂离子[39]。
3.3.3 碱金属和碱土金属加合母离子裂解天然寡糖钠离子加合物通过CID能量活化, 然后发生离子碎裂, 这种碎裂模式与形成质子化母离子后的碎裂模式不同, 如图 3所示[39], 质子化离子的碎裂机制是电荷诱导裂解使糖苷键更易发生断裂, 而对于金属加合离子碎裂机制可能是电荷远程影响的, 电荷远程影响比电荷诱导的寡糖母离子结构稳定, 经CID裂解后, 金属加合离子有3种可能的碎裂途径[54], 第一种为失去金属阳离子碎裂路径, 得到寡糖中性碎片, 无详细的结构信息碎片, 第二种为糖苷键裂解路径, 碱金属阳离子加合的寡糖主要为糖苷键断裂; 第三种为跨环裂解途径, 碱金属阳离子加合的寡糖主要为跨环断裂。若金属离子与糖苷键氧原子缔合, 主要发生糖苷键断裂, 裂解所需能量低, 若金属离子与糖环上多个氧原子缔合, 母离子糖苷键结构变得稳定, 更易发生跨环断裂, 所需裂解能量也越高, 并且金属阳离子越大这种远程影响越强结构越稳定[55, 56], 如低能量CID分析二糖的单锂离子加合物, 显示出丰富的糖苷键裂解碎片离子, 但不能区分寡糖连接异构体, 高能量CID分析二糖的双锂离子加合物, 产生的跨环断裂离子碎片信息可容易地区分连接异构体[57]。

|
图 3 Decomposition of protonated ions (A) and metal-cationized ions (B) |
寡糖碱金属加合离子裂解与分支度相关, 寡糖分支度低碱金属加合离子更稳定, 跨环断裂离子碎片较多, 分支度高糖苷键不稳定, 主要发生糖苷键断裂, 如ESI-Q-TOF-MS分析质子化和钠加合的N-连接寡糖母离子, 后者产生更多结构信息离子碎片, 包含还原末端乙酰葡糖胺(GlcNAc)残基跨环断裂碎片0, 2A和2, 4A片段峰和低丰度的支链上糖残基峰, 而且随着支链上HexNAc残基数量的增加, 跨环断裂离子峰减少[58]。
对于分子量较大的寡糖(三糖以上), 形成金属阳离子加合物所用到的金属阳离子数量更多或者携带电荷数越多, 寡糖糖苷键结构更稳定, 加合离子相对丰度较高, 产生更多的跨环断裂碎片[59], 如碱土金属Mg2+、Ca2+等, 这是由于二价碱土金属离子带电荷能力强, 增强了与寡糖结合能力, 对于这些大分子量寡糖, 一价碱金属离子寡糖加合物碎裂方式仅发生糖苷键裂解。
3.3.4 全甲基化和全乙酰化寡糖的多级质谱研究表明, 无论使用何种电离技术, 通过衍生化降低寡糖极性的方法都会提高质谱图中相关结构离子的信号强度。两种最常见的衍生化方法为寡糖的全甲基化和全乙酰化, 全甲基化通常优于全乙酰化, 因为它有增加较小质量的同时增加较大挥发性的优势, 分析糖肽中的寡糖成分时全甲基化法是首选方法, 如有游离氨基的己糖胺寡糖, 全乙酰化后, 与肽结合的糖残基和乙酰化糖残基难以区分, 而全甲基化后, 含游离氨基的糖残基形成季铵阳离子与N-乙酰基己糖胺残基的质量不同, 并且还产生独特的CID碎片离子。寡糖甲基化后极性降低, 容易分离得到纯度很高的甲基化样品, 降低杂质干扰信号。寡糖甲基化后样品与碱金属离子的亲和力增加, 离子加合物产率增加, 且碱金属离子加合物能够产生相对稳定的碎片离子, 增加质谱检测的灵敏度[27]。全甲基化寡糖和全乙酰化寡糖的[M+H]+母离子很少发生跨环裂解, 因此通过[M+H]+母离子裂解后得到碎片离子峰可区分线性和支链寡糖[60]。
烷基化寡糖金属加合母离子经CID活化, 全烷基化寡糖糖苷键断裂能力随烷基基团增大而增强, 即全甲基化寡糖母离子活化后糖苷键较难断裂, 而全戊基化寡糖母离子活化后糖苷键容易断裂, 原因是较小的烷基可在糖苷键自由旋转, 缓解了寡糖糖苷键结构的空间扭转, 从而使其相对稳定, 较大的烷基不可旋转增加其空间扭转, 糖苷键容易断裂[61]。全烷基化(1-4)-连接寡糖的不同相对构型产生质子化母离子后, CID活化裂解得到的子离子量不同, 这可用于区分α、β构型, 寡糖烷基化后, β-异构体的子离子量高于α-异构体, 原因是β-糖苷键空间结构比α-糖苷键拥挤, 当寡糖戊基化时, 子离子数量差距最明显, 这反映了寡糖构象对寡糖裂解方式的影响, 但由于这种高级构象结构的灵活性, (1-6)-连接寡糖的全烷基化后得到的质谱图无法区分α-和β-糖苷键。
3.3.5 紫外和荧光试剂衍生寡糖的多级质谱带有紫外吸收或者荧光吸收的化合物也通常用于寡糖衍生, 这些衍生化试剂一般带有苯环, 衍生化后糖链极性降低, 可显著改善寡糖混合物的分离与检测, 未知寡糖衍生化后, 通过衍生化寡糖多级质谱数据与标准品寡糖质谱数据比较, 可以快速可靠地解析未知寡糖的结构。常见衍生化类型有还原端衍生化法和非还原端衍生化法, 糖链还原端衍生化试剂主要为带有胺基或氨基的芳香族化合物, 如2-氨基吡啶(2-AP)还原胺化中性糖和氨基糖, 可分离得到较纯的寡糖避免杂质干扰, 增强质谱检测的灵敏度; 4-氨基苯甲酸乙酯(ABEE)可对所有类型糖链衍生化[16], 相比于其他衍生剂, 在ESI和MALDI质谱中均有较高的灵敏度; 荧光试剂2-氨基吖啶(2-AMAC)用于单糖、中性寡糖衍生, 在ESI和MALDI质谱中灵敏度高[62]; 3-乙酰氨基-6-氨基吖啶(AA-AC)用于N-连接糖链衍生在MALDI质谱中灵敏度高[63]; 1-苯基-3-甲基-5-吡唑啉酮(PMP)衍生化寡糖操作简单, 在MALDI-TOF中可明显提高寡糖检测灵敏度; 碳酸氢胺结合酰氯化合物(FMOC-Cl)衍生糖链还原端分离分析后可在酸性条件下回收得到还原性糖; 肼和糖链还原端醛基生成腙可提高寡糖分离和检测灵敏度; 此外氨基糖可根据糖链上胺的性质衍生化, 酸性糖可根据羧基进行衍生化, 如唾液酸化寡糖、神经节苷脂、糖胺聚糖的衍生化。不同衍生化试剂都有它的优点和不足, 选择衍生方式取决于糖的类型、样品复杂性、样品量大小等, 如二糖的不同衍生化产物在多级质谱中糖苷键断裂主要产生Y型及Z型离子, 单糖组成和异头碳的构象不同的二糖, 糖苷键断裂产生的离子类型不同[27]。
尽管衍生化具有优势, 但常规衍生化通常需要苛刻的条件(强碱)和较长的反应时间, 大多数衍生化方法需要清理方法去除非糖化学物质, 从而导致样品损失。因此衍生化方法也在不断改进, 以满足低样本损失和高效率的需求[64]。如通过MALDI-TOF MS对聚糖的唾液酸残基进行固相甲基酯化以进行快速定量的糖谱分析, 可能适用于大规模糖组学研究[65]; Kang等[66]使用固相微型化方法, 在不到一分钟的时间内完成寡糖的定量全甲基化。
3.4 质谱图解析数据分析是解析结构中最重要的一环, 现阶段解析质谱图主要手段为通过计算机软件解析[6], 本文总结了几种有效的糖结构质谱数据分析软件, 第一种为利用寡糖结构质谱信息库, 输入待测寡糖质谱信息搜索得到对应寡糖结构, 如FragLib数据库包含丰富寡糖质谱信息, 且可区分同分异构体, Kameyama's Method软件基于三级质谱数据, 可得到准确的寡糖结构, B2离子库在已知分支情况下解析寡糖的连接位点, 但无法确定结构, GlycosidIQ是糖类二级质谱数据库, 可根据糖苷键裂解碎片离子, 得出寡糖结构, GlycoFragment软件可得到寡糖结构所有离子碎片质量, 并对离子峰进行解析, GlycoSearchMS软件可根据离子碎片找到对应寡糖质谱图和对应结构, GlycanMass可得到寡糖离子碎片质量[67]; 第二种为通过谱峰间m/z差值, 推断寡糖组成单糖的种类与结构特点, 将单糖模拟组装形成理论寡糖结构, 如GlycoMod根据离子碎片推单糖组成, 该程序还可以匹配SWISS-PROT或TrEMBL数据库中的候选寡糖[68]; STAT软件可快速解析聚合度10以内的寡糖, GLYCH数据库主要分析二级质谱信息, 解析直链型、分枝型寡糖和O-连接寡糖结构, Cartoonist Two软件可分析寡糖的单糖组成和寡糖结构, OSCAR基于多级质谱解析聚合度20以内寡糖结构。此外UniCarbKB是用于糖组学研究的在线信息存储和搜索平台[69], 包含聚糖的结构、生物学起源和潜在功能的信息, 并将每个存储聚糖结构与质谱, 色谱法和其他技术的高质量实验数据联系起来; EUROCarbDB基于高效液相色谱、质谱和核磁共振实验的分析数据[70], 提供了各种基于网络的研究工具, 可加快聚糖分析和结构解析速度, 并推荐用于编码糖类结构的格式和术语; GlycReSoft通过对候选聚糖成分进行分组来进行识别[71], 这些候选糖成分汇总了其离子碎片, 加合物和洗脱时间等信息, 可以有效地进行糖谱分析。
在寡糖结构解析过程中往往是多种技术联合使用, 多个质谱解析软件结合使用获得寡糖基本结构, 佐以13C NMR、1H NMR图谱解析结果, 并结合寡糖三维模型数据库, 所有结构信息整合起来, 获得精确的寡糖结构。
4 前景与展望质谱技术目前是寡糖结构解析的主要方法, 通过分离技术与质谱技术的结合, 可以实现对寡糖结构组成进行准确解析, 质谱中的电离活化方法对结构解析具有重要影响, 更高维度串联质谱可以提供更多的寡糖结构细节, 寡糖结构解析专用软件的开发是处理和解释质谱数据的必需工具。总之, 近年来质谱技术的进步促进了寡糖结构的精准分析, 随着更高分辨率和更灵敏的仪器广泛使用, 使复杂寡糖混合物和糖缀合物的结构分析成为可能, 同时寡糖结构上的细微差异以及修饰基团的不稳定性对寡糖分析方法的严格需求, 成为质谱技术进一步发展的动力。
| [1] |
Zhao C, Wu YJ, Liu XY, et al. Functional properties, structural studies and chemo-enzymatic synthesis of oligosaccharides[J]. Trends Food Sci Technol, 2017, 66: 135-145. DOI:10.1016/j.tifs.2017.06.008 |
| [2] |
Kailemia MK, Ruhaak LR, Lebrilla CB, et al. Oligosaccharide analysis by mass spectrometry:a review of recent developments[J]. Anal Chem, 2014, 86: 196-212. DOI:10.1021/ac403969n |
| [3] |
Bode L, Kuhn L, Kim HY, et al. Human milk oligosaccharide concentration and risk of postnatal transmission of HIV through breastfeeding[J]. Am J Clin Nutr, 2012, 96: 831-839. DOI:10.3945/ajcn.112.039503 |
| [4] |
Li JJ. The Study on the Controlled Degradation of Citrus Pectie Polysaeeharide and the Purification and Activity of Oligogalacturonic Acid Fragments (柑橘果胶多糖可控性降解和寡糖片段的分离分析及其活性研究)[D]. Xi'an: Northwest University, 2011.
|
| [5] |
Guo LM, Zhang RX, Jia ZP, et al. Progress in pharmacological study and the underlying mechanism of oligosaccharides[J]. Chin Tradit Pat Med (中成药), 2006, 28: 1353-1356. |
| [6] |
Wang JY, Huang CC, Gao F, et al. Survey on algorithms for tandem mass spectrometry oligosaccharide identification[J]. Prog Biochem Biophys (生物化学与生物物理进展), 2017, 44: 830-847. |
| [7] |
Zhou FJ, Huang M, Peng XJ, et al. Optimization of the extraction technology of oligosaccharides from the flowers of Osmanthus fragrans by uniform design[J]. West China J Pharm Sci (华西药学杂志), 2017, 32: 63-66. |
| [8] |
Liu L, Zhang Y, Zhang MW, et al. Optimization on ultrasonic wave assisted extraction of oligosaccharide from Chinese Yam by response surface methodology[J]. Guangdong Agric Sci (广东农业科学), 2014, 41: 100-105. |
| [9] |
Wang YJ, Xu MD, Yao JY, et al. Influence of microwave on the extraction of soybean germ protein and soybean oligosaccharides[J]. Food Sci Technol (食品科技), 2009, 34: 171-175. |
| [10] |
Huang LH, Wu HJ, Ma YQ, et al. Progress in pectic oligosaccharides[J]. J Food Sci (食品科学), 2015, 36: 277-281. |
| [11] |
Wang GZ. Evaluation of Germplasm Resources on Astragali Radix Based on Saccharide Mapping of Polysaccharide and Structural Analysis Techniques (基于多糖糖谱及结构解析技术的黄芪种质资源评价)[D]. Taiyuan: Shanxi University, 2017.
|
| [12] |
Li XX, Wang D, Wang GZ, et al. Establishment of fingerprint of Astragali Radix polysaccharides based on artificial acidic acid hydrolysis and identification of different germplasm resources of Astragali Radix[J]. Mod Chin Med (中国现代中药), 2018, 20: 152-158. |
| [13] |
Xie JS. Studies on the Activity of Enzymatically Prepared Agar Oligosaccharides (琼胶寡糖的酶解制备及其活性研究)[D]. Fuzhou: Fuzhou University, 2016.
|
| [14] |
Shao M, Liu CH. Recent advances in synthesis of heparin oligosaccharides and their regulation effect on angiogenesis[J]. Pharm Biotechnol (药物生物技术), 2018, 25: 263-267. |
| [15] |
Liu XY. Studies on the Structure and Antitumor Activities of the Neutral Polysaccharides from Dioscorea bulbifera L. (黄药子中性多糖的结构分析及其抗肿瘤活性研究)[D]. Changchun: Northeast Normal University, 2013.
|
| [16] |
Wang ZF, He JY, Wei YH, et al. Progress in precolumn derivatization for HPLC analysis of glycans[J]. Chin J Org Chem (有机化学), 2006, 26: 592-598. |
| [17] |
Peng H, Lin L, Peng T, et al. Preparative separation of high-purity cellooligosaccharides by polyacrylamide gel column[J]. Chem Ind For Prod (林产化学与工业), 2008, 28: 63-66. |
| [18] |
Peng H. Research progress in separation and purification of cellooligosaccharides[J]. Cereals Oilseeds (粮食与油脂), 2011, 7: 41-43. |
| [19] |
Akpinar O, Mcgorrin RJ, Penner MH. Cellulose-based chromatography for cellooligosaccharide production[J]. J Agric Food Chem, 2004, 52: 4144-4148. DOI:10.1021/jf035305m |
| [20] |
Wang ZF, Zhang Y, Lin X, et al. HPLC separation of 1-phenyl-3-methy-5-pyrazolone derivatized oligosaccharides and its matrix-assisted laser desorption/ionization time-of-flight mass spectrometry identification[J]. Acta Chim Sin (化学学报), 2007, 65: 2761-2764. |
| [21] |
Han LJ, Yuan ZH, Fan X, et al. A review of separation and structure determination methods for oligosaccharides[J]. Stud Mar Sin (海洋科学集刊), 2004, 46: 126-133. |
| [22] |
Ding P, Shao YH, Ying G, et al. Study on HPAEC-PAD fingerprint analysis of oligosaccharides from Morinda officinalis[J]. Chin Pharm J (中国药学杂志), 2011, 46: 1707-1711. |
| [23] |
Liu H, Bai XF, Du YG, et al. Progresses on isolation and structural analysis of oligosaccharides[J]. Fine Spec Chem (精细与专用化学品), 2005, 13: 1-5. |
| [24] |
Lang YZ, Liu SL, Wang C, et al. Separation and structural sequence analysis of sialylated HMOs via tandem mass spectrometry[J]. Chem J Chin Univ (高等学校化学学报), 2018, 39: 645-652. |
| [25] |
Sun HH. Analysis of κ-Carrageenan Oligosaccharides by HPCE(κ-卡拉胶寡糖的高效毛细管电泳分析方法的研究)[D]. Qingdao: Ocean University of China, 2006.
|
| [26] |
Zhou YF, Wu YX, Tang QY, et al. Application of bio-mass spectrometry in life science[J]. J Hubei Univ Natl Nat Sci Ed (湖北民族学院学报·自然科学版), 2004, 22: 11-16. |
| [27] |
Wang Z. Preliminary Studies on Derivatived Oligosaccharide Molecular Ions Fragmentation Pattern in ESI-MSn Analysis (寡糖及其衍生物分子离子ESI-MSn碎裂行为初步研究)[D]. Xi'an: Northwest University, 2011.
|
| [28] |
Chen HH, Luan XH, Yu GL. Application of electrospray tandem mass spectrometry in sequence analysis of oligosaccharides[J]. Chem J Chin Univ (高等学校化学学报), 2015, 36: 1-8. |
| [29] |
Karas M, Bahr U, Dulcks T. Nano-electrospray ionization mass spectrometry:addressing analytical problems beyond routine[J]. Fresenius J Anal Chem, 2000, 366: 669-676. DOI:10.1007/s002160051561 |
| [30] |
Lin CQ. The application of mass spectrum on amylose structure analysis[J]. Anal Test Technol Instrum (分析测试技术与仪器), 2005, 11: 221-227. |
| [31] |
Liu F, Zhao SL, Chen CY. Progress on mass spectrometry in structural analysis of polysaccharides[J]. Chin J Pharm Anal (药物分析杂志), 2011, 31: 1612-1617. |
| [32] |
Walker SH, Lilley LM, Enamorado MF, et al. Hydrophobic derivatization of N-linked glycans for increased ion abundance in electrospray ionization mass spectrometry[J]. J Am Soc Mass Spectrom, 2011, 22: 1309-1317. DOI:10.1007/s13361-011-0140-x |
| [33] |
Guo W, Zhang Y, Huang LJ, et al. High performance liquid chromatography/electrospray ionization ion-trap mass spectrometry analysis of chitooligosaccharides by pre-column derivatization with 3-amino-9-ethylcarbazole[J]. Chin J Chromatogr (色谱), 2010, 28: 776-781. DOI:10.3724/SP.J.1123.2010.00776 |
| [34] |
Harvey DJ. Analysis of carbohydrates and glycoconjugates by matrix-assisted laser desorption/ionization mass spectrometry:an update for 2007-2008[J]. Mass Spectrom Rev, 2012, 31: 183-311. DOI:10.1002/mas.20333 |
| [35] |
Domann P, Spencer DIR, Harvey DJ. Production and fragmentation of negative ions from neutral N-linked carbohydrates ionized by matrix-assisted laser desorption/ionization[J]. Rapid Commun Mass Spectrom, 2012, 26: 469-479. DOI:10.1002/rcm.5322 |
| [36] |
Huang CY, Jing Y, Zhan LP, et al. Linkage and sequence analysis of neutral oligosaccharides by negative-ion MALDI tandem mass spectrometry with laser-induced dissociation[J]. Anal Chim Acta, 2019, 1071: 25-35. DOI:10.1016/j.aca.2019.04.067 |
| [37] |
Nishikaze T, Kaneshiro K, Kawabata S, et al. Structural analysis of N-glycans by the glycan-labeling method using 3-aminoquinoline-based liquid matrix in negative-ion MALDI-MS[J]. Anal Chem, 2012, 84: 9453-9461. DOI:10.1021/ac302286e |
| [38] |
Domon B, Costello CE. A systematic nomenclature for carbohydrate fragmentations in FAB-MS/MS spectra of glycoconjugates[J]. Glycoconjugate J, 1988, 5: 397-409. DOI:10.1007/BF01049915 |
| [39] |
Zaia J. Mass spectrometry of oligosaccharides[J]. Mass Spectrom Rev, 2004, 23: 161-227. DOI:10.1002/mas.10073 |
| [40] |
Zaia J, Costello CE. Tandem mass spectrometry of sulfated heparin-like glycosaminoglycan oligosaccharides[J]. Anal Chem, 2003, 75: 2445-2455. DOI:10.1021/ac0263418 |
| [41] |
Jiao J, Zhang H, Reinhold VN. High performance IT-MSn sequencing of glycans:spatial resolution of ovalbumin isomers[J]. Int J Mass Spectrom, 2011, 303: 109-117. DOI:10.1016/j.ijms.2011.01.016 |
| [42] |
Wuhrer M, Andre M, Deelder, et al. Mass spectrometric glycan rearrangements[J]. Mass Spectrom Rev, 2011, 30: 664-680. DOI:10.1002/mas.20337 |
| [43] |
Lee H, An HJ, Lerno LA, et al. Rapid profiling of bovine and human milk gangliosides by matrix-assisted laser desorption/ionization Fourier transform ion cyclotron resonance mass spectrometry[J]. Int J Mass Spectrom, 2011, 305: 138-150. |
| [44] |
Harvey DJ, Rudd PM. Fragmentation of negative ions from N-linked carbohydrates. Part 5:Anionic N-linked glycans[J]. Int J Mass Spectrom, 2011, 305: 120-130. DOI:10.1016/j.ijms.2010.10.016 |
| [45] |
Jeremy JW, Franklin EL, Tatiana NL, et al. Negative electron transfer dissociation of glycosaminoglycans[J]. Anal Chem, 2010, 82: 3460-3466. DOI:10.1021/ac100554a |
| [46] |
Zhao P, Viner R, Teo CF, et al. Combining high-energy C-trap dissociation and electron transfer dissociation for protein O-GlcNAc modification site assignment[J]. J Proteome Res, 2011, 10: 4088-4104. DOI:10.1021/pr2002726 |
| [47] |
Hersberger KE. Characterization of O-sulfopeptides by negative ion mode tandem mass spectrometry:superior performance of negative ion electron capture dissociation[J]. Anal Chem, 2012, 84: 6370-6377. DOI:10.1021/ac301536r |
| [48] |
Zhang Q, Li Z, Wang Y, et al. Mass spectrometry for protein sialoglycosylation[J]. Mass Spectrom Rev, 2017, 37: 652-680. |
| [49] |
Domon B, Costello CE. Structure elucidation of glycosphingolipids and gangliosides using high-performance tandem mass spectrometry[J]. Biochemistry, 1988, 27: 1534-1543. DOI:10.1021/bi00405a021 |
| [50] |
Gao XL, Yu W, Ming Y, et al. Matrix-assisted laser desorption/ionization time-of-flight mass spectrometry analysis of human milk neutral and sialylated free oligosaccharides using Girard's reagent P on-target derivatization[J]. J Agric Food Chem, 2019, 67: 8958-8966. DOI:10.1021/acs.jafc.9b02635 |
| [51] |
Duffin KL, Welply JK, Huang E, et al. Characterization of N-linked oligosaccharides by electrospray and tandem mass spectrometry[J]. Anal Chem, 1992, 64: 1440-1448. DOI:10.1021/ac00037a024 |
| [52] |
Zhao C, Xie B, Chan SY, et al. Collisionally activated dissociation and electron capture dissociation provide complementary structural information for branched permethylated oligosaccharides[J]. J Am Soc Mass Spectrom, 2008, 19: 138-150. DOI:10.1016/j.jasms.2007.10.022 |
| [53] |
Gillece-Castro BL, Burlingame AL. Oligosaccharide characterization with high-energy collision-induced dissociation mass spectrometry[J]. Methods Enzymol, 1990, 193: 689-712. DOI:10.1016/0076-6879(90)93445-Q |
| [54] |
Cancilla MT, Wong AW, Voss LR, et al. Fragmentation reactions in the mass spectrometry analysis of neutral oligosaccharides[J]. Anal Chem, 1999, 71: 3206-3218. DOI:10.1021/ac9813484 |
| [55] |
Yu X, Huang Y, Lin C, et al. Energy-dependent electron activated dissociation of metal-adducted permethylated oligosaccharides[J]. Anal Chem, 2012, 84: 7487-7494. |
| [56] |
Schaller-Duke RM, Bogala MR, Cassady CJ. Electron transfer dissociation and collision-induced dissociation of underivatized metallated oligosaccharides[J]. J Am Soc Mass Spectr, 2018, 29: 1021-1035. DOI:10.1007/s13361-018-1906-1 |
| [57] |
Matthew T, Campbell DC, Gary LG. Distinguishing linkage position and anomeric configuration of glucose-glucose disaccharides by water adduction to lithiated molecules[J]. Anal Chem, 2018, 90: 2048-2054. DOI:10.1021/acs.analchem.7b04162 |
| [58] |
Harvey DJ. Collision-induced fragmentation of underivatized N-linked carbohydrates ionized by electrospray[J]. J Mass Spectrom, 2000, 35: 1178-1190. DOI:10.1002/1096-9888(200010)35:10<1178::AID-JMS46>3.0.CO;2-F |
| [59] |
Mendonca S, Cole RB, Zhu JH, et al. Incremented alkyl derivates enhance collision induced glycosidic bond cleavage in mass spectrometry of disaccharides[J]. J Am Soc Mass Spectrom, 2003, 14: 63-78. DOI:10.1016/S1044-0305(02)00816-4 |
| [60] |
Zhu F, Glover MS, Shi H, et al. Populations of metal-glycan structures influence MS fragmentation patterns[J]. J Am Soc Mass Spectrom, 2015, 26: 25-35. DOI:10.1007/s13361-014-1000-2 |
| [61] |
Jiang K, Zhu H, Li L, et al. Sialic acid linkage-specific permethylation for improved profiling of protein glycosylation by MALDI-TOF MS[J]. Anal Chim Acta, 2017, 981: 53-61. DOI:10.1016/j.aca.2017.05.029 |
| [62] |
Camilleri P, Tolson D, Birrell H. Direct structural analysis of 2-aminoacridone derivatized oligosaccharides by high-performance liquid chromatography/mass spectrometric detection[J]. Rapid Commun Mass Spectrom, 1998, 12: 144-148. DOI:10.1002/(SICI)1097-0231(19980214)12:3<144::AID-RCM133>3.0.CO;2-7 |
| [63] |
Charlwood J, Birrell H, Gribble A, et al. A probe for the versatile analysis and characterization of N-linked oligosaccharides[J]. Anal Chem, 2000, 72: 1453-1461. DOI:10.1021/ac991268f |
| [64] |
Lai YH, Wang YS. Matrix-assisted laser desorption/ionization mass spectrometry:mechanistic studies and methods for improving the structural identification of carbohydrates[J]. Mass Spectrom, 2017, 6: S0072. DOI:10.5702/massspectrometry.S0072 |
| [65] |
Miura Y, Shinohara Y, Furukawa J, et al. Rapid and simple solid-phase esterification of sialic acid residues for quantitative glycomics by mass spectrometry[J]. Chem-Eur J, 2010, 13: 4797-4804. |
| [66] |
Reiding KR, Blank D, Kuijper DM, et al. High-throughput profiling of protein N-glycosylation by MALDI-TOF-MS employing linkage-specific sialic acid esterification[J]. Anal Chem, 2014, 86: 5784-5793. DOI:10.1021/ac500335t |
| [67] |
Jin ZY, Wang JP, Deng L, et al. Databases and web tools on carbohydrates[J]. J Food Sci Biotechnol (食品与生物技术学报), 2008, 27: 1-5. |
| [68] |
Cooper CA, Gasteiger E, Packer NH. GlycoMod:a software tool for determining glycosylation compositions from mass spectrometric data[J]. Proteomics, 2001, 1: 340-349. DOI:10.1002/1615-9861(200102)1:2<340::AID-PROT340>3.0.CO;2-B |
| [69] |
Campbell MP, Hayes CA, Struwe WB, et al. UniCarbKB:putting the pieces together for glycomics research[J]. Proteomics, 2011, 11: 4117-4121. DOI:10.1002/pmic.201100302 |
| [70] |
Claus-Wilhelm VDL, Freire AA, Blank D, et al. EUROCarbDB:an open-access platform for glycoinformatics[J]. Glycobiology, 2011, 21: 493-502. DOI:10.1093/glycob/cwq188 |
| [71] |
Maxwell E, Tan Y, Tan YM, et al. GlycReSoft:a software package for automated recognition of glycans from LC/MS Data[J]. PLoS One, 2012, 7: e45474. DOI:10.1371/journal.pone.0045474 |
 2020, Vol. 55
2020, Vol. 55


