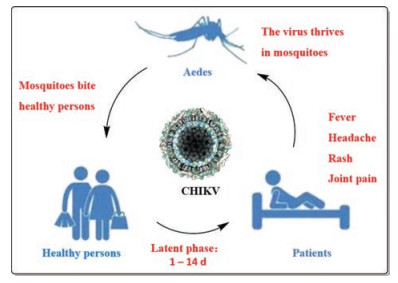
基孔肯雅热(Chikungunya fever, CHIKF)是由基孔肯雅病毒(Chikungunya virus, CHIKV)引起, 经伊蚊传播, 以发热、皮疹及关节疼痛为主要特征的急性感染性疾病(图 1)[1-3]。“基孔肯雅”是坦桑尼亚南部的土语, 意思是“令人弯腰屈背”, 指由于关节炎症状而导致的弯腰姿势[4]。自1953年首次暴发以来, CHIKV在非洲、南亚以及东南亚引起了大量有据可查的疫情和流行病[5-8]。尽管基孔肯雅病致死率很低, 但在蚊媒密度较高的地区容易形成大规模的流行和暴发, 是21世纪初严重影响人类公共卫生安全的虫媒传染病之一[9, 10]。目前尚无疫苗或抗病毒药物可用于预防或治疗CHIKV感染, 因此迫切需要开发安全有效, 具有临床应用价值的抗病毒药物来预防和治疗CHIKV感染。
|
Figure 1 Transmission of Chikungunya virus |
CHIKV属于披膜病毒科(family Togaviridae)、甲病毒属(genus Alphavirus, α virus)、西门利克森林病毒复合群(Semliki forest virus complex, SVF)[11]。病毒为直径60~70 nm的球形颗粒, 由糖蛋白外壳、脂质双分子层包膜以及病毒核心组成[12, 13]。病毒核心外层为二十面体的核衣壳蛋白, 包裹着对病毒复制至关重要的RNA等物质[14, 15]。
CHIKV为单股正链RNA病毒(图 2), 其基因组长度约为11.8 kb, 包含两个开放式阅读框(open reading frame, ORF), 一个5'端ORF (能够编码4种病毒非结构蛋白nsP1~nsP4)和一个3'端ORF (编码病毒结构蛋白, 包括核衣壳蛋白C、两种主要包膜糖蛋白E1和E2, 以及两个较小的辅肽E3和6K)[16-18]。非结构蛋白主要通过介导病毒和宿主细胞成分的相互作用, 参与病毒的发病机制: nsP1通过其鸟嘌呤-7-甲基转移酶和鸟嘌呤转移酶的活性参与病毒的mRNA封顶; nsP2充当蛋白酶和螺旋酶; nsP3是复制酶单元的一部分, 并且属于参与RNA合成的辅助蛋白; nsP4是RNA依赖性RNA聚合酶[19-21]。病毒糖蛋白E1和E2镶嵌在病毒包膜上, 介导病毒吸附宿主细胞并与细胞膜融合[22]。E3蛋白是一种小的A/B蛋白, 由64个氨基酸组成, 具有二硫异构酶活性, 有助于病毒糖蛋白的正确折叠和二硫键形成[19]。6K蛋白是病毒颗粒组装所必需的疏水性小蛋白, 它是处理E1蛋白的信号序列[23]。两个ORF之间的连接区是不可翻译的, 它包含一个内部启动子和转录亚基因组RNA (26S RNA)的起始位点[24]。

|
Figure 2 RNA sequence of Chikungunya virus genome |
CHIKV的复制周期如图 3所示。首先, 病毒表面的包膜糖蛋白E2与靶细胞表面的特异性受体产生相互作用, 促使CHIKV进入宿主细胞[25]。一旦与受体结合, 病毒通过内吞作用迅速内化。在内吞泡内, 由于低pH环境, 病毒包膜糖蛋白发生构象变化, 使得包膜糖蛋白E1与内分体膜融合[26]。致使病毒衣壳释放到宿主细胞质中, 进而被分解并释放病毒RNA基因组。随后, 病毒基因组利用宿主细胞核糖体翻译产生非结构多蛋白, 该多蛋白被切割产生nsP123前体蛋白和游离的nsP4蛋白[27]。两者和宿主蛋白相互作用, 形成初始复制复合物(病毒复制酶), 合成负链RNA[24]。然后以负链RNA作为模板合成正链基因组RNA和26S RNA。其中26S RNA作为合成包含病毒结构蛋白C-pE2-6K-E1多肽片段的mRNA[25]。

|
Figure 3 Replication cycle of Chikungunya virus |
随后, 核衣壳蛋白C通过其自身的保护酶活性从多肽中释放, 而其余的pE2-6K-E1多肽则在内质网中加工[27]。pE2和E1糖蛋白形成异二聚体复合物, 通过高尔基复合体向细胞膜迁移。在向细胞表面迁移的过程中, pE2被宿主细胞的蛋白酶切割, 形成成熟的E2和E3[28]。最后, 核衣壳复合物在出芽时通过宿主细胞膜, 从而获得含有病毒糖蛋白E1-E2异二聚体结构的脂质双层包膜, 最终形成成熟的病毒颗粒[26]。从理论上讲, 病毒生命周期的多个步骤均可作为抑制CHIKV复制的靶点。
目前有几个研究小组正在积极使用各种策略研发不同类型的CHIKV疫苗, 包括灭活病毒疫苗、减毒活病毒疫苗、嵌合病毒疫苗、重组病毒疫苗、重组亚单位疫苗以及最近出现的一种类病毒疫苗[29-32]。2010年报道的一种表达CHIKV复合蛋白的类病毒颗粒疫苗可以诱导机体产生中和抗体, 并保护猴子免受病毒感染[29]; 2011年Plante等[33]开发了一种活病毒疫苗, 它能在小鼠身上引起保护性免疫反应, 目前这种候选疫苗仍在非人类灵长类动物中进行评估。2014年, 一种利用结构蛋白嵌合α病毒的减毒活疫苗在试验中显示出良好的预防CHIKV的前景[34]。尽管针对CHIKV疫苗的开发研究正在不断进行, 但是到目前为止依旧没有取得突破性进展, 全世界仍然面临着CHIKV感染迅速蔓延的威胁, 这表明开发针对该病毒的化学疗法的重要性。
1 CHIKV小分子抑制剂本文将主要篇幅用于描述安全有效、具有临床应用价值的CHIKV抑制剂, 并且概述发现这些化合物的方法与策略。特别是对目前最新抑制剂研究进展及药物研发策略的分析, 对于未来抗CHIKV药物的研发具有指导意义。
1.1 CHIKV侵入抑制剂抑制病毒进入细胞是一种非常有吸引力的治疗策略, 如果病毒无法进入细胞完成复制过程, 可以使毒力因子所造成的损害可以降到最低[35]。
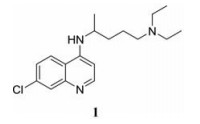
1.1.1 氯喹(chloroquine)氯喹(1)是一种抗疟药, 在体外对多种病毒具有抑制作用, 包括艾滋病毒(human immunodeficiency virus, HIV)、严重急性呼吸综合征(severe acute respiratory syndrome, SARS)冠状病毒以及α病毒[36]。其作用机制为提高内体pH值、阻止E1介导的膜融合过程[37]。氯喹以剂量依赖的方式抑制Vero A细胞中CHIKV的复制(EC50 = 7.0 μmol·L-1)[38]。
|
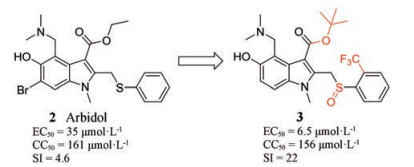
广谱抗病毒药物阿比多尔(2)最初用于预防和治疗急性呼吸道感染, 以及流感[39]。阿比多尔可整合细胞膜, 导致膜结构改变, 从而阻碍CHIKV的膜融合过程[40]。Scuotto等[41]对阿比多尔进行进一步结构优化, 得到化合物3 (图 4), 其抗CHIKV活性以及选择指数都有大幅提升。
|
Figure 4 The discovery of 3 via structural optimization of arbidol (2) |
在病毒基因组和亚基因组RNA进行蛋白质翻译时, 完整的RNA帽状结构可以保护病毒RNA不受宿主细胞5'外切核酸酶的降解, 并保护病毒5'端非蛋白质编码部分, 从而使CHIKV能够逃避宿主的抗病毒反应[42, 43]。病毒基因组的RNA封顶过程十分关键, 而nsP1在RNA帽状结构的形成中起着核心作用[44], 因此, 是十分有前景的抗病毒药物靶标。
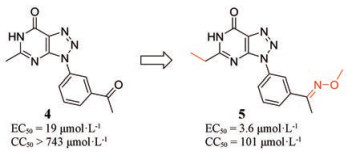
1.2.1 三唑并嘧啶类(triazolopyrimidine)2014年, Gigante等[45]发现了一系列选择性作用于CHIKV nsP1从而抑制病毒复制的小分子。其中, 化合物4被证实可以通过抑制nsP1鸟嘌呤转移酶的活性而影响病毒的复制过程[46]。对其进行结构优化, 将其母核上的甲基变为乙基, 苯乙酮结构中的羰基变为酮邻甲基肟得到的化合物5, 活性提高了5倍[47]。
|
通过高通量筛选技术发现的小分子化合物洛巴酸(6)具有良好的抗CHIKV活性[48]。通过鸟嘌呤化抑制反应以及荧光偏振分析法发现, 洛巴酸可以阻断nsP1与三磷酸鸟苷(guanosine triphosphate, GTP)的相互作用, 从而破坏RNA封顶过程, 阻断蛋白质翻译, 降低基因组稳定性, 有效抑制病毒的成熟过程[49]。因此, 洛巴酸衍生物作为以nsP1为靶点的先导化合物, 值得进一步研发。
|
CHIKV非结构蛋白2是一种半胱氨酸蛋白酶, 在CHIKV非结构多蛋白的裂解和加工过程中起着关键作用, 并且可以抑制Ⅰ/Ⅱ型干扰素的抗病毒作用[37, 50]。抑制nsP2蛋白酶将导致病毒多蛋白链断裂, 从而阻止病毒复制。
1.3.1 硫酸核糖霉素(ribostamycin sulfate)2019年, Kumar等采用“药物再利用”策略[51], 利用计算机辅助药物设计[52-54], 对已知的半胱氨酸蛋白酶抑制剂进行虚拟筛选。结果表明硫酸核糖霉素(7)与CHIKV nsP2蛋白酶(PDB code: 3TRK)相互作用最稳定, 结合能较低(图 5)。因此, 硫酸核糖霉素可以抑制CHIKV蛋白酶的活性, 进而抑制病毒的复制。
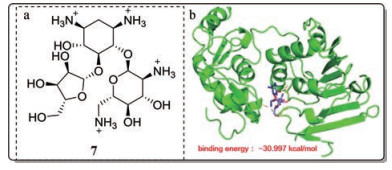
|
Figure 5 (a) The structure of ribostamycin sulfate; (b) Specific mode of action of ribostamycin sulfate (purple) and CHIKV nsP2 (green) (PDB code: 3TRK) |
Saha等[55]利用纯化的nsP2蛋白酶和荧光肽底物, 开发了基于荧光共振能量转移(fluorescence resonance energy transfer, FRET)的高通量筛选分析方法。醋酸锌是FDA批准的治疗威尔逊氏病的药物, 也是CHIKV nsP2蛋白酶抑制剂, 并且在Vero细胞水平具有抗病毒活性[56]。分子对接表明, Zn2+与Cys478、His548以及两个水分子形成配位作用, 水分子则与Asp550产生作用力[55], 为基于结构的合理设计提供了证据(图 6)。
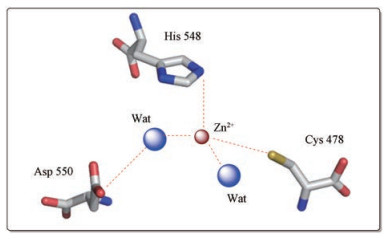
|
Figure 6 The coordination between Zn2+ and the active site of nsP2 (PDB code: 4ZTB) |
CHIKV非结构蛋白3 (nsP3)在病毒复制过程中发挥着极其重要的作用, 在复制前期作为多蛋白nsP123的一部分, 与nsP4一起用于负链RNA的合成, 然后以负链RNA作为模板合成正链基因组RNA和26S RNA[57]。尽管nsP3的具体作用机制尚不明确, 但研究表明nsP3基因突变会显著影响CHIKV的感染能力[58]。
1.4.1 黄岑苷(baicalin)黄芩苷(8)是黄芩素的代谢产物, 可从中药黄芩中提取[59]。2016年, Seyedi等[59]通过虚拟筛选与分子对接技术, 发现黄芩苷对CHIKV nsP3 (PDB code: 3GPO)表现出很强的亲和力(图 7), 有可能抑制CHIKV感染后的病毒基因组的复制。
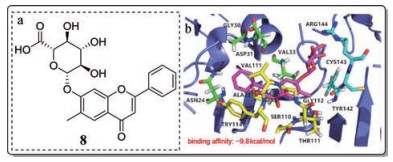
|
Figure 7 (a) The structure of baicalin; (b) Specific mode of action of baicalin (red) and CHIKV nsP3 (blue) (PDB code: 3GPO) |
2019年, Kumar等[60]设计了一种以噁唑烷-2, 4二酮(9)、不同基团取代的苯甲醛(10)以及异氰基乙酸乙酯(11)为原料的三组分反应, 构建了以吡喃唑为母核结构的虚拟化合物库。通过iGemDock虚拟筛选技术发现化合物12 (图 8)对CHIKV nsP3蛋白酶(PDB code: 3GPO)显示出极高的亲和力, 总结合能为-151.024 kcal·mol-1, 优于黄芩苷(总结合能为-134.334 kcal·mol-1)等已报道的化合物, 值得进一步研究。
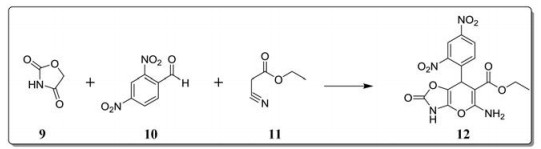
|
Figure 8 Discovery of 12 from compound library via multicomponent reaction |
CHIKV非结构蛋白4是一种RNA依赖性的RNA聚合酶(RNA dependent RNA polymerase, RdRp), 并且可以通过抑制宿主细胞的内质网应激反应, 保证病毒蛋白的正确翻译[25, 61], 是抗病毒药物的新靶点。
2017年, Wada等[62]发现苯并咪唑类化合物13在亚微摩尔浓度下可以抑制CHIKV的感染, 其EC50为0.54 μmol·L-1。作用机制研究发现, 其作用靶点为nsP4上的RdRp功能区, 且该化合物确实具有抑制CHIKV RdRp的功能。
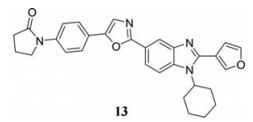
|
CHIKV对宿主细胞的侵袭是在两种病毒糖蛋白E1和E2的作用下完成的[63]。E2与细胞表面受体结合, E1介导膜融合过程[64]。在中性pH下, E1和E2以异二聚体形式存在, 抑制该异二聚体可以阻碍CHIKV进入宿主细胞, 并对病毒感染后期的组装过程产生影响[65]。
2019年, Agarwal等[66]利用CHIKV包膜蛋白(PDB code: 3N41)的晶体结构对FDA批准的药物分子库进行虚拟筛选, 发现几种头孢菌素类药物可以与CHIKV包膜蛋白复合物E1-E2界面的两个位点结合, 且结合能较低。因此, 头孢甲肟(14)等头孢菌素类药物可能会破坏包膜糖蛋白的完整性, 从而导致病毒丧失形成成熟病毒颗粒和进入宿主细胞的能力, 是CHIKV E1-E2异二聚体的潜在抑制剂。
1.7 宿主靶向抑制剂由于病毒复制周期的每一个环节都有细胞因子的参与, 因此将宿主因子作为抗病毒药物的靶点也是一种有前景的方法[67]。
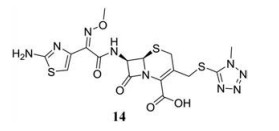
|
奥巴托拉(15)是一种Bcl-2 (B-cell lymphoma-2)蛋白拮抗剂, 可以协助抑制Mcl-1 (myeloid cell leukemia-1)蛋白介导的抗细胞凋亡作用[68]。近期研究证实其具有体外抗CHIKV活性, 其EC50为0.03 μmol·L-1[69]。病毒侵入抑制试验以及中性红保留试验表明奥巴托拉可诱导内溶体酸性环境的快速中和, 从而干扰病毒外膜与宿主细胞膜的融合。
1.7.2 小檗碱(berberine)2016年, Varghese等[70]通过高通量筛选技术, 发现异喹啉类生物碱小檗碱(16)可有效抑制CHIKV的复制, EC50为37.6 μmol·L-1。机制研究表明, 小檗碱在CHIKV感染过程中可以显著影响丝裂原活化蛋白激酶(mitogen-activated protein kinases, MAPK)信号通路, 并显著降低病毒滴度。
1.7.3 12-脱氧佛波醇-13-乙酸(prostratin)蛋白激酶C (protein kinase C, PKC)是一种多功能的丝氨酸/苏氨酸激酶家族, 通过磷酸化信号传导通路调节细胞增殖、分化和凋亡等过程[71]。据Abdelnabi等[9]报道, 12-脱氧佛波醇-13-乙酸(17)可以在体外抑制CHIKV诱导的细胞病变作用。在病毒复制过程中, 该化合物以剂量依赖的方式减少了感染性病毒颗粒的产生和病毒蛋白的积累; 但是在PKC抑制剂的存在下, 其抗病毒活性明显降低。这一结果表明12-脱氧佛波醇-13-乙酸的抗病毒作用是由PKCs的激活引起的, PKCs是抑制CHIKV复制的潜在宿主靶点。
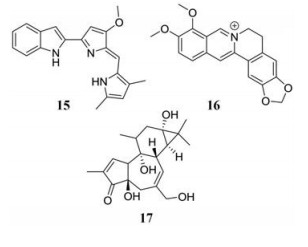
|
病毒基因组复制是病毒生命周期的关键环节, 靶向病毒基因组复制也是抗病毒药物研发的有效途径[72]。
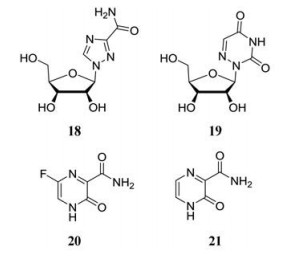
1.8.1 利巴韦林(ribavirin)利巴韦林(18)是一种具有广谱抗病毒活性的合成鸟苷类似物, 被批准用于治疗呼吸道合胞病毒(respiratory syncytial virus, RSV)以及丙型肝炎病毒(hepatitis C virus, HCV)感染, 疗效较好[73]。利巴韦林可以抑制RNA病毒复制过程中的多个阶段, 从而产生广谱抗病毒作用[74]。利巴韦林在体外对CHIKV具有抗病毒活性(EC50 = 341 μmol·L-1), 且与干扰素联合使用时具有协同抑制作用[75]。
1.8.2 6-氮杂尿苷(6-azauridine)6-氮杂尿苷(19)是一种尿苷类似物, 具有抗DNA和RNA病毒的广谱抗病毒活性, 作用机制是通过抑制磷酸脱羧酶从而干扰嘧啶、胞苷的从头合成途径[76]。它在低浓度下显示出对CHIKV的显著抑制作用(EC50 = 0.83 μmol·L-1), 并且与利巴韦林相比对CHIKV更有效[75]。目前该化合物未被批准用于治疗CHIKV。然而, 相应的6-氮杂尿苷三醋酸酯用于治疗其他疾病时, 没有明显的不良反应[77]。
1.8.3 法匹拉韦(favipiravir)法匹拉韦(20)是一种广谱抗病毒药物, 在日本被批准用于治疗流行性感冒[78]。该化合物在细胞内被细胞酶激活, 生成5-三磷酸呋喃核糖酯, 竞争性地抑制了病毒RNA依赖性RNA聚合酶[79]。基于此, Delang等[79, 80]报道法匹拉韦及其类似物T-1105 (21)在体外对CHIKV具有抗病毒活性, 其EC50分别为25和7 μmol·L-1。
|
基孔肯雅热是由基孔肯雅病毒引起, 经伊蚊传播, 以发热、皮疹及关节疼痛为主要特征的急性传染病。近年来, 基孔肯雅病毒的再次流行和全球范围的传播, 使该病毒引发的公共卫生安全问题日趋严峻。目前尚无疫苗或抗病毒药物可用于预防或治疗基孔肯雅病毒感染, 因此迫切需要开发安全有效的抗病毒药物来防治基孔肯雅病毒感染。通过科研人员的不懈努力, 一些有潜力的先导化合物已经被发现。本文对最具代表性的新型基孔肯雅病毒抑制剂进行归纳, 并分析了发现这些化合物的方法与策略, 主要包括:临床药物的重新定位或再利用; 对已知的基孔肯雅病毒抑制剂的结构改造和优化; 依据靶点蛋白三维结构, 基于计算机辅助技术的合理设计; 基于机体细胞机制的抗病毒研究; 生物活性分子库的构建与高通量筛选等。这些成功的案例对未来抗基孔肯雅病毒药物的研发具有指导意义。
虽然现阶段对于基孔肯雅病毒的研究取得了一定进展, 但是仍然还有许多问题亟待解决: ①基孔肯雅病毒在宿主细胞内的受体以及进入人体和虫媒细胞的机制尚不明确; ②基孔肯雅病毒的致病机制还未研究透彻; ③由于缺乏与人体相似的动物模型, 目前大多数基孔肯雅病毒抑制剂的体内疗效无法得到评估; ④当前只有nsP2、nsP3、衣壳蛋白C以及E1-E2复合物的结构被解析出来, 且没有发现其共晶配体, 对于其他蛋白的结构仍然知之甚少; ⑤最近有报道称, 基孔肯雅病毒蛋白的特征是存在无序的蛋白-蛋白相互作用[81], 目前缺乏对于这种相互作用的理解。
诚然, 上述问题的解决依靠结构生物学、生物化学、病毒学、计算化学和药物化学等不同学科研究人员的共同努力, 以获得病毒蛋白准确的结构信息, 并建立合适的体内模型, 为寻找安全有效的抗基孔肯雅病毒感染的抗病毒药物奠定坚实的基础。
| [1] |
Deller JJ Jr, Russell PK. An analysis of fevers of unknown origin in American soldiers in Vietnam[J]. Ann Intern Med, 1967, 66: 1129-1143. DOI:10.7326/0003-4819-66-6-1129 |
| [2] |
McGill PE. Viral infections: alpha-viral arthropathy[J]. Baillieres Clin Rheumatol, 1995, 9: 145-150. |
| [3] |
Ligon BL. Reemergence of an unusual disease: the Chikungunya epidemic[J]. Semin Pediatr Infect Dis, 2006, 17: 99-104. |
| [4] |
Robinson MC. An epidemic of virus disease in Southern Province, Tanganyika Territory, in 1952-53. I. Clinical features[J]. Trans R Soc Trop Med Hyg, 1955, 49: 28-32. |
| [5] |
Lo Presti A, Cella E, Angeletti S, et al. Molecular epidemiology, evolution and phylogeny of Chikungunya virus: an updating review[J]. Infect Genet Evol, 2016, 41: 270-278. DOI:10.1016/j.meegid.2016.04.006 |
| [6] |
Mendoza EJ, Robinson A, Dimitrova K, et al. Combining anti-IgM and IgG immunoassays for comprehensive Chikungunya virus diagnostic testing[J]. Zoonoses Public Health, 2019, 66: 909-917. |
| [7] |
Schwartz O, Albert ML. Biology and pathogenesis of Chikungunya virus[J]. Nat Rev Microbiol, 2010, 8: 491-500. DOI:10.1038/nrmicro2368 |
| [8] |
Weaver SC, Forrester NL. Chikungunya: evolutionary history and recent epidemic spread[J]. Antiviral Res, 2015, 120: 32-39. DOI:10.1016/j.antiviral.2015.04.016 |
| [9] |
Abdelnabi R, Amrun SN, Ng LFP, et al. Protein kinases C as potential host targets for the inhibition of Chikungunya virus replication[J]. Antiviral Res, 2017, 139: 79-87. DOI:10.1016/j.antiviral.2016.12.020 |
| [10] |
Lo Presti A, Lai A, Cella E, et al. Chikungunya virus, epidemiology, clinics and phylogenesis: a review[J]. Asian Pac J Trop Med, 2014, 7: 925-932. |
| [11] |
Rashad AA, Mahalingam S, Keller PA. Chikungunya virus: emerging targets and new opportunities for medicinal chemistry[J]. J Med Chem, 2014, 57: 1147-1166. DOI:10.1021/jm400460d |
| [12] |
Higashi N, Matsumoto A, Tabata K, et al. Electron microscope study of development of Chikungunya virus in green monkey kidney stable (VERO) cells[J]. Virology, 1967, 33: 55-69. DOI:10.1016/0042-6822(67)90093-1 |
| [13] |
Simizu B, Yamamoto K, Hashimoto K, et al. Structural proteins of Chikungunya virus[J]. J Virol, 1984, 51: 254-258. DOI:10.1128/JVI.51.1.254-258.1984 |
| [14] |
Powers AM, Brault AC, Shirako Y, et al. Evolutionary relationships and systematics of the alphaviruses[J]. J Virol, 2001, 75: 10118-10131. |
| [15] |
BalaMurugan S, Sathishkumar R. Chikungunya infection: a potential re-emerging global threat[J]. Asian Pac J Trop Med, 2016, 9: 933-937. DOI:10.1016/j.apjtm.2016.07.020 |
| [16] |
Enserink M. Infectious diseases-Chikungunya: no longer a Third World disease[J]. Science, 2007, 318: 1860-1861. DOI:10.1126/science.318.5858.1860 |
| [17] |
Seyedi SS, Shukri M, Hassandarvish P, et al. Corrigendum: computational approach towards exploring potential anti-Chikungunya activity of selected flavonoids[J]. Sci Rep, 2016, 6: 26368. DOI:10.1038/srep26368 |
| [18] |
Jain J, Dubey SK, Shrinet J, et al. Dengue Chikungunya co-infection: a live-in relationship?[J]. Biochem Biophys Res Commun, 2017, 492: 608-616. |
| [19] |
Singh SK, Unni SK. Chikungunya virus: host pathogen interaction[J]. Rev Med Virol, 2011, 21: 78-88. |
| [20] |
Russo AT, Malmstrom RD, White MA, et al. Structural basis for substrate specificity of alphavirus nsP2 proteases[J]. J Mol Graph Model, 2010, 29: 46-53. DOI:10.1016/j.jmgm.2010.04.005 |
| [21] |
Bhakat S, Karubiu W, Jayaprakash V, et al. A perspective on targeting non-structural proteins to combat neglected tropical diseases: Dengue, West Nile and Chikungunya viruses[J]. Eur J Med Chem, 2014, 87: 677-702. DOI:10.1016/j.ejmech.2014.10.010 |
| [22] |
Sourisseau M, Schilte C, Casartelli N, et al. Characterization of reemerging Chikungunya virus[J]. PLoS Pathog, 2007, 3: e89. DOI:10.1371/journal.ppat.0030089 |
| [23] |
Mukhopadhyay S, Zhang W, Gabler S, et al. Mapping the structure and function of the E1 and E2 glycoproteins in alphaviruses[J]. Structure, 2006, 14: 63-73. DOI:10.1016/j.str.2005.07.025 |
| [24] |
Solignat M, Gay B, Higgs S, et al. Replication cycle of Chikungunya: a re-emerging arbovirus[J]. Virology, 2009, 393: 183-197. DOI:10.1016/j.virol.2009.07.024 |
| [25] |
Grakoui A, Levis R, Raju R, et al. A cis-acting mutation in the sindbis virus junction region which affects subgenomic RNA-synthesis[J]. J Virol, 1989, 63: 5216-5227. |
| [26] |
Gould EA, Coutard B, Malet H, et al. Understanding the alphaviruses: recent research on important emerging pathogens and progress towards their control[J]. Antiviral Res, 2010, 87: 111-124. DOI:10.1016/j.antiviral.2009.07.007 |
| [27] |
Khan AH, Morita K, Parquet MDC, et al. Complete nucleotide sequence of Chikungunya virus and evidence for an internal polyadenylation site[J]. J Gen Virol, 2002, 83: 3075-3084. DOI:10.1099/0022-1317-83-12-3075 |
| [28] |
Schilte C, Couderc T, Chretien F, et al. Type Ⅰ IFN controls Chikungunya virus via its action on nonhematopoietic cells[J]. J Exp Med, 2010, 207: 429-442. |
| [29] |
Akahata W, Yang ZY, Andersen H, et al. A virus-like particle vaccine for epidemic Chikungunya virus protects nonhuman primates against infection[J]. Nat Med, 2010, 16: 334-338. DOI:10.1038/nm.2105 |
| [30] |
Mallilankaraman K, Shedlock DJ, Bao H, et al. A DNA vaccine against Chikungunya virus is protective in mice and induces neutralizing antibodies in mice and nonhuman primates[J]. PLoS Neglect Trop D, 2011, 5: e928. |
| [31] |
Metz SW, Geertsema C, Martina BE, et al. Functional processing and secretion of Chikungunya virus E1 and E2 glycoproteins in insect cells[J]. Virol J, 2011, 8: 353. |
| [32] |
Wang D, Suhrbier A, Penn-Nicholson A, et al. A complex adenovirus vaccine against Chikungunya virus provides complete protection against viraemia and arthritis[J]. Vaccine, 2011, 29: 2803-2809. DOI:10.1016/j.vaccine.2011.01.108 |
| [33] |
Plante K, Wang E, Partidos CD, et al. Novel Chikungunya vaccine candidate with an IRES-based attenuation and host range alteration mechanism[J]. PLoS Pathog, 2011, 7: e1002142. |
| [34] |
Roy CJ, Adams AP, Wang E, et al. Chikungunya vaccine candidate is highly attenuated and protects nonhuman primates against telemetrically monitored disease following a single dose[J]. J Infect Dis, 2014, 209: 1891-1899. |
| [35] |
Kaur P, Chu JJ. Chikungunya virus: an update on antiviral development and challenges[J]. Drug Discov Today, 2013, 18: 969-983. |
| [36] |
Khan M, Santhosh SR, Tiwari M, et al. Assessment of in vitro prophylactic and therapeutic efficacy of chloroquine against Chikungunya virus in Vero cells[J]. J Med Virol, 2010, 82: 817-824. |
| [37] |
Abdelnabi R, Neyts J, Delang L. Towards antivirals against Chikungunya virus[J]. Antiviral Res, 2015, 121: 59-68. |
| [38] |
Di Mola A, Peduto A, La Gatta A, et al. Structure-activity relationship study of arbidol derivatives as inhibitors of Chikungunya virus replication[J]. Bioorg Med Chem, 2014, 22: 6014-6025. DOI:10.1016/j.bmc.2014.09.013 |
| [39] |
Delogu I, Pastorino B, Baronti C, et al. In vitro antiviral activity of arbidol against Chikungunya virus and characteristics of a selected resistant mutant[J]. Antiviral Res, 2011, 90: 99-107. |
| [40] |
Blaising J, Polyak SJ, Pecheur EI. Arbidol as a broad-spectrum antiviral: an update[J]. Antiviral Res, 2014, 107: 84-94. DOI:10.1016/j.antiviral.2014.04.006 |
| [41] |
Scuotto M, Abdelnabi R, Collarile S, et al. Discovery of novel multi-target indole-based derivatives as potent and selective inhibitors of Chikungunya virus replication[J]. Bioorg Med Chem, 2017, 25: 327-337. |
| [42] |
Strauss JH, Strauss EG. The alphaviruses - gene-expression, replication, and evolution[J]. Microbiol Rev, 1994, 58: 806. DOI:10.1128/MMBR.58.4.806-806.1994 |
| [43] |
Hyde JL, Gardner CL, Kimura T, et al. A viral RNA structural element alters host recognition of nonself RNA[J]. Science, 2014, 343: 783-787. |
| [44] |
Feibelman KM, Fuller BP, Li L, et al. Identification of small molecule inhibitors of the Chikungunya virus nsP1 RNA capping enzyme[J]. Antiviral Res, 2018, 154: 124-131. DOI:10.1016/j.antiviral.2018.03.013 |
| [45] |
Gigante A, Canela MD, Delang L, et al. Identification of [1, 2, 3]triazolo[4, 5-d]pyrimidin-7(6H)-ones as novel inhibitors of Chikungunya virus replication[J]. J Med Chem, 2014, 57: 4000-4008. |
| [46] |
Delang L, Li C, Tas A, et al. The viral capping enzyme nsP1: a novel target for the inhibition of Chikungunya virus infection[J]. Sci Rep, 2016, 6: 31819. |
| [47] |
Gigante A, Gomez-SanJuan A, Delang L, et al. Antiviral activity of [1, 2, 3]triazolo[4, 5-d]pyrimidin-7(6H)-ones against Chikungunya virus targeting the viral capping nsP1[J]. Antiviral Res, 2017, 144: 216-222. DOI:10.1016/j.antiviral.2017.06.003 |
| [48] |
Bullard-Feibelman KM, Fuller BP, Geiss BJ. A sensitive and robust high-throughput screening assay for inhibitors of the Chikungunya virus nsP1 capping enzyme[J]. PLoS One, 2016, 11: e0158923. |
| [49] |
Feibelman KM, Fuller BP, Li LF, et al. Identification of small molecule inhibitors of the Chikungunya virus nsP1 RNA capping enzyme[J]. Antiviral Res, 2018, 154: 124-131. DOI:10.1016/j.antiviral.2018.03.013 |
| [50] |
Bao HH, Ramanathan AA, Kawalakar O, et al. Nonstructural protein 2 (nsP2) of Chikungunya virus (CHIKV) enhances protective immunity mediated by a CHIKV envelope protein expressing DNA vaccine[J]. Viral Immunol, 2013, 26: 75-83. DOI:10.1089/vim.2012.0061 |
| [51] |
Kumar P, Kumar D, Giri R. Targeting the nsp2 cysteine protease of Chikungunya virus using FDA approved library and selected cysteine protease inhibitors[J]. Pathogens, 2019, 8: E128. |
| [52] |
Friesner RA, Murphy RB, Repasky MP, et al. Extra precision glide: docking and scoring incorporating a model of hydrophobic enclosure for protein-ligand complexes[J]. J Med Chem, 2006, 49: 6177-6196. DOI:10.1021/jm051256o |
| [53] |
Vanommeslaeghe K, Hatcher E, Acharya C, et al. CHARMM general force field: a force field for drug-like molecules compatible with the CHARMM all-atom additive biological force fields[J]. J Comput Chem, 2010, 31: 671-690. |
| [54] |
Hanwell MD, Curtis DE, Lonie DC, et al. Avogadro: an advanced semantic chemical editor, visualization, and analysis platform[J]. J Cheminform, 2012, 4: 17. DOI:10.1186/1758-2946-4-17 |
| [55] |
Saha A, Acharya BN, Priya R, et al. Development of nsP2 protease based cell free high throughput screening assay for evaluation of inhibitors against emerging Chikungunya virus[J]. Sci Rep, 2018, 8: 10831. |
| [56] |
Perez-Perez MJ, Delang L, Ng LFP, et al. Chikungunya virus drug discovery: still a long way to go?[J]. Expert Opin Drug Discov, 2019, 14: 855-866. DOI:10.1080/17460441.2019.1629413 |
| [57] |
Gotte B, Liu L, McInerney GM. The enigmatic alphavirus non-structural protein 3 (nsP3) revealing its secrets at last[J]. Viruses-Basel, 2018, 10: 105. |
| [58] |
Abraham R, Hauer D, McPherson RL, et al. ADP-ribosyl-binding and hydrolase activities of the alphavirus nsP3 macrodomain are critical for initiation of virus replication[J]. Proc Natl Acad Sci U S A, 2018, 115: E10457-E10466. DOI:10.1073/pnas.1812130115 |
| [59] |
Seyedi SS, Shukri M, Hassandarvish P, et al. Computational approach towards exploring potential anti-Chikungunya activity of selected flavonoids[J]. Sci Rep, 2016, 6: 24027. DOI:10.1038/srep24027 |
| [60] |
Kumar D, Kumari K, Jayaraj A, et al. Development of a theoretical model for the inhibition of nsP3 protease of Chikungunya virus using pyranooxazoles[J]. J Biomol Struct Dyn, 2019. DOI:10.1080/07391102.2019.1650830 |
| [61] |
Tardif KD, Waris G, Siddiqui A. Hepatitis C virus, ER stress, and oxidative stress[J]. Trends Microbiol, 2005, 13: 159-163. |
| [62] |
Wada Y, Orba Y, Sasaki M, et al. Discovery of a novel antiviral agent targeting the nonstructural protein 4 (nsP4) of Chikungunya virus[J]. Virology, 2017, 505: 102-112. |
| [63] |
Salminen A, Wahlberg JM, Lobigs M, et al. Membrane fusion process of Semliki Forest virus. Ⅱ: Cleavage-dependent reorganization of the spike protein complex controls virus entry[J]. J Cell Biol, 1992, 116: 349-357. DOI:10.1083/jcb.116.2.349 |
| [64] |
Li L, Jose J, Xiang Y, et al. Structural changes of envelope proteins during alphavirus fusion[J]. Nature, 2010, 468: 705-708. DOI:10.1038/nature09546 |
| [65] |
Rashad AA, Keller PA. Structure based design towards the identification of novel binding sites and inhibitors for the Chikungunya virus envelope proteins[J]. J Mol Graph Model, 2013, 44: 241-252. |
| [66] |
Agarwal G, Gupta S, Gabrani R, et al. Virtual screening of inhibitors against envelope glycoprotein of Chikungunya virus: a drug repositioning approach[J]. Bioinformation, 2019, 15: 439-447. |
| [67] |
Ching KC, Ng LFP, Chai CLL. A compendium of small molecule direct-acting and host-targeting inhibitors as therapies against alphaviruses[J]. J Antimicrob Chemoth, 2017, 72: 2973-2989. DOI:10.1093/jac/dkx224 |
| [68] |
Nguyen M, Marcellus RC, Roulston A, et al. Small molecule obatoclax (GX15-070) antagonizes MCL-1 and overcomes MCL-1-mediated resistance to apoptosis[J]. Proc Natl Acad Sci U S A, 2007, 104: 19512-19517. DOI:10.1073/pnas.0709443104 |
| [69] |
Varghese FS, Rausalu K, Hakanen M, et al. Obatoclax inhibits alphavirus membrane fusion by neutralizing the acidic environment of endocytic compartments[J]. Antimicrob Agents Chemother, 2017, 61: e02227-16. |
| [70] |
Varghese FS, Thaa B, Amrun SN, et al. The antiviral alkaloid berberine reduces Chikungunya virus-induced mitogen-activated protein kinase signaling[J]. J Virol, 2016, 90: 9743-9757. DOI:10.1128/JVI.01382-16 |
| [71] |
Bourjot M, Delang L, Nguyen VH, et al. Prostratin and 12-O-tetradecanoylphorbol 13-acetate are potent and selective inhibitors of Chikungunya virus replication[J]. J Nat Prod, 2012, 75: 2183-2187. |
| [72] |
Kaur P, Thiruchelvan M, Lee RCH, et al. Inhibition of Chikungunya virus replication by harringtonine, a novel antiviral that suppresses viral protein expression[J]. Antimicrob Agents Chemother, 2013, 57: 155-167. DOI:10.1128/AAC.01467-12 |
| [73] |
Paeshuyse J, Dallmeier K, Neyts J. Ribavirin for the treatment of chronic hepatitis C virus infection: a review of the proposed mechanisms of action[J]. Curr Opin Virol, 2011, 1: 590-598. |
| [74] |
Debing Y, Emerson SU, Wang YJ, et al. Ribavirin inhibits in vitrohepatitis E virus replication through depletion of cellular GTP pools and is moderately synergistic with alpha interferon[J]. Antimicrob Agents Chemother, 2014, 58: 267-273. DOI:10.1128/AAC.01795-13 |
| [75] |
Briolant S, Garin D, Scaramozzino N, et al. In vitro inhibition of Chikungunya and Semliki Forest viruses replication by antiviral compounds: synergistic effect of interferon-alpha and ribavirin combination[J]. Antiviral Res, 2004, 61: 111-117. DOI:10.1016/j.antiviral.2003.09.005 |
| [76] |
Rada B, Dragun M. Antiviral action and selectivity of 6-azauridine[J]. Ann N Y Acad Sci, 1977, 284: 410-417. DOI:10.1111/j.1749-6632.1977.tb21977.x |
| [77] |
Crutcher WA, Moschella SL. Double-blind controlled crossover high-dose study of azaribine in psoriasis[J]. Br J Dermatol, 1975, 92: 199-205. |
| [78] |
Furuta Y, Gowen BB, Takahashi K, et al. Favipiravir (T-705), a novel viral RNA polymerase inhibitor[J]. Antiviral Res, 2013, 100: 446-454. |
| [79] |
Delang L, Segura Guerrero N, Tas A, et al. Mutations in the Chikungunya virus non-structural proteins cause resistance to favipiravir (T-705), a broad-spectrum antiviral[J]. J Antimicrob Chemother, 2014, 69: 2770-2784. |
| [80] |
Delang L, Abdelnabi R, Neyts J. Favipiravir as a potential countermeasure against neglected and emerging RNA viruses[J]. Antiviral Res, 2018, 153: 85-94. |
| [81] |
Singh A, Kumar A, Uversky VN, et al. Understanding the interactability of Chikungunya virus proteins via molecular recognition feature analysis[J]. Rsc Adv, 2018, 8: 27293-27303. |
 2020, Vol. 55
2020, Vol. 55


