2. 山西农业大学园艺学院, 山西 晋中 030801;
3. 内蒙古医科大学药学院, 内蒙古 呼和浩特 010059
2. College of Horticulture, Shanxi Agricultural University, Jinzhong 030801, China;
3. College of Pharmacy, Inner Mongolia Medical University, Hohhot 010059, China
据2015年版《中华人民共和国药典》记载, 正品药用黄芪为豆科植物蒙古黄芪Astragalus membranaceus (Fisch.) Bge. var. mongholicus (Bge.) Hsiao或膜荚黄芪Astragalus membranaceus (Fisch.) Bge.的干燥根[1]。迄今已有2 000多年的药用历史, 为常用大宗药材, 属于补气药中的一种要药。具有补气健脾、升阳举陷、益卫固表、利尿消肿、托毒生肌的功效[1]。目前以黄芪为原料的中成药多达200余种, 素有“十药八芪”之称。因此, 在医学领域引起了越来越多的关注, 并在临床治疗中得到了广泛的应用。正是这种强劲的需求, 致使近年来黄芪野生资源几近枯竭, 栽培黄芪占据商品黄芪的绝大部分。但在长期的人工栽培与引种驯化过程中, 黄芪种间杂交现象普遍, 其种质资源混杂严重, 且一些混伪品因利益也常流入市场, 影响了正品黄芪的质量和临床疗效。目前市场上多以同科植物红芪(Hedysarum polybotrys Hand.-Mazz.)、蓝花棘豆(Oxytropis coerulea (pall.) DC.)、锦鸡儿(Caragana sinica (Buóhoz) Rehd.)、及锦葵科植物圆叶锦葵(Malva rotundifolia L.)、蜀葵(Althaea rosea L.)等的干燥根来冒充正品药用黄芪[2]。这些混伪品的根茎在外形上与正品黄芪具有相似的特征, 但在药用功效上及主要化学成分上远落后于正品药用黄芪, 部分混伪品甚至还有毒性, 若在临床药用上混用, 难以保障人们的用药安全[2]。因此对药用黄芪植物进行准确鉴别, 对于药用黄芪的质量控制、安全合理利用及开展相关的研究具有重要意义。
DNA条形码(DNA barcoding)分子鉴定技术是依靠物种基因组上一段标准的、具有独特变异的DNA序列来快速、可靠和有效地识别物种的一种分子生物技术[3]。是传统形态学鉴定和理化鉴定方法的有效补充, 广泛运用于真菌属种[4]、植物基源鉴别[5]等。其中ITS2 (internal transcribed spacer)序列作为DNA条形码中的一种, 位于5.8S和28S核rDNA基因之间。该序列长度短, 仅有200~300 bp, 易扩增, 且有良好的物种鉴定能力, 适用于属、种的鉴定[6]。是目前药用植物鉴定、遗传多样性研究、系统演化分析和植物分类常用的分子标记之一[7, 8]。此外相对于ITS2序列(一级结构), ITS2的二级结构是由RNA单链自身回折而形成的规范配对(GC, AU)和不规范但稳定(GU)及不稳定(AC)的未配对碱基交替而成的茎环结构组成[9, 10]。这些配对和非配对的ITS2结构提供了额外的分子形态特征, 可以有效提高分类效率, 具有一定的系统分类价值[11-14]。目前关于黄芪及其混伪品的鉴定研究主要集中在ITS/ITS2核酸序列的分析或进行简单的二级结构预测[2, 15, 16], 但尚无有关结合ITS2核酸序列信息及其二级结构信息进行亲缘关系鉴定方面的研究。
本实验对道地产区山西、内蒙古和吉林等地药用黄芪样品的ITS2序列及其二级结构进行分析, 并从GenBank下载黄芪混伪品, 研究其变异位点、亲缘关系及构建系统发育树, 并在此基础上评价ITS2序列及其二级结构对药用黄芪及混伪品的鉴定能力, 为正品黄芪种质及其易混伪品的鉴定提供一定依据。
材料与方法材料 本研究共收集了32份药用黄芪材料, 其中蒙古黄芪22份, 包括山西代县4份(野生)、山西庞泉沟4份(野生)、吉林珲春6份(野生)以及内蒙古满洲里8份(野生); 膜荚黄芪10份, 包括山西浑源5份(仿野生)及内蒙赤峰5份(野生), 样品均由刘德旺教授鉴定; 另外, 从GenBank数据库下载正品药用黄芪的ITS2序列4条(蒙古黄芪2条、膜荚黄芪2条)及其常见混伪品的ITS2序列12条(蓝花棘豆3条、锦鸡儿3条、红芪3条、蜀葵3条), 一共得到48条ITS2序列用于本次分析。详细信息见表 1、表 2。
| Table 1 Astragalus material information |
| 表 2 GenBank download sample information |
样品DNA的提取 本实验采用CTAB法提取所有黄芪样品基因组DNA, 所得DNA用1%的琼脂糖凝胶电泳进行质量检测, 并用核酸蛋白测定仪进行纯度检测, -20 ℃冰箱中保存备用。
PCR扩增及测序 本研究选用ITS2国际通用引物[6], ITS2扩增引物由北京擎科生物科技有限公司合成。正向引物序列为: 5′-ATGCGATACTTGGTGT GAAT-3′, 反向引物序列为: 5′-GACGCTTCTCCAGA CTACAAT-3′。PCR反应体系为30 μL, 其中2×Taq-mix 15 μL, ddH2O 12 μL, DNA模板及上下引物各1 μL。PCR反应条件为: 95 ℃预变性5 min; 94 ℃变性45 s, 55 ℃退火40 s, 72 ℃延伸10 min (30个循环); 72 ℃延伸10 min。在4 ℃冰箱中保存备用。PCR产物由上海美吉常规测序公司进行双向测序。
数据分析 利用CExpress软件对测序结果进行人工拼接校对, 去除两端引物区及低质量区后, 得到一条完整序列; 将完整序列导入ITS2数据库中(http://its2.bioapps.biozentrum.uni-wuerzburg.de/), 基于隐马尔可夫模型(HMMs)[17]注释的方法去除两端5.8S和28S区域, 即得到标准的ITS2间隔区序列。利用EditSeq软件对48个样本ITS2序列的碱基含量及序列特征进行统计分析; 通过DNAman软件对本实验所有样品的48条标准ITS2间隔区序列进行同源性比对, 分析其变异位点; 对48个样本按种进行分组, 通过MEGA 7[18]软件基于K2-P (Kimura 2-parameter)距离模型计算种内种间的遗传距离, 构建Neighbor-Joining (NJ)系统发育树, 并用bootstrap1000次检验各分支置信度, 评价分支系统发育关系的可靠性; 利用在线网站ITS2数据库[19]对各样本进行二级结构预测, 并针对所有黄芪样本的核酸序列及二级结构的联合矩阵导入4Sale1.7.1软件[20], 进行了共有二级结构的比对分析; 利用ProfdistS软件[21]对得到的黄芪及其混伪品的核酸序列及二级结构的联合矩阵构建剖面邻接PNJ (profile neighbor joining)系统发育树, 并用bootstrap1000次检验各分支置信度。
结果与分析 1 ITS2序列特征及差异性本实验对药用黄芪及其混伪品的ITS2序列特征分别进行分析, 结果如表 3所示:蒙古黄芪、膜荚黄芪及蓝花棘豆的平均ITS2序列长度均为216 bp, G+C含量分别为50.46%、50.00%、51.73%。锦鸡儿、红芪、蜀葵的ITS2序列长度分别为223、220、233 bp, G+C含量为49.21%、54.21%、53.22%。即两种药用黄芪与蓝花棘豆的ITS2序列相似性最高, 与蜀葵存在较大的差异。通过MEGA7.0软件对药用黄芪及其混伪品ITS2序列进行比对分析, 得到种内变异信息位点(表 3), 结果显示蒙古黄芪、膜荚黄芪、蓝花棘豆、锦鸡儿、红芪、蜀葵的种内变异位点数分别为: 27、4、3、22、6、0, 蒙古黄芪相比于膜荚黄芪具有较多变异位点(表 4)。
| Table 3 ITS2 sequence characteristics of Astragalus and its adulterants |
| Table 4 Intraspecific variation sites of Astragalus. *Represents the same base as the first line |
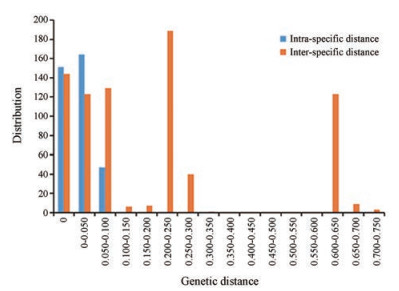
基于MEGA7软件中K2-P (Kimura2-parameter)距离模型, 计算黄芪及其混伪品的种内种间遗传距离可知(表 5), 蒙古黄芪、膜荚黄芪的平均种内遗传距离分别为0.001、0.014, 蜀葵种内遗传距离最小为0, 锦鸡儿种内遗传距离最大为0.073;在各个物种间, 蒙古黄芪与膜荚黄芪间的遗传距离为0.008, 蒙古黄芪、膜荚黄芪与蓝花棘豆间的遗传距离最小, 为0.068、0.066, 与蜀葵遗传距离最大(0.635、0.637)。通过对种间、种内遗传距离柱状分布图得出(图 1), 药用黄芪及其混伪品种内遗传距离主要集中在0.1以内, 而种间遗传距离分布较散, 主要分布在0.000~0.750之间。
| Table 5 Intra-specific and inter-specific genetic distanceof Astragalus and its adulterants. The diagonal line is theintra-specific genetic distance, and the lower triangle is the inter-specificgenetic distance |
|
Figure 1 Distribution map of intra-specific and inter-specific genetic distance of Astragalus and its adulterants |
本实验对所采集的32份药用黄芪的ITS2序列及二级结构序列导入4Sale1.7.1软件中, 得到共有二级结构, 如图 2所示, 其为典型的一个环四个螺旋区(臂)的结构, 与其他臂相比, 臂Ⅲ最长, 其次为臂Ⅰ、Ⅱ、Ⅳ; 臂Ⅱ的结构最保守, 且存在一个高度保守的嘧啶-嘧啶错配的凸起; 臂Ⅳ的变异最大; 臂Ⅲ的5′端近顶端位置序列保守性最高, 存在UGGU (或UGGU、UGG、GGU)的部分; 中间的主环相对较保守, 且富含嘌呤。此ITS2的二级结构特征与多数真核生物的ITS2二级结构相一致。

|
Figure 2 The common secondary structure of the ITS2 for all Astragalus samples. The color of the base in the graph from red to green indicates that its conservatism increases gradually |
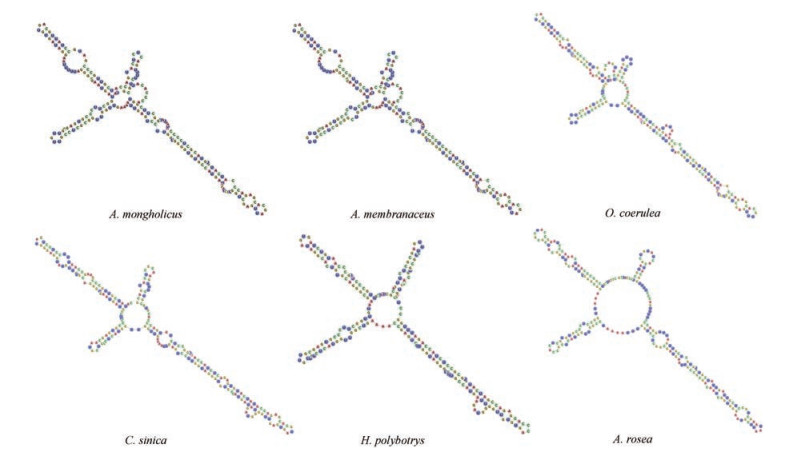
通过对两种药用黄芪和4种混伪品的ITS2二级结构进行预测, 结果如图 3所示:在臂Ⅰ中, 两种黄芪有1个发卡环和1个内环, 而其他4种混伪品均有1个发卡环和2个内环; 在臂Ⅱ中, 药用黄芪、红芪及蜀葵存在嘧啶-嘧啶错配的内环, 且比蓝花棘豆、锦鸡儿的臂Ⅱ相对更长; 在臂Ⅲ、Ⅳ上, 臂的长度、环的数量以及臂的位置上在各个物种上都存在较大的差异, 如红芪的臂Ⅲ中内环较少, 只有1个, 两种药用黄芪以及蓝花棘豆都有4个内环(突环), 锦鸡儿与蜀葵有5个内环(突环), 红芪的臂Ⅳ相对较长, 蓝花棘豆与蜀葵的较短, 且不存在内环或突环。
|
Figure 3 Prediction of secondary structures of Astragalus and its adulterants |
本实验利用ITS2序列(一级结构)和ITS2序列与其二级结构联合矩阵, 分别构建了药用黄芪及其混伪品的的系统发育树。在基于ITS2序列(一级结构)构建的NJ邻接树中显示(图 4A), 药用正品黄芪与蓝花棘豆分为一支, 锦鸡儿、红芪、蜀葵聚为另一支。系统发育树中支持率在50%、70%、90%以上的支系分别为14、8、7个。基于联合ITS2序列与其二级结构构建的PNJ树显示(图 4B), 蜀葵、红芪、锦鸡儿与蓝花棘豆与两种药用黄芪可以明显区分开。两种药用黄芪基本按照不同来源进行了进一步的分类, 其中来自山西浑源的膜荚黄芪(MHY1-5)与吉林哈达门的蒙古黄芪(GHD2-5)聚为一支, 来自内蒙古满洲里的蒙古黄芪(GMZ1-8)与来自内蒙古赤峰林场的膜荚黄芪(MLC1-4)的亲缘关系较近。系统发育树中支持率在50%、70%、90%以上的支系分别为45、25、5个。相比于ITS2序列构建的系统发育树, 其在准确率和支系的支持率上都有所提高。

|
Figure 4 Phylogenetic tree of Astragalus and its adulterants. A: Neighbour joining (NJ) tree built based on the ITS2 sequence; B: Profile neighbour joining (PNJ) phylogenetic tree based on ITS2 sequence and secondary structure joint matrix |
DNA条形码技术因其不受外界因素、生物发育阶段或器官组织的影响, 为基因水平的鉴定提供了一定的基础。目前国际热点的植物DNA条形码候选序列主要包括来自叶绿体基因组的psbA-trnH、matK、rbcL序列以及核基因组ITS2、ITS序列等, 相比于其他4种DNA条形码, ITS2序列因其具有适宜扩增和测序的长度、高度的变异性和相对保守的二级结构、鉴定成功率高等优点, 成为解决较低分类阶元植物的分子鉴定及系统发育问题的有效工具之一[12, 22]。其扩增效率以及鉴定能力已在多个科属中如蔷薇科、菊科、贝母属、百合属、薄荷属等得到验证[23-26]。如Zheng等[23]利用ITS2及psbA-trnH序列对13种贝母属植物通过遗传距离的计算以及系统进化树的构建, 确定了ITS2在贝母属植物的鉴定比psbA-trnH条形码有更大的优势。Han等[24]利用ITS2、psbA-trnH、rbcL和matK四种DNA条形码对唇形科、百合科、芸香科、蔷薇科和菊科药用植物进行了鉴定研究, 表明不管是在扩增成功率还是在鉴定成功率上, ITS2序列要优于其他3种DNA条形码。在本研究中, 对药用黄芪及其4种常见混伪品的ITS2序列进行鉴定分析得到:药用黄芪的ITS2序列特征基本一致, 与蓝花棘豆的ITS2序列的长度、GC含量的相似性最高, 与蜀葵存在较大的差异; 此外对两种药用黄芪及其混伪品的种内、种间遗传距离分析以及构建的NJ系统进化树均显示, 药用黄芪与蓝花棘豆亲缘关系最近, 与蜀葵的亲缘关系最远, 此结果与Shang等[16]利用ITS序列对药用黄芪及其混伪品的亲缘关系鉴定结果一致。表明ITS2序列在药用黄芪及其混伪品鉴定中可以提供一定的依据。
ITS2序列虽被作为植物鉴定的核心DNA条形码, 但由于其存在核酸序列变异较大、缺乏足够的系统发育信息等缺点, 导致其对部分植物分类鉴定中在分辨率、支持率等方面相对较低。如在杜鹃属(Rhododendron L.)、葶苈属(Draba L.)的分辨率仅为21.9%、27.2%[27]。而ITS2二级结构由于其在真核生物内相对较保守, 具有一致的二级结构模型, 结构中保守的“图形”提供了序列比对的模板, 为高分类阶元的序列比对提供了更真实的比对关系, 所以相对于一级结构来说, 二级结构的加入可以更直观地揭示物种进化史[28-30]。且目前已有研究认为加入ITS2二级结构的信息进行比对, 对一级核酸序列的比对有修正优化作用, 从而提高系统发育树重建的准确性和稳健性[12, 31]。
因此, 本实验在ITS2核酸序列的基础上, 对药用黄芪及其混伪品的二级结构进行了预测, 并构建了基于联合ITS2一级序列与二级结构的系统进化树, 研究结果显示, 药用黄芪及其混伪品的二级结构符合大多数真核生物的一环四臂的拓扑结构特点(图 2)。两种药用黄芪具有基本一致的二级结构, 但与混伪品的二级结构上如茎、环的数量、大小等存在较大的差异, 这是由于其存在配对与非配对碱基通过折叠形成的, 正是因为这些差异为药用黄芪及其混伪品的鉴定提供了额外的系统分类信息; 此外, 在本实验中, 联合ITS2序列与二级结构共同构建的剖面邻接(PNJ)发育树, 除了得到与单独ITS2序列构建的系统发育树基本一致的拓补结构外, 还对两种药用黄芪与混伪品的亲缘关系得到了进一步的体现, 将其单独分为不同的支系, 并解决了梳子状的拓扑结构, 使得系统发育树末端分支变多, 且在一些支系的支持率上也有所提高(图 4)。对于两种亲缘关系很近的药用黄芪进一步的分类(图 4B), 其分类结果首先是先按照地理来源, 后以物种进行聚类, 此结果与Zhang[32]利用ISSR分子标记以及Wang等[33]利用RAPD分子标记对两种药用黄芪的分类结果一致。但在本实验中来自山西浑源的膜荚黄芪(MHY1-5)与吉林哈达门的蒙古黄芪(GHD2-5)聚为一支, 来自内蒙古满洲里的蒙古黄芪(GMZ1-8)与来自内蒙古赤峰的膜荚黄芪(MLC1-4)的亲缘关系较近。Zhao[34]对黄芪区系地理分布研究得到内蒙古北部地区同时分布着蒙古黄芪和膜荚黄芪两种物种, 且认为内蒙古北部满洲里地区所分布的物种区别于蒙古黄芪与膜荚黄芪, 但其采集的标本叶片大小、数量、排列等观察研究认为其和蒙古黄芪相近, 应将其定为一个新种-北蒙古黄芪。在本实验中, 把此地区所采集的物种以蒙古黄芪进行分析, 是与膜荚黄芪优先聚类, 理论上是与Zhao的研究有一定差别, 推测原因可能是由于同域分布的物种在长期的历史演化过程中发生了一定的基因渐渗, 基因溯祖可能更偏向于膜荚黄芪; 且表观特征会因为生境的变化而有所改变, 仅用表型数据确定其为新种还有待商榷, 因此此区域所分布的物种分类还有待于进一步考证。
通过对药用黄芪及其混伪品ITS2序列变异分析、遗传距离的计算、以及结合ITS2二级结构信息构建系统发育树等多方面的分析, 认为ITS2序列可准确、有效地区分药用黄芪及其混伪品, 且二级结构的加入, 对一级核酸序列的比对起到修正优化作用, 同时构建的系统发育树不仅包含了基于一级结构得到的所有结论, 而且也解决了梳子状拓扑结构, 增加了ITS2作为药用植物DNA条形码鉴定的可靠性。为药用黄芪及其混伪品的分类与准确鉴定提供了一定的科学依据。
| [1] |
Chinese Pharmacopoeia Committee.Pharmacopoeia of the People's Republic of China. Part 1 (中华人民共和国药典: 一部)[S]. Beijing: China Medical Science Press, 2015: 302.
|
| [2] |
Cui ZH, Li Y, Yuan QJ, et al. Molecular identification of Astragali Radix and its adulterants by ITS sequences[J]. China J Chin Mater Med (中国中药杂志), 2012, 37: 3773-3776. |
| [3] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218 |
| [4] |
Yan DH, Gao Q, Sun XM, et al. ITS2 sequence-structure phylogeny reveals diverse endophytic Pseudocercospora fungi on poplars[J]. Genetica, 2018, 146: 187-198. DOI:10.1007/s10709-018-0011-5 |
| [5] |
Liu Z, Gao Y, Zhou J. Molecular authentication of the medicinal species of Ligusticum (Ligustici Rhizoma et Radix, "Gao-ben") by integrating non-coding internal transcribed spacer 2 (ITS2) and its secondary structure[J]. Front Plant Sci, 2019, 10: 429. DOI:10.3389/fpls.2019.00429 |
| [6] |
Chen S, Yao H, Han J, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5: e8613. DOI:10.1371/journal.pone.0008613 |
| [7] |
Liu Z, Zeng H, Yang D, et al. Applying DNA barcodes for identification of plant species in the family Araliaceae[J]. Gene, 2012, 499: 76-80. DOI:10.1016/j.gene.2012.02.016 |
| [8] |
Kim WJ, Moon BC, Yang S, et al. Rapid authentication of the herbal medicine plant species Aralia continentalis Kitag. and Angelica biserrata C.Q. Yuan and R.H. Shan using ITS2 sequences and multiplex-SCAR markers[J]. Molecules, 2016, 21: 270. DOI:10.3390/molecules21030270 |
| [9] |
Elgavish T, Cannone JJ, Lee JC, et al. A:A and A:G base-pairs at the ends of 16S and 23S rRNA helices[J]. J Mol Biol, 2001, 310: 735-753. DOI:10.1006/jmbi.2001.4807 |
| [10] |
Leontis NB, Westhof E. Geometric nomenclature and classification of RNA base pairs[J]. RNA, 2001, 7: 499-512. DOI:10.1017/S1355838201002515 |
| [11] |
Telford MJ, Wise MJ, Gowri-Shankar V. Consideration of RNA secondary structure significantly improves likelihood-based estimates of phylogeny: examples from the bilateria[J]. Mol Biol Evol, 2005, 22: 1129-1136. DOI:10.1093/molbev/msi099 |
| [12] |
Keller A, Förster F, Müller T, et al. Including RNA secondary structures improves accuracy and robustness in reconstruction of phylogenetic trees[J]. Biol Direct, 2010, 5: 4. DOI:10.1186/1745-6150-5-4 |
| [13] |
Coleman AW. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure[J]. Nucleic Acids Res, 2007, 35: 3322-3329. DOI:10.1093/nar/gkm233 |
| [14] |
Prasad PK, Tandon V, Biswal DK, et al. Phylogenetic reconstruction using secondary structures and sequence motifs of ITS2 rDNA of Paragonimus westermani (Kerbert, 1878) Braun, 1899 (Digenea: Paragonimidae) and related species[J]. BMC Gen, 2009, 10(Suppl3): S25. |
| [15] |
Gao T, Yao H, Ma XY, et al. Identification of Astragalus plants in China using the region ITS2[J]. Mod Tradit Chin Med Mater Med-World Sci Technol (世界科学技术-中医药现代), 2010, 12: 222-227. |
| [16] |
Shang HH, Wu Q, Sun YP. Differential analysis of Astragalus and ITS mix adulterants by DNA molecular identification[J]. Chin J Coal Ind Med (中国煤炭工业医学杂志), 2016, 19: 72-75. |
| [17] |
Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430: 50-57. DOI:10.1016/j.gene.2008.10.012 |
| [18] |
Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33: 1870-1874. DOI:10.1093/molbev/msw054 |
| [19] |
Ankenbrand MJ, Keller A, Wolf M, et al. ITS2 database Ⅴ: twice as much[J]. Mol Biol Evol, 2015, 32: 3030-3032. DOI:10.1093/molbev/msv174 |
| [20] |
Seibel PN, Müller T, Dandekar T, et al. 4SALE-A tool for synchronous RNA sequence and secondary structure alignment and editing[J]. BMC Bioinf, 2006, 7: 498. DOI:10.1186/1471-2105-7-498 |
| [21] |
Wolf M, Ruderisch B, Dandekar T, et al. ProfdistS: (Profile-) distance based phylogeny on sequence-structure alignments[J]. BMC Bioinf, 2008, 24: 2401-2402. DOI:10.1093/bioinformatics/btn453 |
| [22] |
Poczai P, Varga I, Hyvönen J. Internal transcribed spacer (ITS) evolution in populations of the hyperparasitic European mistletoe pathogen fungus, Sphaeropsis visci (Botryosphaeriaceae): the utility of ITS2 secondary structures[J]. Gene, 2015, 558: 54-64. DOI:10.1016/j.gene.2014.12.042 |
| [23] |
Zheng H, Deng KY, Chen AQ, et al. Molecular identification and genetic relationship of Fritillaria cirrhosa and related species based on DNA barcode[J]. Acta Pharm Sin (药学学报), 2019, 54: 2326-2334. |
| [24] |
Han J. Identification of 76 Varieties of Chinese Herbal Medicine in the Lingnan Area by DNA Barcoding Technique (76种岭南中草药的DNA条形码研究)[D]. Guangzhou: Guangzhou University of Chinese Medicine, 2016.
|
| [25] |
Zheng SH, Li YK, Ren WG, et al. Molecular identification in genus of Lilium based on DNA barcoding[J]. Acta Pharm Sin (药学学报), 2014, 49: 1730-1738. |
| [26] |
Wang FF, Liu J, He FY, et al. Comparative study for morphological identification, chemical component analysis, and DNA barcode determination of three plants and their varieties in Genus Mentha[J]. Acta Pharm Sin (药学学报), 2019, 54: 2083-2088. |
| [27] |
Yao H, Song J, Liu C, et al. Use of ITS2 region as the universal DNA barcode for plants and animals[J]. PLoS One, 2010, 5: e13102. DOI:10.1371/journal.pone.0013102 |
| [28] |
Zhao TL. Study on the Sequence and Secondary Structure of Haplorchis taichui 5.8S rRNA from Guangxi广西扇棘单睾吸虫5.8S rRNA序列和二级结构研究)[D]. Guilin: Guangxi Medical University, 2007
|
| [29] |
Wang Y, Liu QY, Li G, et al. Prediction and comparative analysis of the secondary structure of ITS2 in Astereae (Asteraceae)[J]. J South-Cent Univ Natl (中南民族大学学报), 2015, 24: 28-36. |
| [30] |
Liu L. Phylogenetic Relationships of Brassicaceae Species Inferred from Various Genomic Sequences (基于不同基因组序列的十字花科分子系统发育研究)[D]. Wuhan: Wuhan University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10486-1012397353.htm
|
| [31] |
Biffin E, Harrington MG, Crisp MD, et al. Structural partitioning, paired-sites models and evolution of the ITS transcript in Syzygium and Myrtaceae[J]. Mol Phylogenet and Evol, 2007, 43: 124-139. DOI:10.1016/j.ympev.2006.08.013 |
| [32] |
Zhang R. Analysis on Genetic Diversity of Radix Astragali ISSR Marker (药用黄芪ISSR遗传多样性分析)[D]. Jinzhong: Shanxi Agricultural University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10113-1014416886.htm
|
| [33] |
Wang HY, Chen JX, Xu ZM, et al. Genetic diversity analysis of Astragalus by RAPD[J]. J Sichuan Tradit Chin Med (四川中医), 2010, 28: 56-58. |
| [34] |
Zhao YZ. Taxonomy and floristic geographical distribution of the Chinese medicinal Huangqi[J]. Bull Bot Res (植物研究), 2006, 26: 532-538. |
 2020, Vol. 55
2020, Vol. 55


