中介复合体(mediator complex), 简称中介体, 是一种多蛋白复合物, 与RNA聚合酶II (RNA polymerase II, Pol II)、增强子、细胞核受体(nuclear receptors, NRs)和其他转录因子相互作用, 充当转录“枢纽”, 调控转录的多个方面。中介复合体首次在酵母中发现, 随后从哺乳动物细胞中分离发现[1]。中介复合体是由20多种不同蛋白亚基组成的复合体, 广泛存在于各种真核生物细胞中, 并且与RNA聚合酶一起构成RNA聚合酶II全酶。已经证明中介复合体与众多细胞核受体和转录因子以受体-配体的方式结合, 在代谢基因的转录过程中发挥重要作用。骨骼肌是人体重要的运动组织, 同时也是参与机体能量代谢的重要器官。人体共有600多块骨骼肌, 约占全身重量的40%[2]。骨骼肌是重要的能量消耗器官, 可通过运动及分泌肌肉因子等调节骨骼肌本身及全身代谢。中介复合体亚基通过参与骨骼肌中调节代谢的关键基因的转录, 调控骨骼肌糖、脂质代谢水平, 从而影响代谢性疾病的发生与发展[3, 4]。
1 中介复合体 1.1 中介复合体的结构和功能哺乳动物中介复合体由30个子单元组成, 可分为4个不同的区域, 分别为头、中、尾和CDK8激酶模块(图 1)。中介复合体中间模块MED1亚基是第一个被识别出的亚基, 又称PBP、TRAP220、DRIP205或RB18A, 是NRs的主要结合靶点, 参与基因转录的调控[5, 6]。中介复合体尾部模块直接与转录激活子作用, 失去一个或多个尾部亚基会影响激活因子依赖的转录, 影响细胞生长[7]。其中, MED23是尾部模块中最大的亚基[8], MED14是中介体的主干, 与中介复合体的3个模块都有联系[9]。
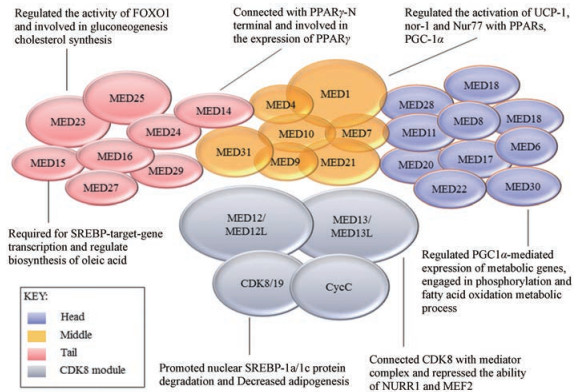
|
Figure 1 Mediator complex composition and regulation of skeletal muscle metabolism. FOXO1: Forkhead box protein O1; PPAR: Peroxisome proliferator-activated receptors; UCP-1: Uncoupling protein 1; Nur 77: Nuclear receptor subfamily 4 group A member 1, NR4A1; PGC1α: PPARγ coactivator-1α; SCREB: Sterol regulatory element-binding protein; NURR-1: Nuclear receptor related factor-1; MEF2: Myocyte enhancer factor 2 |
CDK8激酶子模块由4个亚基组成, 分别是MED12、MED13、CDK8和CycC, 与中介复合体可逆结合, 负责调控中介复合体的转录过程。CDK8激酶既可以通过自身变构与RNA Pol II竞争性结合中介复合体进而抑制转录, 也可以直接促进基因转录, 比如CDK8可通过促进Pol II招募甲状腺激素核受体(thyroid hormone nuclear receptor, TR)靶基因激活依赖TR发生的转录过程[10]。研究发现, 在哺乳动物和其他脊椎动物中, 均存在旁系同源基因激酶模块亚基CDK19、MED12L和MED13L参与转录调控, 但其分子机制尚不清楚。
1.2 中介复合体参与转录因子及信号通路的调控中介复合体在转录调控中发挥重要作用, 转录因子与中介复合体的26个亚基中的一个或多个亚基相连共同控制细胞生长、分化和发育。转录因子是一类存在于细胞内具有识别和结合功能的蛋白质, 可由脂质、类固醇、类维生素A和激素等多种配体激活, 控制机体的发育和新陈代谢[11]。中介复合体与转录因子的活化区域结合, 通过结合后改变自身构象参与转录调控。对于不同的转录因子, 中介复合体的构象改变也是不同的。转录起始复合物(pre-initiation complex, PIC)是一种由转录因子与RNA聚合酶一同结合于转录起始前DNA区域所形成的复合物, 促进基因转录的发生。研究表明, 中介复合体头部模块对PIC的形成至关重要, 它通过与PIC的所有组分相互作用, 调节聚合酶构象并与DNA启动子相互作用影响转录的起始[6]。Carlsten等[12]发现在分裂酵母中MED20的缺失可导致tRNA转录本的异常积累和其他非编码RNA的富集。
中介复合体也参与信号转导过程, 不同的信号通路依赖不同亚基调控基因转录。在人类细胞和秀丽隐杆线虫中, MED15通过参与SREBPs的通路调节脂质稳态[13]。MED12和MED13参与Wnt信号通路, MED23参与RAS/MAPK/ERK信号通路[14]。此外, 中介复合体可以通过自身翻译后修饰转录因子来参与代谢调控, 其机制主要包括中介体亚基的磷酸化和泛素化[15]。
1.3 中介复合体对骨骼肌转录因子的调控作用骨骼肌的代谢功能及肌纤维类型切换均受转录因子调控[16], 骨骼肌中主要表达的转录因子包括过氧化物酶体增殖激活受体(peroxisome proliferator-activated receptors, PPARs)、雌激素相关受体(estrogen-related receptors, ERRs)和甲状腺激素受体(thyroid hormone receptors, TRs)等。在骨骼肌代谢功能上, 中介复合体可通过调控PPAR相关基因的转录, 调节脂质代谢, 影响糖尿病、肥胖的发生发展[4]。PGC1α是核受体的共激活物, PPARβ/δ通过激活PGC1α促进氧化肌肉纤维的形成[17], 骨骼肌中PGC1α可引起慢肌纤维向快肌纤维的转变[18]。MED30参与PGC1α介导的代谢基因的表达, 其调控过程具有心脏和肾脏组织特异性[19]。TR通过调节线粒体基因转录和刺激纤维类型转换, 参与骨骼肌能量代谢[20]。基于体外实验和质谱方法, Belakavadi等[15]发现, 包括MED1在内的几个中介复合体亚基在形成活性转录复合物时经细胞外信号调节激酶磷酸化后引起TR依赖的基因转录增强。
2 中介复合体在骨骼肌代谢过程中的作用机制 2.1 骨骼肌在机体能量代谢中的作用人体运动功能是通过骨骼肌完成的。骨骼肌占哺乳动物总体重的40%, 占健康成人静息代谢率的30%[2]。骨骼肌也是人体最大的糖原储存器官, 是胰岛素刺激条件下处理葡萄糖的主要部位。肌肉收缩将葡萄糖转运体从细胞内转运到质膜和t小管, 增加骨骼肌葡萄糖摄取, 进而控制体内血糖和代谢稳态[21], 在机体能量代谢中发挥重要作用。骨骼肌纤维分为慢肌纤维和快肌纤维两种类型, 二者在收缩蛋白的组成、氧化能力和对产生ATP的底物偏好方面有很大差异。快肌纤维又称“白纤维”, 收缩力大, 氧化能力低, 具有高疲劳性, 并以葡萄糖作为底物通过糖酵解的方式产生ATP; 慢肌纤维收缩力小, 氧化能力高, 具有低疲劳性, 并以脂肪酸作为ATP主要底物[22]。有氧或耐力运动会引起骨骼肌纤维从快肌纤维向慢肌纤维转变。对于胰岛素抵抗患者, 慢肌纤维中I型肌纤维比例减少, IIa型肌纤维比例增加[23]。改善胰岛素抗性训练通过增加肌肉质量和增强快肌纤维的氧化和糖酵解能力来影响骨骼肌代谢[24]。
2.2 中介复合体在骨骼肌葡萄糖代谢过程中的调控机制骨骼肌是葡萄糖摄取的主要来源, 在脂肪组织和肝脏中葡萄糖摄取较少[25]。有多种激酶和肌肉因子参与骨骼肌葡萄糖代谢过程, 如:葡萄糖转运蛋白GLUT4 (glucose transporter member 4)、腺苷酸活化蛋白激酶(AMP-activated protein kinase, AMPK)、钙调素蛋白激酶(Calcium Calmodulin-dependent protein kinase, CaMK)、肝激酶B (liver kinase B1, LKB1)、白介素6 (Interleukin 6, IL-6)和血管生成素样蛋白4 (angiopoietin-like protein 4, ANGPTL4)等。
其中GLUT4是骨骼肌中负责葡萄糖运输的关键葡萄糖转运蛋白, 运输葡萄糖进入肌细胞[26]。运动可通过增强GLUT4转录, 增加骨骼肌GLUT4介导的葡萄糖摄取促进葡萄糖清除[27]。人类GLUT4启动子包含两个高度保守的区域: NRE区域, 位于转录起始位点远端, 与GLUT4增强因子(GLUT4 enhancer factor, GEF)结合; MRE区域, 位于转录起始位点近端, 与肌细胞增强因子2 (myocyte enhancer factor 2, MEF2)结合。MEF2通过调控PPARα和PGC1α的转录活动参与线粒体的生物合成。Krüppel样因子KLF15是肌肉组织中控制GLUT4转录的重要调节因子, 与MEF2A特异性结合, 协同作用激活GLUT4启动子[28]。MEF2活性可被组蛋白去乙酰化酶(histone deacetylase, HDAC)抑制, HDACs参与调控与染色质相关的组蛋白残基乙酰化和去乙酰化的平衡, 这些残基的乙酰化影响转录因子与启动子区域的交互作用[29]。AMPK通过磷酸化II类HDACs, 刺激MEF2依赖的基因表达[5], 进而增加GLUT4表达。CaMK活化可诱导MEF2区域附近组蛋白高度乙酰化, 并增强MEF2与顺式元件的结合[30]。
MED13是中介复合体CDK8激酶子模块的一个组成部分, 通过抑制核受体NURR1和转录因子MEF2的活性来控制骨骼肌胰岛素敏感性、葡萄糖摄取和糖原储存。NURR1是一种细胞核受体, 与MEF2协同作用激活GLUT4启动子。MED13通过调控NURR1基因的表达或与NURR1蛋白直接作用抑制NURR1的活性。骨骼肌MED13缺失增加NURR1表达, 而NURR1过表达促进糖代谢相关基因表达, 促进葡萄糖的摄取和糖原储存, 使骨骼肌对胰岛素作用更加敏感。骨骼肌MED13缺失也会减少肝脏中游离脂肪酸的摄取和脂肪再合成, 这表明MED13亚基介导骨骼肌和肝脏之间的代谢过程[31]。在热量过剩的条件下, 骨骼肌特异性缺失MED13会引起能量物质从肝脏转移到骨骼肌, 从而增强肌肉对葡萄糖的摄取和对肝脂肪变性的抵抗[5]。然而, 果蝇横纹肌MED13缺失却导致果蝇对肥胖和糖尿病的易感性增加[32, 33]。
MED1在骨骼肌葡萄糖代谢中发挥作用。MED1基因敲除的雌性小鼠体内葡萄糖代谢增加, 胰岛素敏感性增强, 并且能够抵抗高脂肪饮食诱导的肥胖[34]。此外, MED1基因敲除小鼠快肌纤维中线粒体数量增加, 线粒体基因表达增加, TR无法激活快肌纤维基因的转录, 引起快肌纤维向慢肌纤维的转换[35]。MED25和MED8也参与调控糖反应过程中的基因转录, 但具体机制仍有待探索[36, 37]。这些研究表明中介复合体通过调控转录因子、激酶和磷酸酶之间的相互作用, 可实现对骨骼肌中GLUT4表达的精细调控。
2.3 中介复合体在骨骼肌脂质代谢过程中的调控机制研究发现, 中介复合体参与脂质代谢的多个方面, 包括脂质合成、脂肪酸氧化、磷脂生物合成和胆汁酸代谢。中介复合体中的多个亚基, 如MED1[3]、MED13[38]、MED14[39]、MED15[40]、MED23[41]、MED30[19]、CDK8[32]、CycC[42]等通过多种机制调节脂质代谢过程。
体外研究表明MED1参与NR4A家族中nor-1和Nur77的活化过程[3]。骨骼肌中NR4A3/nor-1过表达能促进氧化代谢, 增加糖原储存, 增强耐力, 减少脂肪合成。MED1基因敲除的小鼠胚胎成纤维细胞中PPARγ表达缺失。PPARs是脂肪代谢的关键转录因子, MED14将中介复合体与PPARγN端连接, 为PPARγ的转录活动所需[39]。
2.4 中介复合体对骨骼肌线粒体的调控线粒体是骨骼肌进行能量代谢的主要场所之一。骨骼肌代谢高度依赖于线粒体功能, 而线粒体代谢又受多种因素影响, 包括线粒体数量的分裂增殖及影响线粒体功能的线粒体酶活性的调控等。
Amoasii等[31]发现MED13基因敲除小鼠体内线粒体含有完整的功能性电子传递链, 并在含多种底物的反应中均表现为线粒体呼吸能力增强。如在丙酮酸和苹果酸的底物存在下, MED13基因敲除小鼠肝线粒体的氧气消耗率明显增加。MED1基因敲除小鼠快肌纤维中线粒体数量明显增加, 但细胞增殖受到明显抑制, 这与体内编码线粒体蛋白的基因UCP-1和Cidea表达增加以及快肌纤维向慢肌纤维的转换密切相关[35]。此外, MED1基因的下调导致线粒体呼吸谱发生变化, ATP生成显著减少[43]。MED30基因敲除小鼠体内线粒体形态异常, 表现为细胞分裂异常和单个线粒体内形成膜状漩涡, 变化程度不规则。MED30突变小鼠心脏线粒体中电子传递链复合物的酶活性、耗氧量和耦合效率显著下降[44]。目前关于MED30突变的研究多集中于对心肌代谢的影响, 对骨骼肌影响尚无报道。
3 以中介复合体为靶点的药物开发目前尚未有以中介复合体为靶点的药物上市, 但相关靶点的药物设计与开发已逐渐引起重视。类固醇类天然化学产物皮质抑素A (cortistatin A, CA)是最先发现的CDK8/19高亲和力和高选择性的抑制性配体, 其IC50为12 nmol·L-1[45]。研究发现, CA通过靶向CDK8/19抑制急性髓系白血病细胞增殖, 在体内外均具有抗白血病活性。SEL120-34A是基于结构设计发现的一个取代的三环苯并咪唑化合物, 在1~100 nmol·L-1内以剂量依赖的方式与CDK8结合发挥抑制作用, 其IC50为4.4 nmol·L-1[46]。类似的小分子抑制剂还包括氨基喹唑啉类化合物Senexin B和CCT251545[47]。此外, CT251921是一种具有高选择性的, 可口服的, 且对CDK19具有等效亲和力的CDK8抑制剂。Mallinger等[48]发现CT251921可通过抑制CDK8表达减少动物模型中人结直肠癌肿瘤异种移植增殖, 可能为结直肠癌治疗的新靶点。另外, 也可以通过影响参与中介复合体与转录因子结合相关的辅助因子活性从而调控中介复合体与转录因子的相互作用。然而, 目前文献大多报道以针对CDK8激酶为靶点的化合物研究, 关于以其他亚基为靶点的分子设计和药物开发的报道很少, 仍需深入探索, 以期开发出高效、高特异性的新型靶向中介复合体的药物。
4 小结与展望中介复合体可调节多种基因的转录, 尤其是与NRs调控相关的基因, 并且在全身代谢调节过程中具有组织特异性(表 1)[13, 31, 35, 38-42, 44, 49, 50]。深入了解中介复合体亚基在不同组织尤其是骨骼肌能量代谢过程中的作用, 将有利于揭示全身能量代谢的调控机制, 并发现新型抗代谢性疾病的药物靶点。然而, 目前针对中介复合体为靶点开发调节骨骼肌代谢功能的药物研究报道甚少。中介复合体深入作用机制及高分辨率结构信息的研究, 对了解骨骼肌代谢相关的转录因子与中介复合体之间的相互作用, 及相关药物的开发至关重要, 需要进一步研究。
| Table 1 Mediator complex subunits involved in regulation of metabolic processes |
| [1] |
Flanagan PM, Kelleher RJ, Sayre MH, et al. A mediator required for activation of RNA polymerase Ⅱ transcription in vitro[J]. Nature, 1991, 350: 436-438. DOI:10.1038/350436a0 |
| [2] |
Batsis JA, Villareal DT. Sarcopenic obesity in older adults:aetiology, epidemiology and treatment strategies[J]. Nat Rev Endocrinol, 2018, 14: 513-537. DOI:10.1038/s41574-018-0062-9 |
| [3] |
Zhang Y, Xiaoli, Zhao X, et al. The mediator complex and lipid metabolism[J]. J Biochem Pharmacol Res, 2013, 1: 51-55. |
| [4] |
Baskin KK, Winders BR, Olson EN. Muscle as a "mediator" of systemic metabolism[J]. Cell Metab, 2015, 21: 237-248. DOI:10.1016/j.cmet.2014.12.021 |
| [5] |
Amoasii L, Olson EN, Bassel-Duby R. Control of muscle metabolism by the mediator complex[J]. Cold Spring Harb Perspect Med, 2018, 8. |
| [6] |
Poss ZC, Ebmeier CC, Taatjes DJ. The mediator complex and transcription regulation[J]. Crit Rev Biochem Mol Biol, 2013, 48: 575-608. DOI:10.3109/10409238.2013.840259 |
| [7] |
Paul E, Zhu ZI, Landsman D, et al. Genome-wide association of mediator and RNA polymerase Ⅱ in wild-type and mediator mutant yeast[J]. Mol Cell Biol, 2015, 35: 331-342. DOI:10.1128/MCB.00991-14 |
| [8] |
Monté D, Clantin B, Dewitte F, et al. Crystal structure of human mediator subunit MED23[J]. Nat Commun, 2018, 9: 3389. DOI:10.1038/s41467-018-05967-y |
| [9] |
Nozawa K, Schneider TR, Cramer P. Core Mediator structure at 3.4 extends model of transcription initiation complex[J]. Nature, 2017, 545: 248-251. DOI:10.1038/nature22328 |
| [10] |
Fondell JD. The Mediator complex in thyroid hormone receptor action[J]. Biochim Biophys Acta, 2013, 1830: 3867-3875. DOI:10.1016/j.bbagen.2012.02.012 |
| [11] |
Burris TP, Solt LA, Wang Y, et al. Nuclear receptors and their selective pharmacologic modulators[J]. Pharmacol Rev, 2013, 65: 710-778. DOI:10.1124/pr.112.006833 |
| [12] |
Carlsten JO, Zhu X, López MD, et al. Loss of the Mediator subunit Med20 affects transcription of tRNA and other non-coding RNA genes in fission yeast[J]. Biochim Biophys Acta, 2016, 1859: 339-347. DOI:10.1016/j.bbagrm.2015.11.007 |
| [13] |
Yang F, Vought BW, Satterlee JS, et al. An ARC/Mediator subunit required for SREBP control of cholesterol and lipid homeostasis[J]. Nature, 2006, 442: 700-704. DOI:10.1038/nature04942 |
| [14] |
Allen BL, Taatjes DJ. The mediator complex:a central integrator of transcription[J]. Nat Rev Mol Cell Biol, 2015, 16: 155-166. DOI:10.1038/nrm3951 |
| [15] |
Belakavadi M, Pandey PK, Vijayvargia R, et al. MED1 phosphorylation promotes its association with mediator:implications for nuclear receptor signaling[J]. Mol Cell Biol, 2008, 28: 3932-3942. DOI:10.1128/MCB.02191-07 |
| [16] |
Anderson CM, Hu J, Barnes RM, et al. Myocyte enhancer factor 2C function in skeletal muscle is required for normal growth and glucose metabolism in mice[J]. Skelet Muscle, 2015, 5: 7. DOI:10.1186/s13395-015-0031-0 |
| [17] |
Wang YX, Zhang CL, Yu RT, et al. Regulation of muscle fiber type and running endurance by PPARdelta[J]. PLoS Biol, 2004, 2: e294. DOI:10.1371/journal.pbio.0020294 |
| [18] |
Bourdeau JI, Sephton CF, Dutchak PA. Metabolic networks influencing skeletal muscle fiber composition[J]. Front Cell Dev Biol, 2018, 6: 125. DOI:10.3389/fcell.2018.00125 |
| [19] |
Lee YJ, Han ME, Baek SJ, et al. MED30 regulates the proliferation and motility of gastric cancer cells[J]. PLoS One, 2015, 10: e0130826. DOI:10.1371/journal.pone.0130826 |
| [20] |
De Andrade PB, Neff LA, Strosova MK, et al. Caloric restriction induces energy-sparing alterations in skeletal muscle contraction, fiber composition and local thyroid hormone metabolism that persist during catch-up fat upon refeeding[J]. Front Physiol, 2015, 6: 254. |
| [21] |
Goodyear LJ, Kahn BB. Exercise, glucose transport, and insulin sensitivity[J]. Annu Rev Med, 1998, 49: 235-261. DOI:10.1146/annurev.med.49.1.235 |
| [22] |
Schiaffino S, Reggiani C. Fiber types in mammalian skeletal muscles[J]. Physiol Rev, 2011, 91: 1447-1531. DOI:10.1152/physrev.00031.2010 |
| [23] |
Stuart CA, McCurry MP, Marino A, et al. Slow-twitch fiber proportion in skeletal muscle correlates with insulin responsiveness[J]. J Clin Endocrinol Metab, 2013, 98: 2027-2036. DOI:10.1210/jc.2012-3876 |
| [24] |
Duan Y, Li F, Tan B, et al. Metabolic control of myofibers:promising therapeutic target for obesity and type 2 diabetes[J]. Obes Rev, 2017, 18: 647-659. DOI:10.1111/obr.12530 |
| [25] |
Honka MJ, Latva-Rasku A, Bucci M, et al. Insulin-stimulated glucose uptake in skeletal muscle, adipose tissue and liver:a positron emission tomography study[J]. Eur J Endocrinol, 2018, 178: 523-531. DOI:10.1530/EJE-17-0882 |
| [26] |
Richter EA, Hargreaves M. Exercise, GLUT 4, and skeletal muscle glucose uptake[J]. Physiol Rev, 2013, 93: 993-1017. DOI:10.1152/physrev.00038.2012 |
| [27] |
Gurley JM, Griesel BA, Olson AL. Increased skeletal muscle GLUT4 expression in obese mice after voluntary wheel running exercise is posttranscriptional[J]. Diabetes, 2016, 65: 2911-2919. DOI:10.2337/db16-0305 |
| [28] |
Gray S, Feinberg MW, Hull S, et al. The Krüppel-like factor KLF15 regulates the insulin-sensitive glucose transporter GLUT4[J]. J Biol Chem, 2002, 277: 34322-34328. DOI:10.1074/jbc.M201304200 |
| [29] |
Schnyder S, Kupr B, Handschin C. Coregulator-mediated control of skeletal muscle plasticity-a mini-review[J]. Biochimie, 2017, 136: 49-54. DOI:10.1016/j.biochi.2016.12.011 |
| [30] |
Ojuka EO, Goyaram V, Smith JA. The role of CaMKⅡ in regulating GLUT4 expression in skeletal muscle[J]. Am J Physiol Endocrinol Metab, 2012, 303: E322-331. DOI:10.1152/ajpendo.00091.2012 |
| [31] |
Amoasii L, Holland W, Sanchez-Ortiz E, et al. A MED13-dependent skeletal muscle gene program controls systemic glucose homeostasis and hepatic metabolism[J]. Genes Dev, 2016, 30: 434-446. DOI:10.1101/gad.273128.115 |
| [32] |
Nautiyal J, Christian M, Parker MG. Distinct functions for RIP140 in development, inflammation, and metabolism[J]. Trends Endocrinol Metab, 2013, 24: 451-459. DOI:10.1016/j.tem.2013.05.001 |
| [33] |
Harfmann BD, Schroder EA, Kachman MT, et al. Muscle-specific loss of Bmal1 leads to disrupted tissue glucose metabolism and systemic glucose homeostasis[J]. Skelet Muscle, 2016, 6: 12. DOI:10.1186/s13395-016-0082-x |
| [34] |
Youn DY, Xiaoli AM, Pessin JE, et al. Regulation of metabolism by the mediator complex[J]. Biophys Rep, 2016, 2: 69-77. DOI:10.1007/s41048-016-0031-6 |
| [35] |
Chen W, Zhang X, Birsoy K, et al. A muscle-specific knockout implicates nuclear receptor coactivator MED1 in the regulation of glucose and energy metabolism[J]. Proc Natl Acad Sci U S A, 2010, 107: 10196-10201. DOI:10.1073/pnas.1005626107 |
| [36] |
Marette A, Liu Y, Sweeney G. Skeletal muscle glucose metabolism and inflammation in the development of the metabolic syndrome[J]. Rev Endocr Metab Disord, 2014, 15: 299-305. DOI:10.1007/s11154-014-9296-6 |
| [37] |
Seguela-Arnaud M, Smith C, Uribe MC, et al. The mediator complex subunits MED25/PFT1 and MED8 are required for transcriptional responses to changes in cell wall arabinose composition and glucose treatment in Arabidopsis thaliana[J]. BMC Plant Biol, 2015, 15: 215. DOI:10.1186/s12870-015-0592-4 |
| [38] |
Baskin KK, Grueter CE, Kusminski CM, et al. MED13-dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver[J]. EMBO Mol Med, 2014, 6: 1610-1621. DOI:10.15252/emmm.201404218 |
| [39] |
Gr ntved L, Madsen MS, Boergesen M, et al. MED14 tethers mediator to the N-terminal domain of peroxisome proliferator-activated receptor gamma and is required for full transcriptional activity and adipogenesis[J]. Mol Cell Biol, 2010, 30: 2155-2169. DOI:10.1128/MCB.01238-09 |
| [40] |
Kim MJ, Jang IC, Chua NH. The mediator complex MED15 subunit mediates activation of downstream lipid-related genes by the WRINKLED1 transcription factor[J]. Plant Physiol, 2016, 171: 1951-1964. DOI:10.1104/pp.16.00664 |
| [41] |
Chu Y, Gómez RL, Huang P, et al. Liver Med23 ablation improves glucose and lipid metabolism through modulating FOXO1 activity[J]. Cell Res, 2014, 24: 1250-1265. DOI:10.1038/cr.2014.120 |
| [42] |
Zhao X, Feng D, Wang Q, et al. Regulation of lipogenesis by cyclin-dependent kinase 8-mediated control of SREBP-1[J]. J Clin Invest, 2012, 122: 2417-2427. DOI:10.1172/JCI61462 |
| [43] |
Yu J, Xiao Y, Liu J, et al. Loss of MED1 triggers mitochondrial biogenesis in C2C12 cells[J]. Mitochondrion, 2014, 14: 18-25. DOI:10.1016/j.mito.2013.12.004 |
| [44] |
Krebs P, Fan W, Chen YH, et al. Lethal mitochondrial cardiomyopathy in a hypomorphic Med30 mouse mutant is ameliorated by ketogenic diet[J]. Proc Natl Acad Sci U S A, 2011, 108: 19678-19682. DOI:10.1073/pnas.1117835108 |
| [45] |
Cee VJ, Chen DY, Lee MR, et al. Cortistatin A is a high-affinity ligand of protein kinases ROCK, CDK8, and CDK11[J]. Angew Chem Int Ed Engl, 2009, 48: 8952-8957. DOI:10.1002/anie.200904778 |
| [46] |
Rzymski T, Mikula M, Żyłkiewicz E, et al. SEL120-34A is a novel CDK8 inhibitor active in AML cells with high levels of serine phosphorylation of STAT1 and STAT5 transactivation domains[J]. Oncotarget, 2017, 8: 33779-33795. |
| [47] |
Rzymski T, Mikula M, Wiklik K, et al. CDK8 kinase--an emerging target in targeted cancer therapy[J]. Biochim Biophys Acta, 2015, 1854: 1617-1629. DOI:10.1016/j.bbapap.2015.05.011 |
| [48] |
Mallinger A, Schiemann K, Rink C, et al. Discovery of potent, selective, and orally bioavailable small-molecule modulators of the mediator complex-associated kinases CDK8 and CDK19[J]. J Med Chem, 2016, 59: 1078-1101. DOI:10.1021/acs.jmedchem.5b01685 |
| [49] |
Jia Y, Viswakarma N, Reddy JK. Med1 subunit of the mediator complex in nuclear receptor-regulated energy metabolism, liver regeneration, and hepatocarcinogenesis[J]. Gene Expr, 2014, 16: 63-75. DOI:10.3727/105221614X13919976902219 |
| [50] |
Lee JH, Bassel-Duby R, Olson EN. Heart- and muscle-derived signaling system dependent on MED13 and Wingless controls obesity in Drosophila[J]. Proc Natl Acad Sci U S A, 2014, 111: 9491-9496. DOI:10.1073/pnas.1409427111 |
 2020, Vol. 55
2020, Vol. 55


