叶绿体普遍存在于高等植物、藻类等自养生物细胞中, 是光合作用的主要场所, 同时执行着合成淀粉、脂肪酸、蛋白质及色素等功能[1]。作为细胞内半自主性的细胞器, 叶绿体拥有自身完整的一套遗传系统[2], 其遗传物质叶绿体基因组在裸子植物中一般为父系遗传, 而在被子植物中以母系遗传为主[3]。典型的被子植物叶绿体基因组为共价闭合环状双链DNA分子, 具有高度保守的四分体结构, 包括一个大单拷贝区(large single copy, LSC)、一个小单拷贝区(small single copy, SSC)和两个反向重复区(inverted repeats, IRa/b), 其大小一般为115~165 kb, 编码约110~130个基因[4]。
1986年, 烟草Nicotiana tabacum L.[5]和地钱Marchantia polymorpha L.[6]叶绿体全基因组的成功测序和解析标志着叶绿体基因组研究的开始。新一代测序技术和生物信息学分析方法的快速发展, 加之测序成本的不断降低, 极大地推动了叶绿体基因组研究的发展, 而“本草基因组学”[7]的提出进一步使得药用植物叶绿体基因组研究成为热点。由于叶绿体基因组序列组成和结构相对保守, 碱基变异速率适中, 基因组相对较小, 易于测序, 且在细胞中常存在多个拷贝[4], 同时具有单亲遗传等特征, 在植物分类与物种水平的系统发育等研究中具有明显优势, 叶绿体系统发育基因组学分析已成为解决药用植物物种鉴定问题的有效途径[8-10]。
美丽芍药Paeonia mairei H. Lév.为芍药科Paeoniaceaeae芍药属Paeonia多年生草本植物, 为中国特有物种, 主要分布于云南东北部、贵州西部、四川中南部、湖北西南部、陕西南部和甘肃南部等地[11]。其根可入药, 具有行瘀活血、止痛之功效, 在我国四川等一些地区, 美丽芍药的根亦被作为中药材赤芍(Paeoniae Radix Rubra)的来源之一[12]。目前, 芍药属芍药P. lactiflora Pall.[13]、川赤芍P. anomala subsp. Veitchii (Lynch) D. Y. Hong & K. Y. Pan[14]、凤丹P. ostii T. Hong & J. X. Zhang[15]、紫斑牡丹P. rockii (S. G. Haw & Lauener) T. Hong & J. J. Li ex D. Y. Hong[16]、四川牡丹P. decomposita Hand.-Mazz.[17]、矮牡丹P. jishanensis T. Hong & W. Z. Zhao[18]、以及大花黄牡丹P. ludlowii (Stern & G. Taylor) D. Y. Hong和滇牡丹P. delavayi Franch.[19]等多种植物的叶绿体基因组已经得到解析, 并基于这些叶绿体基因组序列开展了不同阶元的系统发育研究。然而, 美丽芍药的叶绿体基因组相关研究还未见报道, Stern[20]和Hong[21]的分类系统都曾将芍药属下的物种划分为3个组, 并将美丽芍药归于其中的芍药组Sect. Paeonia, 但美丽芍药在芍药组内的系统发育位置还尚未解决。
本研究基于二代高通量测序技术和生物信息学分析方法, 对美丽芍药开展叶绿体全基因组测序、组装、注释, 及序列变异和结构特征解析, 并将其与芍药属已报道的其他11种植物的叶绿体基因组序列进行比较基因组学和系统发育分析。旨在为美丽芍药的保护遗传学和资源开发利用、以及芍药属植物种间系统发育关系、物种鉴定等研究奠定基础。
材料与方法实验材料 美丽芍药新鲜叶片材料采自陕西省宝鸡市太白县(34°5′35″N, 107°26′35″E), 经陕西中医药大学标本馆王继涛高级实验师鉴定, 凭证标本保存于陕西中医药大学药学院中药资源学与鉴定学科实验中心。
基因组DNA提取与测序 取新鲜幼嫩叶片, 利用植物基因组DNA提取试剂盒(TIANGEN, 北京)提取总DNA, 琼脂糖凝胶电泳和Qubit 3.0 (Thermo Fisher Scientific, USA)荧光定量仪检测DNA质量及浓度。检测合格后的总DNA被随机打断, 构建长度约350 bp的插入片段文库, 利用Illumina HiSeq X Ten平台进行双端测序, 测序读长为150 bp。测序工作由华大基因完成。
叶绿体基因组序列组装及注释 测序完成后得到的原始序列(raw reads)首先利用NGS QC ToolKit[22]过滤去除接头及两端的低质量序列(参数设置为cut-off value for percentage of read length = 80, cut-off value for PHRED quality score = 30), 得到高质量待分析序列(即clean reads)。在Linux系统下利用GetOrganelle程序包[23]对clean reads进行组装, 序列延伸的种子(seed)为GetOrganelle默认seed, 获得序列重叠群(contigs), 再通过Bandage软件[24]进行可视化编辑, 将contigs拼接成环并去除冗余序列, 初步完成叶绿体全基因组序列的从头组装(De novo assembly)。之后, 再利用Bowtie2软件[25]将原始序列重新映射(mapping)到组装完成的叶绿体基因组序列上, 检查验证LSC/IRa、IRa/SSC、SSC/IRb和IR/LSC四个节点及各contigs拼接处的正确性, 并修补有缺口(gaps)的位点, 最终获得美丽芍药完整的叶绿体全基因组序列。
以NCBI数据库中收录的同属植物芍药的叶绿体全基因组注释结果(GenBank登录号: NC_040983)作为参考, 利用PGA (Plastid Genome Annotator)软件[26]对组装得到的叶绿体全基因组序列进行注释, 在Geneious软件[27]中对注释结果进行手动校正, 调整蛋白编码基因的起始、终止位置和基因内含子、外显子边界, 并利用tRNAscan-SE软件[28]确定tRNA基因的边界。最后, 利用Organellar Genome DRAW (OGDRAW)软件[29]绘制叶绿体全基因组图谱。组装并注释完成后的叶绿体全基因组序列提交至NCBI数据库, 获得登录号MN061945。
叶绿体全基因组重复序列检测 叶绿体全基因组中的散在重复序列(dispersed repeats)利用REPute软件(https://bibiserv.cebitec.uni-bielefeld.de/reputer)进行检测, 参数设置为最小重复序列长度= 30 bp, 重复序列间的相似度 > 90%[30]。串联重复序列(tandem repeats)利用Tandem repeats finder软件(https://tandem.bu.edu/trf/trf.html)进行检测, 参数设置选择默认值[31]。简单重复序列(simple sequence repeats, SSRs)检测通过MISA软件[32]完成, 参数设置为单核苷酸重复单元不少于10个, 二核苷酸重复单元不少于5个, 三核苷酸和四核苷酸重复单元不少于4个, 五核苷酸和六核苷酸重复单元不少于3个, 且两个SSRs之间的最小距离为100 bp。
芍药属种间叶绿体基因组比较分析 目前, NCBI数据库共收录芍药属11种植物的叶绿体全基因组序列数据, 即草芍药P. obovata Maxim. (GenBank登录号: NC_026076)、芍药(NC_040983)、川赤芍(NC_032401)、紫斑牡丹(MF488719)、大花黄牡丹(NC_035623)、滇牡丹(NC_035718)、凤丹(NC_036834)、矮牡丹(MG991935)、四川牡丹(NC_039425)、北美芍药P. brownii Douglas ex Hook. (MH191385)和牡丹P. suffruticosa Andr. (MH191384)。利用mVISTA软件[33]基于LAGAN模型对美丽芍药与这11种植物进行叶绿体全基因组比较分析。此外, 统计比较包括美丽芍药在内的所有12个物种的叶绿体全基因组长度、各个区域长度及其GC含量, 并利用CORREL函数分别计算全基因组长度变化与LSC区、SSC区和IR区长度变化之间的相关性。
系统发育分析 下载NCBI数据库中收录的芍药属上述所有11种植物的叶绿体全基因组序列, 与本研究获得的美丽芍药叶绿体全基因组序列共同构建序列矩阵, 同时选择芍药属的近缘类群虎耳草目Saxifragales虎耳草科Saxifragaceae秦岭岩白菜Bergenia scopulosa T. P. Wang和毛茛目Ranunculales毛茛科Ranunculaceae黄连Coptis chinensis Franch.作为外类群, 利用MAFFT软件[34]完成序列比对, 利用RAxML[35]以最大似然法(Maximum Likelihood, ML)进行系统发育关系重建分析, 核苷酸替换模型选择GTR+G, 其他参数选择默认设置, 进行1 000次自展重复分析, 计算系统发育树各节点的靴带支持率(Bootstrap values, BS)。
结果 1 叶绿体全基因组测序、组装及注释测序共获得80 175 282条读长为150 bp的raw reads, 质控除去接头和低质量序列之后得到79 009 120条clean reads, 即clean reads占raw reads的比例为98.55%。将clean reads组装、拼接后获得一条完整的环状叶绿体基因组序列。美丽芍药叶绿体全基因组序列总长度为152 731 bp, 包括一个长度为84 402 bp的大单拷贝区、一个16 969 bp的小单拷贝区和一对长度分别为25 680 bp的反向重复区(图 1)。基因组整体GC含量为38.4%, SSC、LSC和IR区的GC含量依次为32.7%、36.7%和43.1%。
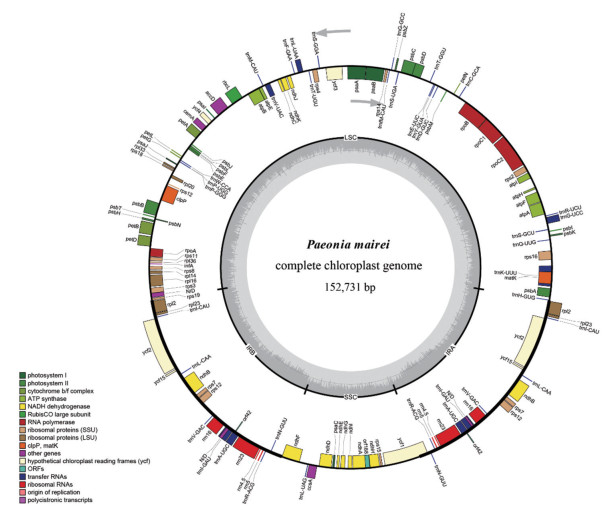
|
图 1 Chloroplast genome map of P. mairei. Genes inside and outside the circle are transcribed in a clockwise and counter clockwise direction, respectively. Genes are color-coded based on their functions. The grey area in the inner circle indicates the GC content of the chloroplast genome. |
共注释得到136个基因, 包括90个蛋白编码基因(ycf68和rpl22为假基因)、38个tRNA基因和8个rRNA基因。其中, 7个蛋白编码基因、7个tRNA基因和4个rRNA基因分别在反向重复区发生了一次重复(表 1)。在这些基因中, 有6个tRNA基因(trnI-GAU、trnA-UGC、trnV-UAC、trnL-UAA、trnG-UCC、trnK-UUU)和12个蛋白编码基因(rpl2、rpl16、rpoC1、rps16、atpF、ndhB、ndhA、petD、petB、ycf3、clpP和rps12)包含内含子, 其中, 除ycf3、clpP和rps12各包含2个内含子外, 其余均只包含1个内含子(表 2)。此外, 蛋白编码基因matK位于trnK-UUU基因的内含子区域之内, rps12基因具反式剪切(trans-spliced)结构, 其5′端和3′端分别位于LSC区和IR区, 被分成2个独立的转录单元。
| 表 1 Gene composition in the chloroplast genome of P. mairei. Bold font indicates genes duplicated in the IR regions; a represents pseudogenes; b and c indicate genes with one and two introns, respectively; d indicates the gene has 2 separate transcription units; e indicates the gene spans the large single copy region and the IR region |
| 表 2 List of genes with introns identified in the chloroplast genome of P. mairei |
美丽芍药叶绿体全基因组的蛋白编码基因序列总长度为78 273 bp, 约占叶绿体基因组总长度的51.2%, 由26 091个密码子组成。其中, 编码亮氨酸Leu的密码子使用频率最高, 共2 735次, 约占总密码子使用率的10.5%, 而编码半胱氨酸的密码子使用率最低, 仅296次, 占总密码子使用率的1.1%。
2 重复序列检测共检测到28个同时满足长度不小于30 bp且重复序列间相似度大于90%两个条件的散在重复序列, 包括14个回文重复(palindromic repeats, P)、12个正向重复(forward repeats, F)、1个互补重复(complement repeats, C)和1个反向重复(reverse repeats, R), 其长度范围为30~64 bp。检测到10条长度范围为18~33 bp的串联重复序列。串联重复序列在4个区域均有分布, 其中, LSC区和SSC区各2条, 重复次数均为2次, 两个IR区各3条, 重复次数分别为2、3和4次。共检测到64个SSRs, 包括44个单核苷酸、13个双核苷酸和7个三核苷酸SSRs, 四核苷酸、五核苷酸和六核苷酸SSR均未检测到。其中, 有11个SSRs分别分布于蛋白编码基因matK、rpoC2、atpB、cemA、rps3和ycf1的编码区内, 其余均分布于非编码区(包括基因间区和内含子区)。
3 芍药属种间叶绿体基因组比较分析美丽芍药与芍药属其他11种植物叶绿体全基因组序列间的比较分析结果见图 2。整体上, 12个物种的叶绿体基因组基因构成和顺序高度一致, 同时, LSC区和SSC区序列相比于IR区存在较多变异, 并且变异主要出现在非编码序列中。此外, 美丽芍药与其他11种植物叶绿体基因组序列在rps16-trnQ、psaA-ycf3、trnL-trnF、petA-psbJ、ndhF-trnL、rps15-ycf1等基因间区, 以及rpoC1、rpl16、ycf1基因的内含子区域存在明显差异。12个物种叶绿体全基因组、LSC、SSC和IR区的长度变化范围分别为152 153~154 405 bp、84 365~86 057 bp、16 679~17 059 bp和24 863~25 745 bp, GC含量变化范围分别为38.3%~38.5%、36.6%~36.8%、32.6%~33.0%和43.1%~43.2% (表 3)。此外, CORREL函数计算得到种间叶绿体全基因组长度变化与LSC、SSC和IR区长度变化之间的相关性系数分别为0.657 4、0.360 7和0.371 8。
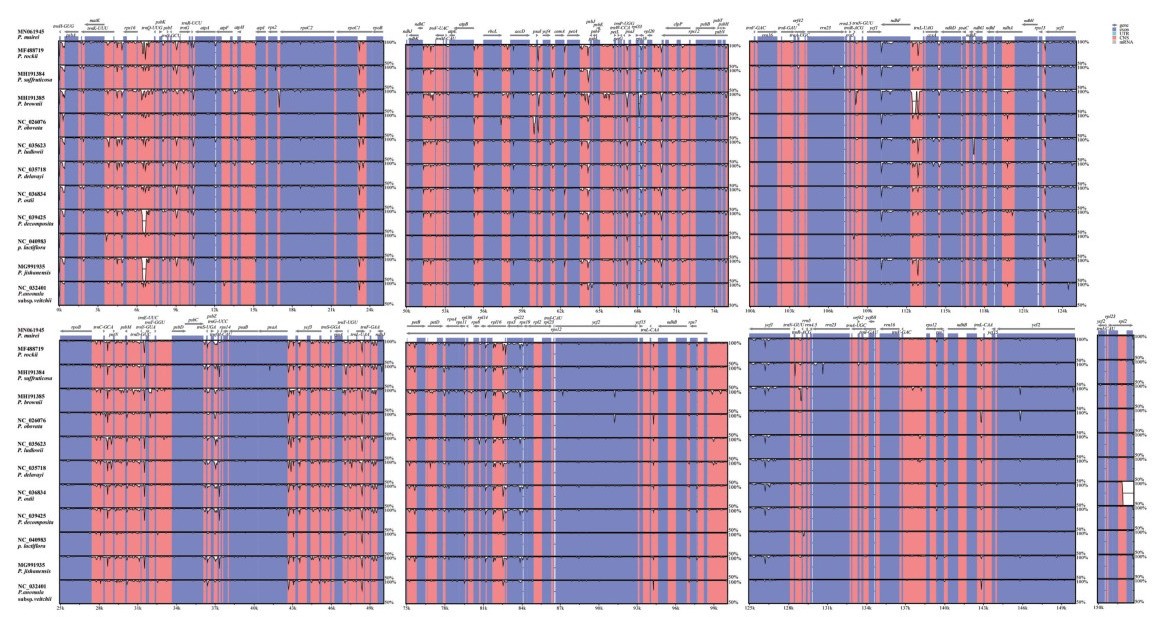
|
图 2 Identity plot comparing the chloroplast genomes of P. mairei and other 11 Paeonia species. P. mairei was used as a reference. Coding and noncoding regions are colored in blue and red, respectively |
| 表 3 Comparison of chloroplast sequences of P. mairei and other 11 species from Paeonia |
系统发育分析结果显示(图 3), 包括美丽芍药在内的芍药属12个物种以100%的支持率构成为一个独立的单系分支, 与毛茛科、虎耳草科区分开来。同时, 这12个物种可以进一步划分为3个次级单系分支, 其中, 同来自芍药组的草芍药、川赤芍、芍药和美丽芍药4个物种构成一个单系的支持率为100%, 并且美丽芍药和芍药的亲缘关系最近, 构成姐妹类群。来自牡丹组sect. Moutan的7个物种亦以100%的支持率聚为一支, 并与北美芍药构成姐妹关系。在整个系统发育树上, 除芍药组-北美芍药组sect. Onaepia、凤丹-牡丹两个节点的支持率分别为59%和66%之外, 其余节点的支持率均为100%。
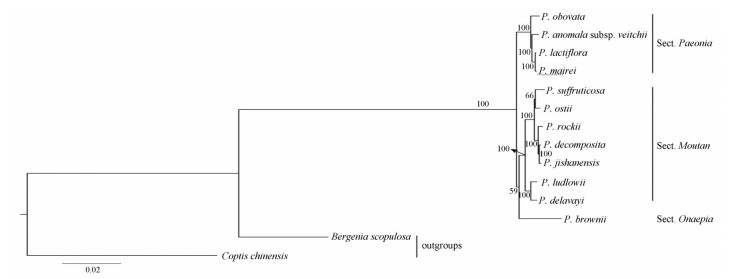
|
图 3 Maximum likelihood phylogenetic tree of 12 species from Paeonia inferred using dadaset of the whole chloroplast genome sequences. Numbers beside each node are bootstrap values (BS) |
作为芍药属中国特有物种之一, 美丽芍药曾被《中国物种红色名录》列为近危种[36], 化学成分研究表明, 美丽芍药根部含芍药苷等多种芍药属植物所具有的特征化合物, 并且具有较强的抗氧化活性, 可以作为抗菌抗氧化药物新资源[37], 因而具有重要的保护、研究和开发利用价值。本研究基于二代高通量测序技术和生物信息学分析方法首次对美丽芍药叶绿体全基因组进行了分析研究。结果显示美丽芍药叶绿体基因组具有被子植物叶绿体基因组典型的四分体结构, 并且其大小、结构、基因组成和顺序、CG含量等与芍药属其他已报道的植物高度相似[13, 15-19]。此外, 分析发现, 包括美丽芍药在内的芍药属12个物种叶绿体全基因组、LSC、SSC和IR区的长度差异范围分别为5~2 252 bp、0~1 792 bp、2~380 bp和0~882 bp, 说明芍药属植物叶绿体基因组在进化过程中高度保守。种间叶绿体全基因组长度变化与LSC、SSC和IR区长度变化之间的相关性系数为LSC (0.657 4) > IR (0.371 8) > SSC (0.360 7), 说明不同物种间叶绿体全基因组长度的变化主要受LSC区长度变化影响, 而IR区和SSC区长度变化相对保守。
本文所研究的芍药属12种植物中, 叶绿体基因组序列变异模式在不同区域表现出明显的差异(图 2)。总体上, LSC区和SSC区序列变异高于IR区, IR区序列高度保守, 这一结果与Guo等[15]对芍药属5种植物叶绿体全基因组的比较分析结果相同, 也与其他被子植物类群叶绿体基因组序列变异模式一致[9, 38-40]。同时, LSC区和SSC区所呈现出的高变异主要来自于非编码序列(主要为基因间区序列, 其次为部分基因的内含子序列), 说明在芍药属中, 非编码序列的进化速率快于蛋白编码基因序列, 与多数被子植物叶绿体基因组序列进化式样一致[38, 40, 41]。此外, 美丽芍药与芍药属其他11种植物叶绿体基因组序列在rps16-trnQ、psaA-ycf3、trnL-trnF、petA-psbJ、ndhF-trnL、rps15-ycf1等基因间区, 以及rpoC1、rpl16、ycf1基因的内含子区均存在明显差异, 这些区域可以为芍药属物种鉴定特异性DNA条形码的筛选提供参考。
叶绿体基因组中的SSRs因含量丰富、多态性高, 且兼具单亲遗传模式等优点而被作为分子标记广泛用于群体遗传学[42]、谱系地理学[43]等研究。本研究从美丽芍药叶绿体全基因组中共检测出44个单核苷酸、13个双核苷酸和7个三核苷酸SSRs。这些SSRs可以为美丽芍药的遗传多样性及保护遗传学等相关研究提供候选分子标记。在这些SSRs中, 单核苷酸和双核苷酸的重复序列类型分别以A/T (约占单核苷酸SSRs总数的95.45%)和AT/AT (约占双核苷酸SSRs总数的92.30%)为主, 三核苷酸SSRs的重复单元全部由A、T碱基组合(AAT、ATA、TAT、TTA、TAA)构成, 这也与其他被子植物叶绿体基因组中的SSRs序列构成一致[39, 40, 44], 进一步支持了叶绿体SSRs主要是由短的polyA和polyT重复所构成, 而较少含有C或G串联重复的观点[45]。此外, 这些SSRs主要分布在基因组中的LSC区(50个), 其次为SSC区(10个), 两个IR区分别仅检测到2个重复次数分别为10和12的A、T单核苷酸SSRs, 因此, 推测这些高A/T含量的SSRs可能是引起叶绿体基因组中CG含量偏低及各区域碱基含量偏差的原因之一(本研究中美丽芍药叶绿体基因组中平均GC含量为38.4%, LSC、SSC、和IR区的GC含量分别为36.7%、32.7%和43.1%)。
芍药属是一个分类与系统发育研究中较为复杂的类群[46]。依据形态特征和分子系统学等研究结果, 属下物种被划分为3个组, 即牡丹组、芍药组和北美芍药组[20, 21], 但由于经历了复杂的网状进化及多倍化演化历程, 有限的DNA片段(ITS、matK、psbA-trnH及trnL-trnF等)未能提供足够的系统发育信息来有效解决其种间关系[46-48]。本研究基于叶绿体全基因组序列构建的系统发育树将芍药属12个物种明确划分为3个独立的单系分支, 与Stern[20]和Hong等[21]划分的3个组分别对应, 并且各分支内部不同物种间的系统发育关系也得到较好解决(除凤丹-牡丹之间姐妹关系的支持率为66%外, 其余所有种间关系的支持率均为100%)。说明叶绿体全基因组序列包含丰富的物种演化信息, 是解决芍药属种间系统发育关系的一个有效途径。
| [1] |
Neuhaus HE, Emes MJ. Nonphotosynthetic metabolism in plastids[J]. Annu Rev Plant Physiol Plant Mol Biol, 2000, 51: 111-140. DOI:10.1146/annurev.arplant.51.1.111 |
| [2] |
McFadden GI. Primary and secondary endosymbiosis and the origin of plastids[J]. J Phycol, 2001, 37: 951-959. DOI:10.1046/j.1529-8817.2001.01126.x |
| [3] |
Corriveau JL, Coleman AW. Rapid screening method to detect potential biparental inheritance of plastid DNA and results for over 200 angiosperm species[J]. Am J Bot, 1988, 75: 1443-1458. |
| [4] |
Jansen RK, Ruhlman TA. Plastid Genomes of Seed Plants, in Genomics of Chloroplasts and Mitochondria[M]. Dordrecht: Springer Netherlands, 2012.
|
| [5] |
Sugiura M, Shinozaki K, Zaita N, et al. Clone bank of the tobacco (Nicotiana tabacum) chloroplast genome as a set of overlapping restriction endonuclease fragments: mapping of eleven ribosomal protein genes[J]. Plant Sci, 1986, 44: 211-217. DOI:10.1016/0168-9452(86)90093-2 |
| [6] |
Ohyama K, Fukuzawa H, Kohchi T, et al. Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA[J]. Nature, 1986, 322: 572-574. DOI:10.1038/322572a0 |
| [7] |
Chen SL, Song JY. Herbgenomics[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 3881-3889. |
| [8] |
Zhang N, Erickson DL, Ramachandran P, et al. An analysis of Echinacea chloroplast genomes: implications for future botanical identification[J]. Sci Rep, 2017, 7: 216. DOI:10.1038/s41598-017-00321-6 |
| [9] |
Zhu S, Niu Z, Xue Q, et al. Accurate authentication of Dendrobium officinale and its closely related species by comparative analysis of complete plastomes[J]. Acta Pharm Sin B, 2018, 8: 969-980. DOI:10.1016/j.apsb.2018.05.009 |
| [10] |
Fu CN, Wu CS, Ye LJ, et al. Prevalence of isomeric plastomes and effectiveness of plastome super-barcodes in yews (Taxus) worldwide[J]. Sci Rep, 2019, 9: 2773. DOI:10.1038/s41598-019-39161-x |
| [11] |
Hong DY, Pan KY, Turland NJ. Paeoniaceae in Flora of China[M]. Beijing: Science Press/St. Louis: Missouri Botanical Garden Press, 2001.
|
| [12] |
Editorial Committee of Chinese Materia Medica. Chinese Materia Medica (中华本草)[M]. Shanghai: Shanghai Science and Technology Press, 1999.
|
| [13] |
Lee M, Park JH, Gil JS, et al. The complete chloroplast genome of Paeonia lactiflora Pall. (Paeoniaceae)[J]. Mitochondr DNA Part B Resour, 2019, 4: 2715-2716. DOI:10.1080/23802359.2019.1644548 |
| [14] |
Zhang G, Sun J, Li YM, et al. The complete chloroplast genome of Paeonia anomala subsp. Veitchii[J]. Mitochondr DNA Part B Resour, 2016, 1: 191-192. DOI:10.1080/23802359.2015.1137838 |
| [15] |
Guo S, Guo L, Zhao W, et al. Complete chloroplast genome sequence and phylogenetic analysis of Paeonia ostii[J]. Molecules, 2018, 23: 246. DOI:10.3390/molecules23020246 |
| [16] |
Bai GQ, Guo HW, Zhao N, et al. The complete chloroplast genome of Paeonia rockii (Paeoniaceae), an endangered endemic species to China[J]. Conserv Genet Resour, 2018, 10: 453-456. DOI:10.1007/s12686-017-0847-5 |
| [17] |
Chen YM, Zhou QJ, Sun LJ, et al. The chloroplast genome of Paeonia decomposita (Paeoniaceae), an endangered wild tree peony from Sichuan, China[J]. Conserv Genet Resour, 2019, 11: 59-61. DOI:10.1007/s12686-017-0968-x |
| [18] |
Zhou XJ, Song LL, Peng ZF, et al. The complete chloroplast genome sequence of Paeonia jishanensis (Paeoniaceae), a rare wild tree peony[J]. Mitochondr DNA Part B Resour, 2019, 4: 503-504. DOI:10.1080/23802359.2018.1551077 |
| [19] |
Li HE, Guo QQ, Zheng WL. Characterization of the complete chloroplast genomes of two sister species of Paeonia: genome structure and evolution[J]. Conserv Genet Resour, 2018, 10: 209-212. DOI:10.1007/s12686-017-0800-7 |
| [20] |
Stern FC. A Study of the Genus Paeonia[M]. London: The Royal Horticulture Society, 1946.
|
| [21] |
Hong DY. Peonies of the World: Taxonomy and Phytogeography[M]. Richmond: Royal Botanic Gardens, 2010.
|
| [22] |
Patel RK, Jain M. NGS QC Toolkit: a toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7: e30619. DOI:10.1371/journal.pone.0030619 |
| [23] |
Jin JJ, Yu WB, Yang JB, et al. GetOrganelle: a simple and fast pipeline for de novo assembly of a complete circular chloroplast genome using genome skimming data[J]. BioRxiv, 2018, 256479. |
| [24] |
Wick RR, Schultz MB, Zobel J, et al. Bandage: interactive visualization of de novo genome assemblies[J]. Bioinformatics, 2015, 31: 3350-3352. DOI:10.1093/bioinformatics/btv383 |
| [25] |
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9: 357-359. DOI:10.1038/nmeth.1923 |
| [26] |
Qu XJ, Moore MJ, Li DZ, et al. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes[J]. Plant Methods, 2019, 15: 50. DOI:10.1186/s13007-019-0435-7 |
| [27] |
Kearse M, Moir R, Wilson A, et al. Geneious Basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics, 2012, 28: 1647-1649. DOI:10.1093/bioinformatics/bts199 |
| [28] |
Lowe TM, Chan PP. tRNAscan-SE on-line: integrating search and context for analysis of transfer RNA genes[J]. Nucleic Acids Res, 2016, 44: W54-W57. DOI:10.1093/nar/gkw413 |
| [29] |
Greiner S, Lehwark P, Boc R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1: expanded toolkit for the graphical visualization of organellar genomes[J]. Nucleic Acids Res, 2019, 47: W59-W64. DOI:10.1093/nar/gkz238 |
| [30] |
Kurtz S, Choudhuri JV, Ohlebusch E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Res, 2001, 29: 4633-4642. DOI:10.1093/nar/29.22.4633 |
| [31] |
Benson G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Res, 1999, 27: 573-580. DOI:10.1093/nar/27.2.573 |
| [32] |
Beier S, Thiel T, Munch T, et al. MISA-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33: 2583-2585. DOI:10.1093/bioinformatics/btx198 |
| [33] |
Frazer KA, Pachter L, Poliakov A, et al. VISTA: computational tools for comparative genomics[J]. Nucleic Acids Res, 2004, 32(suppl_2): W273-W279. |
| [34] |
Katoh K, Toh H. Parallelization of the MAFFT multiple sequence alignment program[J]. Bioinformatics, 2010, 26: 1899-1900. DOI:10.1093/bioinformatics/btq224 |
| [35] |
Stamatakis A. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies[J]. Bioinformatics, 2014, 30: 1312-1313. DOI:10.1093/bioinformatics/btu033 |
| [36] |
Wang S, Xie Y. China Species Eed List (中国物种红色名录)[M]. Beijing: Higher Education Press, 2004.
|
| [37] |
Shi Y. Study on Chemical Compositions of Paeonia mairei Levl. and Their Bioactivities (美丽芍药化学成分及其生物活性分析)[D]. Xi'an: Shaanxi University of Science and Technology, 2015.
|
| [38] |
Shen XF, Wu ML, Liao BS, et al. Complete chloroplast genome sequence and phylogenetic analysis of the medicinal plant Artemisia annua[J]. Molecules, 2017, 22: E1330. DOI:10.3390/molecules22081330 |
| [39] |
Guo HJ, Liu JS, Luo L, et al. Complete chloroplast genome sequences of Schisandra chinensis: genome structure, comparative analysis, and phylogenetic relationship of basal angiosperms[J]. Sci China C (中国科学:生命科学), 2017, 47: 728-739. |
| [40] |
Yang QQ, Jiang M, Wang LQ, et al. Complete chloroplast genome of Allium chinense: comparative genomic and phylogenetic analysis[J]. Acta Pharm Sin (药学学报), 2019, 54: 173-181. |
| [41] |
Zhang MY, Fritsch PW, Ma PF, et al. Plastid phylogenomics and adaptive evolution of Gaultheria series Trichophyllae (Ericaceae), a clade from sky islands of the Himalaya-Hengduan Mountains[J]. Mol Phylogenet Evol, 2017, 110: 7-18. DOI:10.1016/j.ympev.2017.01.015 |
| [42] |
Du Q, Wang B, Wei Z, et al. Genetic diversity and population structure of Chinese white poplar (Populus tomentosa) revealed by SSR markers[J]. J Hered, 2012, 103: 853-862. DOI:10.1093/jhered/ess061 |
| [43] |
Chmielewski M, Meyza K, Chybicki I, et al. Chloroplast microsatellites as a tool for phylogeographic studies: the case of white oaks in Poland[J]. iForest, 2015, 8: 765-771. DOI:10.3832/ifor1597-008 |
| [44] |
Tian N, Han LM, Chen C, et al. The complete chloroplast genome sequence of Epipremnum aureum and its comparative analysis among eight Araceae species[J]. PLoS One, 2018, 13: e0192956. DOI:10.1371/journal.pone.0192956 |
| [45] |
Kuang DY, Wu H, Wang YL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics[J]. Genome, 2011, 54: 663-673. DOI:10.1139/g11-026 |
| [46] |
Sun YL, Hong SK. Phylogenetic relationship and evolution analysis of the peony Paeonia species using multi-locus deoxyribonucleic acid (DNA) barcodes[J]. J Med Plants Res, 2012, 6: 5048-5058. DOI:10.5897/JMPR11.1733 |
| [47] |
Sang T, Crawford DJ, Stuessy TF. Documentation of reticulate evolution in peonies (Paeonia) using internal transcribed spacer sequences of nuclear ribosomal DNA: implications for biogeography and concerted evolution[J]. Proc Natl Acad Sci U S A, 1995, 92: 6813-6817. DOI:10.1073/pnas.92.15.6813 |
| [48] |
Sang T, Crawford D, Stuessy T. Chloroplast DNA phylogeny, reticulate evolution, and biogeography of Paeonia (Paeoniaceae)[J]. Am J Bot, 1997, 84: 1120-1136. DOI:10.2307/2446155 |
 2020, Vol. 55
2020, Vol. 55


