2. 福建农林大学园林学院, 福建 福州 350002
2. College of Landscape, Fujian Agriculture and Forestry University, Fuzhou 350002, China
在植物中, MYB转录因子是一个庞大的且功能多样化的转录因子家族, 其N端氨基酸残基具有保守的DNA结合结构域, 从而与基因的启动子区结合, 调控基因的表达。根据MYB转录因子N端保守的DNA结合结构域的重复个数, 将其分为4类: 1R-MYB (包含MYB-related)、R2R3-MYB、3R-MYB及4R-MYB[1, 2]。在过去几十年里, 从少量的MYB转录因子的发现鉴别再到基于基因组数据大量发掘各个植物中的MYB转录因子家族, 对MYB转录因子的分类和功能研究已经有了更全面的了解, 如双子叶植物拟南芥[2, 3]、毛果杨[4]及葡萄[5], 单子叶植物:玉米[6]、水稻[3]等。在4类MYB转录因子中R2R3-MYB的数量最多, 主要参与调控植物所特有的生长和代谢途径, 并且前人根据多个已测序物种的R2R3-MYB转录因子蛋白的C端保守的氨基酸基序的分析, 功能相似的序列聚为一类, 将其划分为28个不同的亚家族[7]。在以后各种植物R2R3-MYB转录因子的鉴定和功能注释的过程中, 都是通过构建系统进化树的方法与拟南芥的R2R3-MYB进行对比分析, 从毛果杨和葡萄基因组中也鉴定到了几个在拟南芥中没有的R2R3-MYB转录因子。R2R3-MYB转录因子的主要功能: ①初级和次级代谢物的调节, 如S7、S6和S2亚家族分别对黄酮醇[8]、花青素及原花青素[9]的调控; 也有其他亚家族主要参与调控次生细胞壁的合成, 如S3在纤维和导管的木质素合成, S21在次生细胞壁增厚方面起作用[10, 11]; ②调控细胞的生命周期; ③植物的生长发育调节; ④生物或非生物胁迫响应, 在干旱胁迫条件下, AtMYB2和AtMYB102的表达量显著提高[12, 13], S1亚家族的AtMYB96和AtMYB60通过脱落酸信号级联反应调控植物的气孔运动、干旱胁迫和抗病性[14-16], 。R2R3-MYB除了可以作为正调控转录因子外, 也可起到负调控的作用, 如AtMYB4在野生型拟南芥中通过抑制肉桂酸-4-羟基化酶基因(C4H)的表达, 阻止了芥子酸酯的合成, 而在紫外线的照射下, AtMYB4基因的表达量下降, 从而解除了对C4H的抑制作用, 使芥子酸酯的含量在植物体内有所增加, 在拟南芥AtMYB4突变体植株上, 相较于野生型植物, 对紫外线的抗性能力也大幅增强[17]。MYB转录因子除了可以单独调控基因的转录表达外, 在类黄酮(flavonoids)的合成过程中, 主要以转录因子复合物(transcriptional complex, MBW complex)的形式发挥功能, 其由bHLH转录因子、WD-repeat (WDR)转录因子和MYB转录因子构成[18-20]。MYB转录因子除了可以调控基因的转录表达, 其本身也受到microRNAs和ta-siRNAs的调控, 如miRNA159对参与花药和花粉发育的AtMYB33、AtMYB35、AtMYB65和AtMYB101起调控作用[21]。
巴戟天(Morinda officinalis How)作为一种藤本类药用植物, 其根部含有蒽醌类、环烯醚萜类等多种次生代谢产物。其中茜草素型蒽醌类有机物C环的合成和萜类化合物的合成都需要经过甲基赤藓醇磷酸途径(MEP), 并且1-脱氧-D-木酮糖5-磷酸还原异构酶基因(DXR)和1-脱氧-D-木酮糖5-磷酸合成酶基因(DXS)都是MEP途径的限速酶[22, 23]。有学者通过对青蒿的DXS基因的启动子区的克隆研究发现, 在上游调控序列中含有与MYB转录因子相结合的DNA结合位点[24]。本实验在前期的研究中也克隆了3个DXS基因和1个DXR基因及其启动子区序列, 通过对启动子序列的分析发现巴戟天3个DXS基因和DXR基因上游调控序列中也都含有多个与MYB转录因子相结合的DNA结合位点(未发表), 这说明MYB转录因子可能参与对DXS基因的调控, 除了对MEP途径关键基因进行调控外, MYB转录因子还参与调控苯丙烷代谢合成途径中关键酶基因的转录表达, 从而对植物的生长发育、胁迫响应、次级代谢产物的合成进行调节。但是针对巴戟天植物的MYB转录因子的研究还未见报道, 有必要对巴戟天的MYB转录因子进行研究和鉴定。通过巴戟天的根茎叶3个不同组织的转录组数据, 筛选并鉴定巴戟天MYB转录因子, 并对其进行GO功能分类、保守结构域和进化树分析, 以及巴戟天R2R3-MYB基因在根茎叶中的表达差异分析和验证。
材料与方法材料 2年生巴戟天植株栽植于20 cm (直径)×15 cm (高)的花盆中, 在福建农林大学园林学院温室大棚内培养2个月后, 选取9棵生长良好的巴戟天植株用于试验研究, 任意挑选3棵植株组成1个重复, 共3个重复, 其中每一个重复内的3棵植株的根、茎、叶3个组织部位于2019年6月22日8:00~10:00AM取样, 材料采集后立即放入液氮中冷冻, 存放在-80 ℃冰箱。栽植的2年生巴戟天植株经福建农林大学林学院邹小兴教授鉴定为茜草科植物巴戟天M. officinalis。
试剂 总RNA提取试剂盒(RNAprep Pure Plant Kit, DP441)购自天根生化科技有限公司, qRT-PCR的TB Green Premix Ex Taq试剂盒和PrimeScriptTM Ⅱ 1st Strand cDNA Synthesis kit反转录试剂盒均购自TaKaRa公司。
根、茎和叶总RNA提取及转录组测序 采用天根总RNA提取试剂盒分别提取巴戟天根、茎、叶3个不同组织的总RNA, 用Nanodrop 2000分光光度计测定其总RNA的浓度, 并用琼脂糖凝胶电泳检测总RNA的完整性。将合格的总RNA经反转录后合成cDNA第一链, 放于-20 ℃备用。将存放在-80 ℃冰箱的巴戟天根、茎、叶3个组织部位的样品送至北京诺禾致源科技股份有限公司进行转录组测序, 后续试验都是依据此测序结果进行分析。
巴戟天转录因子家族的筛选和鉴定 将巴戟天转录组数据中的unigene数据提交至CD-HIT Suite在线数据库(http://weizhong-lab.ucsd.edu/cdhit_suite/cgi-bin/index.cgi)去除冗余的核酸片段, 将输出的去除冗余后unigene数据提交至plantTFDB (http://planttfdb.cbi.pku.edu.cn/)进行转录因子筛选与鉴定。
巴戟天MYB转录因子家族鉴定、理化性质与功能注释 从鉴定的巴戟天转录因子数据库中挑选出MYB转录因子序列, 通过NCBI的ORFfinder在线软件分析预测各个MYB基因的开放阅读框并进行SmartBLAST与相应的蛋白数据库进行蛋白相似性比对分析鉴定。从plantTFDB (http://planttfdb.cbi.pku.edu.cn/)数据库中下载拟南芥MYB转录因子序列, 并根据Stracke和Dubos对拟南芥MYB转录因子家族的分类方法(MYB转录因子N端所含的DNA结合结构域的数目)将其编码后并分为4大类, 分别为1R-MYB (含MYB-related)、R2R3-MYB、3R-MYB、4R-MYB。使用ExPASy-pROSITE在线软件预测巴戟天MYB转录因子N端所含的DNA结合结构域的数目, 并对巴戟天MYB转录因子进行分类。运用在线软件Prot Comp 9.0 (http://linux1.softberry.com/all.htm)和SignalP (http://www.cbs.dtu.dk/services/SignalP-4.1/)对预测的巴戟天MYB蛋白进行亚细胞定位和信号肽分析。将筛选好的巴戟天MYB转录因子的蛋白序列以fasta格式用Blast2GO软件进行GO功能分类和注释。
巴戟天R2R3-MYB转录因子家族保守结构域和进化树分析 从已分类的巴戟天MYB转录因子数据库中筛选出R2R3-MYB类型, 运用MEME在线软件数据库(http://meme-suite.org/tools/meme)分别对巴戟天R2R3-MYB转录因子的R2和R3保守结构域进行分析。以拟南芥R2R3-MYB类型为参考序列, 通过MEGA7.0软件的ClustalW多序列比对功能将拟南芥的R2R3-MYB转录因子与预测的巴戟天R2R3-MYB转录因子进行比对, 后运用比邻法(Neighbor-Joining), 泊松模型(Poisson model), Bootstrap方法参数设为1000, 构建系统进化树。
巴戟天R2R3-MYB转录因子基因的表达模式分析和验证 从巴戟天转录组数据库中分别提取R2R3-MYB转录因子在根、茎、叶中的FPKM值, 由于不同的R2R3-MYB基因在不同组织的FPKM值相差较大, 在使用软件处理之前需要将这些数据用对数函数归一化处理, 后采用Heml软件对巴戟天R2R3-MYB转录因子在根、茎、叶的表达量进行分析。共选取8个在不同组织中有明显差异表达的R2R3-MYB基因进行RT-qPCR验证(表 1), 前期通过对巴戟天内参基因的选择与验证, 选择扩增效率97.887%的elongation elongation factor1α (ef1α)作为巴戟天的内参基因。以巴戟天根、茎、叶3个组织部位反转录合成的cDNA第一链为材料, 扩增体系和反应程序按照TaKaRa公司的TB Green Premix Ex Taq试剂盒的说明操作, 扩增体系(20 μL): TB Green Premix Ex Taq 10 μL, 正反引物各0.8 μL, RoxII 0.4 μL, cDNA 2 μL (100 ng·μL-1), ddH2O 6 μL。扩增程序: 95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 共40个循环; 溶解曲线: 95 ℃ 15 s, 60 ℃ 1 min, 95℃, 15 s。每个样品做3个平行实验, 并做3个生物学重复, 仪器为ABI公司的7500 Real Time PCR System。后用Ct(2-ΔΔCt)法计算巴戟天R2R3-MYB转录因子在巴戟天根、茎、叶3个不同组织的中的相对表达量。
| 表 1 Primer sequences |
将巴戟天根茎叶3个组织部位的unigenes经过CD-HIT去除冗余后, 提交至plantTFDB数据库, 与其他植物的转录因子比对, 最终得到54种共1 154个转录因子(图 1), 涉及的MYB (含MYB-related)转录因子家族的数量为128个, 其次为bHLH、C2H2、ERF、NAC、bZIP和WRKY等转录因子家族, 图 1仅列出了部分巴戟天转录因子家族的数量统计。
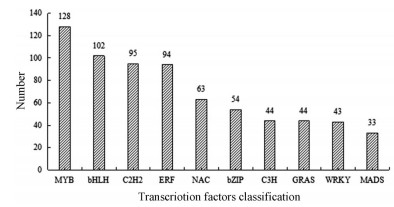
|
图 1 Excavation of transcriptional factors from Morinda officinalis transcriptome data |
初步筛选得到的128个巴戟天MYB转录因子的核酸序列中有109个序列具有MYB转录因子保守域的蛋白序列, 并随机对这109个巴戟天MYB转录因子序列进行编码MoMYB1~109, 并参照Stracke和Dubos对拟南芥MYB转录因子家族的分类方法将其分为4类: 1R-MYB (含MYB-related)、R2R3-MYB、3R-MYB、4R-MYB, 其数量分别为: 53、51、3和2个。亚细胞定位结果显示有81个序列定位于细胞核(nucleus), 其余28个则位于细胞外基质(extracellular)。信号肽分析表明有7个序列的N端有信号肽剪切位点, 如: MoMYB2 (1R)、MoMYB26 (R2R3)、MoMYB30 (1R)、MoMYB46 (R2R3)、MoMYB50 (R2R3)、MoMYB97 (1R)和MoMYB107 (1R)。Blast2GO软件处理最终结果显示, 注释到的序列涉及到427个GO terms, 其中分子功能(molecular function, MF)、生物过程(biological process, BP)和细胞组成(cellular component, CC)涉及到的GO terms分别为: 112、76和239个。MF中注释最多的是binding, 数量为109条, 表明所筛选的MYB转录因子N端都具有DNA结合结构域, 通过与所调控基因的启动子区结合, 进而控制生物体的生长发育和代谢产物的产生。BP中注释到参与细胞过程(cellular process)的序列较多, 而与代谢过程的序列数量也有5个, 都是R2R3-MYB类型, 如: MoMYB18、MoMYB45、MoMYB50 (R2R3)、MoMYB53和MoMYB65 (图 2)。CC中注释最多的是细胞组分、细胞器和细胞。
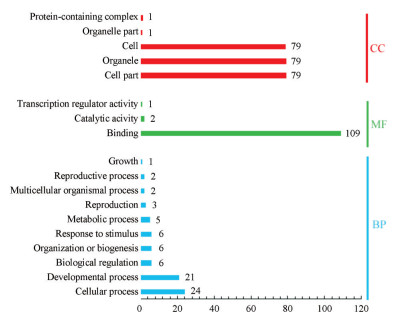
|
图 2 GO categorization and annotation of 109 MoMYB genes |
由图 3可知: 51个巴戟天R2R3-MYB转录因子中的R2-MYB和R3-MYB的保守结构域基序分别为: -W-(X19)-W-(X19)-W-、-F-(X18)-W-(X18)-W-。巴戟天R2-MYB基序中含有3个保守的色氨酸残基(W, Trp), 每个W之间有19个氨基酸残基相隔。而巴戟天R3-MYB基序中第一个保守的色氨酸残基被苯丙氨酸(F, Phe)、异亮氨酸(I, Ile)或亮氨酸(L, Leu)所取代, 并且苯丙氨酸和色氨酸残基之间有18个氨基酸残基相隔。但是在巴戟天R3-MYB中, 相对于异亮氨酸和亮氨酸残基, 苯丙氨酸取代色氨酸残基的频率更大。在巴戟天R2或R3-MYB保守结构域基序中, 除了极度保守的色氨酸或苯丙氨酸残基外, R2-MYB中靠近第三个色氨酸残基前6位的氨基酸残基也相对保守且集中, 如:赖氨酸(K, Lys)、丝氨酸(S, Ser)、半胱氨酸(C, Cys)、精氨酸(R, Arg)及亮氨酸残基(L, Leu)。R3-MYB中靠近第3个色氨酸残基前的氨基酸大部分也表现出高度保守, 如苏氨酸(T, Thr)、天冬氨酸(D, Asp)、天冬酰胺(N, Asn)及谷氨酸(E, Glu)。
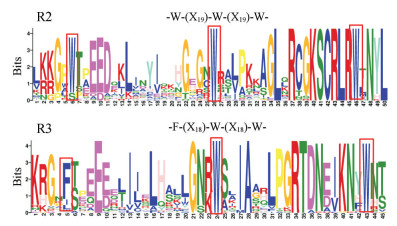
|
图 3 The conservative motif of R2R3-MYB of Morinda officinalis. Illustration showing the primary structures of a typical R2R3-MYB, including R2-type and R3-type MYB repeat. W: Tryptophan; F: Phenylalanine; X: Amino acid |
巴戟天R2R3-MYB转录因子中的R2-MYB和R3-MYB的保守结构域基序的特征与拟南芥的R2-MYB和R3-MYB结构特征相一致, 这也证明了从巴戟天转录组数据中筛选到的R2R3-MYB转录因子的正确性, 同时也说明R2R3-MYB转录因子N端保守的重复序列能够形成H (螺旋)-T (转角)-H (螺旋)结构, 使R2R3-MYB转录因子蛋白与基因启动子区的结合, 从而控制基因的转录水平。
在植物中, 对拟南芥的R2R3-MYB转录因子的功能注释和分类研究的比较透彻, 将拟南芥的R2R3-MYB转录因子与预测的巴戟天R2R3-MYB转录因子的氨基酸序列进行多序列比对后, 通过构建系统进化树的方式对巴戟天的R2R3-MYB转录因子的功能进行注释, 系统进化树的结果显示:巴戟天的51个R2R3-MYB转录因子分散在拟南芥R2R3-MYB转录因子的亚家族中, 但是除了在S10、S19和S21亚家族中未发现R2R3-MoMYB外, 其他亚家族中都有分布(图 4)。
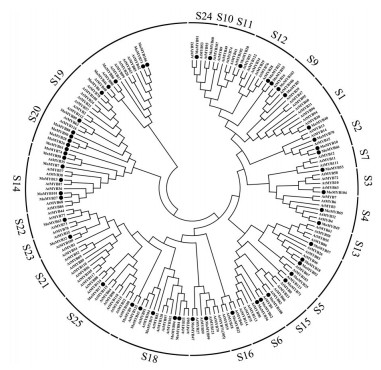
|
图 4 Phylogenetic tree analysis of R2R3-MYB between Morinda officinalis and Arabidopsis thaliana |
通过对巴戟天根、茎和叶3个组织部位中51个R2R3-MYB转录因子基因的表达量进行分层聚类分析, 由图 5可知, 大部分R2R3-MYB转录因子基因在3个组织部位中的表达差异较显著, 在叶组织中, 有6个基因显著表达, 且表达量明显高于根和茎组织, 如: MoMYB9、MoMYB40、MoMYB43和MoMYB53等, 但有5个基因在叶中不表达。在茎组织中, 有13个基因的表达量显著高于叶和根, 如: MoMYB18、MoMYB25、MoMYB57和MoMYB73等, 但有1个基因在茎中没有表达。而有17个基因在根组织中的表达量明显高于叶和茎, 如: MoMYB6、MoMYB27、MoMYB60、MoMYB61、MoMYB63和MoMYB70等, 但也有4个基因没有在根中表达。通过RT-qPCR的方法验证巴戟天部分R2R3-MYB基因在根茎叶3个组织部位的差异表达, 由图可知, MoMYB6、MoMYB27和MoMYB60基因在根中的表达量最高; MoMYB40、MoMYB53和MoMYB104在叶中的表达量最高; MoMYB57基因在茎中的表达量较根和茎中高。且这7个基因在3个组织部位中的表达趋势与转录组数据相一致。但是MoMYB63基因在根茎叶中的表达量差异不大, 在叶中的表达量最高, 但与转录组数据中根的表达量最高(图 6)。
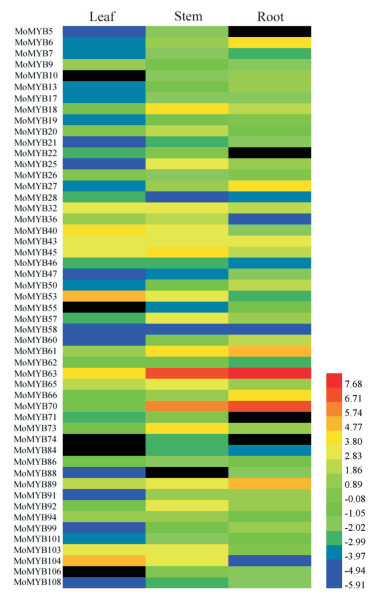
|
图 5 The pattern of expression of R2R3-MYB genes in three different tissues(root, stem, leaf) of Morinda officinalis. The black rectangle indicates that the FPKM |
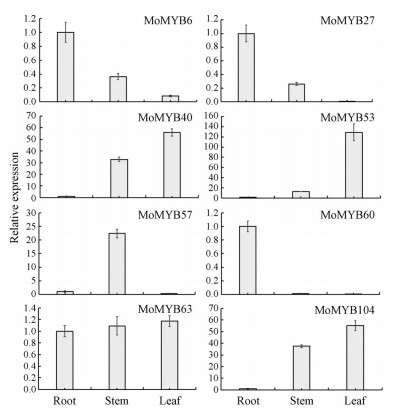
|
图 6 RT-PCR analysis of several R2R3-MYB genes of Morinda officinalis |
巴戟天是一种藤本类非模式植物, 因其根部能够产生重要的次生代谢产物而广泛种植于南方地区。科研工作者们通过对相关的次生代谢合成途径上的关键基因进行克隆分析, 并利用过表达和基因敲除的方式对限速酶关键基因的功能进行验证或提高某些次生代谢产物的含量, 但是植物的代谢途径是多步骤的连锁反应, 有时对单个基因进行调控无法取得应有的效果, 这时通过调控转录因子与代谢途径中相关酶基因启动子区的顺式作用元件的结合, 在转录水平上同时调控某些代谢途径的关键酶基因的表达水平, 从而影响相应次生代谢产物的含量。MYB类转录因子在植物生长发育过程中起到重要的作用并广泛参与植物苯丙烷类代谢途径的调节, 在苯丙烷代谢途径上的一些关键酶基因的启动子区均含有MYB结合位点, 从而显著提高苯丙烷类化合物的产量提供了可能。前人对拟南芥中MYB转录因子尤其是R2R3-MYB的研究较为透彻, 其主要参与初级或次生代谢, 发育过程及对生物或非生物胁迫的响应。
本研究通过巴戟天根、茎、叶3个组织部位的转录组数据, 最终筛选到了109个MYB转录因子序列, 其中有51个R2R3-MYB, 这些MYB转录因子的N端都具有与拟南芥MYB转录因子相同或相似的保守结构域与基序, 这些特征是判定筛选到的序列是否为MYB转录因子的前提条件, 但是这些特征并不是说在所有植物中都是一成不变的, 如对黑果枸杞R2R3-MYB转录因子的研究中, 其R2-MYB基序中除了包含3个保守的色氨酸残基外, 但是这3个色氨酸残基之间的间隔分别为23和19个氨基酸残基, 这与拟南芥和巴戟天鉴定和预测的R2-MYB转录因子3个色氨酸残基之间的间隔都是19个碱基有所不同, 作者推测这插入的4个碱基可能是黑果枸杞R2R3-MYB类转录因子分子进化和功能分化的活跃区[25]。
通过与拟南芥R2R3-MYB转录因子的氨基酸序列进行比对并构建系统进化树, 能够推测巴戟天预测的MYB蛋白的功能, 因为在进化树上位于相邻位置上的蛋白, 其功能具有一致性[26]。巴戟天MoMYB53与拟南芥S7亚家族下的AtMYB11、AtMYB12和AtMYB111相邻, 这3个基因功能相似, 主要是调控黄酮醇的生物合成, AtMYB11主要在根、茎的分生组织和花原基中表达, AtMYB12和AtMYB111分别调控根和子叶中黄酮醇的合成, 所以推测MoMYB53也可能参与调控黄酮醇的生物合成[8, 27-29]。MoMYB36与拟南芥MYB转录因子S6亚家族的几个基因(AtMYB75/90/113/114)位于同一个进化枝上, 可能参与了巴戟天花青素生物代谢合成[9, 26, 30]。除了拟南芥S6亚家族外, MoMYB71与S5亚家族的AtMYB123 (TT2)具有相似的蛋白结构, 可能通过调控花青素还原酶(anthocyanidin reductase, ANR)的转录水平, 从而控制原花青素(proanthocyanidin, PA)的代谢合成[9, 31]。MoMYB45和MoMYB65与R2R3-MYB转录因子的S4亚家族位于同一进化树分枝, 功能相似, S4亚家族在其C端含有保守的EAR基序[32], 被认为在苯丙烷代谢途径中作为一种转录因子抑制剂抑制一些关键酶基因的表达, 如AtMYB3需要转录共抑制因子LNK1和LNK2共同作用于肉桂酸-4-羟基化酶基因(C4H)[33]; AtMYB4转录因子也抑制C4H基因的表达, 从而减少芥子酸酯的含量[34]; AtMYB7抑制黄酮醇生物合成, 其作用的主要靶标为二氢黄酮醇-4-还原酶(DFR)和类黄酮-3-O-糖苷转移酶(UFGT)[35]; AtMYB32则降低了花粉的木质素和类黄酮的含量[36, 37]。
在拟南芥细胞壁合成调控网络中涉及到一系列的R2R3-MYB转录因子, 如AtMYB85和S3亚家族的AtMYB58、AtMYB63能够促进纤维素和导管中木质素的合成[10, 11], 但S14亚家族的AtMYB68负调控木质素在拟南芥根中的沉积[38]。AtMYB46转录因子蛋白是控制整个次生细胞壁生物合成的关键因子, MYB46基因的表达受到SND1的调控, 过表达MYB46基因能够激活纤维素、木聚糖和木质素的合成[39]。AtMYB42、AtMYB43、AtMYB20、AtMYB103和S21亚家族的AtMYB52、AtMYB54、AtMYB69也与拟南芥次生细胞壁增厚有直接联系[10]。S13亚家族的AtMYB61转录因子具有多重效应, 并推测它可能通过调节拟南芥的碳分配从而作用在不同代谢途径的上游基因从而发挥作用, 如木质素沉积、黏液的产生和气孔开度[40-42]。巴戟天的转录因子MoMYB18、MoMYB92、MoMYB101、MoMYB27、MoMYB25、MoMYB57、MoMYB73、MoMYB106和MoMYB104与上述拟南芥中的调控木质素合成和次生细胞壁增厚的转录因子位于同一亚家族, 遗传进化关系较近, 所以推测巴戟天中这些转录因子也有相似的功能。将巴戟天转录组数据中的MYB转录因子与拟南芥的MYB转录因子通过构建进化树的方式, 对其进行功能注释, 这也说明了MYB转录因子的保守性。
| [1] |
Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis[J]. Trends Plant Sci, 2010, 15: 573-581. DOI:10.1016/j.tplants.2010.06.005 |
| [2] |
Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana[J]. Curr Opin Plant Biol, 2001, 4: 447-456. DOI:10.1016/S1369-5266(00)00199-0 |
| [3] |
Chen YH, Yang XY, He K, et al. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family[J]. Plant Mol Biol, 2006, 60: 107-124. DOI:10.1007/s11103-005-2910-y |
| [4] |
Wilkins O, Nahal H, Foong J, et al. Expansion and diversification of the Populus R2R3-MYB family of transcription factors[J]. Plant Physiol, 2009, 149: 981-993. DOI:10.1104/pp.108.132795 |
| [5] |
Matus JT, Aquea F, Arce-Johnson P. Analysis of the grape MYB R2R3 subfamily reveals expanded wine quality-related clades and conserved gene structure organization across Vitis and Arabidopsis genomes[J]. BMC Plant Biol, 2008, 8: 83-97. DOI:10.1186/1471-2229-8-83 |
| [6] |
Du H, Feng B, Yang S, et al. The R2R3-MYB transcription factor gene family in maize[J]. PLoS One, 2012, 7: e37463. DOI:10.1371/journal.pone.0037463 |
| [7] |
Liu J, Osbourn A, Ma P. MYB transcription factors as regulators of phenylpropanoid metabolism in plants[J]. Mol Plant, 2015, 8: 689-708. DOI:10.1016/j.molp.2015.03.012 |
| [8] |
Stracke R, Ishihara H, Huep G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J]. Plant J, 2007, 50: 660-677. DOI:10.1111/j.1365-313X.2007.03078.x |
| [9] |
Heppel SC, Takos AM, Schellmann S, et al. Identification of key amino acids for the evolution of promoter target specificity of anthocyanin and proanthocyanidin regulating MYB factors[J]. Plant Mol Biol, 2013, 82: 457-471. DOI:10.1007/s11103-013-0074-8 |
| [10] |
Zhong RQ, Lee CH, Zhou JL, et al. A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis[J]. Plant Cell, 2008, 20: 2763-2782. DOI:10.1105/tpc.108.061325 |
| [11] |
Zhou J, Lee C, Zhong R, et al. MYB58 and MYB63 are transcriptional activators of the lignin biosynthetic pathway during secondary cell wall formation in Arabidopsis[J]. Plant Cell, 2009, 21: 248-266. DOI:10.1105/tpc.108.063321 |
| [12] |
Abe H, Urao T, Ito T, et al. Arabidopsis AtMYC2 (bHLH) and AtMYB2 (MYB) function as transcriptional activators in abscisic acid signaling[J]. Plant Cell, 2003, 15: 63-78. DOI:10.1105/tpc.006130 |
| [13] |
Denekamp M, Smeekens SC. Integration of wounding and osmotic stress signals determines the expression of the AtMYB102 transcription factor gene[J]. Plant Physiol, 2003, 132: 1415-1423. DOI:10.1104/pp.102.019273 |
| [14] |
Cominelli E, Galbiati M, Vavasseur A, et al. A guard-cell-specific MYB transcription factor regulates stomatal movements and plant drought tolerance[J]. Curr Biol, 2005, 15: 1196-1200. DOI:10.1016/j.cub.2005.05.048 |
| [15] |
Seo PJ, Xiang F, Qiao M, et al. The MYB96 transcription factor mediates abscisic acid signaling during drought stress response in Arabidopsis[J]. Plant Physiol, 2009, 151: 275-289. DOI:10.1104/pp.109.144220 |
| [16] |
Seo PJ, Lee SB, Suh MC, et al. The MYB96 transcription factor regulates cuticular wax biosynthesis under drought conditions in Arabidopsis[J]. Plant Cell, 2011, 23: 1138-1152. DOI:10.1105/tpc.111.083485 |
| [17] |
Jin H, Cominelli E, Bailey P, et al. Transcriptional repression by AtMYB4 controls production of UV-protecting sunscreens in Arabidopsis[J]. EMBO J, 2000, 19: 6150-6161. DOI:10.1093/emboj/19.22.6150 |
| [18] |
Zhang B, Hülskamp M. Evolutionary analysis of MBW function by phenotypic rescue in Arabidopsis thaliana[J]. Front Plant Sci, 2019, 10: 375. DOI:10.3389/fpls.2019.00375 |
| [19] |
Grotewold E, Sainz MB, Tagliani L, et al. Identification of the residues in the Myb domain of maize C1 that specify the interaction with the bHLH cofactor R[J]. Proc Natl Acad Sci U S A, 2000, 97: 13579-13584. DOI:10.1073/pnas.250379897 |
| [20] |
Zimmermann IM, Heim MA, Weisshaar B, et al. Comprehensive identification of Arabidopsis thaliana MYB transcription factors interacting with R/B-like BHLH proteins[J]. Plant J, 2004, 40: 22-34. DOI:10.1111/j.1365-313X.2004.02183.x |
| [21] |
Allen RS, Li J, Stahle MI, et al. Genetic analysis reveals functional redundancy and the major target genes of the Arabidopsis miR159 family[J]. Proc Natl Acad Sci U S A, 2007, 104: 16371-16376. DOI:10.1073/pnas.0707653104 |
| [22] |
Han Y, van der Heijden R, Lefeber AWM, et al. Biosynthesis of anthraquinones in cell cultures of Cinchona 'Robusta' proceeds via the methylerythritol 4-phosphate pathway[J]. Phytochemistry, 2002, 59: 45-55. DOI:10.1016/S0031-9422(01)00296-5 |
| [23] |
Han YS, Van der Heijden R, Verpoorte R, et al. Biosynthesis of anthraquinones in cell cultures of the Rubiaceae[J]. Plant Cell Tiss Org Culture, 2001, 67: 201-220. DOI:10.1023/A:1012758922713 |
| [24] |
Liu W. Study on Functional Differentiation of AaDXS Gene Family and Molecular Mechanism of Low Temperature Improving Artemisinin Production in Artemisim annua L. (青蒿DXS基因家族功能分化及低温促进青蒿素合成的分子机制研究)[D]. Chongqing: Chongqing University. 2016.
|
| [25] |
Yan L, Wang CP, Chen JW, et al. Analysis of MYB transcription factor family based on transcriptome sequencing in Lycium ruthenicum Murr[J]. Sci Agric Sin (中国农业科学), 2017, 50: 3991-4002. |
| [26] |
Zuluaga D, Gonzali SLE, Pucciariello C, et al. Arabidopsis thaliana MYB75/PAP1 transcription factor induces anthocyanin production in transgenic tomato plants[J]. Funct Plant Biol, 2008, 35: 606-618. DOI:10.1071/FP08021 |
| [27] |
Mehrtens F, Kranz H, Bednarek P, et al. The Arabidopsis transcription factor MYB12 is a flavonol-specific regulator of phenylpropanoid biosynthesis[J]. Plant Physiol, 2005, 138: 1083-1096. DOI:10.1104/pp.104.058032 |
| [28] |
Petroni K, Falasca G, Calvenzani V, et al. The AtMYB11 gene from Arabidopsis is expressed in meristematic cells and modulates growth in planta and organogenesis in vitro[J]. J Exp Bot, 2008, 59: 1201-1213. DOI:10.1093/jxb/ern027 |
| [29] |
Zhang SS. Preliminary Study on the MYB111 and MYB148 Transcription Factors Regulating the Phenylpropanoid Metabolism in Maize (玉米MYB111和MYB148转录因子调控苯丙烷代谢途径的初步研究)[D]. Chengdu: Sichuan Agricultural University, 2015.
|
| [30] |
Jeff V, Cahid C, Cazzonelli CI. A spontaneous dominant-negative mutation within a 35S::AtMYB90 transgene inhibits flower pigment production in tobacco[J]. PLoS One, 2010, 5: e9917. DOI:10.1371/journal.pone.0009917 |
| [31] |
Hou ZP, Wang SP, Wei SD, et al. Anthocyanin biosynthesis and regulation in plants[J]. Guihaia (广西植物), 2017, 37: 1603-1613. |
| [32] |
Kagale S, Rozwadowski K. EAR motif-mediated transcriptional repression in plants: an underlying mechanism for epigenetic regulation of gene expression[J]. Epigenetics, 2011, 6: 141-146. DOI:10.4161/epi.6.2.13627 |
| [33] |
Zhou M, Zhang K, Sun Z, et al. LNK1 and LNK2 co-repressors interact with the MYB3 transcription factor in phenylpropanoid biosynthesis[J]. Plant Physiol, 2017, 174: 1348-1358. DOI:10.1104/pp.17.00160 |
| [34] |
Jin H, Cominelli E, Bailey P, et al. Transcriptional repression by AtMYB4 controls production of UV-protecting sunscreens in Arabidopsis[J]. EMBO J, 2000, 19: 6150-6161. DOI:10.1093/emboj/19.22.6150 |
| [35] |
Fornale S, Lopez E, Salazar-Henao J, et al. AtMYB7, a new player in the regulation of UV-sunscreens in Arabidopsis thaliana[J]. Plant Cell Physiol, 2013, 55: 507-516. |
| [36] |
Preston J, Wheeler JJ, Li S, et al. AtMYB32 is required for normal pollen development in Arabidopsis thaliana[J]. Plant J, 2010, 40: 979-995. |
| [37] |
Ma D, Constabel CP. MYB repressors as regulators of phenylpropanoid metabolism in plants[J]. Trends Plant Sci, 2019, 24: 275-289. DOI:10.1016/j.tplants.2018.12.003 |
| [38] |
Feng C, Andreasson E, Maslak A, et al. Arabidopsis MYB68 in development and responses to environmental cues[J]. Plant Sci, 2004, 167: 1099-1107. DOI:10.1016/j.plantsci.2004.06.014 |
| [39] |
Zhong RQ, Richardson EA, Ye ZH. The MYB46 transcription factor is a direct target of SND1 and regulates secondary wall biosynthesis in Arabidopsis[J]. Plant Cell, 2007, 19: 2776-2792. DOI:10.1105/tpc.107.053678 |
| [40] |
Liang YK, Dubos C, Dodd IC, et al. At MYB61, an R2R3-MYB transcription factor controlling stomatal aperture in Arabidopsis thaliana[J]. Curr Biol, 2005, 15: 1201-1206. DOI:10.1016/j.cub.2005.06.041 |
| [41] |
Newman LJ, Perazza DE, Lusanda J, et al. Involvement of the R2R3-MYB, AtMYB61, in the ectopic lignification and dark-photomorphogenic components of the det3 mutant phenotype[J]. Plant J Cell Mol Biol, 2010, 37: 239-250. |
| [42] |
Penfield S, Meissner RC, Shoue DA, et al. MYB61 is required for mucilage deposition and extrusion in the Arabidopsis seed coat[J]. Plant Cell, 2001, 13: 2777-2791. DOI:10.1105/tpc.010265 |
 2020, Vol. 55
2020, Vol. 55


