2. 中国中医科学院中药资源中心, 道地药材国家重点实验室培育基地, 北京 100700;
3. 呼伦贝尔蒙医医院, 内蒙古 呼伦贝尔 021000
2. State Key Laboratory of Dao-di Herbs, National Resource Center for Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China;
3. Hulunbeier Mongolian Medical Hospital, Hulunbeier 021000, China
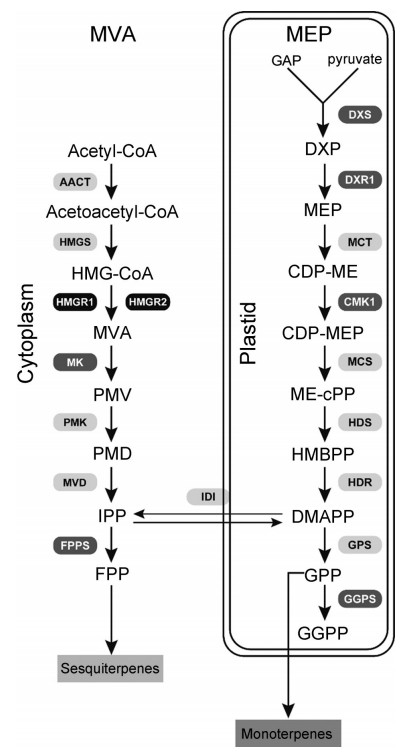
香樟(Cinnamum camphora (L.) Presl)是我国的珍贵药用和经济树种, 从其根、枝、叶和种子中提取的挥发油成分被广泛应用于化工、医药、食品等工业[1]。根据香樟精油中所含主要成分的不同, 可以把香樟划分为油樟、异樟、芳樟、脑樟和龙脑樟5种化学类型[2], 其主要成分分别为桉叶素、异橙花叔醇、芳樟醇、樟脑和龙脑。香樟精油已被证实具有抑菌[3, 4]、抗氧化[5, 6]、抗炎[7, 8]、抗癌[9]、镇痛[10]、止痒[11]等重要的药理作用。目前, 樟属植物沉水樟(Cinnamomum micranthum f. kanehirae)的基因组已经被报道[12], 同时江香梅等[13]和陈彩慧等[14]也对不同化学类型的香樟叶片转录组进行了测序, 初步推测了香樟挥发油等萜类成分的合成通路。已报道的香樟萜类物质合成通路关键酶相关基因, 包括MK基因[15]、DXS基因[16]、DXR基因[17]、CMK基因[18], FPPS基因[19]、GGPPS基因[20]等, 但尚未见香樟HMGR基因克隆和表达分析的报道, 见图 1。
|
图 1 Terpenoids biosynthesis pathway in C. camphora. The round boxs represent the key enzymes in the terpenoids biosynthesis pathway. Light gray represents unpublished, gray represents published, and black represents the research in this figure. AACT: Acetyl-CoA C-acetyltransferase; HMGS: Hydroxymethylglutaryl-CoA synthase; HMGR: Hydroxymethylglutaryl-CoA reductase (NADPH); MK: Mevalonate kinase; PMK: Phosphomevalonate kinase; MVD: Diphosphomevalonate decarboxylase; FPPS: Farnesyl diphosphate synthase; DXS: 1-Deoxy-D-xylulose-5-phosphate synthase; DXR: 1-Deoxy-D-xylulose-5-phosphate reductoisomerase; MCT: 2-C-Methyl-D-erythritol 4-phosphate cytidylyltransferase; CMK: 4-Diphosphocytidyl-2-C-methyl-D-erythritol kinase; MCS: 2-C-Methyl-D-erythritol 2, 4-cyclodiphosphate synthase; HDS: (E)-4-Hydroxy-3-methylbut-2-enyl-diphosphate synthase; HDR: 4-Hydroxy-3-methylbut-2-en-1-yl diphosphate reductase; IDI: Isopentenyl-diphosphate Delta-isomerase; GPS: Geranyl diphosphate synthase; GGPS: Geranylgeranyl diphosphate synthase |
香樟精油主要是由单萜(monoterpenes)和倍半萜类(sesquiterpenes)化合物组成。萜类前体物质是通过存在于细胞质的甲羟戊酸(MVA)途径和质体的2-C-甲基-D-赤藓糖醇-4-磷酸(MEP)途径[21]合成(图 1)。HMGR是MVA途径上游的第一个限速酶, 也是细胞质体内萜类化合物代谢的重要调控位点, 它含有两个HMGH-CoA功能结合基序(motif)和两个NADPH功能结合基序, 该酶催化3-羟基-3-甲基戊二酸单酰辅酶A (HMG-CoA)产生MVA, 这一步反应是需要依赖于NADP的不可逆转的过程, 因此萜类物质的产生受到限速酶活性的严格调控[22]。
在药用植物中过量表达HMGR, 可以显著提高其萜类物质的积累。在雷公藤悬浮细胞中过表达TwHMGR基因, 导致二萜化合物雷公藤甲素及三萜化合物雷公藤红素含量的上升[23]; 转HMGR基因的青蒿中, HMGR基因最高表达量是野生型的2.8倍, 而且最高青蒿素含量约是对照植株的1.8倍[24]; 将人参PgHMGR基因转入桔梗毛状根中, 使其三萜皂苷和甾醇的积累量增加[25]; 过表达丹参的SmHMGR1[26]和SmHMGR2[27]基因, 丹参毛根中二萜和三萜等的含量显著提高。因此, HMGR表达量的高低决定了萜类物质合成的共同前体IPP量, 从而对萜类化合物的合成起到十分重要的作用。
本研究以香樟为材料, 在前期转录组测序的基础上, 扩增得到香樟CcHMGR1和CcHMGR2基因cDNA的全长, 对氨基酸序列进行生物信息学分析, 并对香樟不同化学型叶片及不同组织中CcHMGRs基因的表达特性进行分析, 为香樟中萜类物质生物合成途径的解析及提高精油类化合物的含量提供了一定的参考。
材料与方法材料 香樟(Cinnamum camphora), 采集自江西省龙脑樟繁殖基地, 栽于中国中医科学院温室, 其中包括油樟、异樟、芳樟、脑樟、龙脑樟5种化学型。新鲜的根、枝和叶, 采集后立即用液氮速冻, -80 ℃保存, 用于提取总RNA。
试剂 超快新型植物RNA提取试剂盒购自北京华越洋生物科技有限公司; 焦碳酸二乙脂(DEPC)购自于西格玛奥德里奇(上海)贸易有限公司, Revert Aid Reverse Transcriptase, RiboLock RNase Inhibitor和10 mmol·L-1 dNTP购自赛默飞世尔科技(中国)公司; Prime STAR HS DNA Polymerase, Taq® DNA Polymerase和DL2000 DNA Marker和TB Green™ Premix Ex Taq™ Ⅱ购自宝生物工程(大连)有限公司; pEASY-Blunt Zero Cloning Kit和Trans T1感受态细胞购自于北京全式金生物技术有限公司; 所有引物均由生工生物工程(上海)股份有限公司合成。
仪器 MM400冷冻混合研磨仪(Retsch, 德国), 5810R型低温冷冻离心机(Eppendorf, 德国), NanoDrop2000分光光度计(Thermo Fisher Scientific, 美国), 9700 PCR仪(ABI, 美国), LightCycler480型Realtime PCR System (Roche, 瑞士), 紫外凝胶成像分析仪(Gene, 中国)。
总RNA的提取 香樟根、枝及叶总RNA的提取采用华越洋核酸提取试剂盒中的方法。实验中所用玻璃器皿、离心管和去离子水均用焦炭酸二乙酯(DEPC)浸泡处理, 以变性灭活RNase。将提取到的总RNA进行1%非变性琼脂糖凝胶电泳检测, 判断RNA的完整性; 使用分光光度计, 测定吸光度(A)值和A260/A280、A260/A230的比值, 以确定所提取总RNA的浓度及纯度; 并依照测定浓度确定下一步实验中合成cDNA所需模板的用量, 其余RNA置于-80 ℃冰箱中保存。
RNA的反转录 混合1 μg RNA, 1 μL 10 μmol·L-1 oligo(dT)18, 加DEPC水至12 μL, 65 ℃加热5 min, 产物置于冰上冷却; 待产物冷却后, 加入4 μL 5×Reaction Buffer, 2 μL 10 mmol·L-1 dNTP, 1 μL RevertAid Reverse Transcriptase和1 μL RiboLock RNase Inhibitor混匀, 42 ℃加热60 min, 70 ℃加热5 min, 产物置于冰上冷却, 获得模板香樟cDNA。
cDNA全长的获得 二代转录组测序得到5个被注释为HMGR的unigene, 经比对筛选后发现仅存在两个不一致的HMGR, 根据已知的ORF序列片段, 设计特异性引物(表 1)。用序列特异引物, 按以下体积进行扩增反应: 0.5 μL PrimeSTAR HS DNA Polymerase, 10 μL 5×PrimeSTAR Buffer, 4 μL 2.5 mmol·L-1 dNTP, 1 μL cDNA, 1 μL Primer L, 1 μL Primer R, 加ddH2O至50 μL, 反应条件为: 98 ℃预变性5 min, 然后进行35个循环: 98 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 2 min; 循环结束后72 ℃延伸10 min, 4 ℃保温; 对PCR产物进行1%非变性琼脂糖电泳检测。
| 表 1 Sequences of primers used in the CcHMGRs cloning and expression |
克隆载体的构建、转化及鉴定 PCR产物与pEASY-Blunt Zero载体连接体系包括: pEASY-Blunt Zero载体1 μL (50 ng)、PCR产物2 μL、无菌去离子水2 μL, 室温孵育15 min。连接产物全部加入到50 μL置于冰上的感受态Trans T1感受态细胞中, 冰浴30 min; 42 ℃热激45 s; 冰浴3 min。加入500 μL预热至室温的液体LB培养基, 摇菌1 h (37 ℃, 250 r·min-1)。取50 μL菌液涂布于含有卡那霉素的LB固体培养基上, 37 ℃培养过夜。次日挑选单菌落, 进行菌落PCR检测, 阳性克隆送至美吉生物医药科技有限公司测序。
生物信息学分析 利用NCBI的ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/)分析CcHMGR1和CcHMGR2基因的开放阅读框(ORF); 利用ProtParam tool (http://web.expasy.org/protparam/)分析蛋白的理化性质; 利用TMHMM Server v.2.0 (http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构; 利用SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析; 利用Bioeditor进行多重序列比对; 利用MEGA6中Neighbor-joining进行系统进化树的构建, 选用Jones-Taylor-Thornton (JTT)模型, bootstrap重复1 000次。
qRT-PCR检测 Realtime PCR测定香樟植株不同化学型及不同组织的叶片中CcHMGRs基因的表达量。采用引物设计工具Primer Premier 5设计Realtime PCR引物, 见表 1。反应体系: cDNA 1 μL, TB Green™ Premix Ex Taq™ Ⅱ 5 μL, Primer-L 0.2 μL, Primer-R 0.2 μL, ddH2O 3.6 μL, PCR反应条件为:预变性95 ℃预变性30 s, 然后进行40个循环: 95 ℃ 5 s, 60 ℃ 34 s, 每组设置3次技术重复, 数据采用2-△△CT公式进行分析。
结果与分析 1 香樟总RNA的提取及检测在对比多种RNA提取方法[28, 29]后, 选用北京华越洋RNA快速提取试剂盒进行RNA提取。RNA非变性琼脂糖凝胶电泳结果显示: 28S rRNA和18S rRNA条带亮度的比例大约为2:1, 说明提取的总RNA的完整性较好; 经分光光度计测定A260/A280平均值为2.01, 表明总RNA的纯度较高, 可用于全长扩增及之后的Realtime PCR实验。
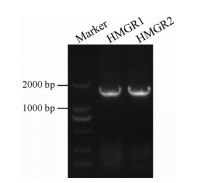
2 CcHMGRs基因的cDNA全长的克隆为深入研究香樟精油的生物合成机制, 前期利用二代测序技术对4种化学型香樟叶片进行转录组测序及组装注释分析, 并且对单独拼接的Unigenes和混拼的Unigenes均进行了分析, 得到了多条注释为HMGR的Unigenes, 但对其进行两两比对发现, 多数Unigenes为同一个基因, 筛选后有且仅有两条HMGRs, 这也与其近缘种沉水樟只有两条HMGRs基因的结果一致。这2条包含完整ORF序列的HMGRs基因, 长度分别为1 689 bp和1 683 bp。因此, 根据CcHMGR基因的ORF区设计该基因的上下游特异引物, 以5种化学型混合cDNA为模板进行PCR扩增, 结果如图 2。将PCR产物连接到pEASY-Blunt Zero克隆载体上, 转化大肠杆菌Trans T1, 经测序, 得出2条长度分别与转录组中HMGRs长度一致的编码区序列。序列比对分析后, 发现2条序列编码的均为3-羟基-3-甲基戊二酰辅酶A还原酶(CcHMGR), 在NCBI网站的GenBank数据库中进行注册, CcHMGR1登录号为MN163055, CcHMGR 2为MN163056。
|
图 2 Amplification products of CcHMGRs gene |
运用在线工具ProtParam tool对CcHMGR1和CcHMGR2蛋白的理化性质进行分析, 预测其分别编码562和560个氨基酸, 推测其分子式分别为C2638H4238N728O788S33和C2641H4244N720O776S28, 对应分子质量为59.819 kDa和59.397 kDa; 等电点为8.20和8.61;该蛋白的不稳定系数(instability index)为45.47和44.94, 说明其较为不稳定; 脂肪系数(aliphatic index)为91.46和96.04;亲水性平均系数(grand average of hydropathicity, GRAVY)为0.154和0.232, 说明其为疏水性蛋白(GRAVY为负值, 蛋白亲水)。利用TMHMM Server v.2.0预测跨膜结构, 结果表明CcHMGR1存在2个跨膜结构, 分别位于39位和61位之间以及74位和96位之间的氨基酸; CcHMGR2存在与CcHMGR1类似, 也存在2个跨膜结构分别位于41位和63位之间以及76位和98位之间的氨基酸。利用SignalP 4.1 Server进行信号肽分析, 结果表明其为非分泌蛋白, 不存在信号肽。
3.2 CcHMGRs序列比对及结构域分析对克隆得到的CcHMGRs基因编码的氨基酸进行初步的序列分析, 发现CcHMGR1和CcHMGR2蛋白相似度在75%以上, 主要差异位置存在于1~70和110~200位之间的氨基酸, 其余位置的氨基酸具有较高的保守性。Blastp结果显示, 与同属植物沉水樟仅有的两个HMGR相比, 香樟的CcHMGR1与沉水樟CkHMGR1 (AAY87014.1)的同源性最高(98.4%), 香樟的CcHMGR2与沉水樟CkHMGR2 (RWR72749.1)同源性最高为98.6%。
而CcHMGR1与丹参SmHMGR1 (ACD37361.1)同源性为70.18%, 与SmHMGR2 (ACT65734.1)同源性为69.46%, 与SmHMGR3 (AEZ55672.1)同源性为67.70%, 与蓖麻RcHMGR (XP_002510732.1)同源性为76.0%。CcHMGR2与丹参SmHMGR1同源性为73.2%, 与SmHMGR2同源性为71.8%, 与SmHMGR3同源性为69.4%, 与蓖麻RcHMGR同源性为77.1%。
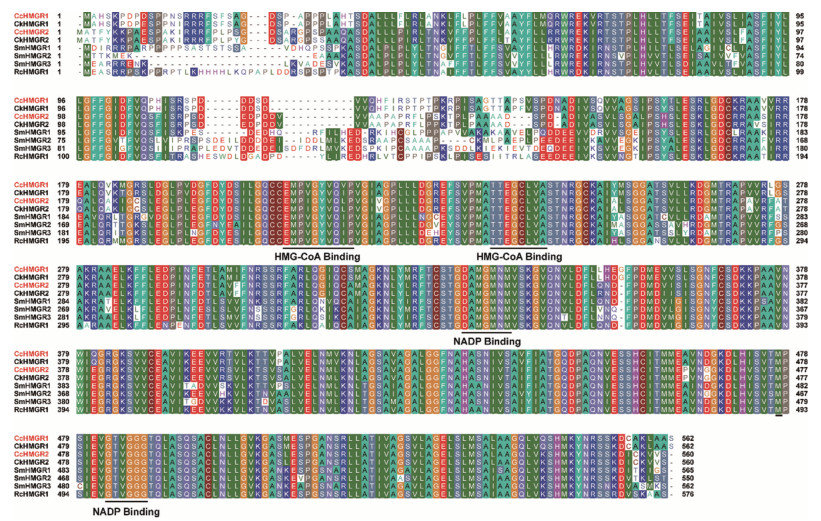
将上述HMGRs与CcHMGRs氨基酸序列进行多重序列比对(图 3), 发现这两个CcHMGRs蛋白存在2个保守结构域HMG-CoA结合基序EMPIGYVQIP和TTEGCLVA, 2个NADP (H)结合基序DAMGMNM和GTVGGGT (图 3), 这4个保守结构域的功能是在NADPH的存在下以HMG-CoA为底物催化产生MVA。结果表明, 除N末端氨基酸序列存在较大的差异外, 不同物种间HMGR蛋白的氨基酸序列均具有较高的保守性。
|
图 3 Multiple comparison of amino acid sequence of HMGRs in C. camphora and other plant species |
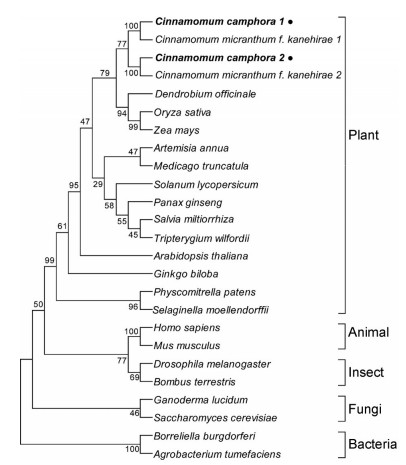
为了进一步探索CcHMGRs与不同物种间的进化关系, 从NCBI的非冗余蛋白数据库(Nr)中选取来自于细菌、真菌、昆虫、动物和植物的23条HMGR蛋白与CcHMGRs蛋白进行系统进化分析。利用MEGA 6.0软件构建氨基酸序列的进化树, 结果(图 4)显示, 在进化过程中, 所有的HMGR都有共同的起源, 香樟的两个CcHMGRs与沉水樟的两个CkHMGRs分别聚在一起, 并且这4个蛋白与单子叶植物水稻、玉米和铁皮石斛聚在一起, 表现出较高的同源性, 表明其亲缘关系较近, 可能具有相似的功能。
|
图 4 Phylogenetic analysis tree of CcHMGRs with other plant HMGRs The sequences were obtained from the GenBank database. NP 000850.1: Homo sapiens; AAH85083.1: Mus musculus; NP 732900.1: Drosophila melanogaster; NP 001295235.1: Bombus terrestris; ABY84848.1: Ganoderma lucidum; AAB67527.1: Saccharomyces cerevisiae; WP 106022668.1: Borreliella burgdorferi; WP 080865132.1: Agrobacterium tumefaciens; XP 024387921.1: Physcomitrella patens; XP 002988517.1: Selaginella moellendorffii; AAU89123.1: Ginkgo biloba; AGA18962.1: Dendrobium officinale; AAD38873.1: Oryza sativa; NP 001146733.1: Zea mays; AAF16652.1: Arabidopsis thaliana; AAD47596.1: Artemisia annua; ABY20976.1: Medicago truncatula; AAB62581.1: Solanum lycopersicum; AIX87980.1: Panax ginseng; ACT65734.1: Salvia miltiorrhiza; ALU11310.1 Tripterygium wilfordii; MN163055: Cinnamomum camphora 1; MN163056: AAY87014.1: Cinnamomum camphora 2; Cinnamomum micranthum f. kanehirae 1; RWR72749.1: Cinnamomum micranthum f. kanehirae 2 |
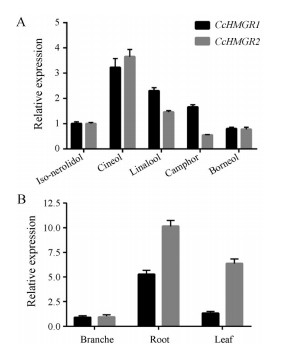
首先对Realtime PCR引物进行了设计及筛选, 最终选取CcHMGR1开放阅读框的1055~1214位和CcHMGR2开放阅读框的1050~1213位作为荧光定量扩增片段, 两对引物相似度低, 可很好的分别表征两基因的相对表达量, 引物见表 1。扩增曲线显示Actin[30]和CcHMGRs引物的扩增效率良好, Ct值在24~29;熔解曲线分析也充分表示扩增曲线有良好的特异性, 表现为单峰, 无非特异性荧光峰。分析油樟、异樟、芳樟、脑樟和龙脑樟5种化学型叶片中CcHMGRs相对表达量, 得出CcHMGR1和CcHMGR2在各化学型叶片中均有表达, 且表达量相似, 以油樟中表达量最高, 分别是异樟3~4倍、芳樟的1.5~2倍、脑樟的2~3倍和龙脑樟的3~4倍(图 5A)。以油樟为研究对象, 分析香樟根、枝、叶组织中CcHMGRs相对表达量的结果显示, CcHMGRs在植株各组织中均有表达, 且均在根中最高, 枝中最低, 其中CcHMGR1在根中的相对表达量是枝和叶的5倍左右(图 5B), CcHMGR2根是枝的10倍, 是叶的1.5倍(图 5B)。这一结果与油樟中精油在根中含量最高, 在枝中最低的结果一致, 推测CcHMGRs基因很可能是调控香樟精油的生物合成过程中关键催化节点。
|
图 5 Relative expression levels of CcHMGRs. A: Analysis of the expression level of CcHMGRs gene in different chemotypes of C. camphora. B: Relative expression levels of CcHMGRs in branches, roots and leaves of C. camphora |
单萜类化合物是构成香樟精油的主要成分, 也是中药冰片(龙脑, borneol)的主要来源, 解析香樟单萜类化合物的生物合成途径是阐明香樟精油产生机制和提高精油产量的分子基础[31]。随着对植物次生代谢研究的深入, 发现HMGR在植物萜类和甾醇合成途径中起着至关重要的调节作用, 是催化MVA途径中一步重要的限速反应, 该基因表达的上调会明显促进植物萜类物质含量的增加[17-21, 32]。
本文首次在香樟中克隆到2条CcHMGRs基因, 并通过多序列比对, 发现CcHMGRs与同属植物沉水樟的序列相似度高达95%以上, 与其他物种相似度高达70%左右, 其编码的CcHMGRs蛋白具有该家族非常保守的HMG-CoA和NADP(H)结合基序, 推测其具有HMGR蛋白的催化功能。通过系统进化分析, 发现双子叶植物香樟的HMGRs与单子叶植物水稻、玉米和铁皮石斛的HMGRs聚在一起, 出现这一现象的原因可能是香樟在进化中的特殊位置造成的。香樟属于木兰类(Magnoliids)中的樟目, 根据最新APG Ⅳ (被子植物种系发生)分类法, 木兰类较为原始, 在进化上要更靠近并且早于单子叶植物[33], 更早于真双子叶植物。所以相比真双子叶植物, 香樟的HMGRs更容易和单子叶植物聚为一支。
实验室前期工作发现油樟精油含量最高(4.05%), 脑樟其次(3.26%), 然后是芳樟(2.17%)、龙脑樟(2.07%)、异樟最低(1.25%)[34]。而本研究检测了CcHMGRs基因在5种香樟化学型中表达量, 发现油樟中CcHMGRs基因的表达量高于另外4种化学型香樟, 这一结果与前期的化学结果具有一致性。HMGR是萜类化合物生物合成MVA途径上游的第一个限速酶, 在药用植物中过量表达HMGR, 可以显著提高其萜类物质的积累[17-21], 说明不同化学型香樟中HMGRs的表达量可能与萜类终产物的含量具有相关性。HMGRs基因在植物中的表达具有组织特异性, 例如其在鱼腥草的花中[35], 金钗石斛的叶中[36], 丹参的叶中[37], 白木香的根中[38]等的表达量均高于其他组织及器官。而在香樟中, CcHMGRs在根中表达量最高, 叶中其次, 枝条中表达量最低, 这与根中精油的含量最高, 叶中其次, 枝条中精油含量最低的结果具有一致性[2], 说明CcHMGRs基因具有组织特异性的表达模式的同时, 也反映了CcHMGRs基因表达量的高低与香樟精油含量的高低呈正相关性。
综合本文研究, 发现CcHMGR1和CcHMGR2的蛋白的序列、理化性质及其基因的表达模式都具有较高的一致性, 推测CcHMGR1和CcHMGR2在香樟中是具有相同功能, 互为冗余基因, 更有利于面对复杂多变的生物及非生物胁迫[39]。
| [1] |
Chen SY, Zhao LH, Xu XJ, et al. Linalool natural resources and its exploitation and utilization[J]. Chin Fore Sci Technol (林业科技开发), 2013, 27: 13-17. |
| [2] |
Xu YM, Jiang ZH, Bao CH, et al. Comparative studies on differences of oil contents and wood properties among five clones of camphor trees[J]. J Huazhong Agric Univ (华中农业大学学报), 2001, 20: 484-488. |
| [3] |
Singh P, Srivastava B, Kumar A, et al. Fungal contamination of raw materials of some herbal drugs and recommendation of Cinnamomum camphora oil as herbal fungitoxicant[J]. Microb Ecol, 2008, 56: 555-560. DOI:10.1007/s00248-008-9375-x |
| [4] |
Herman A, Tambor K, Herman A. Linalool affects the antimicrobial efficacy of essential oils[J]. Curr Microbiol, 2016, 72: 165-172. DOI:10.1007/s00284-015-0933-4 |
| [5] |
Luo SH, Huang T, Shi ZP, et al. A comparative study on antioxidant activities of essential oils from four Cinnamomum plants[J]. J Chongqing Norm Univ Nat Sci Ed (重庆师范大学·自然科学版), 2018, 35: 111-116. |
| [6] |
Zan LX. Optimization of the extraction technology of camphor pericarp anthocyanins by response surface method[J]. Guihaia (广西植物), 2015, 35: 603-608. |
| [7] |
Wu XH. Comparative analysis of anti-inflammatory and analgesic effects of extracts from the stems and branches of camphor trees with different diameters and camphor roots[J]. Chin J Mod Drug Appl (中国现代药物应用), 2017, 11: 197-198. |
| [8] |
Ye XL, Zhao Y, Zhang SY, et al. Study on the anti-inflammatory activity of camphor volatile oil in Meizhou[J]. Guangdong Chem Ind (广东化工), 2018, 45: 49-50. |
| [9] |
Miyashita M, Sadzuka Y. Effect of linalool as a component of Humulus Lupulus on doxorubicin-induced antitumor activity[J]. Food Chem Toxicol, 2013, 53: 174-179. DOI:10.1016/j.fct.2012.11.035 |
| [10] |
Granger RE, Campbell EL, Johnston GA. (+)- And (-)-borneol: efficacious positive modulators of GABA action at human recombinant α1β2γ2L GABAA receptors[J]. Biochem Pharmacol, 2005, 69: 1101-1111. DOI:10.1016/j.bcp.2005.01.002 |
| [11] |
Yang JQ, Yang NY. Antipruritic effects of essential oil of Cinnamomum camphora leaves[J]. Tianjin J Tradit Chin Med (天津中医药), 2019, 36: 300-303. |
| [12] |
Chaw SM, Liu YC, Wu YW, et al. Stout camphor tree genome fills gaps in understanding of flowering plant genome evolution[J]. Nat Plants, 2019, 5: 63-73. DOI:10.1038/s41477-018-0337-0 |
| [13] |
Jiang XM, Wu YF, Xiao FM, et al. Transcriptome analysis for leaves of five chemical types in Cinnamomum camphora[J]. Heredity (遗传), 2014, 36: 58-68. |
| [14] |
Chen CH, Zheng YJ, Zhong YD, et al. Transcriptome analysis and identification of genes related to terpenoid biosynthesis in Cinnamomum camphora[J]. BMC Gen, 2018, 19: 550. DOI:10.1186/s12864-018-4941-1 |
| [15] |
Cao XS, Wang J, Zhang YY, et al. Cloning and bioinformatic analysis of MK gene from Cinnamomum camphora[J]. Chin J Trop Crop (热带作物学报), 2017, 38: 2302-2309. |
| [16] |
Zhang YY, Liu W, Cao XS, et al. Cloning and expression analysis of DXS gene in Cinnamomum camphora [J/OL]. Gen Appl Biol (基因组学与应用生物学), 2019[2019-04-24]. http://kns.cnki.net/kcms/detail/45.1369.Q.20190423.1645.002.html.
|
| [17] |
Zheng H, Jing L, Yao N, et al. Cloning and expression analysis of 1-deoxy-D-xylulose-5-phosphate reductoisomerase gene (CcDXR1) in Cinnamomum camphora (L.) Presl[J]. Acta Pharm Sin (药学学报), 2016, 51: 1494-1501. |
| [18] |
Jing L, Zheng H, Yao N, et al. Cloning and expression analysis of 4-diphosphocytidyl-2-C-methyl-D-erythritol kinase gene in Cinnamomum camphora[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 1578-1584. |
| [19] |
Zhang YY, Song L, Liu W, et al. Cloning and bioinformatic analysis of FPPS gene from Cinnamomum camphora[J]. Mol Plant Breed (分子植物育种), 2018, 16: 6276-6281. |
| [20] |
Cao XS, Wang J, Zhang YY, et al. Cloning and bioinformatics analysis of GGPPS gene from Cinnamomum camphora[J]. Genom Appl Biol (基因组学与应用生物学), 2018, 37: 3466-3472. |
| [21] |
Vranová E, Coman D, Gruissem W. Network analysis of the MVA and MEP pathways for isoprenoid synthesis[J]. Annu Rev Plant Biol, 2013, 64: 463-476. |
| [22] |
Chen Y, Xie QD, Tang YQ, et al. Advances in synthetic metabolic pathways and rate-limiting enzymes of plant terpene[J]. Mol Plant Breed (分子植物育种), 2018, 16: 2371-2379. |
| [23] |
Wang JD, Zhao YJ, Zhang YF, et al. The effects of TwHMGR overexpression on the biosynthesis of triptolide and celastrol in Tripterygium wilfordii[J]. Acta Pharm Sin (药学学报), 2018, 53: 1225-1232. |
| [24] |
Wang Y, Jing F, Yu S, et al. Co-overexpression of the HMGR and FPS genes enhances artemisinin content in Artemisia Annua L.[J]. J Med Plants Res, 2011, 5: 3396-3403. |
| [25] |
Kim Y, Kim JK, Kim YB, et al. Enhanced accumulation of phytosterol and triterpene in hairy root cultures of Platycodon grandiflorum by overexpression of Panax ginseng 3-hydroxy-3-methylglutaryl-coenzyme a reductase[J]. J Agric Food Chem, 2013, 61: 1928-1934. DOI:10.1021/jf304911t |
| [26] |
Shi M, Luo X, Ju G, et al. Increased accumulation of the cardio-cerebrovascular disease treatment drug tanshinone in Salvia miltiorrhiza hairy roots by the enzymes 3-hydroxy-3-methylglutaryl CoA reductase and 1-deoxy-d-xylulose 5-phosphate reductoisomerase[J]. Funct Integr Gen, 2014, 14: 603-615. DOI:10.1007/s10142-014-0385-0 |
| [27] |
Dai Z, Cui G, Zhou S, et al. Cloning and characterization of a novel 3-hydroxy-3-methylglutaryl coenzyme a reductase gene from Salvia Miltiorrhiza involved in diterpenoid tanshinone accumulation[J]. J Plant Physiol, 2011, 168: 148-157. DOI:10.1016/j.jplph.2010.06.008 |
| [28] |
Wu YF, Xu HN, Song XC, et al. Comparative isolation methods for total RNA from different tissues and examination with RT-PCR in Cinnamomum camphora[J]. Jiangxi Forest Sci Technol (江西林业科技), 2013, 5: 1-4. |
| [29] |
Wu YF, Xu HN, Jiang XM, et al. Comparative research on extraction methods for total RNA from leaves of five chemotypys of Cinnamomum camphora[J]. Chin Forest Sci Technol (林业科技开发), 2013, 27: 75-79. |
| [30] |
Zhang LW, Li YP, Yao Y, et al. Cloning and expression analysis of Actin gene in Cinnamomum camphora[J]. J Cent South Univ Forest Technol (中南林业科技大学学报), 2015, 31: 120-127. |
| [31] |
Huang LQ, Gao W, Zhou YJ. Application of synthetic biology to sustainable utilization of Chinese materia medica resources[J]. Acta Pharm Sin (药学学报), 2014, 49: 37-43. |
| [32] |
Zhu CS, Chen MM, Liu Y, et al. Effect of over-expressing TwHMGR and TwDXR on the biosynthesis of terpenoids in Tripterygium wilfordii[J]. Chin J Agric Biotechol (农业生物技术学报), 2018, 26: 940-948. |
| [33] |
Liu WZ, Zhao P. A preliminary study on the application of APGⅣ system in botany teaching[J]. High Edu Sci (高等理科教育), 2017, 4: 104-109. |
| [34] |
Luo YJM. Study on the Characteristic Chinese Medicinal Materials of Jiangxi Province: Sarcandra glabra and Cinnamomum camphora (江西特色中药材草珊瑚和樟的研究)[D]. Beijing: China Academy of Chinese Medical Sciences, 2004.
|
| [35] |
Wei L, Li XY, Liu SG, et al. Molecular cloning, sequence identification, and expression analysis of HMGR gene in Houttuynia cordata[J]. Chin Tradit Herb Drugs (中草药), 2017, 48: 3815-3819. |
| [36] |
Li Q, Li B, Guo SX. Cloning and expression analysis of HMGR gene in Dendrobium nobile in response to mycorrhizal fungal inoculation[J]. Chin Pharm J (中国药学杂志), 2017, 52: 1976-1982. |
| [37] |
Zhang XN, Guo J, Shen Y, et al. Cloning and expression analysis of a new 3-hydroxy-3-methylglutarylcoenzyme A reductase gene from Salvia miltiorrhiza (SmHMGR3)[J]. China J Chin Mater Med (中国中药杂志), 2012, 37: 2378-2382. |
| [38] |
Xu YH, Yang X, Zhang Z, et al. Cloning and expression analysis of HMG-CoA reductase from Aquilaria sinensis (Lour.) Gilg[J]. Acta Pharm Sin (药学学报), 2013, 48: 953-959. |
| [39] |
Cai KZ. Functional redundancy of gene[J]. J Ecol (生态学杂志), 2001, 20: 61-64. |
 2020, Vol. 55
2020, Vol. 55

 ,
黄璐琦2
,
黄璐琦2