2. 青海绿康生物开发有限公司, 青海 西宁 810003;
3. 阿坝州食品药品检验所, 四川 阿坝 624000
2. Qinghai Lukang Biological Development Co., Ltd., Xining 810003, China;
3. Aba Food and Drug Inspection, Aba 624000, China
川贝母为百合科贝母属植物川贝母(Fritillaria cirrhosa)、暗紫贝母(Fritillaria unibracteata P.K.Hsiao & K.C.Hsia)、甘肃贝母(Fritillaria przewalskii)、梭砂贝母(Fritillaria delavayi)、太白贝母(Fritillaria taipaiensis P.Y.Li)或瓦布贝母(Fritillaria unibracteata Hsiaoet K. C. Hsia var. wabuensis)的干燥鳞茎, 按性状不同分别习称“松贝”、“青贝”与“炉贝”。川贝母治疗劳虚咳嗽、吐痰咳血等症状效果显著, 其疗效远超过平贝母、浙贝母与伊贝母等, 因此被誉为“止咳圣药”[1, 2]。由于川贝母的疗效显著, 市场需求量大, 然而川贝母多为野生, 资源有限, 导致市场上出现了大量的近缘物种混伪品。这些品种与川贝母的形态特征近似, 仅凭传统生药学鉴定难度较大, 因此开展川贝母及其近缘种的分子鉴定与亲缘关系研究已迫在眉睫。
2015版《中国药典》补充本增加川贝母的PCR-RFLP分子鉴别方法, 其原理是川贝母的ITS1区域含有1个SmaI酶切位点, 因此能够对PCR产物进行切割, 而浙贝母与伊犁贝母等品种不含有SmaI酶切位点, 所以根据是否在100~250 bp间出现两条条带来判断药材的真伪, 然而该方法是否适用于所有川贝母的近缘种鉴定还有待进一步研究。DNA条形码是一种利用基因组中一段公认标准的短序列来进行种间亲缘关系及物种鉴定的方法, 它是建立在PCR技术和DNA测序技术上的一种新型分子鉴定技术[3-6]。ITS2片段在物种水平的变异较快, 有更多的突变位点以区分不同的物种[7]。2010年, 陈士林提出了用ITS2序列作为药用条形码的通用序列[3]。Zhu等[7]采用ITS2序列对百合科重楼属植物取得了100%的鉴定效率, 推测ITS2可能同样适用于百合科贝母属植物的分子鉴定。Hao等[8]通过对181个甘草样品的ITS2序列进行遗传距离及NJ进化树分析, 初步鉴定了国内疑似变异甘草植株和国外未鉴定到种的甘草样品与我国药用甘草的亲缘关系较近。psbA-trnH为叶绿体基因组中psbA基因与trnH基因的间隔区。Yang等[9]使用psbA-trnH序列对8种肉桂类药材进行鉴定, 结果发现使用psbA-trnH序列能够准确将肉桂及其近缘种鉴定出来。Xu等[10]使用psbA-trnH序列对34种稻属禾本科进行鉴定, 结果发现栽培稻的psbA-trnH序列几乎没有碱基变异, 而野生稻相对变异丰富, 说明psbA-trnH序列不能将近缘物种分开, 而是更适用于属间的鉴别。Chen等[11]首次提出并建立了以ITS2为核心, psbA-trnH为辅的植物类药材的DNA条形码鉴定体系。由此, 本实验进一步获得了上述样本的ITS2与psbA-trnH序列, 以期寻找到能够用于川贝母及其近缘物种的分子鉴定方法, 并借此探讨川贝母及其近缘物种的亲缘关系, 为川贝母品种资源的合理利用奠定基础。
材料与方法材料 本实验的13种贝母属植物分别为暗紫贝母(F. unibracteata)、太白贝母(F. taipaiensis)、卷叶贝母(F. cirrhosa)、梭砂贝母(F. delavayi)、瓦布贝母(F. wabuensis)、甘肃贝母(F. przewalskii)、康定贝母(F. cirrhosa var. ecirrhosa Franch)、浓蜜贝母(F. mellea)、中华贝母(F. sinica)、长腺贝母(F. unibracteata var. longinectarea)、浙贝母(F. thunbergii)、平贝母(F. ussuriensis)、伊犁贝母(F. pallidiflora)共34份样品(表 1)。所有样品由西南交通大学生命科学与工程学院宋良科副教授与周嘉裕副教授鉴定。标本保存于西南交通大学生命科学与工程学院。
| Table 1 Total materials for experiment |
试剂 北京天根生化有限公司购买的植物基因组DNA提取试剂盒; 宝日医生物技术有限公司购买的SmaI限制内切酶; 北京擎科新业生物技术有限公司购买的PCR扩增试剂盒(TaqPCR Master Mix); 大连宝生物工程有限公司购买的DL2000 Marker。
仪器 Effendorf公司的台式高速离心机, 型号为5415R; ABI公司的PCR仪, 型号为720; Bio-Rad公司的凝胶成像仪, 型号为Universal Hood Ⅱ。
DNA提取 新鲜叶片样品称取100 mg, 干燥鳞茎称取50~60 mg, 用75%的酒精和无菌超纯水擦拭干净, 加入液氮研磨成极细粉, 采用北京天根生化有限公司的植物基因组DNA提取试剂盒提取样品总DNA。
RFLP反应 按照2015版《中国药典》川贝母聚合酶链式反应-限制性内切酶长度多态性(PCR-RFLP)方法中的鉴别引物合成引物序列:正向引物: 5'-CGTA ACAACGTTCCGTAGGTGAA-3';反向引物: 5'-GCTA CGTTCTTCATCGAT-3'。反应总体积为50 μL, 反应体系包括TaqPCR 2×Master Mix 25 μL, 正反引物各1 μL, 模板DNA溶液2 μL, 加入无菌超纯水21 μL, 将离心管放入PCR仪, PCR扩增条件为: 95 ℃预变性4 min, 循环反应30次(95 ℃变性30 s, 55 ℃复性30 s, 72 ℃延伸30 s), 72 ℃延伸5 min。使用北京天根生化有限公司的DNA纯化回收试剂盒对PCR扩增产物进行切胶回收, 并使用微分光度计测纯化产物的浓度。
在200 μL离心管中进行反应, 反应总体系为20 μL, 反应体系包括10×酶切缓冲液2 μL, SmaI (10 U·μL-1) 0.5 μL, 加入浓度为700 ng的纯化产物, 用无菌超纯水将体系补充到20 μL, 酶切反应在30 ℃水浴反应2 h。PCR-RFLP反应产物使用琼脂糖凝胶电泳法进行检测, 胶的浓度为1.5%, 电泳结束后使用凝胶成像仪进行观察。
PCR扩增体系及反应条件 PCR扩增反应体系及条件参考陈士林等[7]方法。ITS2序列扩增引物为: ITS2-F: 5'-ATGCGATACTTGGTGTGAAT-3'; ITS2-R: 5'-GACGCTTCTCCAGACTACAAT-3'。psbA-trnH序列扩增引物为: psbA-F: 5'-GTTATGCATGAACGTAATG CTCPCR-3'; psbA-R: 5'-CGCGCATGGTGGATTCACA TCC-3'。反应体系包括: TaqPCR 2×Master Mix 12.5 μL, 正反引物各1 μL, 模板DNA溶液2 μL, 加入无菌超纯水至25 μL。ITS2序列的PCR扩增条件为: 94 ℃预变性4 min, 循环反应30次(94 ℃变性1 min, 59 ℃退火30 s, 72 ℃延伸1 min), 72 ℃延伸7 min。psbA-trnH序列的PCR扩增条件为: 94 ℃预变性5 min, 循环反应30次(94 ℃变性30 s, 58 ℃退火30 s, 72 ℃延伸2 min), 72 ℃延伸7 min。PCR扩增结束后, 将PCR扩增产物使用浓度为1%的琼脂糖凝胶电泳法进行检测。将PCR产物送到成都擎科梓熙生物技术有限公司进行双向测序。
DNA条形码数据处理及分析 对所获得的原始序列, 采用CExpressi软件对序列拼接并校对。采用基于隐马尔科夫模型的HMMer注释方法去除5.8S和28S序列, 得到准确的ITS2间隔区序列[12]; 测序所得的psbA-trnH序列峰图使用Seqman软件进行校对拼接, 去除引物区和低质量区, 获得psbA-trnH序列。使用MEGA 5.0软件进行序列对比分析, 比较全部样品的序列的保守位点、变异位点、简约信息位点, 基于K2P (Kimura-2-Parameter)双参数模型计算种内种间距离, 使用NJ (Neighbor Joining)法构建系统进化树, 设置bootstrap为1 000次重复。
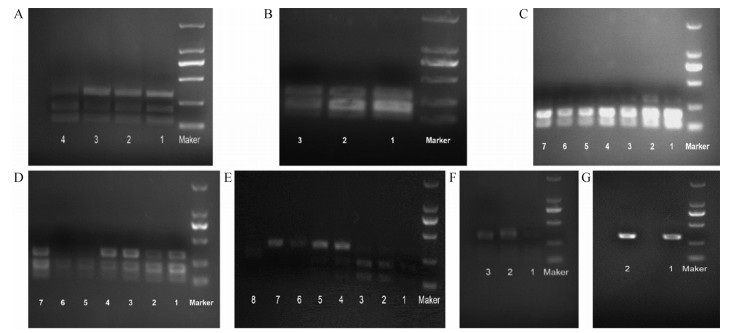
结果与分析 1 RFLP-PCR测定如图 1所示, 浙贝、平贝与伊犁贝母的PCR产物经SmaI酶切后未在100~250 bp间出现2条片段条带; 6种川贝母基原植物的PCR产物酶切后得到2条片段条带, 其分子量处在100~250 bp之间, 能够有效区分川贝母与浙贝、平贝及伊犁贝母。然而, 康定贝母、长腺贝母、中华贝母与浓蜜贝母也同样显示出2条条带, 且片段大小与6种川贝母基原植物几乎相等(图 1中C图的1、2、3、6、7号; D图中的1、2号; E图中的4、5号)。尽管部分片段因为含量高低在琼脂糖凝胶成像中显示模糊, 但并不影响观察。本实验随后对康定贝母、长腺贝母、中华贝母及浓蜜贝母的PCR扩增产物测序, 发现它们在第102位与107位碱基之间均含有Sma I酶切位点(图 2), 从而导致它们与川贝母具有相同的酶切图谱。
|
Figure 1 PCR-RFLP results of different Fritillaria plants. A: 1-2: F. cirrhosa, 3-4: F. taipaiensis; B: 1-3: F. przewalskii; C: 1-3: F. cirrhosa var. ecirrhosa Franch, 4-5: F. unibracteata, 6-7: F. unibracteata var. longinectarea; D: 1-2: F. sinica, 3-4: F. delavayi, 5-7: F.wabuensis; E: 1-3: F. unibracteata, 4-5: F. mellea, 6-8: F. pallidiflora; F: 1-3: F. ussuriensis; G: 1-2: F. thunbergii |

|
Figure 2 The red region was the SmaI cleavage site marker of F. unibracteata var. longinectarea, F. cirrhosa var.ecirrhosa Franch, F. mellea and F. sinica |
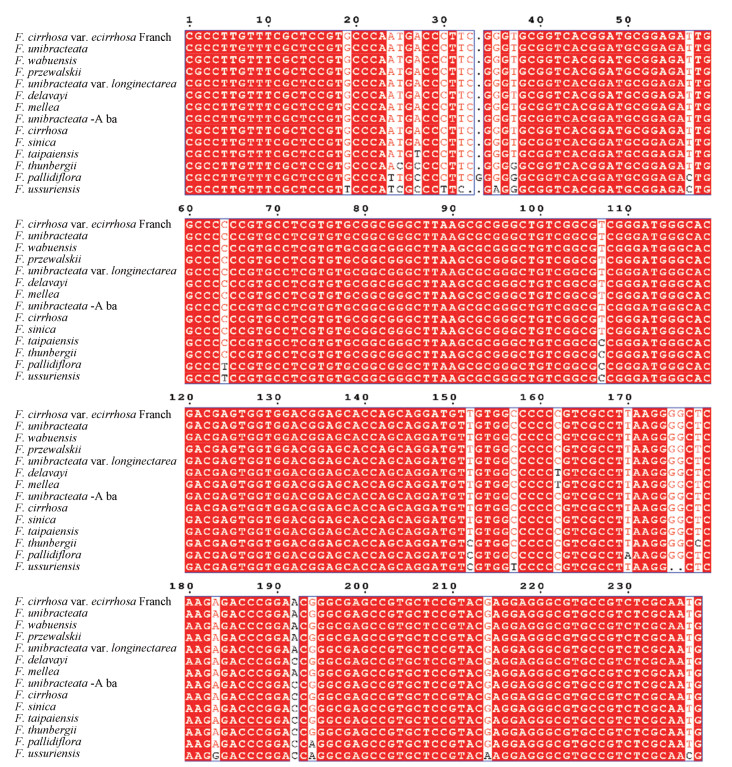
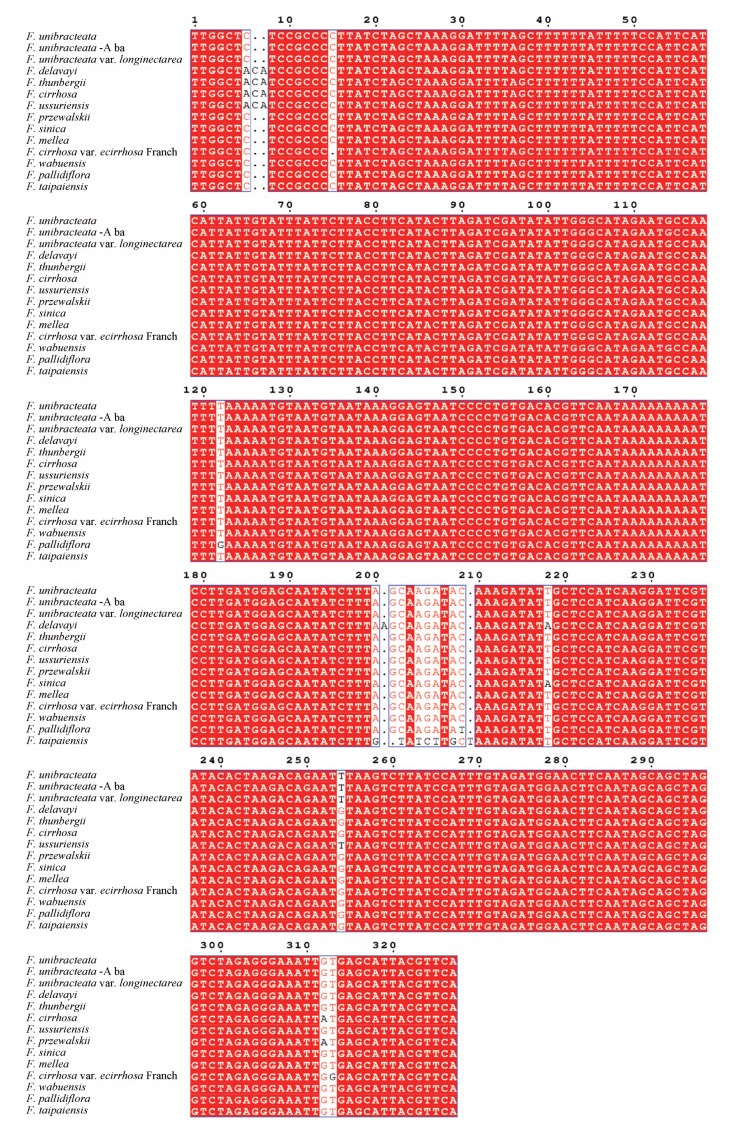
全部样品的ITS2序列长度为235~239 bp, GC含量为67%~72%, psbA-trnH序列的长度为337~373 bp, GC含量为33%~36%。应用MEGA5.0软件对所有样品的ITS2和psbA-trnH序列进行多序列对比, ITS2序列保守位点219个, 变异位点20个, 简约信息位点18个, 分别占总序列长度的91.63%、8.37%和7.53%; psbA-trnH序列保守位点为354个, 变异位点112个, 简约信息位点106个, 分别占总序列长度的74.44%、23.53%和22.27%。
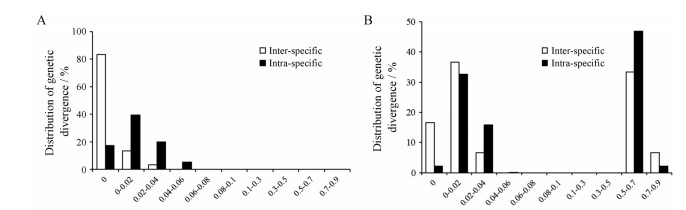
参照陈士林等[3]的方法, 使用MEGA5.0软件计算所有ITS2和psbA-trnH序列的种间、种内的遗传距离, 基于K2-P (Kimura2-parameter)距离模型, 计算种间、种内遗传距离, ITS2序列的种间平均遗传距离为0.022, 种内平均遗传距离为0.001; psbA-trnH序列的种间平均距离为0.329, 种内平均遗传距离为0.263。统计分析样品种间及种内遗传距离, 画出遗传距离分布柱状图(图 3), 从该柱状图可以看出ITS2序列的遗传距离集中在0~0.06, 种内遗传距离高度保守, 在0区域占84%。而psbA-trnH序列的种内与种间距离分布重叠度较高, 在0.5~0.9区域仍有分布, 在0区域仅占17%。所以ITS2序列保守稳定, 更适合作为贝母属植物鉴定的条形码序列。
|
Figure 3 Intraspecific and interspecific genetic distance distribution of ITS2 (A) and psbA-trnH (B) |
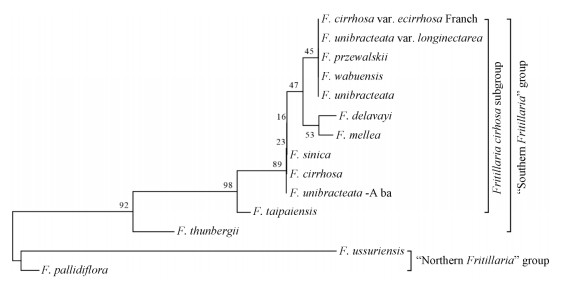
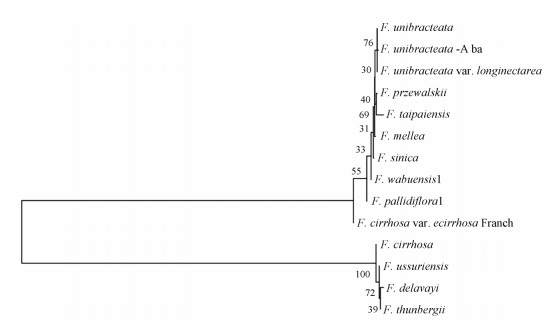
分别构建不同种属不同产地贝母样品的ITS2序列与psbA-trnH序列系统发育树并对建树所用的序列进行对比, 见图 4~7。ITS2序列进化树将所有贝母样品分为南方贝母群与北方贝母群等2个簇, 南方贝母群包括暗紫贝母、长腺贝母、康定贝母、甘肃贝母、瓦布贝母、梭砂贝母、浓蜜贝母、川贝母、中华贝母、浙贝母, 北方贝母群包括伊犁贝母和平贝母; 南方贝母群又分为浙贝母与川贝母复合群, 川贝母复合群中太白贝母单独为一支, 其他贝母为另一分支。ITS2构建的进化树能将川贝母复合群与伊犁贝母、浙贝母、平贝母有效分离鉴定, 同时指示出康定贝母、长腺贝母、中华贝母、浓蜜贝母与川贝母的亲缘关系较近。psbA-trnH序列进化树也将贝母样品分为两大簇, 第一大簇包括暗紫贝母、长腺贝母、太白贝母、甘肃贝母、中华贝母、伊犁贝母、浓蜜贝母和康定贝母, 第二簇包括瓦布贝母、平贝母、浙贝母、川贝母、梭砂贝母等, psbA-trnH序列构建的进化树对贝母样品的分离效果不是很好。
|
Figure 4 NJ tree based on ITS2 sequence |
|
Figure 5 The sequence alignment of ITS2 from different Fritillaria plants |
|
Figure 6 NJ tree based on psbA-trnH sequence |
|
Figure 7 The sequence alignment of psbA-trnH from different Fritillaria plants |
从分子遗传学角度来看, 物种表现型的差异归根结底应追溯到DNA上的差异, 而且这种差异不受外界环境和个体差异的影响。因此, 比较DNA序列差异为植物鉴定与遗传多样性研究提供了本质依据。随着生命科学技术不断取得重大突破, 应用分子标记技术鉴定中药基原植物及饮片取得了快速发展。
本文首先采用《中国药典》分析13种贝母属植物的PCR-RFLP结果, 结果显示该方法对平贝母、浙贝母与伊犁贝母的鉴定效果符合预期。然而, 长腺贝母、康定贝母、中华贝母与浓蜜贝母均含有SmaI酶切位点, 致使它们与6种川贝母基原植物的PCR-RFLP图谱相同, 无法区分。由于SmaI该酶切位点及其上下游DNA序列的保守性, 推测长腺贝母、康定贝母、中华贝母、浓蜜贝母与6种川贝母基源植物可能有较近的亲缘关系。为了验证这一推测, 并探讨这13种贝母属植物的遗传差异, 本实验进一步扩增出ITS2与psbA-trnH序列, 计算遗传距离, 并构建系统进化树。其结果表明, 与psbA-trnH条形码比较, ITS2条形码能准确区分川贝母、浙贝母、平贝母与伊犁贝母, 并将6种不同的川贝母基原植物:卷叶贝母、暗紫贝母、甘肃贝母、梭砂贝母、太白贝母、瓦布贝母聚类在一起, 验证了药典的正确性, 表明ITS2在贝母属植物的鉴定中有更大的优势。ITS2将不同贝母属植物区分为“北方贝母群”与“南方贝母群”, 前者包括平贝母与伊犁贝母, 后者主要由川贝母与浙贝母组成, 这个结果与Yu等[13]的结果吻合。伊犁贝母与川贝母复合群的亲缘关系最远, 平贝母稍近, 浙贝母最近, Lu等[14]的研究也支持上述结果。在“南方贝母群”中, 川贝母形成了一个庞大的分类复合群, 包括了长腺贝母、康定贝母、中华贝母、浓蜜贝母与6种川贝母基原植物, 多个川贝母物种呈多物种交错排列, 推测此川贝母复合群可能是处于激烈分化的物种形成阶段, 其遗传变异可能受到了地理分布、品质与形态的影响, 此推论与Xiao等[15]的讨论相符。在川贝母复合群中, 太白贝母与其他川贝母物种并无聚类, 单独成为一支, 该分类结果验证了Luo等[16]的推测, 太白贝母与川贝母亲缘关系虽近, 但却是单独一个物种。暗紫贝母与长腺贝母靠得最近, 表明它们亲缘关系最近, 这一结果与《四川植物志》记载一致, 长腺贝母属于暗紫贝母的变种, 其主要特征与暗紫贝母大部分一致, 仅蜜腺稍长。Xu等[17]认为康定贝母与暗紫贝母或卷叶贝母有相近的亲缘关系, 本文的聚类结果显示康定贝母与暗紫贝母、长腺贝母更为接近, 而距卷叶贝母较远, 造成这一结果的原因可能有2个, 第1个原因是康定贝母与暗紫贝母亲缘关系更近, 从形态上比较, 康定贝母与暗紫贝母有明显相似的特征, 例如它们均含1个苞片、叶尖不卷曲或少卷曲, 而卷叶贝母含3个苞片、叶尖卷曲。第2个可能原因是由于地理分布因素, 本文中卷叶贝母采集于青海省西宁市, 康定贝母采集于四川省康定市, 两地距离较远, 造成了基因流向阻碍, 产生了单独进化, 导致遗传相似度下降。系统进化树还显示, 甘肃贝母与暗紫贝母高度相似, 但它们之间有一些过渡群, 例如长腺贝母与康定贝母, 这一结果与Li等[18]的推测一致, 他们比较了甘肃贝母、暗紫贝母与卷叶贝母的鳞茎形态与大小, 认为甘肃贝母与暗紫贝母更相近。瓦布贝母显示出与甘肃贝母及暗紫贝母具有较近的亲缘关系, 其结果与Li等[19]的川贝母RADP分析结果相同。Su等[20]发现ISSR将浓蜜贝母与梭砂贝母聚在一起, 本研究中亦发现浓蜜贝母与梭砂贝母聚在一起。然而, 梭砂贝母在形态上与浓蜜贝母变化很大, 这可能是由于ITS2序列与贝母形态的形成与发生无直接关联。Xiao等[15]亦发现贝母属植物的遗传关系很复杂, 极有可能出现形态学变化较大但化学成分或遗传物质比较一致的现象。本研究首次发现中华贝母出现在川贝母复合群中, 尽管其与梭砂贝母、卷叶贝母及暗紫贝母联在一起, 但仍有较大的遗传距离, 这可能与它们均处在横断山区有关(肖培根等2007)。
ITS2序列不仅能够用于不同贝母属植物物种的鉴定, 还能够反映不同居群间的遗传多样性。例如, 来自青海西宁的暗紫贝母与四川阿坝的暗紫贝母形成了2个居群, 它们存在较大的遗传变异, 在进化树中距离较远。
本研究利用PCR-RFLP与DNA条形码鉴定方法对34份贝母样品进行鉴定, 发现长腺贝母、康定贝母、浓蜜贝母与中华贝母位于川贝母复合群中, 与6种川贝母基原植物有较近的亲缘关系, 无法通过PCR-RFLP鉴定, 急需补充其他类型分子标记; ITS2能够将川贝母、浙贝母、平贝母与伊犁贝母分开, 并且有效聚类6种川贝母基原植物, 其鉴定效果优于psbA-trnH序列; 然而ITS2尚无法完成暗紫贝母、甘肃贝母、瓦布贝母、川贝母的分子鉴定, 例如暗紫贝母1、暗紫贝母2、暗紫贝母3、甘肃贝母1、甘肃贝母2、甘肃贝母3与瓦布贝母1、瓦布贝母2、瓦布贝母3的序列相似度为100%, 川贝母1、川贝母2、梭砂贝母2与暗紫贝母4和暗紫贝母5的序列相似度为100%;推测贝母属植物可能起源于同一祖先物种, 该祖先物种在进化过程中首先分化产生“北方贝母群”与“南方贝母群”, 随后后者分化产生浙贝与“川贝母复合群”。由于“北方贝母群”与“南方贝母群”的分歧时间较早, ITS2序列变异较大; 而“川贝母复合群”分歧时间较近, 导致部分川贝母的ITS2变异程度较低甚至未发生变异。由此, 多分子标记的联合使用是一种可行解决方法; 同时, 依托川贝母功能基因组信息, 以筛选特异性更高的分子标记也是本实验室下阶段的研究重点。另外, 本文发现康定贝母与暗紫贝母亲缘关系更近, 该结果与前人研究和传统分类标准相异, 还有待进一步研究。
致谢: 感谢青海绿康生物开发有限公司及阿坝州食品药品检验所提供的帮助。
| [1] |
Xu SC, Xiao PG. Medicinal history and development direction of traditional Chinese medicine Fritillaria[J]. China J Chin Mater Med (中国中药杂志), 1990, 15: 6-8. |
| [2] |
Zhang ZY, Yang J, Qi ZM. Research progress of Fritillaria[J]. Jiangsu Agric Sci (江苏农业科学), 2017, 45: 9-13. |
| [3] |
Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5: e8613. DOI:10.1371/journal.pone.0008613 |
| [4] |
Shi ZH, Chen SL, Yao H, et al. DNA barcode identification of original species in Aesculus Linn[J]. Chin Tradit Herb Drugs (中草药), 2013, 44: 2593-2599. |
| [5] |
Casiraghi M, Labra M, Ferri E, et al. DNA barcoding: a six-question tour to improve users' awareness about the method[J]. Brief Bioinform, 2010, 11: 440-453. DOI:10.1093/bib/bbq003 |
| [6] |
Sun ZY, Chen SL, Yao H, et al. Identification of Notopterygii Rhizoma et Radix and its adulterants using DNA barcoding method based on ITS2 sequence[J]. Chin Tradit Herb Drugs (中草药), 2012, 43: 568-571. |
| [7] |
Zhu YJ, Chen SL, Yao H, et al. DNA barcoding the medicinal plants of the genus Paris[J]. Acta Pharm Sin (药学学报), 2010, 45: 376-382. |
| [8] |
Hao J, Cai YP, Liu G, et al. Identification of genetic relationships between medicinal Licorices based on ITS2 sequences[J]. Chin Pharm J (中国药学杂志), 2018, 53: 411-417. |
| [9] |
Yang P, Zhou H, Ma SJ, et al. Authenticatio of raw material for edible and medicinal Cinnamon based on plastid intergenic region psbA-trnH[J]. Chin Pharm J (中国药学杂志), 2015, 50: 1496-1499. |
| [10] |
Xu J, Xu T, Xiao ZK, et al. Analysis of psbA-trnH gene sequences of Granineae and its application in identification of Oryza punctata[J]. Biotechnol Bull (生物技术通报), 2011(02): 85-92. |
| [11] |
Chen SL, Yao H, Han JP, et al. Principles for molecular identification of traditional Chinese materia medica using DNA barcoding[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 141-148. |
| [12] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731-2739. DOI:10.1093/molbev/msr121 |
| [13] |
Yu C, Liang XQ, Chen JJ, et al. Identification of plants in Fritillariae L. by DNA barcoding technology[J]. Chin Tradit Herb Drugs (中草药), 2014, 45: 1613-1619. |
| [14] |
Lu H, Zhu SH, Zhou SJ, et al. Specific RAPD screening of breeds in Fritillaria thunbergii and genetic diversity analysis of five species of Fritillaria[J]. J Ningbo Univ (Nat Sci Eng Ed) (宁波大学学报自然科学与工程版), 2009, 2: 44-47. |
| [15] |
Xiao PG, Jang Y, Li P, et al. The botanical origin and pharmacophylogenetic treatment of Chinese materia medica Beimu[J]. Acta Phytotaxon Sin (植物分类学报), 2007, 45: 473-487. DOI:10.1360/aps06113 |
| [16] |
Luo YB, Chen XQ. A revision of Fritillaria L. (Liliaceae) in the Hengduan Mountains and adjacent regins, China(l)-a study of Fritillaria cirrhosa D. Don and ITS related species[J]. Acta Phytotaxon Sin (植物分类学报), 1996, 34: 304-312. |
| [17] |
Xu Y, Zhang JZ, Cheng CJ, et al. Study on the botanical origin of Chinese materia medica Bei-mu from Western Sichuan Plateau[J]. J Southwest Minzu Univ (Nat Sci Ed) (西南民族大学学报自然科学版), 2011, 37: 617-620. |
| [18] |
Li KQ, Wu W. A karyological study of Fritillaria from Sichuan Province[J]. Hubei Agric Sci (湖北农业科技), 2016, 55: 4224-4229. |
| [19] |
Li Q, Chen X, Wang S. Study on the relative in molecular biology among Fritillariae of Sichuan Species[J]. West China J Pharm Sci (华西药学杂志), 2010, 25: 140-143. |
| [20] |
Su P, Hu L, Dong PL. Identification of eight varieties of bullbus Fritillaria cirrhosa[J]. Southwest China J Agric Sci (西南农业学报), 2014, 27: 2559-2563. |
 2019, Vol. 54
2019, Vol. 54


