苦参(Sophora flavescens Ait.)为豆科槐属(Sophora)多年生落叶亚灌木或多年生草本植物[1], 其干燥根入药, 具有清热、燥湿、杀虫、利尿等功效[2]。苦参的主要化学成分为奎诺里西啶生物碱和黄酮类化合物[3], 苦参除用于中药方剂外, 还是制药工业中常用的制剂、日用品、生物农药和兽药的主要原料, 如:苦参碱注射液[4]、苦参碱滴眼液[5]、苦参碱杀虫剂[6]等。
叶绿体是细胞内半自主性的细胞器, 自身拥有相对独立的遗传物质, 即叶绿体基因组。叶绿体基因组所包含的遗传信息虽远小于核基因组, 但叶绿体基因组结构稳定、高度保守[7], 在植物系统发育、物种鉴定和遗传转化等研究中都具有重要意义[8-12]。近年来, 随着测序技术的发展和叶绿体基因组生物信息学分析技术的成熟, 叶绿体基因组的研究也得到了迅速发展。自1986年首次测序获得烟草(Nicotiana tabacum)[13]和地钱(Marchantia polymorpha)[14]叶绿体基因组的完整序列以来, 叶绿体基因组数据库迅速增加充实。之后, 不断有新物种的叶绿体基因组被测得。叶绿体基因组多是环式双链结构, 典型的环式双链叶绿体基因组结构包括4个部分:大单拷贝区(large single copy, LSC)和小单拷贝区(small single copy, SSC), 以及两个反向重复区(inverted repeats, IRs)将其分隔开[15]。
目前已有多种豆科植物的叶绿体基因组被报道[16-18], Zhang等[19]对苦参叶绿体基因组测序并进行遗传进化分析, 但并未对其叶绿体基因组进行详细论述。本研究通过对苦参叶绿体基因组测序, 从SSR、密码子偏好性、比较基因组学等方面对其进行分析, 以期为苦参的遗传变异、育种以及豆科植物的系统进化发育和药用植物的分子系统发育提供重要的理论基础和依据。
材料与方法材料 本研究所用苦参种植于山西农业大学生命科学学院药用植物园, 于2018年7月选取一年生植株的新鲜叶片, 液氮速冻后于-80 ℃保存备用。
DNA的提取、测序 用改良的CTAB法提取叶片DNA, 琼脂糖凝胶电泳和NANODROP 2000C (Thermo Fisher Scientific, 美国)检测DNA质量和浓度。DNA检测合格后, 用机械打断(超声波)的方法将DNA片段化, 经过片段纯化、末端修复、3'端加A、连接测序接头, PCR扩增等获得测序文库, 质检合格后用Illumina HiSeq (陕西博瑞德生物科技有限公司)进行测序。
叶绿体基因组的注释和比较基因组学分析 运用MITObim软件[20]对叶绿体基因组进行组装, 参考序列为Sophora alopecuroides L. (NC_036102), 将获得的叶绿体基因组用GeSeq软件[21]进行基因注释并将最终结果提交至GenBank (MN205931)。利用OGDRAW在线软件[22]绘制苦参叶绿体基因组图谱, 并比较和分析两个不同地区苦参的叶绿体基因组。
SSR分析 利用MISA软件[23]对苦参Unigene序列进行分析, 参数设置为:单核苷酸重复次数 > 10, 二核苷酸重复次数 > 6, 三核苷酸、四核苷酸、五核苷酸和六核苷酸重复次数 > 5; 2个SSR之间的最小距离设置为100 bp。
密码子偏好性分析 苦参叶绿体基因组的密码子组成分析运用软件CodonW1.4.2 (availableonline: downloads.fyxm.net/CodonW-76666.html)[24], 获得相关参数:有效密码子数(effective number of codon, Nc)、相对同义密码子使用情况(relative synonymous codon usage, RSCU)、GC含量和第三位上的GC碱基的含量(GC3s)。
系统发育分析 利用BioEdit软件[25]对17个豆科物种的叶绿体基因组序列进行比对, 将比对结果采用MEGA7.0软件[26]ML法构建系统进化树。
结果与分析 1 叶绿体基因组的基本特征采用Illumina HiSeq高通量测序平台对苦参叶绿体基因组测序, 经过测序质量控制[27], 样品Q30碱基百分比不小于90%, 利用MITObim软件对其组装得到苦参的叶绿体全基因组。苦参的叶绿体基因组呈现典型的四段式结构(图 1), 包括大单拷贝区(LSC)、小单拷贝区(SSC), 以及两个大小相同、方向相反的反向重复区(IRA, IRB)。

|
Figure 1 Chloroplast genome map of Sophora flavescens. Genes drawn inside the circle are transcribed clockwise, genes outside are transcribed counter clockwise. Genes are color coded by their function in the legend. The area in darker gray and lighter gray in the inner circle indicates GC content and AT content, respectively |
苦参的叶绿体基因组全长154 165 bp (表 1), 其中LSC、SSC、IR区域分别为84 413 bp、18 056 bp、51 696 bp, GC含量为36.5%, 其中LSC和SSC区域分别为34.1%、30.1%, 而IR区域的GC含量为42.7%。苦参叶绿体基因组的基因含量和序列较为保守, 共注释123个基因(表 2), 包括77个蛋白质编码基因(62.6%)、38个tRNA基因(30.9%)以及8个rRNA基因(6.5%)。按照功能可将这些基因分为4类:其中atpA、ndhB、psaI、rbcL等44个基因与光合作用相关; rpl2、rps2、trnA-UGC等57个基因与自我复制相关; 还有其他基因和功能未知的基因。其中有13个基因含有内含子, ycf3、clpP以及trnI-GAU含有两个内含子。
| Table 1 Statistics on the chloroplast gene annotation of Sophora flavescens |
| Table 2 Functional classification of Sophora flavescens chloroplast gene. *Indicate the intron-containing genes |
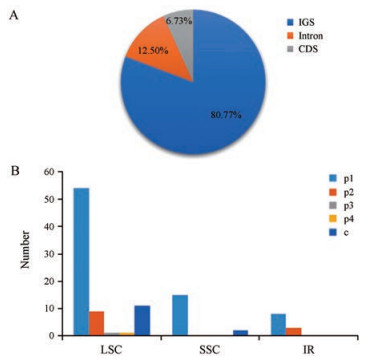
利用MISA软件对苦参Unigene序列进行分析, 共检测到104个SSR位点, 大部分的SSR位于基因间隔区(intergenic spacer, IGS), 共有84个(80.77%); 而分布于基因编码区(coding sequence, CDS)的SSR最少, 只有7个(6.73%) (图 2A)。同时发现大部分的SSR均位于LSC区域内, 只有少数位于SSC和IR区域内, 其中位于IR区的SSR最少。SSR位点包括单核苷酸重复、双核苷酸重复、三核苷酸重复、四核苷酸重复以及复合SSR。其中单核苷酸重复最多, 主要分布在LSC区域, 而三核苷酸重复和四核苷酸重复数量最少, 分布于LSC区域内(图 2B)。苦参叶绿体基因组中的SSR主要是由A和T组成的, 其中大部分是以A/T碱基构成的单核苷酸重复, 共检测到73条, 其次是AT/AT组成的二核苷酸重复序列, 二者合计占所有重复序列的90%以上。
|
Figure 2 SSR distribution of Sophora flavescens chloroplast genome. A: Distribution of SSR in the coding region, intron region, and intergenic region of the gene; B: Distribution of various types of SSRs in LSC, SSC and IR regions |
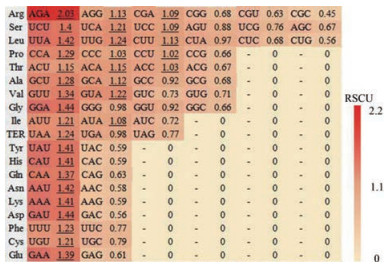
利用CodonW1.4.2对苦参叶绿体基因组的密码子组成进行分析。结果表明(表 3), 苦参叶绿体基因组的Nc值为55.34, 说明该叶绿体基因组的密码子偏好性较弱。苦参叶绿体基因组的GC3s含量为35.4%, GC含量也小于50%, 说明苦参叶绿体基因的密码子偏向使用A和T这两种碱基。对苦参叶绿体基因组的RSCU进行分析(图 3), RSCU > 1的密码子有32个, 其中以A/T碱基结尾的有27个, 占84.38%, 表明这些密码子偏向以A/T结尾。
| Table 3 Overall characteristics of codon usage of Sophora flavescens chloroplast genome |
|
Figure 3 Relative synonymous codon usage of Sophora flavescens chloroplast genome. The value of the relative synonymous codon usage (RSCU > 1) is shown in underline |
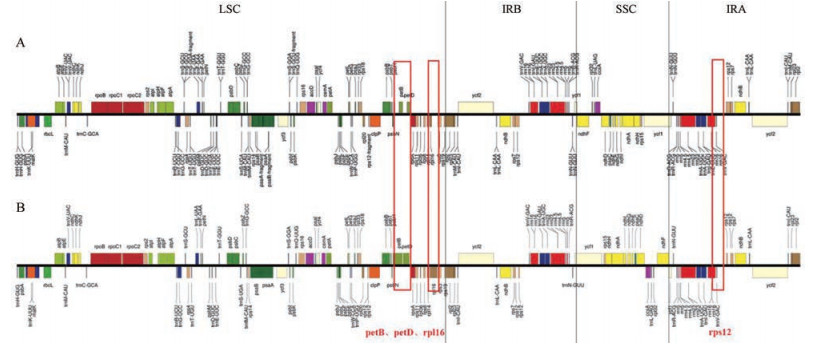
将已测序苦参叶绿体基因组作为参考, 与贵阳地区的苦参叶绿体基因组序列[19]进行相似性比较分析(图 4)。结果表明, 两个不同地区苦参叶绿体基因组序列整体上高度相似, 但其仍存在明显差异。二者的叶绿体基因组长度不同, 相差213 bp, 且其在LSC区和IR区有明显区别, 主要表现为petB、petD、rpl16和rps12等基因的扩张和收缩。在两个叶绿体基因组的LSC/IR和SSC/IR边界区, 由于IR区正常拷贝时的不完整, 产生了rps19和ycf1两个假基因。其中rps19基因正好位于IRb/LSC边界区并截止于LSC区。而ycf1基因位于IR/SSC边界区, 横跨了IR区和SSC区。
|
Figure 4 Comparison of chloroplast genomes of Sophora flavescens in different regions. A: Sophora flavescens that have been sequenced in this study; B: Sophora flavescens in another different region |
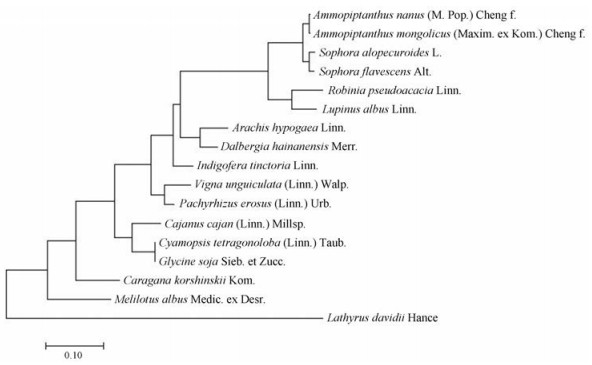
为了鉴定苦参在豆科中的进化位置, 本研究以苦参叶绿体基因组和已公布的16种豆科植物叶绿体基因组序列为研究对象, 利用BioEdit软件对17个物种的叶绿体全基因组序列进行比对, 将比对结果采用MEGA7.0软件ML法(maximum likelihood, 最大似然法)构建系统进化树(图 5)。结果显示, 苦参与同属植物苦豆子(Sophora alopecuroides L.)的亲缘关系最近, 二者的遗传距离为0.019。同时位于山黧豆属的大山黧豆(Lathyrus davidii)与其他16个物种的亲缘关系最远, 遗传距离均大于0.75, 为单系分支。
|
Figure 5 The phylogenetic tree was constructed based on the chloroplast genome of 17 legumes using the maximum likelihood method |
苦参是一种重要的药用植物, 但是目前对苦参叶绿体基因组方面的研究还很有限。而叶绿体基因组高度保守, 含有大量的遗传信息, 对研究苦参的遗传变异、育种及该科、属内物种的系统进化发育等具有重要意义。
本研究通过测序获得苦参叶绿体基因组序列, 并对其进行分析。SSR位点分析对研究苦参的物种进化、遗传和变异具有重要的意义[28]。经分析共鉴定出104个SSR, 多分布于LSC区, 主要由碱基A和T组成。由于AT键相对于GC键少了一个氢键, 其断裂所需的能量远小于GC键, 相对于GC键而言更易断裂发生变动, 因此在叶绿体基因组中含AT重复基序结构的SSR重复类型出现的可能性更大, 这与Kuang和Qian等的研究结果一致[29, 30]。大部分的SSR位于IGS区, 而分布于CDS区的SSR最少, 这是由于生物体的外显子区域相对较保守, 而内含子区和非编码区的保守性比外显子区要弱, 因此SSR在这两个区域出现的可能性增加[31]。
对苦参叶绿体基因组的密码子进行分析, 获得相关参数Nc、RSCU、GC和GC3s。Nc值的范围在20~61之间, 越靠近20表明密码子的偏好性越强, 反之, 越偏向61密码子的偏好性越弱[32]。经分析发现苦参叶绿体基因组的Nc值大于40, 说明苦参叶绿体基因组的密码子使用偏好性较弱。RSCU=1, 表明该密码子的使用没有偏好性, RSCU > 1, 说明该密码子的使用频率较高, 反之则较低[33]。发现RSCU > 1的密码子更多, 且偏向以A/T结尾。GC和GC3s含量在密码子的偏好性分析中也具有重要意义, 经测定苦参叶绿体基因组的GC含量和GC3s含量均小于50%, 说明苦参叶绿体基因组的密码子偏好使用A和T碱基。
植物叶绿体基因组的扩张和收缩是植物比较基因组学中的重要数据, 它们被认为是叶绿体基因组变异的主要机制[34, 35]。本研究对两个不同地区的苦参叶绿体基因组进行比较分析, 发现二者的叶绿体基因组序列整体上高度相似, 但其仍存在明显差异, 主要表现为petB、petD、rpl16和rps12等基因的扩张和收缩。根据这些基因区域的变化可进行DNA条形码的开发, 有望利用DNA条形码区分不同地区苦参, 以此来优化苦参种质资源并进行苦参的良种繁育。基于整个叶绿体基因组的系统发育分析显示, 苦参与同属的苦豆子的亲缘关系最近。利用叶绿体基因组可为解决豆科槐属内植物的分类问题提供参考, 但是已发表的槐属植物叶绿体基因组数据有限, 因此, 还需要获得更多槐属植物的叶绿体基因组才能更好的研究该属植物的系统发育关系。
结论本文通过对苦参叶绿体基因组进行测序、注释、分析和预测, 获得其全长154 165 bp, 共注释到123个基因, 筛选了104个SSR位点。同时分析发现其基因组密码子偏好性较弱。与另一不同地区苦参叶绿体基因组进行比较分析后, 发现二者在LSC区有较大差异。经系统进化分析可知, 苦参与同属植物苦豆子的亲缘关系最近。本研究对苦参的系统进化发育以及遗传变异、育种等方面都具有重要作用, 同时也为苦参和叶绿体基因组未来的研究奠定了重要基础。
| [1] | He TN. Gentianaceae in Flora Reipublicae Popularis Sinicae: Vol 73(中国植物志: 73卷)[M]. Beijing: Science Press, 1983. |
| [2] | Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China: Vol I (中华人民共和国药典: 一部)[S]. Beijing: China Medical Science Press, 2015: 202-203. |
| [3] | Liu G, Dong J, Wang H, et al. Characterization of alkaloids in Sophora flavescens Ait. by high-performance liquid chromatography-electrospray ionization tandem mass spectrometry[J]. J Pharm Biomed Anal, 2011, 54: 1065–1072. DOI:10.1016/j.jpba.2010.12.024 |
| [4] | Dong Y, Li YX, Yan X, et al. Protection effect of matrine against precancerous lesion of gastric cancer in model rats[J]. China Pharm (中国药房), 2010, 21: 1357–1359. |
| [5] | Zhang JR. Effects of matrine on cisplatinum sensitivity of epithelial ovarian cancer and analysis of its potential mechanism[J]. Guide China Med (中国医药指南), 2010, 8: 26–28. |
| [6] | Zhang JH, Zhao YY, Liu QX, et al. Studies on the chemical constituents from Sophora flavescens Ait[J]. China J Chin Mater Med (中国中药杂志), 2000, 25: 37–39. |
| [7] | Meng J, Li XP, Li HT, et al. Comparative analysis of the complete chloroplast genomes of four aconitum medicinal species[J]. Molecules, 2018, 23: 1015–1017. DOI:10.3390/molecules23051015 |
| [8] | Tonti-Filippini J, Nevill PG, Dixon K, et al. What can we do with 1000 plastid genomes?[J]. Plant J, 2017, 90: 808–818. DOI:10.1111/tpj.13491 |
| [9] | Zhou J, Chen X, Cui Y, et al. Molecular structure and phylogenetic analyses of genomes of two aristolochia medicinal species[J]. Int J Mol Sci, 2017, 18: 1839. DOI:10.3390/ijms18091839 |
| [10] | Yu XQ, Drew BT, Yang JB, et al. Comparative chloroplast genomes of eleven Schima (Theaceae) species: insights into DNA barcoding and phylogeny[J]. PLoS One, 2017, 12: e0178026. DOI:10.1371/journal.pone.0178026 |
| [11] | Xu C, Dong W, Li W, et al. Comparative analysis of six lagerstroemia complete chloroplast genomes[J]. Front Plant Sci, 2017, 8: 15. |
| [12] | Gu C, Tembrock LR, Zheng S, et al. The complete chloroplast genome of Catha edulis: a comparative analysis of genome features with related species[J]. Int J Mol Sci, 2018, 19: 525. DOI:10.3390/ijms19020525 |
| [13] | Shinozaki K, Ohme M, Tanaka M, et al. The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression[J]. Plant Mol Biol Rep, 1986, 5: 2043–2049. |
| [14] | Ohyama K, Fukuzawa H, Kohchi T, et al. Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA[J]. Nature, 1986, 322: 572–574. DOI:10.1038/322572a0 |
| [15] | Mu XP, Wang PF, Du JJ, et al. The chloroplast genome of Cerasus humilis: genomic characterization and phylogenetic analysis[J]. PLoS One, 2018, 13: e0196473. DOI:10.1371/journal.pone.0196473 |
| [16] | Deng CY, Xin GL, Zhang JQ, et al. Characterization of the complete chloroplast genome of Dalbergia hainanensis (Leguminosae), a vulnerably endangered legume endemic to China[J]. Conserv Gen Res, 2018, 11: 105–108. |
| [17] | Tao X, Ma L, Zhang Z, et al. Characterization of the complete chloroplast genome of alfalfa (Medicago sativa) (Leguminosae)[J]. Gene Rep, 2016, 6: 67–73. |
| [18] | Sakai M, Kanazawa A, Fujii A, et al. Phylogenetic relationships of the chloroplast genomes in the genus Glycine inferred from four intergenic spacer sequences[J]. Plant System Evol, 2003, 239: 29–54. DOI:10.1007/s00606-002-0226-9 |
| [19] | Zhang WL, Li L, Li GH. Characterization of the complete chloroplast genome of shrubby sophora (Sophora flavescens Ait.)[J]. Mitochondrial DNA Part B, 2018, 3: 1282–1283. DOI:10.1080/23802359.2018.1532839 |
| [20] | Freyer R, Hoch B, Neckermann K, et al. RNA editing in maize chloroplasts is a processing step independent of splicing and cleavage to monocistronic mRNAs[J]. Plant J, 1993, 4: 621–629. DOI:10.1046/j.1365-313X.1993.04040621.x |
| [21] | Tillich M, Lehwark P, Pellizzer T, et al. GeSeq-versatile and accurate annotation of organelle genomes[J]. Nucleic Acids Res, 2017, 45: W6–W11. DOI:10.1093/nar/gkx391 |
| [22] | Lohse M, Drechsel O, Kahlau S, et al. Organellar Genome DRAW—a suite of tools for generating physical maps of plastid and mitochondrial genomes and visualizing expression data sets[J]. Nucleic Acids Res, 2013, 41: W575–W581. DOI:10.1093/nar/gkt289 |
| [23] | Thiel T, Michalek W, Varshney R, et al. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.)[J]. Theo Appl Gen, 2003, 106: 411–422. DOI:10.1007/s00122-002-1031-0 |
| [24] | Shields DC, Sharp PM. Synonymous codon usage in Bacillus subtilis reflects both translational selection and mutational biases[J]. Nucleic Acids Res, 1987, 15: 8023–8040. DOI:10.1093/nar/15.19.8023 |
| [25] | Tippmann HF. Analysis for free: comparing programs for sequence analysis[J]. Briefings Bioinf, 2004, 5: 82. DOI:10.1093/bib/5.1.82 |
| [26] | Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33: 1870. DOI:10.1093/molbev/msw054 |
| [27] | Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet J, 2011. DOI:10.14806/ej.17.1.200 |
| [28] | Hu J, Gui S, Zhu Z, et al. Genome-wide identification of SSR and SNP markers based on whole-genome re-sequencing of a Thailand wild sacred lotus (Nelumbo nucifera)[J]. PLoS One, 2015, 10: e0143765. DOI:10.1371/journal.pone.0143765 |
| [29] | Kuang DY, Wu H, Wang YL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics[J]. Genome, 2011, 54: 663–673. DOI:10.1139/g11-026 |
| [30] | Qian J, Song J, Gao H, et al. The complete chloroplast genome sequence of the medicinal plant Salvia miltiorrhiza[J]. PLoS One, 2013, 8: e57607. DOI:10.1371/journal.pone.0057607 |
| [31] | Li XY, Xiao BG, Gao YL, et al. SSR locus analysis of chloroplast genome and mitochondrial genome of tobacco[J]. Acta Bot Sin, 2011, 31: 2399–2405. |
| [32] | Ikemura T. Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes: a proposal for a synonymous codon choice that is optimal for the E. coli translational system[J]. J Mol Biol, 1981, 151: 389–409. DOI:10.1016/0022-2836(81)90003-6 |
| [33] | Sharp PM, Tuohy TMF, Mosurski KR. Codon usage in yeast: cluster analysis clearly differentiates highly and lowly expressed genes[J]. Nucleic Acids Res, 1986, 14: 5125–5143. DOI:10.1093/nar/14.13.5125 |
| [34] | Chumley TW, Palmer JD, Mower JP, et al. The complete chloroplast genome sequence of Pelargonium hortorum: organization and evolution of the largest and most highly rearranged chloroplast genome of land plants[J]. Mol Biol Evol, 2006, 23: 2175–2190. DOI:10.1093/molbev/msl089 |
| [35] | Yang M, Zhang X, Liu G, et al. The complete chloroplast genome sequence of date palm (Phoenix dactylifera L.)[J]. PLoS One, 2010, 5: e12762. DOI:10.1371/journal.pone.0012762 |
 2019, Vol. 54
2019, Vol. 54


