信使RNA (messenger RNA, mRNA)是联系DNA与蛋白质的核心分子, 通过密码子方式指导蛋白的合成, 从而参与遗传信息传递过程, 是生命的核心进程。1974年首次发现信使RNA (mRNA)存在N6-甲基腺苷(N6-methyladenosine, m6A)[1], 此外mRNA上还存在N7-甲基鸟苷(7-methylguanosine, m7G)和N6-2-氧-二甲基腺苷(N6-2-O-dimethyladenosine, m6Am)等多种甲基化修饰[2]。m6A是大多数真核生物mRNA中丰度最高的化学修饰[3], 由于当时技术限制, 关于mRNA修饰的作用及功能研究进展缓慢。2011年, 芝加哥大学何川教授实验室[4]报道了第一个mRNA去甲基化酶脂肪和肥胖相关蛋白(fat-mass and obesity-associated protein, FTO), 其可特异性将mRNA上的m6A转化为A, 表明mRNA上甲基化修饰是动态可逆过程, 从而拉开RNA表观遗传学/表观转录组学的研究热潮。
根据中心法则, 遗传信息从DNA传递给RNA, 再从RNA传递给蛋白质, 从而完成遗传信息的转录和翻译。由于RNA的生物学行为涉及转录、剪切、出入核、翻译及降解等众多步骤, 其调控机制备受关注。作为mRNA上丰度最高的修饰, m6A广泛存在于原核和真核生物中。Dominissini等[5]通过m6A测序(m6A sequencing, m6A-seq)发现人的mRNA上有超过12 000个m6A修饰位点, 其主要分布于终止密码子附近和长外显子内, 并且修饰位点的分布在人鼠之间高度保守。随后, Meyer等[6]通过甲基化RNA免疫沉淀反应测序(methylated RNA immunoprecipitation sequencing, MeRIP-seq)发现7 676个哺乳类基因的mRNA上存在m6A修饰, 主要分布于终止密码子附近和3'-非翻译区(3'-untranslated region, 3'UTR)。由于RNA在基因表达中的中心作用, 越来越多的证据支持RNA生物学在肿瘤中发挥着重要的功能[7]。本文综述了m6A调控mRNA的生物学行为, 研究其在肿瘤细胞增殖、转移、能量代谢和血管生成等恶性生物学行为中的作用, 阐述其在急性白血病等多种肿瘤中的作用及机制, 探讨其相应的靶向治疗策略, 并评价其作为肿瘤治疗靶点的可行性。
1 m6A在mRNA修饰中的作用及生物学意义 1.1 m6A及相关酶与DNA和组蛋白甲基化相类似, RNA m6A甲基化修饰是一个动态可逆的表观调控过程[4], 主要由甲基化酶(writers)、去甲基化酶(erasers)和识别蛋白(readers)所调控[3, 8]。其中m6A甲基转移酶如甲基转移酶样因子3/14 (methyltransferase like 3/14, METTL3/14)和/或Wilm'肿瘤1相关蛋白(Wilm's tumor 1-associated protein, WTAP)[9]和去甲基化酶FTO[4]和AlkB同源物5 (AlkB homolog 5, ALKBH5)[10]分别负责细胞核内mRNA的甲基化及去甲基化。而识别蛋白如YT521-B同源性(YT521-B homology, YTH)家族蛋白YTHDF1~3和核成员YTHDC1~2、异质核核糖核蛋白(heterogeneous nuclear ribonucleoprotein, hnRNPs)等则可识别mRNA的m6A从而调控mRNA生物学行为(图 1)。

|
Figure 1 The m6A modification, related proteins and biological functions. m6A modification is dynamically reversible in mammalian cells and regulated by m6A methyltransferase (writers), demethylase (erasers), and "reader" proteins. m6A can regulate various biological processes of mRNA such as RNA splicing, nuclear export, protein translation and degradation |
m6A甲基化酶是将甲基化修饰“写入”RNA从而介导RNA甲基化修饰过程, 由METTL3、METTL14和WTAP复合物组成[9]。该多组分甲基转移酶复合物最初是从HeLa细胞核提取物中分离出来的[11, 12]。蛋白METTL3是该复合物的催化核心酶, METTL14是METTL3的结构支持伴侣, 两者可以1:1形成稳定的复合物[13]。敲低METTL3及METTL14均可导致细胞内mRNA的总m6A水平下调[9]。两者的复合体可进一步与锌指CCCH域蛋白13 (zinc finger CCCH domain-containing protein 13, Zc3h13)、WTAP、Virilizer、Hakai及RNA结合基序蛋白15 (RNA binding motif protein 15, RBM15)等形成甲基转移酶复合体[14, 15]。WTAP最初被鉴定为METTL3同源蛋白的伴侣蛋白[16], 后续研究发现WTAP缺乏保守的催化甲基化域, 其本身不具有甲基化活性[9], 但WTAP可与METTL3/METTL14复合体结合并相互作用, 在细胞内与核斑点中的METTL3-METTL14异二聚体共定位, 共同参与m6A修饰调控[17]。敲低WTAP可显著降低细胞中m6A水平, 其可能与WTAP蛋白招募METTL3/METTL14复合体作用于目标mRNA的功能有关[9]。
m6A去甲基化酶主要包括FTO (也称ALKBH9)[4]和ALKBH5[10]。两者均为FeII/α-酮戊二酸依赖性双加氧酶AlkB亚家族成员, 具有FeII和α-酮戊二酸依赖性。FTO最早是通过全基因组关联研究(genome-wide association study, GWAS)分析鉴定到的与肥胖相关的基因[18]。2011年芝加哥大学何川教授课题组[4]发现FTO是RNA的m6A去甲基化酶, 并开启了m6A研究热潮的序幕。2017年, 康奈尔大学Samie Jaffrey教授课题组[19]报道FTO通过调节mRNA的m6Am水平, 影响mRNA的稳定性和翻译效率。m6A和m6Am两者结构非常类似, 差别仅仅在于核糖2'-O上的甲基修饰。2018年, 何川教授课题组[20]对FTO介导的RNA去甲基化进行了更全面的解析, 发现细胞核的FTO介导m6A的去甲基化, 细胞质中的FTO介导m6Am和m6A的去甲基化, 且FTO可以结合转运体RNA (transfer RNA, tRNA)发挥tRNA的N1-甲基腺嘌呤(N1-methyladenosine, m1A)去甲基化酶作用, 并进一步影响蛋白的翻译速度。2019年, Samie Jaffrey教授课题组[21]发现FTO可作用于核内小RNA (small nuclear RNA, snRNA)的m6Am修饰并影响mRNA的可变剪接。ALKBH5定位于细胞核, 沉默或过表达ALKBH5均可改变细胞中m6A的水平[10]。m6A的动态修饰状态依赖于甲基转移酶和去甲基化酶之间的功能相互作用。
m6A识别蛋白可通过读取m6A甲基化修饰信息从而调控mRNA的生物学行为及功能, 主要包括YTH家族成员及相关RNA结合蛋白。具有RNA结合结构域的YTH结构域家族是第一个被发现直接识别的“阅读器”[22], YTH (YT521-B同源性)家族蛋白YTHDF1~3和核成员YTHDC1~2可直接与含有m6A的RNA结合。近期发现胰岛素样生长因子2 mRNA结合蛋白(insulin-like growth factor 2 mRNA-binding proteins, IGF2BPs)可识别共有序列GG(m6A)C从而靶向mRNA转录本[23]。在哺乳动物细胞中发现与m6A关联的蛋白还有核RNA结合蛋白(HuR)和hnRNP等[5]。
1.2 m6A调控mRNA的生物学行为及可能机制近年来研究表明, m6A甲基化修饰几乎参与调控mRNA生物学行为的全过程, 包括mRNA剪切、从细胞核向细胞质输出、翻译起始及延伸、降解等[3]。此外, m6A在长链非编码RNA (long non-coding RNA, lncRNA)和微小RNA (microRNA, miRNA)的生成及功能等方面也发挥了重要调控作用[24]。
1.2.1 m6A影响mRNA剪切及成熟从前体mRNA (precursor mRNA, pre-mRNA)到成熟mRNA的过程包括3个主要步骤: 5'-加帽、3'-多聚腺苷酸化和剪接。Pre-mRNA剪接是基因表达重要过程, 涉及内含子的精确切除和外显子的连接, 可增加基因产物的多样性[25]。m6A最初被认为是一种剪接调节因子, 早期研究发现它在pre-mRNA中比在成熟mRNA中更丰富[26], 且许多m6A位点集中于内含子[27, 28]。FTO敲除可显著调节pre-mRNA剪切过程中的外显子跳跃事件并上调3'末端外显子表达[29]。甲基转移酶样因子16 (methyltransferase like 16, METTL16)最近被鉴定为RNA甲基化酶, 其可甲基化mRNA的3'UTR和U6的43A从而调控其与mRNA 5'剪切位点结合并影响其剪切[30]。YTHDC1可与剪切因子富含丝氨酸和精氨酸的剪接因子3 (serine and arginine rich splicing factor 3, SRSF3)结合并促进pre-mRNA向成熟mRNA的剪切[31]。hnRNPC及hnRNPG可识别mRNA m6A并负责pre-mRNA加工和mRNA成熟[32]。
1.2.2 m6A介导mRNA从细胞核向细胞质的输出mRNA核输出是连接细胞核内转录和加工与细胞质内表达的关键过程[33], 可选择性地调控基因表达。研究表明, mRNA的m6A可促进mRNA的核输出, METTL3的缺失可抑制mRNA核输出[34]。细胞内ALKBH5的敲除可加速目标RNA从细胞核向细胞质的转运[10]。YTHDC1可识别m6A甲基化mRNA并促进其与转运适配器SRSF3及核RNA输出因子1 (nuclear RNA export factor 1, NXF1)的结合从而促进核输出[35]。
1.2.3 m6A调节mRNA的翻译m6A可通过多种机制促进翻译, 主要包括: ①识别蛋白介导的mRNA翻译: YTHDF1通过结合m6A修饰的mRNA并招募翻译真核起始因子3 (eukaryotic initiation factor 3, eIF3)及真核起始因子4A3 (eukaryotic initiation factor 4A3, eIF4A3), 显著提高帽子(Cap)依赖翻译的效率, 促进m6A甲基化mRNA的翻译[8]。而YTHDF3可通过与YTHDF1及eIF4A3结合促进翻译起始[36]。识别蛋白IGF2BP1/2/3可通过提高mRNA的稳定性及翻译效率从而促进mRNA翻译[23]; ② METTL3可通过与核糖体结合促进翻译起始复合物组装从而促进癌基因如表皮生长因子受体(epidermal growth factor receptor, EGFR)、转录辅激活剂与PDZ结合基序(transcriptional coactivator wit PDZ-binding motif, TAZ)等的翻译并促进肿瘤恶性进展[37], 进一步研究表明, METTL3还可通过与eIF3h的结合促进mRNA翻译环(loop)的形成、核糖体循环利用(ribosome recycling)及翻译加速[38]; ③本课题组最近研究表明, 在肿瘤转移过程中关键转录因子Snail mRNA的编码序列(coding sequence, CDS)区而非3'UTR区的m6A修饰, 可促进其mRNA的翻译延伸, 其可能机制是通过YTHDF1与真核延伸因子2 (eukaryotic elongation factor 2, eEF-2)的共结合促进多聚核糖体对其翻译翻译延伸的作用[39]。
1.2.4 m6A调控mRNA的降解m6A与mRNA稳定性降低有关。目前研究表明, YTHDF2可结合m6A从而促进翻译后的mRNA通过P-body等形式进行降解[22]。敲低METTL3/METTL14可增加其靶基因mRNA的表达[9]。最近研究发现, IGF2BP蛋白介导的m6A在促进mRNA的稳定性方面具有独特的功能, 表现为IGF2BP可能与mRNA稳定蛋白如ELAV样蛋白1 (ELAVL1)、基质蛋白3 (matrin 3, MATR3)、胞质多聚(A)结合蛋白1 [cytoplasmic poly(A)-binding protein 1, PABPC1]的结合从而提高其靶标mRNA的稳定性[23]。
1.2.5 m6A对非编码RNA的生成及功能影响除mRNA外, 非编码RNA也受m6A调控。lncRNA和miRNA是两大类非编码RNA。m6A结合蛋白hnRNPA2B1可与初级miRNA (pri-miRNA)剪切复合物DGCR8蛋白相互作用促进miRNA成熟[40]。METTL3可增加pri-miRNAs的m6A修饰, 增强DGCR8对pri-miRNAs的识别及剪切, 从而促进miRNAs生成[41]。同时, hnRNPC以“m6A开关”调控的方式结合lncRNA MALAT1的2577-m6A发夹, 从而调控MALAT1的功能, 促进癌细胞增殖和侵袭的作用[40]。本课题组近期研究表明, m6A修饰的lncRNA RP11 (RP11-138J23.1)可通过与核内RNA结合蛋白hnRNPA2B1结合从而降低负调控E盒结合锌指蛋白1 (zinc finger E-box-binding protein 1, ZEB1)蛋白稳定性[42]。此外, m6A还可以改变RNA的折叠和结构及其与RNA结合蛋白的结合[32, 43]、影响tRNA对mRNA密码子的选择性和配对速率[44]及miRNA与靶标mRNA的配对结合从而发挥作用。
综上所述, m6A在甲基化酶的作用下对目标mRNA进行m6A修饰, 通过不同的识别蛋白及相应的分子机制来调控mRNA加工、核输出、翻译和降解等生物学行为, 最后依靠去甲基化的作用让m6A修饰这一过程变得动态可逆, 从而起到通过m6A修饰动态调控mRNA行为的作用, 也正是这种模式使得m6A甲基化修饰在生理过程中发挥了重要的作用。
2 m6A在肿瘤细胞生物学行为中的作用及机制随着研究的深入, 近期发现m6A修饰在人类疾病如脂肪代谢、神经发育及紊乱、免疫功能调节、病毒复制及感染等诸多疾病密切相关[3]。肿瘤细胞具有独特的恶性生物学特征, 表现为自给自足生长信号、抗生长信号的不敏感、抵抗细胞死亡、潜力无限的复制能力、持续的血管生成、组织浸润和转移、免疫逃逸、促进肿瘤的炎症、细胞能量异常、基因组不稳定和突变等生物学现象[45]。本文将围绕m6A在肿瘤恶性生物学行为中的作用及可能机制展开探讨。
2.1 m6A与肿瘤上皮间质转化(epithelial-mesenchymal transition, EMT)及侵袭转移EMT是肿瘤细胞侵袭转移的最重要步骤之一, 指的是在特定生理病理情况下, 具有极性的上皮细胞向间质化细胞转化的现象[46]。其主要特征是上皮细胞表型分子如E-钙黏素(E-Cad)的丧失和间质细胞特性如波形蛋白(Vim)的获得, 从而使肿瘤细胞摆脱细胞间黏附, 表现得更为疏松且细胞更具侵袭与转移能力[46]。有研究表明抑制METTL3表达可降低E-Cad表达并促进间质标志物Vim等的表达从而促进细胞EMT[47]。Snai1被认为是EMT过程中最重要的调控因子之一, 其与E-Cad基因启动子上E-box (5'-CAGGTG-3')作用元件特异性结合并抑制其转录, 从而启动EMT的过程[48]。
本课题组前期研究发现, 在肿瘤细胞EMT过程中, mRNAs的m6A修饰增加。甲基转移酶METTL3的缺失使得m6A水平下调, 并抑制癌细胞在体外和体内的迁移、侵袭和EMT过程。m6A-seq和功能实验表明, EMT的关键转录因子Snail的表达受m6A调控, 且过表达Snail能逆转METTL3缺失导致的细胞EMT抑制。进一步研究表明, Snail mRNA的CDS区而非3'UTR区的m6A修饰, 可促进其mRNA的翻译延伸, 其可能机制是通过YTHDF1与eEF-2的共结合促进多聚核糖体对其的翻译作用[39]。同时, 还发现m6A甲基化可导致lncRNA RP11表达上调从而促进细胞的迁移、侵袭和EMT发生[42]。
以上结果提示, m6A对肿瘤细胞EMT及侵袭转移有重要的作用, 其相关机制亟待进一步研究。
2.2 m6A与肿瘤耐药在肿瘤的治疗过程中, 化学药物及放射治疗有着不可替代的作用。耐药已经成为恶性肿瘤治疗失败的主要因素, 研究肿瘤耐药的机制及逆转方法对恶性肿瘤的治疗有着重要意义。近期研究表明, m6A在肿瘤耐药中发挥着重要作用, 有望成为肿瘤耐药治疗的新靶点。m6A通过稳定FZD10上调Wnt/β-catenin通路, 促进BRAC缺乏而使卵巢上皮癌(epithelial ovarian cancers, EOC)细胞的PARP抑制剂耐药[49]。m6A甲基化酶METTL3促进胰腺癌细胞的化疗和放疗耐药, 敲低METTL3可显著增加胰腺癌细胞对抗癌药物5-氟尿嘧啶(5-fluorouracil, 5-FU)、顺铂(cis-platinum, CDDP)及放疗的敏感性[50]。FTO在宫颈鳞状细胞癌(cervical squamous cell carcinoma, CSCC)组织中高表达, 其可通过降低β-catenin的m6A从而降低其表达并提高CSCC细胞的化疗敏感性[51]。敲除YTHDF1可抑制结直肠癌细胞的增殖并增强其对化疗药物如5-FU和顺铂的敏感性, 同时MYC (myelocytomatosis oncogene)的高表达可促进YTHDF1的表达从而促进CRC恶性进展[52]。
肿瘤干细胞(cancer stem cells, CSCs)通常是调节治疗耐药性的关键因素, 而研究发现m6A修饰对胶质母细胞瘤干细胞(GCSs)的自我更新和肿瘤发生具有重要作用[53]。在神经胶质瘤干细胞样细胞(glioma stem-like cells, GSCs)中, METTL3高表达并上调SOX2 (sex determining region Y box 2, SOX2) mRNA 3'UTR的m6A甲基化从而上调其稳定性和表达, 从而降低其放疗敏感性并提高DNA修复效率[52]。
2.3 m6A与细胞能量代谢近年来, 肿瘤代谢越来越受到研究者的重视。作为肿瘤细胞的标志, Warburg效应指的是肿瘤细胞即使在氧供应充分的条件下也主要以糖酵解获取能量[54]。FTO最早是通过GWAS分析鉴定到的与肥胖相关的基因[18]。在小鼠研究模型中, FTO的缺失可导致脂肪组织和体重的显著降低, 其机制是通过增强能量消耗及交感神经活化[55]。此外, FTO介导的m6A去甲基化控制着脂肪调节因子RUNX1T1的剪接[56]。在乳腺癌细胞MDA-MB-231中, 靶向抑制FTO可降低乳酸和ATP的生成, 抑制丙酮酸激酶与己糖激酶的活性, 并导致AKT失活, 过表达FTO则可促进肿瘤细胞糖酵解能力及PI3K/AKT通路的活化[57]。然而, 目前关于m6A其他相关酶对肿瘤细胞代谢的影响尚知之甚少。
2.4 m6A与血管生成近期研究提示, m6A调控mRNA在血管生成中发挥重要作用。FTO在衰竭的哺乳动物心脏和缺氧心肌细胞中表达减少, 增加衰竭小鼠心脏中FTO的表达可减轻缺血引起的m6A升高和心肌收缩功能下降, 在心肌梗死小鼠模型中, FTO过表达可降低心肌细胞纤维化并促进血管生成[58]。通过数据库分析发现FTO的表达可调控EGFR相关通路并调控血管生成信号, 过表达FTO可抑制体内肿瘤生长[59]。在肿瘤细胞中, METTL14/ALKBH5可通过调控血管生成相关基因如TGF-β表达从而发挥促癌作用[60]。
3 m6A修饰调控肿瘤体内发生发展越来越多的研究发现, 恶性肿瘤发生发展与m6A修饰存在诸多潜在的联系。m6A作为mRNA重要表观标记, 可实现对转录本的标记及分组, 从而快速跟踪并调控mRNA的生物学功能和代谢, 实现对细胞功能和分化的调控。如在肿瘤EMT过程中mRNAs的m6A显著上调, 且可调控侵袭转移及细胞黏连相关基因[39]。此外, m6A还可通过影响miRNA[40, 41]及lncRNA[42, 61]的生成、生物学功能及稳定性从而调控肿瘤进展。接下来, 本文将根据不同的肿瘤类型来综述m6A与肿瘤之间的关系(表 1)[37, 39, 53, 62-76]
| Table 1 The biological functions of m6A on cancer progression and its related mechanisms. METTL3: Methyltransferase like 3; METTL14: Methyltransferase-like 14; FTO: Fat-mass and obesity-associated protein; YTHDF2: YTH N6-methyladenosine RNA binding protein 2; ALKBH5: Alk B homolog 5; MYC: Myelocytomatosis oncogene; BCL2: B cell leukaemia 2; RARA: Retinoic acid receptor alpha; m6A: N6-Methyladenosine; EGFR: Epidermal growth factor receptor; TAZ: Transcriptional coactivator wit PDZ-binding motif; USP7: Ubiquitin specific protease 7; MZF1: Myeloid zinc finger 1; SUMO1: Small ubiquitin-like modifier 1; CDS: Coding sequence; KLF4: Kruppel like factor 4; NANOG: Nanog homeobox; EMT: Epithelial-mesenchymal transition; DGCR8: DiGeorge syndrome critical region 8; NSCLC: Nonsmall-cell lung carcinoma; ZNF217: Zinc finger protein 217; ADAM19: A disintegrin and metallopeptidase domain 19; EPHA3: EPH receptor A3; BNIP3: BCL2 interacting protein 3; FOXM1: Forkhead box M1; MMP2: Matrix metalloproteinase 2 |
与正常造血祖细胞相比, METTL3和METTL14在急性髓系白血病(acute myelocytic leukemia, AML)细胞中呈高表达[62, 63]。METTL3敲除可上调磷酸化AKT水平并诱导细胞分化及凋亡, 并抑制AML的体内进展, 单碱基精度的m6A测序发现m6A可调控c-myc、BCL2及磷酸酶和紧张素同源物(phosphatase and tensin homolog, PTEN)等癌基因的表达[62]。同时METTL3可结合于基因启动子区域并通过mRNA m6A从而促使其与核糖体结合并翻译, 促进AML恶化[64]。METTL14也可通过调控MYB (myeloblastosis oncogene)及MYC的m6A从而促进骨髓细胞的生成及白血病发生[63]。
FTO在t(11q23)/MLL重排及t(15;17)/PML-RARA、FLT3-ITD、NPM1突变的AML细胞中高表达, 可增强癌基因诱导的细胞分化及白血病生成, 并通过降低ASB2及RARA (retinoic acid receptor alpha, RARA)等基因的m6A抑制其表达从而抑制ATRA诱导AML细胞分化[64]。此外, 异柠檬酸脱氢酶1/2 (IDH1/2)突变体代谢产物R-2-羟基戊二酸盐(R-2HG)可抑制FTO活性, 从而增加R-2HG敏感性白血病细胞中m6A修饰, 进而降低MYC/CEBPA转录本的稳定性, 导致相关通路的抑制。R-2HG通过抑制白血病细胞增殖/存活、促进细胞周期阻滞和凋亡, 在体内外均具有广泛的抗白血病活性[77]。
YTHDF2在多种AML细胞中高表达并介导AML的发生, 其可识别转录本m6A并降低其半衰期从而增强白血病干细胞(leukemic stem cells, LSCs)的功能, 调控的靶基因主要包括受体Tnfrsf2等。同时, YTHDF2的表达对正常造血干细胞(hematopoietic stem cells, HSC)的功能维系并非必需条件, 相反其缺失甚至有利于增强HSC的活性[65]。
这些发现提示, m6A可通过调控AML关键基因的mRNA生物学功能从而调控其发生发展, 通过靶向m6A动态调控相关蛋白, 有望开发出针对AML的治疗方法及药物用于临床治疗。
3.2 m6A与肺癌肺癌是全球最常见的肿瘤, 也是导致肿瘤死亡的主要原因。接受标准治疗的患者在大多数国家的5年生存率低于15%[78, 79]。因此, 探索新的肺癌治疗靶点具有重要意义。肿瘤基因组图谱(TCGA)数据表明, METTL3 mRNA在人肺腺癌(LUAD)中的表达显著升高, 其可增加EGFR和TAZ的表达, 促进细胞生长、存活和侵袭[37]。肺癌组织中FTO过表达, 可降低USP7 m6A水平, 提高其mRNA的稳定性, 并通过增加其表达促进非小细胞肺癌(non-small cell lung carcinoma, NSCLC)细胞的生长[66]。此外, FTO还可通过降低MZF1的m6A水平从而提高其mRNA稳定性并促进肺癌增殖与生长[67]。在NSCLC中, METTL3的赖氨酸残基K177/211/212/215 SUMO化可抑制其m6A甲基化酶活性, 从而降低细胞mRNA m6A水平, 并抑制肺癌细胞的增殖及克隆形成能力[68]。此外, miR-33a通过靶向METTL3 mRNA的3'UTR, 可以减弱NSCLC细胞的增殖[69]。这些研究为了解METTL3和FTO在肺癌中的重要作用提供了依据。
3.3 m6A与肝癌研究表明, mRNA m6A及其相关蛋白在肝癌的恶性进展中发挥了重要作用。METTL3与肝癌患者预后不良有关, 表现为METTL3在肝癌组织中较癌旁组织表达显著升高, 其不仅可以通过YTHDF2沉默SOCS2的mRNA从而抑制其表达[70]。临床分析表明, 肝癌组织中METTL3和YTHDF1表达高于癌旁组织, 其上调是肝癌患者总体生存率(overall survival, OS)不良预后因素[39]。同时有研究表明, 肝癌细胞中m6A甲基化修饰水平显著降低, 下调METT14的表达可促进肿瘤的发生以及肝细胞癌的转移, 其分子机制在于通过与DGCR8的相互作用从而促使前期miR-126向成熟体的生成, 靶向抑制miR-126则可逆转METTL14抑制肝癌进展的作用[71]。综上所述, METTL3上调或METTL14下调可预测肝癌患者预后不良, 并可导致HCC进展和转移[70, 71]。此外, 肝癌细胞中miR-145通过靶向YTHDF2 mRNA的3'UTR下调其表达并上调细胞中m6A甲基化修饰水平[72]。这些研究为肝癌进展过程中的RNA表观遗传改变提供了新的研究成果及发展方向。
3.4 m6A与其他肿瘤研究还发现m6A参与乳腺癌的恶性进展。低氧刺激促进依赖低氧因子(HIF)的ALKBH5高表达, 从而导致NANOG基因mRNA上m6A的去甲基化并增强其mRNA的稳定性, 进而促进乳腺肿瘤干细胞的维持和转移[73]。同样, 缺氧可诱导乳腺癌干细胞形成, 在此过程中伴随着ALKBH5及锌指蛋白217 (zinc finger protein 217, ZNF217)的表达上升, 而抑制ALKBH5可抑制乳腺癌细胞从乳腺向肺的体内转移[80]。FTO可通过对BNIP3的3'UTR去甲基化从而导致其降解并促进乳腺癌的生长及转移[74]。
m6A RNA甲基化下调显著促进恶性胶质瘤干细胞(GSCs)的生长、自我更新和肿瘤发生。GSCs对放化疗具有耐药性, 研究发现分化的GSC细胞株m6A水平升高而原代GSC细胞株m6A水平较低, METTL3或METTL14的下调可诱导原癌基因分解素和金属肽酶结构域19 (a disintegrin and metallopeptidase domain 19, ADAM19)、EPHA3和KLF4 mRNA的表达, 促进GSCs的生长和自我更新[53]。ALKBH5可去除去甲基叉头核蛋白M1 (forkhead box M1, FOXM1)的新生成mRNA的m6A从而提高其稳定性并促进CSC细胞增殖[75]。METTL3在黑色素瘤中上调, 并通过基质金属蛋白酶2 (matrix metalloproteinase 2, MMP2)在侵袭/迁移中发挥作用, 表明METTL3抑制剂在治疗黑色素瘤中可能具有良好前景[76]。子宫内膜癌中, m6A修饰可调控AKT通路活性从而对肿瘤细胞的增殖及肿瘤进展进行调节[81]。本课题组前期研究还发现, m6A可导致lncRNA RP11在CRC细胞核中的积累并促进CRC细胞的迁移、侵袭和EMT发生[42]。
目前关于m6A调控肿瘤进展还存在不一致的研究结论。以AML为例, 甲基化酶METTL3可结合于基因启动子区域并通过mRNA m6A从而促使其与核糖体结合并翻译, 促进AML恶化[64]。去甲基化酶FTO在AML中表达水平升高, 并可降低抑癌基因ASB2和RARA促进AML细胞增殖[82]。其可能原因在于METTL3及FTO可通过作用于不同的靶标mRNA并调控其生物学功能, 从而在肿瘤进展中共同发挥促进作用。
以上研究表明, m6A可调控包括白血病、乳腺癌、肝癌、肺癌、结直肠癌、黑色素瘤等多种肿瘤的发生及发展, 靶向抑制m6A相关蛋白对相关肿瘤的治疗具有良好的应用前景和临床价值。同时, m6A在肿瘤恶性生物学行为中的作用可能与肿瘤细胞种属、分化及生理状态等多种因素有关, 相关机制亟待进一步阐明。
4 基于m6A的相关靶向治疗策略针对靶点的特异性小分子抑制剂开发具有巨大的科学意义及应用价值。一方面, 小分子抑制剂可大大推动其靶点的生物学功能及分子机制研究。以溴结构域蛋白4 (BRD4)为例, 其特异性抑制剂JQ1的开发[83]使得对其功能及机制研究出现井喷式增长; 另一方面, 小分子抑制剂的研发为临床药物提供了候选化合物。根据药渡数据, 目前有13个以BRD4为靶点的药物已经进入临床试验研究阶段。
基于表观遗传分子机制开发的药物目前广泛应用于肿瘤治疗, 主要代表性药物有DNA甲基化酶抑制剂阿扎胞苷(azacitidine)、组蛋白去乙酰化酶抑制剂伏立诺他(vorinostat)及其他表观遗传相关蛋白特异性的抑制剂, 在临床上发挥显著作用[84]。鉴于表观遗传均基于对转录及转录后产物进行调控的分子机制, 且前期研究结果表明m6A在肿瘤的发生、侵袭转移、分化、能量代谢及血管生成等恶性生物学行为中均发挥重要作用, 因此m6A相关蛋白的特异性抑制剂开发具有巨大的科学意义和临床价值, 可在肿瘤治疗中发挥重要作用(表 2)[85-91]。
| Table 2 The inhibitors of m6A related enzymes. *IC50 represents the value of in vitro reaction. N-CDPCB: N-(5-Chloro-2, 4-dihydroxyphenyl)-1-phenylcyclobutanecarboxamide; CHTB: 4-Chloro-6-(6'-chloro-7'-hydroxy-2', 4', 4'-trimethyl-chroman-2'-yl)benzene-1, 3-diol; IOX3: [(1-Chloro-4-hydroxy-isoquinoline-3-carbonyl)-amino]-acetic acid |
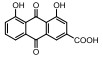
目前基于m6A相关酶抑制剂的开发主要集中于首个发现的RNA去甲基化酶FTO。FTO是一类保守的2OG加氧酶, 通用抑制剂NOG可抑制FTO的活性。中国科学院上海药物所杨财广课题组[85]通过虚拟筛选发现大黄酸是FTO竞争性抑制剂, 在大黄酸存在的情况下FTO的去甲基化酶活性并未因Fe2+浓度加倍而恢复, 表明大黄酸并没有去螯合金属离子, 其机制在于通过与单链RNA (single stranded RNA, ssRNA)底物竞争性结合催化域抑制FTO, 可体内外抑制FTO对mRNA上m6A的去甲基化活性, 从而提高细胞中m6A的水平。随后, 其发现荧光素衍生物可同时抑制及标记FTO蛋白[92], 进一步研究发现甲氯芬那酸(meclofenamic acid, MA)是一种选择性较强的FTO抑制剂, 能与FTO结合并稳定FTO, 而不影响ALKBH5的去甲基化酶活, 可显著提高细胞m6A的水平, 但对于FTO缺失的细胞m6A水平无显著影响[86]。这项研究还发现FTO/MA复合物的晶体结构清晰地展现了FTO中β3i和β4i可形成特异性识别MA的疏水口袋, 而ALKBH5缺乏相应的结构, 该研究为开发特异性的FTO抑制剂提供了化学基础。
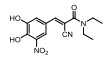
此外, Aik等[93]发现了一系列化合物可通过金属离子螯合基团与2OG竞争抑制FTO的活性, 但此类抑制剂开发药物时需避免抑制其他2OG加氧酶, 提高特异性。Zheng等[94]发现的化合物能有效抑制其2OG加氧酶家族的FTO, 而且IC50值和广谱性2OG酶抑制剂NOG接近。郑州大学常俊标课题组[87, 88]发现根赤壳菌素(radicicol)、N-CDPCB及CHTB等化合物可作为FTO抑制剂。
2019年, 杨财广课题组[95]根据FTO识别m6A修饰底物的分子机制等特点, 应用基于晶体结构的化合物设计、合成优化等手段, 获得FTO小分子抑制剂。该化合物选择性抑制AML细胞中FTO的去甲基化功能, 上调AML关键基因mRNA上m6A修饰, 增加抑癌蛋白质例如ASB2和RARA的丰度, 降低促癌蛋白质如MYC和CEBPA的丰度, 从而抑制AML细胞增殖, 并且在PDX小鼠模型上展现抗白血病的治疗效果, 本研究指明了分子靶向性干预m6A修饰从而影响基因表达来实现抗肿瘤的研究新方向。
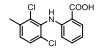
最近, 黄牛课题组[89]基于结构的虚拟筛选以及一系列体内外生物活性实验, 发现之前被FDA批准上市的恩他卡朋(entacapone)可以作为一个FTO的特异性抑制剂, FTO抑制后m6A修饰水平在FOXO1信使RNA上升高, 引起了FOXO1蛋白表达下调, 从而调控糖异生关键基因G6PC的表达, 小鼠在服用恩他卡朋后表现出体重显著减少、血糖水平明显降低以及脂肪组织产热明显增强等特征。本研究进一步揭示了FTO在能量代谢方面调控的分子机制, 并且FTO的抑制剂恩他卡朋的发现对肥胖患者的临床用药具有一定指导意义。
作为最早是通过GWAS分析鉴定到的与肥胖相关的基因[18], FTO不仅与肥胖及肿瘤密切相关, 其常见变异rs9939609可能与中枢神经系统疾病如脑容量下降、酒精依赖等有关[96]。因此FTO抑制剂的潜在用途除抗肿瘤及减肥外, 还可能被研发为神经类疾病药物。
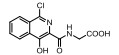
4.2 m6A相关其他蛋白抑制剂及化学干预ALKBH5和FTO均属于Fe(Ⅱ)/2-氧戊二酸盐(2OG)双加氧酶超家族的AlkB亚家族。2OG双加氧酶超家族成员作用于多种底物, 参与调控蛋白质的生物合成。IOX3也可以与ALKBH5共价结合[90]。在ALKBH5结晶条件下, 柠檬酸盐在烷烃的活性部位与2OGs和Mn(ii)竞争, 可以作为ALKHB5的抑制剂发展和改造的方向[97]。在甲基化酶中, METTL3是主要的活性催化位点, 而METTL14在底物识别过程起到关键作用。由于对甲基化酶识别和催化RNA甲基化机制认知有限, 其抑制剂开发相对进展较慢。到目前为止, 仅发现脱氮腺苷(3-deazaadenosine, DAA)对METTL3有抑制作用[34], 但是DAA的作用属于广谱作用, 抑制所有RNA甲基化酶的活性, 没有针对m6A甲基化酶的特异性。
此外, 利用化学方法对m6A进行去甲基化反应也是另外一种重要的化学干预策略。周翔课题组[98]在H2O2条件下利用化学氧化的方法得到可对mRNA的m6A进行39%的特异性去除。程靓课题组[99]利用外源性光敏分子核黄素(riboflavin)可在LED蓝光的催化下可对mRNA的m6A进行选择性高效氧化, 从而降低mRNA的m6A修饰。在此基础上, 该课题组进一步开发出可在活细胞上进行mRNA的m6A修饰去除的化合物黄素单核苷酸(flavin mononucleotide, FMN), 其可在LED蓝光的催化下在细胞内进行化学去m6A修饰, 从而实现利用化合物在活体细胞内进行mRNA m6A特异性去甲基化作用[91]。
5 结论和展望近年来, 随着研究的深入, m6A甲基化修饰相关酶体系、m6A在mRNA修饰中的作用和生物学意义以及m6A在恶性肿瘤中的调控机制均得到了揭示。m6A被甲基化酶、去甲基化酶及识别蛋白精确调控, 几乎涉及mRNA生物学从生成到降解的每个步骤, 并参与调控miRNA、lncRNA的生成及生物学功能。m6A在多种恶性肿瘤的代谢、耐药和转移等方面发挥重要作用, 表明m6A修饰可以作为预防和治疗人类肿瘤的靶点。
研究发现的m6A相关因子(如FTO、ALKBH5和HIF)抑制剂, 为实现肿瘤基因靶向治疗提供了帮助。到目前为止, 已经报道的多种FTO抑制剂(大黄酸、甲氯芬那酸和IOX3等), 大部分不具有特异性。甲氯芬那酸(MA)作为选择性FTO抑制剂之一, 是一种与FTO结合位点竞争的非甾体类抗炎药物[86]。FTO抑制剂MA2 (MA的乙酯衍生物)延长了GSC移植小鼠的寿命, 这意味着m6A修饰可能是抑制肿瘤进展和逆转胶质母细胞瘤放疗和化疗耐药性的靶点[53]。此外, 贝克曼研究所陈建军课题组[77]发现, 异柠檬酸脱氢酶(IDH)突变造成的代谢产物R-2HG通过抑制FTO/m6A/MYC/CEBPA信号发挥抗白血病的作用, 为临床治疗白血病的用药提供了新的靶点。虽然已经发现了许多针对m6A去甲基酶的抑制剂, 但它们的作用在体内证据有限, m6A相关蛋白抑制剂的开发及临床适应症选择仍有广泛探索空间。
同时, m6A调控肿瘤进展及其靶向治疗也存在着诸多尚待解决的问题:首先, RNA修饰具有时空动态性和组织特异性, 其调控相关机制亟待单碱基精度的高分辨测序与测量技术, 目前主流的抗体捕获技术尚不能满足m6A的单碱基精度定位; 其次, 目前尚未有技术可针对特异性基因实现其转录本的m6A单基因编辑技术, 本课题组在前期工作中结合PspCas13b/gRNA系统, 构建出PspCas13b-Alkbh5融合蛋白, 并针对目的基因设计gRNA, 实验验证其可特异性去除目的基因mRNA的甲基化修饰, 从而提供一种基于PspCas13b-Alkbh5单基因特异性m6A修饰编辑方法[100], 后续工作中可进一步优化提高其编辑效率及特异性; 此外, 虽然发现了一些去甲基化酶抑制剂, 并且为肿瘤用药提供了新的靶点, 但在体内外作用及具体机制尚不完全清楚, 并且缺乏特异性。因此, 研究者们期望获得更多针对m6A相关蛋白的抑制剂, 尤其是特异性更强的抑制剂, 为指导基于RNA表观遗传的肿瘤靶向治疗带来新的曙光。
靶向DNA甲基化酶或者组蛋白修饰酶的数个新药获批用于治疗肿瘤并获得良好的治疗效果和巨大的经济效益。基于表观遗传的化学干预研究已经成为国际上药物新靶标研究的活跃领域。由于起步时间较晚, 目前以m6A修饰为核心的RNA表观遗传研究尚处于早期阶段。由于靶向RNA表观转录组包括其编辑、降解、转运及翻译在安全性及时效性等方面存在良好的优势, 因此发现高质量化学探针及特异性抑制剂, 开发单基因特异性编辑技术, 靶向性干预m6A修饰, 不仅可推动相关领域的基础研究, 也可在肿瘤治疗等疾病相关领域展现出巨大的应用前景, 在生命科学和新药发现领域同时展现重要科学意义。
| [1] | Desrosiers R, Friderici K, Rottman F, et al. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells[J]. Proc Natl Acad Sci U S A, 1974, 71: 3971–3975. DOI:10.1073/pnas.71.10.3971 |
| [2] | Wei CM, Gershowitz A, Moss B, et al. Methylated nucleotides block 5' terminus of HeLa cell messenger RNA[J]. Cell, 1975, 4: 379–386. DOI:10.1016/0092-8674(75)90158-0 |
| [3] | Roundtree IA, Evans ME, Pan T, et al. Dynamic RNA modifications in gene expression regulation[J]. Cell, 2017, 169: 1187–1200. DOI:10.1016/j.cell.2017.05.045 |
| [4] | Jia G, Fu Y, Zhao X, et al. N6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO[J]. Nat Chem Biol, 2011, 7: 885–887. DOI:10.1038/nchembio.687 |
| [5] | Dominissini D, Moshitch-Moshkovitz S, Schwartz S, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq[J]. Nature, 2012, 485: 201–206. DOI:10.1038/nature11112 |
| [6] | Meyer KD, Saletore Y, Zumbo P, et al. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons[J]. Cell, 2012, 149: 1635–1646. |
| [7] | Sullenger BA, Nair S. From the RNA world to the clinic[J]. Science, 2016, 352: 1417–1420. DOI:10.1126/science.aad8709 |
| [8] | Wang X, Zhao BS, Roundtree IA, et al. N6-Methyladenosine modulates messenger RNA translation efficiency[J]. Cell, 2015, 161: 1388–1399. DOI:10.1016/j.cell.2015.05.014 |
| [9] | Liu JZ, Yue YN, Han DL, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N-6-adenosine methylation[J]. Nat Chem Biol, 2014, 10: 93–95. DOI:10.1038/nchembio.1432 |
| [10] | Zheng G, Dahl JA, Niu Y, et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility[J]. Mol Cell, 2013, 49: 18–29. DOI:10.1016/j.molcel.2012.10.015 |
| [11] | Narayan P, Rottman FM. An in vitro system for accurate methylation of internal adenosine residues in messenger RNA[J]. Science, 1988, 242: 1159–1162. DOI:10.1126/science.3187541 |
| [12] | Bokar JA, Rath-Shambaugh ME, Ludwiczak R, et al. Characterization and partial purification of mRNA N6-adenosine methyltransferase from HeLa cell nuclei. Internal mRNA methylation requires a multisubunit complex[J]. J Biol Chem, 1994, 269: 17697–17704. |
| [13] | Wang P, Doxtader KA, Nam Y. Structural basis for cooperative function of METTL3 and METTL14 methyltransferases[J]. Mol Cell, 2016, 63: 306–317. DOI:10.1016/j.molcel.2016.05.041 |
| [14] | Wen J, Lv R, Ma H, et al. Zc3h13 regulates nuclear RNA m6A methylation and mouse embryonic stem cell self-renewal[J]. Mol Cell, 2018, 69: 1028–1038.e6. DOI:10.1016/j.molcel.2018.02.015 |
| [15] | Knuckles P, Lence T, Haussmann IU, et al. Zc3h13/Flacc is required for adenosine methylation by bridging the mRNA-binding factor Rbm15/Spenito to the m6A machinery component Wtap/Fl(2)d[J]. Genes Dev, 2018, 32: 415–429. DOI:10.1101/gad.309146.117 |
| [16] | Zhong S, Li H, Bodi Z, et al. MTA is an Arabidopsis messenger RNA adenosine methylase and interacts with a homolog of a sex-specific splicing factor[J]. Plant Cell, 2008, 20: 1278–1288. DOI:10.1105/tpc.108.058883 |
| [17] | Ping XL, Sun BF, Wang L, et al. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase[J]. Cell Res, 2014, 24: 177–189. DOI:10.1038/cr.2014.3 |
| [18] | Frayling TM, Timpson NJ, Weedon MN, et al. A common variant in the FTO gene is associated with body mass index and predisposes to childhood and adult obesity[J]. Science, 2007, 316: 889–894. DOI:10.1126/science.1141634 |
| [19] | Mauer J, Luo X, Blanjoie A, et al. Reversible methylation of m6Am in the 5' cap controls mRNA stability[J]. Nature, 2017, 541: 371–375. DOI:10.1038/nature21022 |
| [20] | Wei J, Liu F, Lu Z, et al. Differential m6A, m6Am, and m1A demethylation mediated by FTO in the cell nucleus and cytoplasm[J]. Mol Cell, 2018, 71: 973–985.e5. DOI:10.1016/j.molcel.2018.08.011 |
| [21] | Mauer J, Sindelar M, Despic V, et al. FTO controls reversible m6Am RNA methylation during snRNA biogenesis[J]. Nat Chem Biol, 2019, 15: 340–347. DOI:10.1038/s41589-019-0231-8 |
| [22] | Wang X, Lu Z, Gomez A, et al. N6-Methyladenosine-dependent regulation of messenger RNA stability[J]. Nature, 2014, 505: 117–120. DOI:10.1038/nature12730 |
| [23] | Huang H, Weng H, Sun W, et al. Recognition of RNA N6-methyladenosine by IGF2BP proteins enhances mRNA stability and translation[J]. Nat Cell Biol, 2018, 20: 285–295. DOI:10.1038/s41556-018-0045-z |
| [24] | Coker H, Wei G, Brockdorff N. m6A modification of non-coding RNA and the control of mammalian gene expression[J]. Biochim Biophys Acta Gene Regul Mech, 2019, 1862: 310–318. DOI:10.1016/j.bbagrm.2018.12.002 |
| [25] | Baralle FE, Giudice J. Alternative splicing as a regulator of development and tissue identity[J]. Nat Rev Mol Cell Biol, 2017, 18: 437–451. DOI:10.1038/nrm.2017.27 |
| [26] | Salditt-Georgieff M, Jelinek W, Darnell JE, et al. Methyl labeling of HeLa cell hnRNA: a comparison with mRNA[J]. Cell, 1976, 7: 227–237. DOI:10.1016/0092-8674(76)90022-2 |
| [27] | Carroll SM, Narayan P, Rottman FM. N6-Methyladenosine residues in an intron-specific region of prolactin pre-mRNA[J]. Mol Cell Biol, 1990, 10: 4456–4465. DOI:10.1128/MCB.10.9.4456 |
| [28] | Stoltzfus CM, Dane RW. Accumulation of spliced avian retrovirus mRNA is inhibited in S-adenosylmethionine-depleted chicken embryo fibroblasts[J]. J Virol, 1982, 42: 918–931. |
| [29] | Bartosovic M, Molares HC, Gregorova P, et al. N6-Methyladenosine demethylase FTO targets pre-mRNAs and regulates alternative splicing and 3'-end processing[J]. Nucleic Acids Res, 2017, 45: 11356–11370. DOI:10.1093/nar/gkx778 |
| [30] | Shima H, Matsumoto M, Ishigami Y, et al. S-Adenosylmethionine synthesis is regulated by selective N6-adenosine methylation and mRNA degradation involving METTL16 and YTHDC1[J]. Cell Rep, 2017, 21: 3354–3363. DOI:10.1016/j.celrep.2017.11.092 |
| [31] | Xiao W, Adhikari S, Dahal U, et al. Nuclear m6A reader YTHDC1 regulates mRNA splicing[J]. Mol Cell, 2016, 61: 507–519. DOI:10.1016/j.molcel.2016.01.012 |
| [32] | Liu N, Dai Q, Zheng G, et al. N6-Methyladenosine-dependent RNA structural switches regulate RNA-protein interactions[J]. Nature, 2015, 518: 560–564. DOI:10.1038/nature14234 |
| [33] | Wickramasinghe VO, Laskey RA. Control of mammalian gene expression by selective mRNA export[J]. Nat Rev Mol Cell Biol, 2015, 16: 431–442. DOI:10.1038/nrm4010 |
| [34] | Fustin JM, Doi M, Yamaguchi Y, et al. RNA-methylation-dependent RNA processing controls the speed of the circadian clock[J]. Cell, 2013, 155: 793–806. DOI:10.1016/j.cell.2013.10.026 |
| [35] | Roundtree IA, Luo GZ, Zhang Z, et al. YTHDC1 mediates nuclear export of N6-methyladenosine methylated mRNAs[J]. Elife, 2017, 6: e31311. DOI:10.7554/eLife.31311 |
| [36] | Shi H, Wang X, Lu Z, et al. YTHDF3 facilitates translation and decay of N6-methyladenosine-modified RNA[J]. Cell Res, 2017, 27: 315–328. DOI:10.1038/cr.2017.15 |
| [37] | Lin SB, Choe J, Du P, et al. The m6A methyltransferase METTL3 promotes translation in human cancer cells[J]. Mol Cell, 2016, 62: 335–345. DOI:10.1016/j.molcel.2016.03.021 |
| [38] | Choe J, Lin S, Zhang W, et al. mRNA circularization by METTL3-eIF3h enhances translation and promotes oncogenesis[J]. Nature, 2018, 561: 556–560. DOI:10.1038/s41586-018-0538-8 |
| [39] | Lin X, Chai G, Wu Y, et al. RNA m6A methylation regulates the epithelial mesenchymal transition of cancer cells and translation of Snail[J]. Nat Commun, 2019, 10: 2065. DOI:10.1038/s41467-019-09865-9 |
| [40] | Alarcon CR, Goodarzi H, Lee H, et al. HNRNPA2B1 is a mediator of m6A-dependent nuclear RNA processing events[J]. Cell, 2015, 162: 1299–1308. DOI:10.1016/j.cell.2015.08.011 |
| [41] | Alarcon CR, Lee H, Goodarzi H, et al. N6-Methyladenosine marks primary microRNAs for processing[J]. Nature, 2015, 519: 482–485. DOI:10.1038/nature14281 |
| [42] | Wu Y, Yang X, Chen Z, et al. m6A-induced lncRNA RP11 triggers the dissemination of colorectal cancer cells via upregulation of Zeb1[J]. Mol Cancer, 2019, 18: 87. DOI:10.1186/s12943-019-1014-2 |
| [43] | Liu N, Zhou KI, Parisien M, et al. N6-Methyladenosine alters RNA structure to regulate binding of a low-complexity protein[J]. Nucleic Acids Res, 2017, 45: 6051–6063. DOI:10.1093/nar/gkx141 |
| [44] | Choi J, Ieong KW, Demirci H, et al. N6-Methyladenosine in mRNA disrupts tRNA selection and translation-elongation dynamics[J]. Nat Struct Mol Biol, 2016, 23: 110–115. DOI:10.1038/nsmb.3148 |
| [45] | Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation[J]. Cell, 2011, 144: 646–674. DOI:10.1016/j.cell.2011.02.013 |
| [46] | Brabletz T, Kalluri R, Nieto MA, et al. EMT in cancer[J]. Nat Rev Cancer, 2018, 18: 128–134. DOI:10.1038/nrc.2017.118 |
| [47] | Li X, Tang J, Huang W, et al. The m6A methyltransferase METTL3: acting as a tumor suppressor in renal cell carcinoma[J]. Oncotarget, 2017, 8: 96103–96116. |
| [48] | Stemmler MP, Eccles RL, Brabletz S, et al. Non-redundant functions of EMT transcription factors[J]. Nat Cell Biol, 2019, 21: 102–112. DOI:10.1038/s41556-018-0196-y |
| [49] | Fukumoto T, Zhu H, Nacarelli T, et al. N6-Methylation of adenosine of FZD10 mRNA contributes to PARP inhibitor resistance[J]. Cancer Res, 2019, 79: 2812–2820. |
| [50] | Taketo K, Konno M, Asai A, et al. The epitranscriptome m6A writer METTL3 promotes chemo- and radioresistance in pancreatic cancer cells[J]. Int J Oncol, 2018, 52: 621–629. |
| [51] | Zhou S, Bai ZL, Xia D, et al. FTO regulates the chemo-radiotherapy resistance of cervical squamous cell carcinoma (CSCC) by targeting beta-catenin through mRNA demethylation[J]. Mol Carcinog, 2018, 57: 590–597. DOI:10.1002/mc.22782 |
| [52] | Nishizawa Y, Konno M, Asai A, et al. Oncogene c-Myc promotes epitranscriptome m6A reader YTHDF1 expression in colorectal cancer[J]. Oncotarget, 2018, 9: 7476–7486. |
| [53] | Cui Q, Shi H, Ye P, et al. m6A RNA methylation regulates the self-renewal and tumorigenesis of glioblastoma stem cells[J]. Cell Rep, 2017, 18: 2622–2634. DOI:10.1016/j.celrep.2017.02.059 |
| [54] | Warburg O. On the origin of cancer cells[J]. Science, 1956, 123: 309–314. DOI:10.1126/science.123.3191.309 |
| [55] | Fischer J, Koch L, Emmerling C, et al. Inactivation of the FTO gene protects from obesity[J]. Nature, 2009, 458: 894–898. DOI:10.1038/nature07848 |
| [56] | Zhao X, Yang Y, Sun BF, et al. FTO-dependent demethylation of N6-methyladenosine regulates mRNA splicing and is required for adipogenesis[J]. Cell Res, 2014, 24: 1403–1419. DOI:10.1038/cr.2014.151 |
| [57] | Liu Y, Wang R, Zhang L, et al. The lipid metabolism gene FTO influences breast cancer cell energy metabolism via the PI3K/AKT signaling pathway[J]. Oncol Lett, 2017, 13: 4685–4690. DOI:10.3892/ol.2017.6038 |
| [58] | Mathiyalagan P, Adamiak M, Mayourian J, et al. FTO-dependent N6-methyladenosine regulates cardiac function during remodeling and repair[J]. Circulation, 2019, 139: 518–532. DOI:10.1161/CIRCULATIONAHA.118.033794 |
| [59] | Rong ZX, Li Z, He JJ, et al. Downregulation of fat mass and obesity associated (FTO) promotes the progression of intrahepatic cholangiocarcinoma[J]. Front Oncol, 2019, 9: 369. DOI:10.3389/fonc.2019.00369 |
| [60] | Panneerdoss S, Eedunuri VK, Yadav P, et al. Cross-talk among writers, readers, and erasers of m6A regulates cancer growth and progression[J]. Sci Adv, 2018, 4: eaar8263. DOI:10.1126/sciadv.aar8263 |
| [61] | Yang D, Qiao J, Wang G, et al. N6-Methyladenosine modification of lincRNA 1281 is critically required for mESC differentiation potential[J]. Nucleic acids research, 2018, 46: 3906–3920. DOI:10.1093/nar/gky130 |
| [62] | Vu LP, Pickering BF, Cheng Y, et al. The N6-methyladenosine (m6A)-forming enzyme METTL3 controls myeloid differentiation of normal hematopoietic and leukemia cells[J]. Nat Med, 2017, 23: 1369–1376. DOI:10.1038/nm.4416 |
| [63] | Weng H, Huang H, Wu H, et al. METTL14 inhibits hematopoietic stem/progenitor differentiation and promotes leukemogenesis via mRNA m6A modification[J]. Cell Stem Cell, 2018, 22: 191–205.e9. DOI:10.1016/j.stem.2017.11.016 |
| [64] | Barbieri I, Tzelepis K, Pandolfini L, et al. Promoter-bound METTL3 maintains myeloid leukaemia by m6A-dependent translation control[J]. Nature, 2017, 552: 126–131. DOI:10.1038/nature24678 |
| [65] | Paris J, Morgan M, Campos J, et al. Targeting the RNA m6A reader YTHDF2 selectively compromises cancer stem cells in acute myeloid leukemia[J]. Cell Stem Cell, 2019, 25: 137–148.e6. DOI:10.1016/j.stem.2019.03.021 |
| [66] | Li J, Han Y, Zhang H, et al. The m6A demethylase FTO promotes the growth of lung cancer cells by regulating the m6A level of USP7 mRNA[J]. Biochem Biophys Res Commun, 2019, 512: 479–485. DOI:10.1016/j.bbrc.2019.03.093 |
| [67] | Liu J, Ren D, Du Z, et al. m6A demethylase FTO facilitates tumor progression in lung squamous cell carcinoma by regulating MZF1 expression[J]. Biochem Biophys Res Commun, 2018, 502: 456–464. DOI:10.1016/j.bbrc.2018.05.175 |
| [68] | Du Y, Hou G, Zhang H, et al. SUMOylation of the m6A-RNA methyltransferase METTL3 modulates its function[J]. Nucleic Acids Res, 2018, 46: 5195–5208. DOI:10.1093/nar/gky156 |
| [69] | Du M, Zhang Y, Mao Y, et al. MiR-33a suppresses proliferation of NSCLC cells via targeting METTL3 mRNA[J]. Biochem Biophys Res Commun, 2017, 482: 582–589. DOI:10.1016/j.bbrc.2016.11.077 |
| [70] | Chen M, Wei L, Law CT, et al. RNA N6-methyladenosine methyltransferase-like 3 promotes liver cancer progression through YTHDF2-dependent posttranscriptional silencing of SOCS2[J]. Hepatology, 2018, 67: 2254–2270. DOI:10.1002/hep.29683 |
| [71] | Ma JZ, Yang F, Zhou CC, et al. METTL14 suppresses the metastatic potential of hepatocellular carcinoma by modulating N6-methyladenosine-dependent primary microRNA processing[J]. Hepatology, 2017, 65: 529–543. DOI:10.1002/hep.28885 |
| [72] | Yang Z, Li J, Feng G, et al. MicroRNA-145 modulates N6-methyladenosine levels by targeting the 3'-untranslated mRNA region of the N6-methyladenosine binding YTH domain family 2 protein[J]. J Biol Chem, 2017, 292: 3614–3623. DOI:10.1074/jbc.M116.749689 |
| [73] | Zhang C, Samanta D, Lu H, et al. Hypoxia induces the breast cancer stem cell phenotype by HIF-dependent and ALKBH5-mediated m6A-demethylation of NANOG mRNA[J]. Proc Natl Acad Sci U S A, 2016, 113: 2047–2056. DOI:10.1073/pnas.1521518113 |
| [74] | Niu Y, Lin Z, Wan A, et al. RNA N6-methyladenosine demethylase FTO promotes breast tumor progression through inhibiting BNIP3[J]. Mol Cancer, 2019, 18: 46. DOI:10.1186/s12943-019-1004-4 |
| [75] | Zhang S, Zhao BS, Zhou A, et al. m6A demethylase ALKBH5 maintains tumorigenicity of glioblastoma stem-like cells by sustaining FOXM1 expression and cell proliferation program[J]. Cancer Cell, 2017, 31: 591–606.e6. DOI:10.1016/j.ccell.2017.02.013 |
| [76] | Dahal U, Kang L, Gupta M. RNA m6A methyltransferase METTL3 regulates invasiveness of melanoma cells by matrix metallopeptidase 2[J]. Melanoma Res, 2019, 29: 382–389. DOI:10.1097/CMR.0000000000000580 |
| [77] | Su R, Dong L, Li C, et al. R-2HG exhibits anti-tumor activity by targeting FTO/m6A/MYC/CEBPA signaling[J]. Cell, 2018, 172: 90–105.e23. DOI:10.1016/j.cell.2017.11.031 |
| [78] | Chen W, Zheng R, Baade PD, et al. Cancer statistics in China[J]. Cancer J Clin, 2016, 66: 115–132. DOI:10.3322/caac.21338 |
| [79] | Siegel RL, Miller KD, Jemal A. Cancer statistics[J]. CA Cancer J Clin, 2016, 66: 7–30. DOI:10.3322/caac.21332 |
| [80] | Zhang C, Zhi WI, Lu H, et al. Hypoxia-inducible factors regulate pluripotency factor expression by ZNF217- and ALKBH5-mediated modulation of RNA methylation in breast cancer cells[J]. Oncotarget, 2016, 7: 64527–64542. |
| [81] | Liu J, Eckert MA, Harada BT, et al. m6A mRNA methylation regulates AKT activity to promote the proliferation and tumorigenicity of endometrial cancer[J]. Nat Cell Biol, 2018, 20: 1074–1083. DOI:10.1038/s41556-018-0174-4 |
| [82] | Li Z, Weng H, Su R, et al. FTO plays an oncogenic role in acute myeloid leukemia as a N6-methyladenosine RNA demethylase[J]. Cancer Cell, 2017, 31: 127–141. DOI:10.1016/j.ccell.2016.11.017 |
| [83] | Filippakopoulos P, Qi J, Picaud S, et al. Selective inhibition of BET bromodomains[J]. Nature, 2010, 468: 1067–1073. DOI:10.1038/nature09504 |
| [84] | Pfister SX, Ashworth A. Marked for death: targeting epigenetic changes in cancer[J]. Nat Rev Drug Discov, 2017, 16: 241–263. DOI:10.1038/nrd.2016.256 |
| [85] | Chen B, Ye F, Yu L, et al. Development of cell-active N6-methyladenosine RNA demethylase FTO inhibitor[J]. J Am Chem Soc, 2012, 134: 17963–17971. DOI:10.1021/ja3064149 |
| [86] | Huang Y, Yan J, Li Q, et al. Meclofenamic acid selectively inhibits FTO demethylation of m6A over ALKBH5[J]. Nucleic Acids Res, 2015, 43: 373–384. DOI:10.1093/nar/gku1276 |
| [87] | Wang R, Han Z, Liu B, et al. Identification of natural compound radicicol as a potent FTO inhibitor[J]. Mol Pharm, 2018, 15: 4092–4098. DOI:10.1021/acs.molpharmaceut.8b00522 |
| [88] | He W, Zhou B, Liu W, et al. Identification of a novel small-molecule binding site of the fat mass and obesity associated protein (FTO)[J]. J Med Chem, 2015, 58: 7341–7348. DOI:10.1021/acs.jmedchem.5b00702 |
| [89] | Peng S, Xiao W, Ju D, et al. Identification of entacapone as a chemical inhibitor of FTO mediating metabolic regulation through FOXO1[J]. Sci Transl Med, 2019, 11: eaau7116. DOI:10.1126/scitranslmed.aau7116 |
| [90] | Han X, Wang N, Li J, et al. Identification of nafamostat mesilate as an inhibitor of the fat mass and obesity-associated protein (FTO) demethylase activity[J]. Chem Biol Interact, 2019, 297: 80–84. DOI:10.1016/j.cbi.2018.10.023 |
| [91] | Xie LJ, Yang XT, Wang RL, et al. Identification of flavin mononucleotide as a cell-active artificial N6-methyladenosine RNA demethylase[J]. Angew Chem Int Ed Engl, 2019, 58: 5028–5032. DOI:10.1002/anie.201900901 |
| [92] | Wang T, Hong T, Huang Y, et al. Fluorescein derivatives as bifunctional molecules for the simultaneous inhibiting and labeling of FTO protein[J]. J Am Chem Soc, 2015, 137: 13736–13739. DOI:10.1021/jacs.5b06690 |
| [93] | Aik W, Demetriades M, Hamdan MK, et al. Structural basis for inhibition of the fat mass and obesity associated protein (FTO)[J]. J Med Chem, 2013, 56: 3680–3688. DOI:10.1021/jm400193d |
| [94] | Zheng G, Cox T, Tribbey L, et al. Synthesis of a FTO inhibitor with anticonvulsant activity[J]. ACS Chem Neurosci, 2014, 5: 658–665. DOI:10.1021/cn500042t |
| [95] | Huang Y, Su R, Sheng Y, et al. Small-molecule targeting of oncogenic FTO demethylase in acute myeloid leukemia[J]. Cancer Cell, 2019, 35: 677–691.e10. DOI:10.1016/j.ccell.2019.03.006 |
| [96] | Milaneschi Y, Lamers F, Mbarek H, et al. The effect of FTO rs9939609 on major depression differs across MDD subtypes[J]. Mol Psychiatry, 2014, 19: 960–962. |
| [97] | Xu C, Liu K, Tempel W, et al. Structures of human ALKBH5 demethylase reveal a unique binding mode for specific single-stranded N6-methyladenosine RNA demethylation[J]. J Biol Chem, 2014, 289: 17299–17311. DOI:10.1074/jbc.M114.550350 |
| [98] | Wu J, Xiao H, Wang T, et al. N6-Hydroperoxymethyladenosine: a new intermediate of chemical oxidation of N6-methyladenosine mediated by bicarbonate-activated hydrogen peroxide[J]. Chem Sci, 2015, 6: 3013–3017. DOI:10.1039/C5SC00484E |
| [99] | Xie LJ, Wang RL, Wang D, et al. Visible-light-mediated oxidative demethylation of N6-methyl adenines[J]. Chem Commun (Camb), 2017, 53: 10734–10737. DOI:10.1039/C7CC05544G |
| [100] | Li JX, Chen ZJ, Chen F, et al. Targeted mRNA demethylation using an engineered dCas13b-ALKBH5 fusion protein[J]. bioRxiv, 2019. DOI:10.1101/614859 |
 2019, Vol. 54
2019, Vol. 54