2. 中国中医科学院中药资源中心, 北京 100700;
3. 甘肃省农业工程技术研究院, 甘肃 武威 733000
2. National Resource Center for Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China;
3. Gansu Institute of Agricultural Engineering Technology, Wuwei 733000, China
罂粟(Papaver somniferum L.)与虞美人(Papaver rhoeas L.)均是罂粟科罂粟属植物, 为一年生草本植物。罂粟是一种重要的药源植物, 含有100多种生物碱, 已分离的生物碱有20多种, 是麻醉性镇痛药物吗啡、可待因和半合成类似物(如羟考酮、氢可酮和纳曲酮)的主要原料[1]。其同属近源种虞美人中也含有多种生物碱, 如阿朴啡、丽春花碱、原阿片碱和海罂粟胺等, 在民间被用来治疗咳嗽、腹泻等症, 但研究表明虞美人中不含有吗啡和可待因生物碱[2-4]。
CODM基因是苄基异喹啉类生物碱可待因、吗啡生物合成过程中的关键酶基因。研究表明, CODM基因可竞争性利用可待因生物合成中的上游底物蒂巴因合成吗啡, 同时又可促进可待因向吗啡的转化[5]。如Wijekoo等[6]发现降低CODM水平则引起吗啡含量的下降, 而可待因成分积累。Hagel等[5]发现不同罂粟资源中CODM转录水平的差别可导致吗啡、可待因含量的差异。
本文以3个不同罂粟资源及不含吗啡、可待因成分的虞美人为研究对象, 克隆其CODM基因, 并对二者的序列特征进行比较, 以期为进一步开展罂粟生物碱类成分的合成与调控机制研究奠定基础。
材料与方法植物材料和试剂 罂粟和虞美人叶片均由甘肃省农业工程技术研究院提供, 叶片样品自然干燥后放于密封袋中, 室温保存。
Plant Genomic DNA Kit (TIANGEN公司)、pEASY-Blunt Zero Cloning Kit (Trans公司)、Gel Extraction Kit (OMEGA公司)、15k DNA Marker (TaKaRa公司)、5k DNA Marker (TaKaRa公司)、Fast pfu Fly聚合酶(Trans公司)、2×T5 Super PCR Mix (北京擎科新业生物技术有限公司)、Trans1-T1感受态细胞(Trans公司), 引物合成及测序服务由北京睿博兴科生物技术有限公司完成。
仪器 Veriti 96孔梯度PCR扩增仪(Applied Biosystem公司)、VORTEX-2 GENIE漩涡震荡仪(Scientific industries公司)、SYNGENE凝胶成像系统(GENE公司)、Nanodrop 2000微量核酸定量分析仪(Thermo Fisher公司)、电热恒温培养箱(上海一恒科学仪器有限公司)。
基因组DNA提取 所有样品各取20 mg, 放入2 mL EP管中, 液氮研磨。使用植物DNA提取试剂盒, 依据操作说明提取, DNA溶解于60 μL灭菌蒸馏水中, 使用核酸蛋白仪测定DNA浓度和纯度后, 于-20 ℃保存备用。
引物设计与PCR扩增 以NCBI公布的罂粟全基因组(BioProject Accession: PRJNA492326)为参考, 找到CODM基因(Gene ID: 113311621)及其上下游各1 000 bp的序列, 利用Primer Premier 5软件分别设计CODM基因的外侧全长扩增引物(2001/2002)及内侧测通引物(2003/2004)并合成(表 1)。使用引物2001/2002对不同样品DNA进行扩增, 50 μL PCR反应体系包括5×Buffer 10 μL、dNTPs 6 μL、上游及下游引物各1.5 μL、Fast pfu Fly DNA聚合酶1 μL、DNA模板2 μL, 加灭菌蒸馏水补足至50 μL。反应程序为95 ℃预变性2 min; 95 ℃变性20 s, 54℃退火20 s, 72 ℃延伸2 min, 共38个循环; 72 ℃终延伸5 min。扩增产物用溴化乙锭(EB)染色的1.0%琼脂糖凝胶电泳检测, SYNGENE凝胶成像系统成像、观察。
| Table 1 Primer sequences |
CODM基因的克隆、测序 使用琼脂糖凝胶回收试剂盒回收PCR扩增片段, 切胶回收片段与克隆载体pEASY-Blunt Zero进行连接, 并转化至Trans1-T1感受态细胞中, 用含有氨苄抗性的LB平板在37 ℃下过夜培养, 通过菌落PCR鉴定阳性转化子。PCR检测体系包括2×T5 Super PCR Mix 12.5 μL、引物M13F和M13R各1 μL、菌液1 μL, 加灭菌蒸馏水补足至25 μL。反应程序为98 ℃预变性10 min; 98 ℃变性10 s, 54 ℃退火10 s, 72 ℃延伸15 s, 共30个循环; 72 ℃终延伸2 min。扩增产物用溴化乙锭染色的1.0%琼脂糖凝胶电泳检测, SYNGENE凝胶成像系统成像、观察。通过鉴定的阳性转化子送至北京睿博兴科生物技术有限公司进行测序, 测序引物为通用引物M13F、M13R及引物2003、2004。
CODM蛋白的生物信息学分析 利用ExPASY ProtParam tool (http://web.expasy.org/protparam/)对CODM的理化性质进行预测; Protscale (http://web.expasy.org/protscale/)对其亲水性和疏水性进行分析; 使用CFSSP (http://swissmodel.expasy.org/)进行蛋白质二级结构分析; Signal (http://www.cbs.dtu.dk/services/SignalP/)分析CODM蛋白信号肽; TargetP (http://www.cbs.dtu.dk/services/TargetP/)对CODM蛋白进行亚细胞定位; TMHMM Server (http://www.cbs.dtu.dk/services/TMHMM-1.0/tmhmm1.0.html)预测跨膜结构域; 使用SWISS-MODEL (https://swissmodel.expasy.org/)基于同源建模法对该蛋白的三级结构进行预测; 通过MEGA 7.0构建Neighbor-joining系统进化树[7]。
结果与分析 1 CODM基因的克隆使用试剂盒提取的罂粟及虞美人样品基因组DNA的浓度均在110 mg·L-1以上, A260/280值均在1.8~1.9之间, 表明其DNA质量较好。用1.0%的琼脂糖凝胶电泳检查DNA质量, 所提取的DNA条带清晰, 符合PCR扩增反应的要求。以虞美人和罂粟DNA为模板进行PCR扩增, 产物经1.0%琼脂糖凝胶电泳检测, 在2 500 bp附近有明亮单一的条带, 与所设计引物对应的CODM基因片段大小一致。
基因测序结果表明, 3个不同罂粟资源中CODM均有3种不同基因型(分别标注为PsCODM.1、PsCODM.2和PsCODM.3), 虞美人样品CODM基因有5种不同基因型(分别标注为PrCODM.1、PrCODM.2、PrCODM.3、PrCODM.4和PrCODM.5)。
2 CODM基因序列分析通过DNAMAN软件对罂粟和虞美人CODM基因进行多序列比对, 发现其核苷酸序列的一致性达到99.89%。BLAST分析发现, 所有CODM基因核苷酸序列与MG995581.1的同源性为100%, 与KY688198.1的同源性为87%;与XM_026560449.1的同源性为75%;与GQ500141.1的同源性为73%。
使用DNAMAN软件将CODM核苷酸序列翻译为氨基酸序列, 并进行多序列比对, 结果发现存在3种氨基酸序列, 即PsCODM.1、PsCODM.2、PsCODM.3、PrCODM.1、PrCODM.2与PrCODM.3的氨基酸序列一致(共同标注为CODM.n), 均编码360个氨基酸; PrCODM.4编码360个氨基酸; PrCODM.5编码373个氨基酸, 3种氨基酸序列的一致性达到了96.46%。其中, CODM.n氨基酸序列与已公布的罂粟全基因组序列一致; PrCODM.4的氨基酸序列由于1283位点碱基C突变为A, 导致密码子由Asp (天冬氨酸)变成了Glu (谷氨酸); PrCODM.5氨基酸序列由于1665位点由碱基G变成了T, 导致终止密码子变为了Leu (亮氨酸), 增加了12个氨基酸(图 1)。
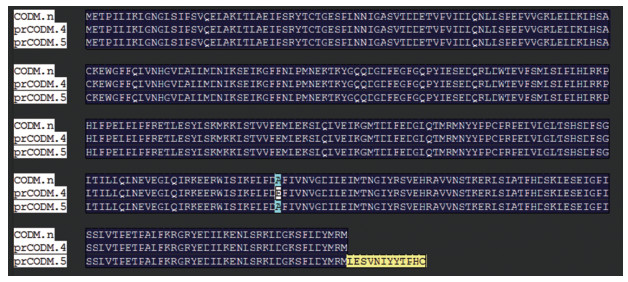
|
Figure 1 Clustalx W analyses of CODM proteins from Papaver somniferum and Papaver rhoeas |
使用ExPASY软件对罂粟和虞美人CODM进行理化性质预测, 结果显示CODM.n、CODM.4及CODM.5编码蛋白的分子质量分别为40.85 kD、40.91 kD和42.37 kD, 理论PI值分别为5.04、5.00和5.05。CODM.n和PrCODM.5编码的蛋白的负电荷残基总数(Asp+Glu)和正电荷残基总数(Arg+Lys)均相同, 分别为53和38个, 而PrCODM.4为54和38个。不稳定指数均大于40, 属于不稳定蛋白。使用ProtScale软件进一步分析表明, 这些氨基酸序列中疏水最大值均为1.900, 最小值为-2.689。利用SOPMA软件预测罂粟和虞美人CODM二级结构, 结果显示其蛋白由无规则卷曲、α-螺旋、延伸链和β-折叠构成, 其中比例最高的是无规则卷曲, β-折叠最少(图 2)。
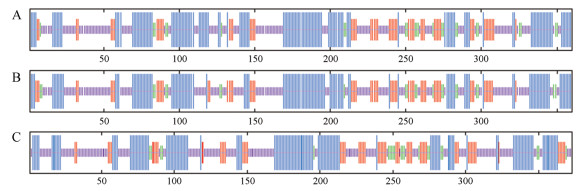
|
Figure 2 Predicts secondary structure of CODM from the amino acid sequence. A: CODM.n; B: PrCODM.4; C: PrCODM.5.-Helix; -Turn; -Sheet; -Coli |
亚细胞定位预测结果表明, 虞美人和罂粟CODM存在于线粒体的可能性为0.043, 细胞质基质为0.169, 其他细胞器为0.842, 故推定该蛋白存在于其他细胞器中。利用Signal和TMHMM Server软件分别预测该蛋白的信号肽和跨膜结构, 得出这些蛋白均不含信号肽序列, 属于非分泌蛋白。保守结构域分析结果显示, 3种CODM蛋白均具有Pcbc结构域, 由Pcbc superfamily超基因家族组成。
利用SWISS-MODEL在线软件对虞美人和罂粟的蛋白序列进行三级结构预测, 结果显示, CODM.n蛋白与SWISS-MODEL数据库目标蛋白5o7y.1.A (T6ODM)的序列相似度为73.61%, QMEAN值是-2.23; PrCODM.4蛋白SWISS-MODEL数据库目标蛋白5o7y.1.A的序列相似度为73.33%, QMEAN值是-2.25; PrCODM.5蛋白SWISS-MODEL数据库目标蛋白5o7y.1.A的序列相似度为73.61%, QMEAN值是-2.45 (图 3)。
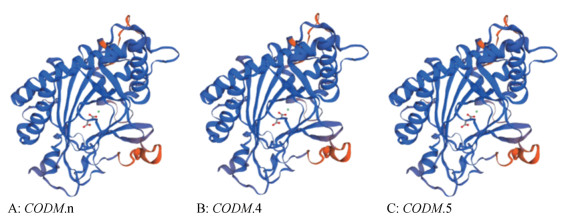
|
Figure 3 Predicts tertiary structure of CODM from the amino acid sequence |
以CODM.n氨基酸序列作为参考, 利用Blast软件搜索与CODM蛋白同源序列, 并利用MEGA 7.0进行系统发育分析, 结果表明所有氨基酸序列大致分为2大类, 第1类包括罂粟(XP_026416223.1)、博落回(OVA18943.1)和克隆得到的所有序列; 第2类是小麦(EMS50518.1)和拟南芥(XP_020869163.1) (图 4)。
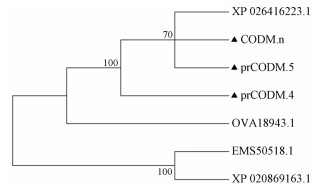
|
Figure 4 Phylogenetic analysis of CODM proteins from plants using NJ method |
本课题组保存了不同罂粟资源的转录组数据, 对CODM基因的转录水平进行分析, 结果发现不同罂粟资源中CODM基因的表达量存在明显差异(表 2), 且其表达量与吗啡和可待因生物碱含量呈正相关关系, 暗示其在吗啡和可待因生物合成中发挥重要作用。
| Table 2 Transcriptome data of CODM genes from different poppy resources |
罂粟作为一种重要的药源植物, 在医疗上的应用历史悠久且有着不可替代的作用, 尤其是镇痛作用, 目前仍是麻醉性镇痛药物吗啡、可待因和半合成类似物的唯一商业来源[8]。罂粟壳中可待因含量仅占总生物碱的0~0.70%, 药典收载的罂粟壳中吗啡含量为0.06%~0.40%, 这在国内远远不能满足医药界的需求。因此, 多年来研究人员在阐明苄基异喹啉类生物碱(BIA)代谢的吗啡分支方面做了大量的努力。CODM作为苄基异喹啉类生物碱吗啡和可待因合成过程中的关键酶基因, 它可利用底物蒂巴因合成东罂粟碱再到吗啡, 也可竞争性利用可待因合成吗啡[5], 目前对植物CODM基因的研究较少。本实验基于罂粟全基因数据库中的CODM序列, 从罂粟和虞美人中克隆得到了CODM, 结果发现罂粟中CODM有3个基因型, 虞美人中的CODM有5个基因型, Guo等[9]通过基因组测序表明罂粟CODM基因有3个拷贝, 与本实验结果相符; 而目前尚无公布虞美人的基因组序列, 通过本实验结果, 推测虞美人中CODM基因也存在多个拷贝。多序列比对发现, 这些CODM核苷酸序列的一致性高达99.88%, 氨基酸序列的一致性达到96.46%, 表明该基因在罂粟科植物的不同物种间高度保守。系统发育分析表明, 来自罂粟及虞美人的CODM.n与已公布的罂粟全基因组中的CODM序列聚为一类, 而来自虞美人的PrCODM.4、PrCODM.5基因与博落回聚为一类。表达谱分析结果发现, CODM基因在不同罂粟资源中的转录水平不同, 且在高吗啡和可待因含量的罂粟资源中表达量最高, 暗示CODM可能并不是影响罂粟中吗啡和可待因生物碱含量的唯一因素, CODM基因转录水平变异可能与其非编码区序列有关, 从而影响罂粟和虞美人中生物碱成分的积累。本研究结果不仅为进一步开展罂粟生物碱类成分的合成与调控机制研究奠定了基础, 同时也对创新可待因专用型品种具有参考价值。
| [1] | Hagel JM, Facchini PJ. Benzylisoquinoline alkaloid metabolism: a century of discovery and a brave new world[J]. Plant Cell Physiol, 2013, 54: 647–672. DOI:10.1093/pcp/pct020 |
| [2] | Choe S, Kim S, Lee C, et al. Species identification of Papaver by metabolite profiling[J]. Forensic Sci Int, 2011, 211: 51–60. DOI:10.1016/j.forsciint.2011.04.015 |
| [3] | Coban I, Toplan GG, zbek B, et al. Variation of alkaloid contents and antimicrobial activities of Papaver rhoeas L. growing in Turkey and northern Cyprus[J]. Pharm Biol, 2017, 55: 1894–1898. DOI:10.1080/13880209.2017.1340964 |
| [4] | Oh JH, Ha IJ, Lee MY, et al. Identification and metabolite profiling of alkaloids in aerial parts of Papaver rhoeas by liquid chromatography coupled with quadrupole time-of-flight tandem mass spectrometry[J]. J Sep Sci, 2018, 41: 2517–2527. DOI:10.1002/jssc.v41.12 |
| [5] | Hagel JM, Facchini PJ. Dioxygenases catalyze the O-demethylation steps of morphine biosynthesis in opium poppy[J]. Nat Chem Biol, 2010, 6: 273–275. DOI:10.1038/nchembio.317 |
| [6] | Wijekoon CP, Facchini PJ. Systematic knockdown of morphine pathway enzymes in opium poppy using virus-induced gene silencing[J]. Plant J, 2012, 69: 1052–1063. DOI:10.1111/j.1365-313X.2011.04855.x |
| [7] | Chen TY, Liu J, Yuan Y, et al. Bioinformatics analysis and expressed level of Auxin response factor genes in Scutellaria baicalensis[J]. Acta Pharm Sin (药学学报), 2017, 52: 1770–1776. |
| [8] | Facchini PJ, Park SU. Developmental and inducible accumulation of gene transcripts involved in alkaloid biosynthesis in opium poppy[J]. Phytochemistry, 2003, 64: 177–186. DOI:10.1016/S0031-9422(03)00292-9 |
| [9] | Guo L, Winzer T, Yang X, et al. The opium poppy genome and morphinan production[J]. Science, 2018, 362: 343–347. DOI:10.1126/science.aat4096 |
 2019, Vol. 54
2019, Vol. 54


