2. 北京大学第一医院药剂科, 北京 100034
2. Department of Pharmacy, Peking University First Hospital, Beijing 100034, China
阿尔茨海默病(Alzheimer's disease, AD)是一种以学习记忆能力衰退为主要临床表现的中枢神经系统退行性疾病。2016年国际阿尔茨海默病协会统计, 全球AD患者近5 000万, 预计到2050年可能达到1.3亿[1]。当今社会人口老龄化不断加重, AD不仅严重影响老年患者的生活质量, 也给家庭和社会带来沉重的负担[2]。目前AD的治疗用药主要为乙酰胆碱酯酶(aetylcholinesterase, ACHE)抑制剂和N-甲基-D-天冬氨酸(N-methyl-D-aspartic acid receptor, NMDA)受体拮抗剂, 这些药物仅能缓解临床症状, 但是不能有效延缓疾病进程[3]。曾被寄予厚望的针对Aβ沉积和神经纤维缠结两个特异性病理特征研发的药物, 在临床试验不同阶段均以失败告终, 动摇了Aβ级联假说和tau蛋白过度磷酸化假说在AD发病机制中的主导地位, 同时也说明单一靶点的抗AD药物具有很大的局限性。抗AD新药在临床试验阶段的失败率很高, 最核心的问题还是对AD的病理机制认知深度不够[4]。
为了对抗复杂性疾病, 一种创新策略是寻找能够同时调节多个靶点的单体化合物, 即多靶点定向配体(multi-target-directed ligands, MTDLs)[5]。目前MTDL已成为寻找防治复杂性疾病药物的创新方法而被广泛应用[6, 7]。这是因为很多具有良好临床疗效的药物能够与多个靶标相互作用; 另一方面, 对单个靶标具有高度特异性的选择性配体通常缺乏临床功效[8]。MTDL方法可以通过连接对不同靶标具有高度亲和力的不同分子或者发现能够同时调节多个靶标的单一配体来实现[9-11]。AD的复杂性和临床有效治疗药物的匮乏使得寻找抗AD多靶点定向配体显得尤为重要, 并吸引着越来越多的科研团队寻找该类化合物[12, 13]。
基于不同靶点分别建立药物筛选模型并用于多靶点抗AD药物的发现不仅耗时耗力, 且效率低下。因此, 有必要借助虚拟筛选(visual screening, VS)技术方法预测发现多靶点抗AD药物, 为活性评价提供信息, 以减少盲目性, 降低成本。分子对接, 药效团建模和机器学习方法是虚拟筛选中最常用的方法。机器学习方法通过采用统计算法, 包括线性判别分析[14]、支持向量机[15]、朴素贝叶斯[16]、递归分割[17]、随机森林[18]和人工神经网络[19]等, 建立具有将某靶点抑制剂与非抑制剂分离能力的分类模型。支持向量机和朴素贝叶斯模型已成功应用于丁酰胆碱酯酶等抑制剂的虚拟筛选[16, 20]。本实验室前期利用多种算法建立了拥有52个靶点、208个预测模型的抗AD多靶点预测平台[17], 可用于预测多靶点抗AD单体化合物。
众所周知, 药物研发的周期长、耗资大、风险高。近年来, 通过创新药物投入与产出关系来衡量的药物研发生产率急剧下降[21-23]。许多因素导致药物研发过程中生产率下降, 包括研发成本增加, 重磅药物专利到期导致的一般竞争, 越来越保守的监管政策以及不充分创新等。药物研发投入与产出不平衡的矛盾在需要更多投资的疾病中更为明显, 例如阿尔茨海默病[21]。而药物重定位策略是克服上述诸多问题的重要解决方案之一[24-26]。药物重定位, 又称老药新用, 是指对于已批准上市应用于临床或者未上市但结构明确、生物活性已知的药品, 通过进一步研究发现其新的作用靶点, 扩大其适应症范围[27]。与药物从头研发相比, 由于上市药物已经过长期临床实践, 其毒性和不良反应均已明确, 且生产工艺、质量标准、剂型等完备, 所以基于上市药物的新药研发在时间和经济成本方面都有显著优势, 且成功率大幅度提升(图 1)。

|
Figure 1 A schematic representation of two different drug discovery processes. A: Traditional drug discovery and development; B: Drug repositioning. Traditional de novo drug discovery and development involves a 13-15-year process. Drug repositioning decreases costs and time to launch, and reduces safety and pharmacokinetic uncertainty frequently associated with traditional drug development |
本研究基于药学数据中心开发的全球上市药物数据库(http://pharmdata.ncmi.cn/globaldrugs/index.asp)和实验室前期建立的抗AD多靶点预测平台, 利用平台中所有预测模型进行AD靶点-化合物相互作用预测, 并利用Drugbank数据库(https://www.Drugbank.ca/)对预测结果进行验证。以发现潜在的多靶点防治AD的上市药物, 并针对其作用靶点建立分子对接模型, 以验证该上市药物与AD靶点的作用机制, 为抗AD上市药物重定位的进一步研究提供重要信息依据。
材料与方法上市药物的收集 全球上市药物从Drugbank数据库(https://www.Drugbank.ca/)和国家人口与健康科学数据共享平台药学数据中心的全球上市药物数据库(http://pharmdata.ncmi.cn/globaldrugs/index.asp)中收集。
上市药物分子指纹符计算 在应用抗AD靶点预测系统预测潜在作用靶点之前, 需要对上市药物数据集进行数据准备工作, 分别计算其ECFP_6和MACCS两种分子指纹符。其中ECFP_6分子指纹符属于环形拓扑指纹设计的分子特性, 被广泛应用于分子类似性搜索和定量构效关系模型[28], 在Discovery Studio 2018 (San Diego, CA, USA)软件中计算。MACCS分子指纹符使用MDL结构库, 共包含166种子结构, 在PaDEL Descriptor[29]软件中进行计算。
基于抗AD靶点预测系统的化合物-靶点相互作用预测 课题组前期建立的多靶点抗AD化合物-蛋白相互作用(compound-protein interaction, CPI)预测mt-QSAR模型是由多个AD相关靶点的二分类模型组成的。针对52个AD相关靶点, 分别基于ECFP_6和MACCS分子指纹符, 采用朴素贝叶斯和递归分割两种机器学习算法共构建了208个二分类预测模型, 即针对每个靶点, 共建立了NB (ECFP_6)、NB (MACCS)、RP (ECFP_6)、RP (MACCS) 4个二分类预测模型。经过前期工作分析发现, 在使用ECFP_6分子指纹符时, 采用朴素贝叶斯运算方法构建的分类模型NB (ECFP_6)表现较好。相反, 在使用MACCS分子指纹符时, 采用递归分割运算方法构建的分类模型RP (MACCS)表现更佳[17]。在抗AD靶点预测系统中, 分别采用基于ECFP_6分子指纹符的朴素贝叶斯模型NB (ECFP_6)和基于MACCS分子指纹符的递归分割模型RP (MACCS)预测上市药物对52个AD相关靶点的潜在活性, 当上市药物同时被两个模型预测为阳性时, 则认为该上市药物对该AD相关靶点具有活性。
分子对接验证 将潜在的具有抗AD作用的上市药物挑选出来, 利用上市药物的预测靶点构建分子对接模型。这些靶点蛋白的晶体结构来自PDB (Protein Data Bank)数据库。为了保证分子对接的可靠性, 选择分辨率 < 2.5 Å且具有配体复合物的蛋白晶体结构, 建立分子对接模型。分子对接程序使用Discovery Studio 2018 (San Diego, CA, USA)软件包的Libdock模块。首先去除PDB结构中的水分子, 对接的活性口袋由原始配体分子进行定义。设置对接参数之后, 将晶体结构中的配体分子抽取出来并重新对接至预先定义好的活性口袋, 同时计算对接后的配体分子构象与晶体结构中的初始构象之间的均方根差值(root-mean-square deviation, RMSD), 若RMSD值小于2.5, 则认为分子对接结果是可靠的。在此基础上, 对具有潜在活性的上市药物进行对接分析与验证。
结果 1 上市药物数据集的建立通过对Drugbank数据库和全球上市药物数据库进行搜索、下载合并, 去除重复结构后, 建立全球上市药物数据集, 总计2 217个化合物。
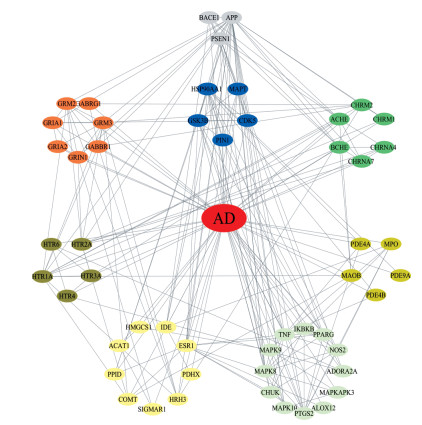
2 抗AD多靶点预测平台本实验室建立的抗AD多靶点预测平台共包含52个蛋白靶点, 利用STRING数据库(https://string-db.org/)对靶点的相互关系进行预测, 构建蛋白质相互作用网络, 如图 2所示; 对52个AD相关靶点进行统计和分类, 如表 1所示。这些靶点相关的功能涉及AD发生发展的各个方面, 包括胆碱能系统功能障碍、β-淀粉样蛋白(Aβ)、tau蛋白过度磷酸化、谷氨酸/GABA系统功能障碍、5-羟色胺能系统功能障碍、氧化应激、神经炎症和线粒体功能障碍等。
|
Figure 2 The analysis of protein-protein network of Alzheimer's disease (AD) related targets by STRING |
| Table 1 Basic information of AD related targets |
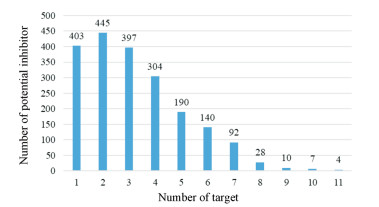
基于上述虚拟筛选平台, 对本文建立的全球上市药物数据集进行活性预测, 并对该数据集中不同作用靶点数目的上市药物数目进行统计, 结果见图 3。大部分上市药物均被预测可作用于该平台中的一个或多个靶点, 其中有49个上市药物可以作用于8个及以上的AD相关靶点, 从中挑选出13个至少作用于1个FDA批准上市抗AD药物相关靶点, 且可以通过多种作用对抗AD的化合物, 应用Cytoscape-3.6.1软件构建了13个上市药物的化学成分-靶点网络, 见图 4。统计这13个上市药物的预测靶点, 部分被Drugbank数据库所验证, 如表 2所示, 未被验证的靶点将为药物的靶点研究提供重要信息。
|
Figure 3 The number of potential inhibitors in database for global drugs on market in different frequency of targets |

|
Figure 4 The constituent-target network for potential anti-AD marketed drugs. The blue rectangle means marketed drugs, and the pink means predicted drug target |
| Table 2 The typical examples of multi-target constituents. The marked targets have been experimentally verified |
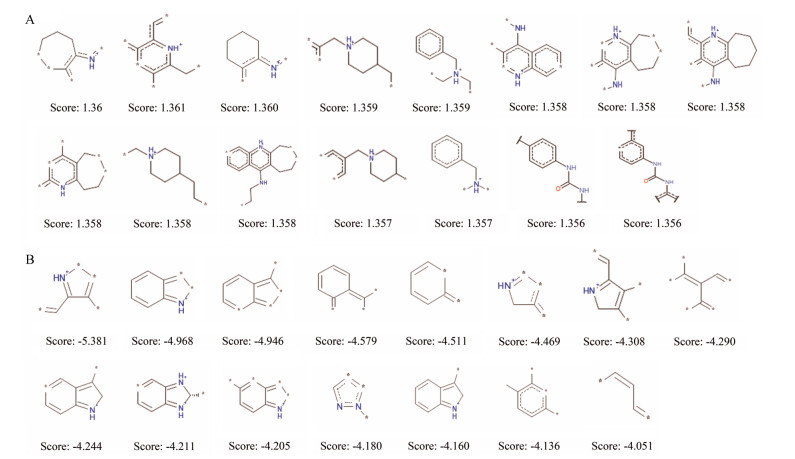
在统计的13个上市药物中, 有12个可以作用于乙酰胆碱酯酶, 观察这些化合物的分子结构发现了一个有趣的现象, 大部分被预测为乙酰胆碱酯酶抑制剂的分子含有一个或多个氮原子。在朴素贝叶斯模型的建立过程中引入ECFP_6分子指纹符, 不仅可以提高模型的预测能力, 同时也可以分析得到对特定化合物成为活性化合物具有较大影响的优势和劣势片段。朴素贝叶斯模型在建立过程中会对每个结构片段进行贝叶斯打分(Bayesian score), 结构片段获得的贝叶斯打分越高, 说明该片段对特定化合物成为活性化合物的贡献越大。本文根据NB (ECFP_6)模型对结构片段的贝叶斯打分总结了针对ACHE抑制剂的15个优势片段和15个劣势片段, 对这些优势片段和劣势片段的分析将有助于指导ACHE抑制剂的合理化药物设计(图 5)。
|
Figure 5 Examples of the top 15 good (A) and bad (B) fragments for ACHE inhibition as estimated by NB(ECFP_6) model. The Bayesian score (score) is given for each fragment |
通过对15个优势片段的分析, 本文发现其中大部分结构片段中都含有一个或多个氮原子。其中一部分氮原子存在六元或七元杂环中, 而另一部分氮原子则与六元环状结构或者羰基结构连接。在15个劣势片段中, 有9个片段中含有一个或多个氮原子, 但是这些
氮原子都没有直接与六元环状结构或者羰基结构连接, 而是存在于五元杂环中, 提示含氮五元环的存在不利于抑制ACHE的活性。
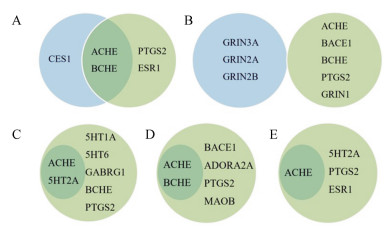
3.3 已上市抗AD药物预测结果分析在Drugbank数据库中收集了FDA批准上市抗AD药物的作用靶点, 并与预测平台的预测结果进行对比, 如图 6所示, 多奈哌齐、艾斯能和加兰他敏的作用靶点全被预测平台命中, 并提示这些药物可能通过作用于其他蛋白靶点改善阿尔茨海默病症状, 他克林和美金刚只有部分靶点被预测平台命中, 其他已报道的靶点为进一步扩大预测平台靶点数目, 提高预测平台的完整性提供了依据。
|
Figure 6 The proved targets and predicted targets of tacrine (A), memantine (B), donepezil (C), rivastigmine (D) and galantamine (E). The blue circle means experimentally validated targets and the green circle means predicted targets |
预测结果表明, 阿戈美拉汀(agomelatine)可以作用于9个AD相关靶点, 包括HTR2A、ADORA2A、ACHE、BACE1、PTGS2、GABRG1、MAOB、SIGMAR1和ESR1, 说明阿戈美拉汀可能通过修复胆碱能系统功能障碍、5-羟色胺能系统功能障碍、谷氨酸/GABA系统功能障碍, 清除β-淀粉样蛋白, 抑制氧化应激、神经炎症来发挥抗AD作用。通过在PDB中检索靶点蛋白, 共获得7个可用于分子对接的靶点蛋白的晶体结构, 包括ADORA2A、ACHE、BACE1、PTGS2、MAOB、SIGMAR1、ESR1, 通过建立分子对接模型, 分析化学成分与靶点的相互作用情况。在化学成分与靶点进行分子对接计算之前, 对靶点晶体结构中的配体进行re-docking验证, 通过计算对接后配体与晶体中的配体的空间坐标, 计算其RMSD值, 当RMSD < 2.5时, 说明对接结果是可靠的。从表 3中的结果可以看出, 其RMSD值均小于2.5, 表明分子对接计算结果是可靠的。Libdock score指示了配体与靶点蛋白晶体的结合程度, 由表 4可知, 阿戈美拉汀与ADORA2A和PTGS2的对接打分值超过了原配体。本文选取了Libdock结果中与阿戈美拉汀对接效果最好的4个靶点蛋白, 分别为ADORA2A、PTGS2、MAOB和SIGMAR1, 用CDocker精准分子对接方法验证了阿戈美拉汀与这4个靶点蛋白的相互作用, 结果如表 4所示, -CDocker energy指示配体与靶点蛋白间的结合能力, 其值越高, 提示结合能力越强, 阿戈美拉汀与ADORA2A的结合能力超过该靶点蛋白的原配体, 与其他3个靶点蛋白也有较好的亲和能力。图 7举例了阿戈美拉汀可以通过氢键、Pi键、离子间的引力等多种方式与多个靶点结合, 证明了预测结果的可靠性。
| Table 3 The target used for docking and their PDB ID, RMSD for validation and Libdock scores of agomelatine and the co-crystallized ligands |
| Table 4 The target used for docking and their PDB ID, RMSD for validation and -CDocker energy of agomelatine and the co-crystallized ligands |

|
Figure 7 The receptor-ligand interaction of agomelatine with the active site of ADORA2A (A, B), PTGS2 (C, D), MAOB (E, F), SIGMAR1 (G, H) |
药物从最初的实验室研究到最终生产上市平均需要花费13~15年的时间, 总平均成本在20亿~30亿美元之间[30], 是一个长期、艰难和昂贵的过程。英国科学家、诺贝尔生理学和医学奖获得者杰姆斯∙布莱克指出: “新药发现最有成效的基础是从旧药开始”[31]。药物重定位相对于传统的药物挖掘来说, 已经通过各项体内外实验、大量毒理研究以及早期的临床研究。因此, 对药物进行重定位可以缩短药物研发时间、减少成本, 很大程度上降低了新药研发的风险并且可以获得更大的经济效益。基于此思路, 本文利用实验室前期建立的多靶点抗AD预测平台, 对全球上市药物数据集进行了预测, 以期发现具有潜在抗AD作用的上市药物, 为上市药物抗AD作用重定位提供思路和依据。
虽然AD发病机制复杂且尚不明确, 但已确定了大量具有潜在治疗作用的生物学靶点[32, 33]。主要包括: β淀粉样蛋白质、tau蛋白、胆碱能受体、谷氨酸受体、5-羟色胺受体、多巴胺受体、去甲肾上腺素受体、乙酰胆碱酯酶、丁酰胆碱酯酶、α-, β-和γ-分泌酶、单胺氧化酶A和单胺氧化酶B等。在AD的病理机制中还涉及许多过程, 被认为是寻找AD治疗药物的希望, 最重要的包括兴奋毒性、氧化应激、神经炎症和线粒体损伤等。鉴于AD发病机制的复杂性及对其认知的有限性, 多靶点定向配体或许会为抗AD药物发现提供新的出路, 目前已经开发出多个可用于治疗阿尔茨海默病的潜在多靶点定向配体[34-37], 为多靶点抗AD药物的研发提供了思路和借鉴。
从本文的预测结果看来, 大部分上市药物均可作用于多靶点抗AD预测平台中的靶点, 从中挑选出13个典型的可以通过作用于多个AD相关病理过程发挥作用的上市药物, 并统计其在Drugbank数据库中收录的作用靶点及预测靶点, 值得注意的是, 这些药物在Drugbank数据库中收录的已报道的作用靶点如果与AD相关且包含在本实验室构建的抗AD靶点预测平台中, 几乎均被预测为阳性, 证明了本实验室构建的多靶点抗AD预测平台的可靠性。其中, 姜黄素(curcumin)被预测出可作用于TNF、MAPT、ALOX12、ESR1、ACHE、APP、PTGS2、GSK3B、PTGS2和MAOB这10个靶点, 提示姜黄素可能通过修复胆碱能系统功能障碍, 减少β-淀粉样蛋白沉积, 抑制tau蛋白过度磷酸化, 减少氧化应激、神经炎症来发挥抗AD作用。近年来有大量关于姜黄素对抗AD的报道, 体外实验[38]发现姜黄素能抑制Aβ的生成和聚积, 使已形成的纤维状Aβ降解, 提示姜黄素可能作用于Aβ生成后的某个环节, 抑制其聚集和沉积; 另有研究报道姜黄素可以发挥单胺氧化酶(monoamine oxidanse, MAO)抑制剂的作用, 增高小鼠不同脑区单胺递质的含量从而改善其痴呆症状[39]; Wang等[40]发现姜黄素可以减少COX-2和胶质原纤维酸性蛋白的表达来发挥PPAR-γ激活剂的作用, 通过增加PPAR-γ的表达和降低NF-κB活性减轻Aβ刺激引起的原代星形胶质细胞的炎症反应, 进而缓解AD患者症状。Zhang等[41]发现姜黄素对Aβ侧脑室内注射所致AD模型大鼠的脑组织具有保护作用, 能增强SOD活性, 提高机体抗自由基氧化的功能, 减少脑组织内NO、MDA的含量, 降低细胞的脂质过氧化程度。大量文献报道的姜黄素抗AD作用与本文的预测结果相符, 提示姜黄素可能被开发为多靶点抗AD药物。
罗格列酮(rosiglitazone)是一种用于改善胰岛素抵抗, 治疗2型糖尿病的上市药物, 被预测出可作用于ADORA2A、ACHE、GSK3B、NOS2、MAOB、CHRM2、GRM2、ESR1和TNF 9个靶点, 提示罗格列酮可能通过修复胆碱能系统功能障碍、谷氨酸/GABA系统功能障碍, 抑制tau蛋白过度磷酸化, 减少氧化应激、神经炎症来发挥抗AD作用。之前对AD的研究大多集中于Aβ沉积和tau蛋白过度磷酸化, 随着研究的深入, 人们逐渐认识到AD可能是一种由脑胰岛素及胰岛素样生长因子抵抗介导的代谢性疾病, AD与胰岛素抵抗所致的疾病有许多共同特征[42]。近年来关于胰岛素抵抗和AD相关的研究和报道越来越多。Xu等[43]发现罗格列酮以剂量依赖的方式减弱Aβ42寡聚体诱导的大鼠记忆障碍, 罗格列酮完全阻止Aβ42寡聚体诱导的炎性细胞因子IL-1β和IFN-γ水平的增加, 提示抑制炎症反应可能是罗格列酮预防由Aβ42寡聚体诱导的记忆缺陷的作用机制的一部分。Song等[44]发现罗格列酮可以改善三重转基因模型小鼠AD样的学习记忆减退和Tau蛋白的过磷酸化表达。罗格列酮有明显的神经保护作用并被认为是潜在的可作为治疗AD的药物[45]。
在13个被预测为多靶点抗AD的上市药物中, 有2个为已上市的抗抑郁药, 阿戈美拉汀和米帕明(imipramine)。目前, 有相关研究表明, 抗抑郁药具有一定改善和预防AD的作用, 近年来临床上也常将抗抑郁药应用于辅助治疗AD, 且取得一定成效[46]。虽然目前还没有关于阿戈美拉汀用于治疗AD的相关研究, 但是从本文的预测结果来看, 阿戈美拉汀作用于HTR2A、ADORA2A、ACHE、BACE1、PTGS2、GABRG1、MAOB、SIGMAR1和ESR1这9个靶点, 说明阿戈美拉汀可能通过修复胆碱能系统功能障碍、5-羟色胺能系统功能障碍、谷氨酸/GABA系统功能障碍, 清除β-淀粉样蛋白, 抑制氧化应激、神经炎症等多靶点多通路来发挥抗AD作用。利用分子对接方法, 本文验证了阿戈美拉汀与预测靶点蛋白的结合情况, 从分子对接的结果看来, 挑选出来的靶点蛋白均可与阿戈美拉汀结合, 且ADORA2A和PTGS2两个蛋白与阿戈美拉汀结合的打分值超过了蛋白的原配体。从该预测结果看来, 阿戈美拉汀可能是一种潜在的可用于治疗AD的多靶点药物, 其体内/体外作用活性还需要进一步验证。
综上所述, 本文利用实验室前期建立的抗AD多靶点预测平台, 对全球上市药物数据集进行了预测, 并从中挑选出具有代表性的可作用于多个靶点的上市药物进行统计分析。研究发现, 阿戈美拉汀可通过多靶点多途径发挥抗AD的作用, 为深入研究提供重要信息依据。同时也为其他上市药物的重定位提供借鉴。
致谢: 感谢国家人口与健康科学数据共享平台提供的全球上市药物数据库(http://pharmdata.ncmi.cn/globaldrugs/index.asp)。
| [1] | Alzheimer's Association. 2016 Alzheimer's disease facts and figures[J]. Alzheimers Dement, 2016, 12: 459–509. DOI:10.1016/j.jalz.2016.03.001 |
| [2] | Karlawish J. How are we going to live with Alzheimer's disease?[J]. Health Affairs, 2014, 33: 541–546. DOI:10.1377/hlthaff.2014.0089 |
| [3] | Berk C, Paul G, Sabbagh M. Investigational drugs in Alzheimer's disease: current progress[J]. Expert Opin Investig Drugs, 2014, 23: 837–846. DOI:10.1517/13543784.2014.905542 |
| [4] | Kumar D, Ganeshpurkar A, Kumar D, et al. Secretase inhibitors for the treatment of Alzheimer's disease: long road ahead[J]. Eur J Med Chem, 2018, 148: 436–452. DOI:10.1016/j.ejmech.2018.02.035 |
| [5] | Cavalli A, Bolognesi ML, Minarini A, et al. Multi-target-directed ligands to combat neurodegenerative diseases[J]. J Med Chem, 2008, 51: 347–372. DOI:10.1021/jm7009364 |
| [6] | Morphy R, Rankovic Z. Designing multiple ligands - medicinal chemistry strategies and challenges[J]. Curr Pharm Des, 2009, 15: 587–600. DOI:10.2174/138161209787315594 |
| [7] | Müller-Schiffmann A, Sticht H, Korth C. Hybrid compounds: from simple combinations to nanomachines[J]. BioDrugs, 2012, 26: 21–31. DOI:10.2165/11597630-000000000-00000 |
| [8] | Espinoza-Fonseca LM. The benefits of the multi‐target approach in drug design and discovery[J]. Bioorg Med Chem, 2006, 37: 896–897. |
| [9] | Nielsch U, Schäfer S, Wild H, et al. One target-multiple indications: a call for an integrated common mechanisms strategy[J]. Drug Discov Today, 2007, 12: 1025–1031. DOI:10.1016/j.drudis.2007.10.004 |
| [10] | Speck-Planche A, Luan F, Cordeiro MN. Role of ligand-based drug design methodologies toward the discovery of new anti-Alzheimer agents: futures perspectives in fragment-based ligand design[J]. Curr Med Chem, 2012, 19: 1635–1645. DOI:10.2174/092986712799945058 |
| [11] | Costantino L, Barlocco D. Designed multiple ligands: basic research vs clinical outcomes[J]. Curr Med Chem, 2012, 19: 3353–3387. DOI:10.2174/092986712801215883 |
| [12] | Tranches DKS, Viegas JC. Multi-target directed drugs: a modern approach for design of new drugs for the treatment of Alzheimer's disease[J]. Curr Neuropharmacol, 2014, 12: 239–254. DOI:10.2174/1570159X1203140511153200 |
| [13] | Guzior N, Ckowska AW, Panek D, et al. Recent development of multifunctional agents as potential drug candidates for the treatment of Alzheimer's disease[J]. Curr Med Chem, 2015, 22: 373–404. |
| [14] | Speck-Planche A, Mn DSC. Speeding up early drug discovery in antiviral research: a fragment-based in silico approach for the design of virtual anti-hepatitis C leads[J]. ACS Comb Sci, 2017, 19: 501–512. DOI:10.1021/acscombsci.7b00039 |
| [15] | Suzuki SD, Ohue M, Akiyama Y. PKRank: a novel learning-to-rank method for ligand-based virtual screening using pairwise kernel and RankSVM[J]. Artif Life Robotics, 2018, 23: 205–212. DOI:10.1007/s10015-017-0416-8 |
| [16] | Lian W, Fang J, Li C, et al. Discovery of influenza A virus neuraminidase inhibitors using support vector machine and naïve Bayesian models[J]. Mol Divers, 2015, 20: 439–451. |
| [17] | Fang J, Li Y, Liu R, et al. Discovery of multitarget-directed ligands against Alzheimer's disease through systematic prediction of chemical-protein interactions[J]. J Chem Inf Model, 2015, 55: 149–164. DOI:10.1021/ci500574n |
| [18] | Ehrman TM, Barlow DJ, Hylands PJ. Virtual screening of Chinese herbs with random forest[J]. J Chem Inf Model, 2007, 47: 264–278. DOI:10.1021/ci600289v |
| [19] | Keserû GM, Molnár L, Greiner I. A neural network based virtual high throughput screening test for the prediction of CNS activity[J]. Comb Chem High Throughput Screen, 2000, 3: 535–540. DOI:10.2174/1386207003331346 |
| [20] | Fang J, Yang R, Gao L, et al. Predictions of BuChE inhibitors using support vector machine and naïve Bayesian classification techniques in drug discovery[J]. J Chem Inf Model, 2013, 53: 3009–3020. DOI:10.1021/ci400331p |
| [21] | Pammolli F, Magazzini L, Riccaboni M. The productivity crisis in pharmaceutical R & D[J]. Nat Rev Drug Discov, 2011, 10: 428–438. DOI:10.1038/nrd3405 |
| [22] | Schulze U, BaedekerM, Chen YT, et al. R & D productivity: on the comeback trail[J]. Nat Rev Drug Discov, 2014, 13: 331–332. DOI:10.1038/nrd4320 |
| [23] | Scannell JW, Blanckley A, Boldon H, et al. Diagnosing the decline in pharmaceutical R & D efficiency[J]. Nat Rev Drug Discov, 2012, 11: 191–200. DOI:10.1038/nrd3681 |
| [24] | Ashburn TT, Thor KB. Drug repositioning: identifying and developing new uses for existing drugs[J]. Nat Rev Drug Discov, 2004, 3: 673–683. DOI:10.1038/nrd1468 |
| [25] | Jin G, Wong ST. Toward better drug repositioning: prioritizing and integrating existing methods into efficient pipelines[J]. Drug Discov Today, 2014, 19: 637–644. DOI:10.1016/j.drudis.2013.11.005 |
| [26] | Mullard A. Drug repurposing programmes get lift off[J]. Nat Rev Drug Discov, 2012, 11: 505–506. DOI:10.1038/nrd3776 |
| [27] | Chong CR, Sullivan DJ. New uses for old drugs[J]. Nature, 2007, 448: 645–646. DOI:10.1038/448645a |
| [28] | Morgan H. The generation of a unique machine description for chemical structures-a technique developed at chemical abstracts service[J]. J Chem Doc, 1965, 5: 107–113. DOI:10.1021/c160017a018 |
| [29] | Yap CW. PaDEL-descriptor: an open source software to calculate molecular descriptors and fingerprints[J]. J Comput Chem, 2011, 32: 1466–1474. DOI:10.1002/jcc.v32.7 |
| [30] | Scannell JW, Blanckley A, Boldon H, et al. Diagnosing the decline in pharmaceutical R & D efficiency[J]. Nat Rev Drug Discov, 2012, 11: 191–200. DOI:10.1038/nrd3681 |
| [31] | Raju TN. The Nobel chronicles. 1997: Stanley Ben Prusine (b1942)[J]. Lancet, 2000, 356: 260. |
| [32] | Khan A, Corbett A, Ballard C. Emerging treatments for Alzheimer's disease for non-amyloid and non-tau targets[J]. Expert Rev Neurother, 2017, 17: 683–695. DOI:10.1080/14737175.2017.1326818 |
| [33] | Jan BG, Florin D. Cognitive decline and dementia in diabetes mellitus: mechanisms and clinical implications[J]. Nat Rev Endocrinol, 2018, 14: 591. DOI:10.1038/s41574-018-0048-7 |
| [34] | Iqbal K, Grundke-Iqbal I. Alzheimer's disease, a multifactorial disorder seeking multitherapies[J]. Alzheimers Dement, 2010, 6: 420–424. DOI:10.1016/j.jalz.2010.04.006 |
| [35] | Calzà L, Baldassarro VA, Giuliani A, et al. From the multifactorial nature of Alzheimer's disease to multitarget therapy: the contribution of the translational approach[J]. Curr Top Med Chem, 2013, 13: 1843–1852. DOI:10.2174/15680266113139990140 |
| [36] | Musiał A, Bajda M, Malawska B. Recent developments in cholinesterases inhibitors for Alzheimer's disease treatment[J]. Curr Med Chem, 2007, 14: 2654–2679. DOI:10.2174/092986707782023217 |
| [37] | Tumiatti V, Minarini A, Bolognesi ML, et al. Tacrine derivatives and Alzheimer's disease[J]. Curr Med Chem, 2012, 17: 1825–1838. |
| [38] | Begum AN, Jones MR, Lim GP, et al. Curcumin structure-function, bioavailability, and efficacy in models of neuroinflammation and Alzheimer's disease[J]. J Pharmacol Exp Ther, 2008, 326: 196–208. DOI:10.1124/jpet.108.137455 |
| [39] | Carradori S, D'Ascenzio M, Chimenti P, et al. Selective MAO-B inhibitors: a lesson from natural products[J]. Mol Divers, 2014, 18: 219–243. DOI:10.1007/s11030-013-9490-6 |
| [40] | Wang HM, Zhao YX, Zhang S, et al. PPARγ agonist curcumin reduces the amyloid-β-stimulated inflammatory responses in primary astrocytes[J]. J Alzheimers Dis, 2010, 20: 1189–1199. DOI:10.3233/JAD-2010-091336 |
| [41] | Zhang X, Zhao Z. Relationship between amelioration of curcumin on learning and memory ability and the contents of NO, SOD, MDA and ChAT in brain of AD rats[J]. J Med Res, 2012, 41: 82–86. |
| [42] | Sm DLM, Tong M. Brain metabolic dysfunction at the core of Alzheimer's disease[J]. Biochem Pharmacol, 2014, 88: 548–559. DOI:10.1016/j.bcp.2013.12.012 |
| [43] | Xu SJ, Guan Q, Wang C, et al. Rosiglitazone prevents the memory deficits induced by amyloid-beta oligomers via inhibition of inflammatory responses[J]. Neurosci Lett, 2014, 578: 7–11. DOI:10.1016/j.neulet.2014.06.010 |
| [44] | Song JZ, Sun J, Jin DC, et al. Rosiglitazone improves learning and memory impairment of 3 x Tg mice[J]. Acta Pharm Sin (药学学报), 2014, 49: 807–812. |
| [45] | Luconi M, Cantini G, Serio M. Peroxisome proliferator-activated receptor gamma (PPARgamma): is the genomic activity the only answer?[J]. Steroids, 2010, 75: 585–594. DOI:10.1016/j.steroids.2009.10.012 |
| [46] | Sheline YI, West T, Yarasheski K, et al. An antidepressant decreases CSF Aβ production in healthy individuals and in transgenic AD mice[J]. Sci Transl Med, 2014, 6: 236re4. DOI:10.1126/scitranslmed.3008169 |
 2019, Vol. 54
2019, Vol. 54