2. 海军军医大学药学院, 上海 200433
2. Pharmacy College, Second Military Medical University, Shanghai 200433, China
红花(Carthamus tinctorius L.)是一种重要的经济作物。其花冠具有活血化瘀功效, 同时还是提取色素的原料[1, 2]; 而红花籽油亚油酸含量高达71%~75%[3], 是一种高档的食用油。据报道, 2014年全球红花种植面积约为116.8万公顷, 花冠产量为2.5万吨, 种子产量为176万吨, 可以生产31.6万吨红花籽油[4]。但其产量尚不能满足日益增长的市场需求。新疆是我国红花药材的主产区, 其产量也一直偏低, 每公顷产量约为180~225 kg。众所周知, 花冠的长度是影响红花药材产量的一个重要因素, 因此, 挖掘与花冠伸长相关的基因, 研究其特征和功能对于提高红花药材的产量具有重要意义。
据报道, 在棉纤维细胞伸长过程中, 其长度通常会伸长约1 000倍[5]。而棉纤维细胞的快速伸长是由胀压驱动的并且受到植物细胞壁的限制[6]。植物细胞壁为植物细胞提供支持和保护作用, 主要由纤维素、半纤维素和果胶多糖组成。其中半纤维素是植物细胞壁的基质多糖, 负责构建细胞壁、调节细胞生长过程[7]。植物细胞壁受到多种因素的调节如转录因子等[8, 9], 且现有研究认为木葡聚糖内切转葡糖基/水解酶基因(xyloglucan endotransglycosylases/hydrolases, XTHs)和膨胀素(expansin, EXPs)是两类直接参与细胞壁松弛与重构的基因[10, 11]。这两类基因在植物种子萌发、根系建成、花和果实的发育中具有重要的作用。尤其是在花的发育过程中, 一些EXPs可能参与花原基的形成和花瓣开放过程中的快速伸长阶段。Zenoni等[12]发现PhEXP1基因在花开放过程中高表达, 进一步的功能研究发现PhEXP1反义植株在花瓣区域表现出较小的花瓣细胞。同时在Dianthus caryophyllus[13]和Cymbidium ensifolium[14]中也发现了类似的结果。最近在菊花上进行的研究发现, CmXTH1/2/3/4基因参与菊花花序的开放和花瓣伸长过程[15]。上述研究表明, XTHs和EXPs基因在花发育过程中具有重要作用, 其可能在药用植物特别是花类药用植物育种上具有较高价值。因此, 本研究从课题组构建的红花花冠转录组数据库筛选XTHs和EXPs基因, 通过PEARSON相关分析、生物信息学分析以及表达模式分析筛选出与红花花冠伸长相关的基因, 并通过过表达的红花遗传转化对其进行了功能验证。
材料与方法材料 实验所用红花为云南巍山红花ZHH0119品系, 由第二军医大学生药学教研室郭美丽教授鉴定。所有红花均种植于第二军医大学药学院红花专用温室, 25 ℃、16 h光照8 h黑暗, 常规水肥管理。实验涉及的花及其他组织材料采集后立即在液氮中速冻, -80 ℃保存。
基因筛选 在前期构建的红花花冠转录组数据库[16]中筛选XTHs和EXPs基因, 基于花冠表达谱芯片数据和花冠长度的相关分析进行筛选, 并通过qRT-PCR对其进行验证。qRT-PCR操作步骤如下:采集不同花冠发育期红花花冠组织样品, 使用TransZol Up Plus RNA Kit (Transgen Biotech, Beijing, China)提取红花总RNA, 使用TransScript®One-Step gDNA Removal and cDNA Synthesis SuperMix (TransGen Biotech, Beijing, China)合成红花cDNA。使用ABI 7500 (ABI, USA), TransStart Top Green qPCR SuperMix (+ Dye II) (TransGen Biotech, Beijing, China)进行qRT-PCR。反应体系按产品说明书进行, 反应条件为95 ℃预变性30 s, 95 ℃变性10 s, 58 ℃退火15 s, 72 ℃延伸30 s, 进行30个循环, 60 ℃退火1 min, 95 ℃解链30 s, 在72 ℃及最后95 ℃解链时采集荧光信号。在qRT-PCR之前通过普通PCR确认引物的特异性, 60S (60S酸性核糖体蛋白)作为内参基因[17]。使用ΔCt方法计算基因的相对表达水平, 重复3次计算标准偏差。
全长克隆 基于红花花冠转录组数据库中的相关基因序列设计基因特异性引物, 用Clontech SmartTM RACE cDNA试剂盒(Clontech, USA)通过RACE法克隆5'和3'未知序列。PCR产物在1.5%琼脂糖凝胶(120 V, 200 mA, 1 h)中电泳, 用QIAquick® Gel Extraction Kit (Qiagen, Germany)回收目标条带, 并将其克隆至pMD-19T载体(TaKaRa, Dalian, China)中进行测序。根据测序结果进行拼接并据此设计全长引物, 以红花花冠cDNA为模板, 使用高保真KOD-Plus-Neo聚合酶(Toyobo, 日本)进行全长扩增。PCR产物经1.2%琼脂糖凝胶电泳后(120 V, 200 mA, 1 h), 切取目的条带胶块进行胶回收。胶回收产物克隆至pEASY-Blunt Zero平末端载体(pEASY-Blunt Zero Cloning Kit, TransGen, China)中进行测序。
生物信息学分析 获得XTHs和EXPs基因全长后, 对获得的基因序列进行系统的生物信息学分析。首先, 使用Open Reading Frame Finder (ORF Finder, http://www.ncbi.nlm.nih.gov/gorf/gorf.html/)预测出各基因的开放阅读框; 使用ExPASy服务器上的ExPASyProtParam工具(http://web.expasy.org/compute/)预测理论等电点(pI)、分子量(Mw)和分子式。同时为了解蛋白的潜在功能, 使用Simple Modular Architecture Research Tool (SMART, http://smart.embl-heidelberg.de/)进行蛋白结构功能域分析。使用ProtScale程序(http://us.expasy.org/cgi-bin/protscale.pl)和TMHMM Server v.2.0 (http://www.cbsdtu.dk/services/TMHMM/)分析这些蛋白的疏水性和跨膜区域, 使用SignaIP 4.1程序(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽序列。之后, 使用ClustalX2.1将红花XTHs和EXPs分别与拟南芥XTHs和EXPs进行多序列比对, 并通过MEGA5.0使用邻接法(N-J)构建系统发育树[18], 1 000个自引重复确保发育树的可靠性。最后, 使用PBIL LYON-GERLAND数据库(https://npsaprabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnnhtml), 通过Hopfield Neural Network (HNN)方法预测蛋白二级结构, 并采用线程建模方法, 使用phyre2.0服务器(http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)预测蛋白三级结构[19]。
表达模式分析 为了解与花冠伸长相关基因的表达模式, 在盛花期, 采集了根、茎、叶、苞片、花以及幼嫩种子, 通过qRT-PCR方法测定了各基因在不同器官中的表达量。
红花遗传转化 使用高保真KOD-Plus-Neo (Toyobo, Japan)聚合酶克隆目标基因的CDS区。之后, 通过In-Fusion HD无缝克隆试剂盒(Toyobo, Japan), 将胶回收产物重组至线性化的真核表达载体pMT39, 形成pMT39-XTH重组载体。热激法转化大肠杆菌DH-5α并测序验证后, 使用液氮冻融法, 将验证正确的质粒转化至根癌农杆菌GV3101菌株中备用。按文献报道的方法[16], 使用农杆菌介导的花粉管通道法进行红花遗传转化工作。通过PCR检测阳性转化植株并测序验证。同时, 将pMT39空载体的阳性转化植株作为对照(CK)。
转基因植株CtXTH1基因转录水平检测、农艺性状及显微结构分析 在开花期, 对转基因阳性植株(OVX)和空载对照(CK)植株, 每株选择3个相同部位的花球, 用游标卡尺测量花球直径, 测量其中30个小花的花冠长度, 计数每个花球的小花数并称重。通过qRT-PCR检测CtXTH1的转录水平。待种子成熟后, 计数每个果球的种子数并称重。同时为探究XTHs基因如何影响花冠的发育, 对花冠组织结构进行了显微观察。具体操作如下:在开花期间从收集的花中随机选择3个小花, 使用FAA固定48 h (在此期间更换2~3次FAA固定液)后, 使用酒精进行梯度脱水并采用二甲苯透明。所有样品包埋于蜡块中进行切片, 花冠裂片采用横切, 花冠管状部分采用纵切。脱蜡后, 番红-O染色, 然后使用显微镜(DM2500, Leica, Germany)观察拍照并保存。
UPLC/Q-TOF/MS检测转基因红花指标成分 采用课题组建立的UPLC/Q-TOF/MS法检测过表达植株和空载对照植株花冠中的7种主要黄酮类成分和苯丙烷类代谢途径的起始分子[20]。依次为:芦丁(rutin)、山柰酚(kaempferol)、羟基红花黄色素A (hydroxysafflor yellow A)、山柰酚-3-O-β-芸香糖苷(kaempferol-3-O-β-rutinoside)、山柰酚-3-O-β-D-葡萄糖苷(kaempferol-3-O-β-D-glucoside)、红花黄色素(carthamin)、野黄芩苷(scutellarin)和D-苯丙氨酸(D-phenylalanine)。
数据分析 使用SPSS 21.0 (IBM, New York, USA)对数据进行分析。所有数值均以平均值±标准差或标准误表示。通过独立样本T检验或ANOVA检验处理数据, 显著性水平设定为*P < 0.05, **P < 0.01。
结果 1 基因筛选在红花花冠转录组数据库中, 通过基因注释共筛选出13个XTHs (注: CtXTH1与CtXTH13为同一基因)和8个EXPs基因。根据红花花冠表达谱芯片与花冠长度的Pearson相关分析发现其中有7个XTHs和3个EXPs可能与花冠伸长相关(图 1)。之后通过qRT-PCR进行验证后, 最终筛选出5个XTHs和1个EXP基因。通过RACE法获得基因全长并分别命名为CtXTH1 (GenBank登录号MK103355)、CtXTH2 (GenBank登录号MK103356)、CtXTH3 (GenBank登录号MK103357)、CtXTH4 (GenBank登录号MK103358)和CtEXP1 (GenBank登录号MK103359)。
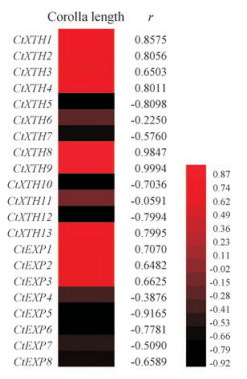
|
Figure 1 Heatmap for Pearson correlation analysis between the signal values by gene chip and corolla length with the floret development. The color key was set from -1 to +1. r≥0.60 |
从红花花冠克隆到的与花冠伸长相关的4个CtXTHs全长为1 107~1 353 bp, 预测的CDS区长度为864~1 050 bp, 编码287~349个氨基酸, 蛋白分子质量为32.6~40.6 kDa, pI值为8.18~8.86, CtXTH1的pI为5.98。CtEXP1全长1 158 bp, 预测的CDS区为780 bp, 编码259个氨基酸, 蛋白分子质量为27.8 kDa, pI值为7.56。具体见表 1。
| Table 1 Gene characteristics of four CtXTHs and CtEXP1 proteins from safflower |
蛋白结构功能域分析显示4个CtXTHs基因都含有Glyco_hydro_16功能域和XET_C功能域, 表明筛选出的CtXTHs基因属于XET亚家族, CtXTH1和CtXTH2含有跨膜区, 推测其属于膜蛋白, CtXTH3和CtXTH4含有信号肽, 推测其属于分泌蛋白。CtEXP1含有DPBB_1和Pfam功能域, 推测其属于花粉过敏原类, 禾本科植物的I组花粉过敏原与膨胀素具有序列同源性, 已被归为α-膨胀素, 因此CtEXP1应属于α-膨胀素基因亚家族。CtEXP1同样含有信号肽, 因此推测其也属于分泌蛋白。根据SMART分析结果, 对信号肽、疏水性和跨膜区进行了额外的预测。结果显示CtXTH3、CtXTH4和CtEXP1具有信号肽, CtXTH1和CtXTH2无信号肽; 且所有基因均在N-末端具有疏水区域, 在C-末端具有亲水区域; 跨膜区分析显示CtXTH1、CtXTH2和CtEXP1均具有TMhelix结构, CtXTH3和CtXTH4仅具有outside结构。二级结构预测显示它们均只包含3种二级结构: α螺旋、无规卷曲和延伸链, 并且无规卷曲的比例 > 50%, 而该结构通常被认为是蛋白分子活性位点的重要构象。通过DNAMAN多序列比对显示了XTHs和EXPs基因家族的保守位点和保守区, 并使用phyre 2.0预测了蛋白三级结构。基于三级结构发现CtXTHs基因具有相对保守的折叠方法, 其蛋白三级结构骨架一致, 同时可识别出典型的双-psiβ-桶(DPBB)结构。系统发育树结果显示(图 2) 4个CtXTHs被划分到3个XTHs基因亚家族[21], CtXTH1和CtXTH3属于亚家族Ⅰ, 与拟南芥AtXTH6、AtXTH7具有高度同源性, 而AtXTH6、AtXTH7在拟南芥花、茎和叶中高表达, 可能参与植株生长的多个阶段, 尤其在花发育中[15, 22, 23]。CtXTH4属于亚家族Ⅱ, 与拟南芥AtXTH15、AtXTH16具有高度同源性, AtXTH15、AtXTH16在拟南芥根、子叶脉和心皮中高表达, 可能与根系发育有关[23]。CtXTH2属于亚家族Ⅲ, 与拟南芥AtXTH29、AtXTH30具有高度同源性, AtXTH29、AtXTH30在拟南芥胚芽、种皮、下胚轴和花药中具有高表达, 推测与花药开裂和花粉释放有关[23-25]。拟南芥AtEXPs基因被划分为3个亚家族[26], CtEXP1属于EXPA亚家族, 与拟南芥AtEXP2、AtEXP8具有高度同源性, AtEXP2、AtEXP8在发芽的种子中高表达, 可能与种子萌发及盐胁迫有关[27, 28]。
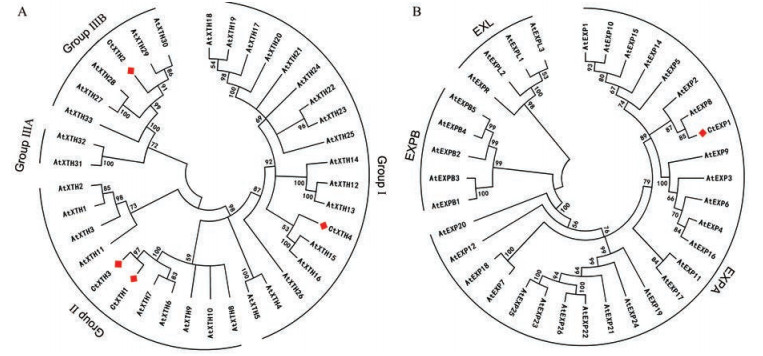
|
Figure 2 A: Phylogenetic tree constructed by the neighbor-joining method using amino acids of XTHs from safflower and Arabidopsis thaliana. B: Phylogenetic tree constructed by the neighbor-joining method using amino acids of EXPs from safflower and Arabidopsis thaliana |
根据生物信息学分析, 认为筛选到的4个CtXTHs和CtEXP1基因功能存在分化, 因此, 进一步对其组织表达特征进行分析。结果表明CtXTHs和CtEXP1在不同组织中存在差异表达, 表现出不同的表达模式(图 3)。CtXTH1在花中表达量最高, 约为根中的73.4倍, 其次在苞片中表达量较高, 约为根中的23.86倍, 而在茎、叶和幼嫩种子中的表达水平与根中相似。CtXTH2在除花和幼嫩种子外的其他部位均有较高表达, 在叶片中表达量最高为根中表达量的3.01倍, 在幼嫩种子中表达量最低, 约为根中表达量的1/45, 花中的表达量约为根中的1/3。对于CtXTH3、CtXTH4以及CtEXP1, 它们在根中均具有较高的表达水平, CtXTH4在花中的表达水平次之, 但仅为根中表达量的1/11。苞片中的表达最低。对于CtXTH3, 苞片和花中的表达小于根中表达量, 约为1/4。在幼嫩种子中, 表达水平最低, 约为根中表达量的1/82。对于CtEXP1, 除了在根中具有最高表达外, 在叶、苞片和花中具有相对较高的表达, 约为根中表达的1/3至1/2, 在幼嫩种子中的表达水平最低, 仅为根中表达水平的1/36。因此, 推测CtXTH1可能在花发育中发挥关键作用, 同样CtXTH4可能在根系发育中起重要作用, CtXTH2可能在营养生长阶段发挥作用, 尤其是在叶片发育过程中, CtXTH3可能在根系发育中起主要作用, 并且可能影响花的发育, 而对于CtEXP1, 其功能类似于CtXTH3。
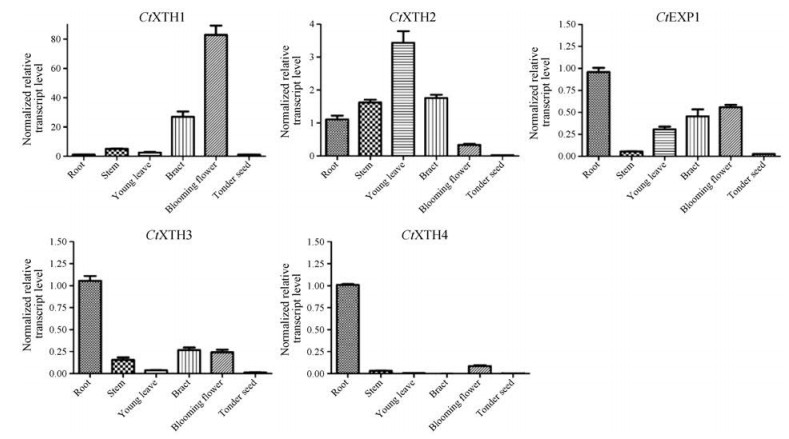
|
Figure 3 The expression patterns of XTHs and EXPs in safflower |
综合考虑Pearson相关分析、生物信息学分析和表达模式分析结果, 认为CtXTH1可能是影响花冠伸长的关键基因。因此将其作为目标基因, 通过红花遗传转化方法进一步探究其功能。
4 转基因红花的鉴定及CtXTH1转录水平测定通过根瘤农杆菌介导的花粉管通道法转化pMT39-CtXTH1载体。采用PCR结合测序从24株潜在个体中筛选出8株独立的阳性转基因植株。之后采用qRT-PCR方法确定各阳性植株与对照组CtXTH1基因的转录水平。根据qRT-PCR结果筛选出5株阳性植株(OVX)和5株对照植株(CK)用于后续分析。如图 4A所示, CtXTH1基因的相对表达量在OVX中发生明显的增加, 约为CK的2~3倍, 说明CtXTH1基因在获得的阳性植株中得到了过表达。
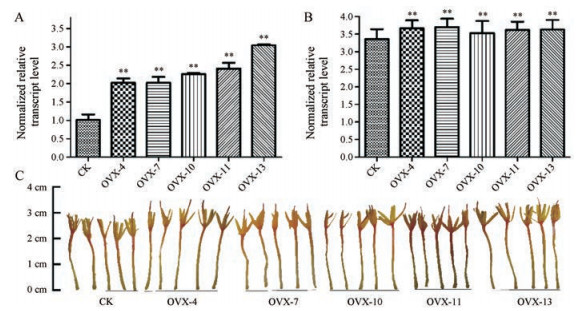
|
Figure 4 A: Transcript levels in negative control (CK) and Contig41 over-expression safflower as determined by qRT-PCR. Each over-expression safflower line vs CK-group, error bar is mean ± SD, **P < 0.01. B: The corolla length in negative control (CK) and Contig41 over-expression safflower. Each over-expression safflower line vs CK-group, error bar is mean ± SD, **P < 0.01. C: The abridged general view of inflorescences in negative control (CK) line and CtXTH1 over-expression safflower lines |
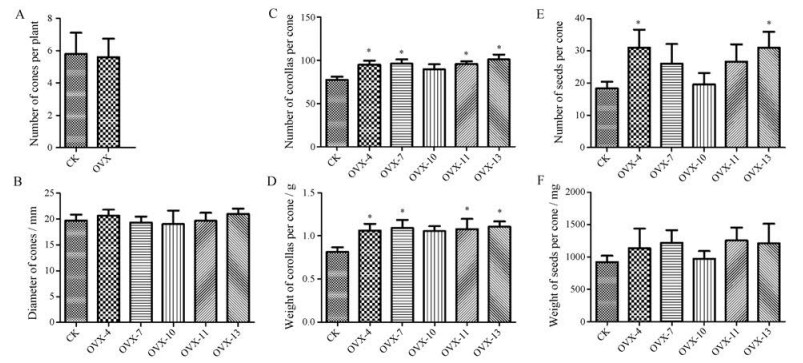
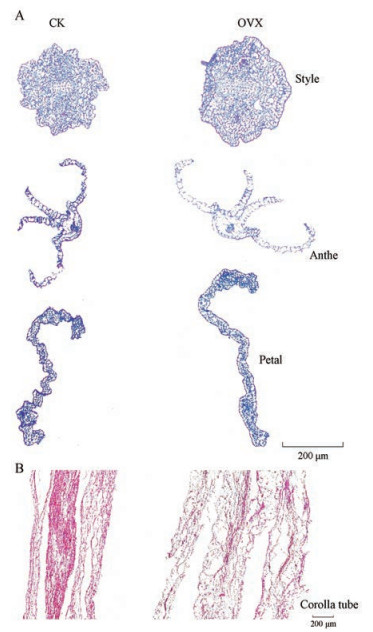
为探究CtXTH1过表达对花和种子的影响, 对过表达组和对照组中的每株花球数、花球直径、花冠长度、小花和种子数量及重量进行了比较。如图 4B、4C, 在过表达组中, 花冠长度发生了显著的伸长, 增长约5.34%~10.25%, 尤其是在OVX-7中, 平均花冠长度为3.70 cm, 比对照组的3.35 cm长约10.25%。对于每株花球数以及花球直径, 两组之间没有显著性差异(图 5A、5B)。对于花, 每花球的平均小花数量与重量均比对照组大, 分别大约15.95%~30.60%和30%~36%, 尤其在OVX-13中, 平均小花数量101, 比对照多30.60%, 小花重量为1.1061 g, 比对照重36%, 除OVX-10外其余各株与对照均显示出显著差异(图 5C、5D)。对于种子, 过表达系在每果球种子数和种子重上均比对照组大, 分别大约7.27%~69%和6.09%~36.53%, 但除了OVX-4, -13在种子数目上与对照组具有显著差异外, 其余均没有显著性差异(图 5E、5F)。在对转基因植株与对照组植株花冠的显微结构观察结果发现, 与对照组相比, 过表达植株花粉囊似乎没有明显变化, 但在花柱及花瓣上均表现出增大的趋势, 并在花冠管状部分的纵切面, 过表达植株组织结构表现出不规则的特征, 并且似乎也更为松散(图 6A、6B)。
|
Figure 5 Flower and fruit traits in negative control (CK) and CtXTH1 over-expression safflower. A: The number of cones per plant in negative control (CK) and CtXTH1 over-expression safflower. B: The diameter of cones in negative control (CK) and CtXTH1 over-expression safflower lines. C and D: The number and weight of corollas per cone in negative control (CK) and CtXTH1 over-expression safflower lines. E and F: The number and weight of seeds per cone in negative control (CK) and CtXTH1 over-expression safflower lines. For A, over-expression safflower group vs CK-group; For B-F, each over-expression safflower line vs CK-group. error bar is mean ± SEM, for CK group SEM means biological duplication, while over-expression safflower lines mean technical duplication, *P < 0.05 |
|
Figure 6 A: Transverse sections of the tissue in negative control (CK) and CtXTH1 over-expression safflower. B: Longitudinal sections of the tissue in negative control (CK) and CtXTH1 over-expression safflower |
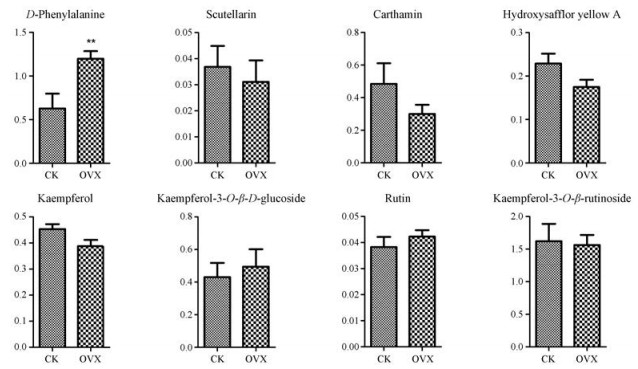
通过UPLC/Q-TOF/MS检测过表达植株和对照植株花冠中的7种主要黄酮类成分和苯丙烷类代谢途径的起始物质D-苯丙氨酸, 发现与对照组相比, 野黄芩苷、红花红色素、羟基红花黄色素A、芦丁、山柰酚、山柰酚-3-O-β-D-葡萄糖苷、山柰酚-3-O-β-芸香糖苷均无显著差异, 但D-苯丙氨酸发生显著积累(图 7)。
|
Figure 7 Levels of flavonoid compounds in negative control (CK) and CtXTH1 over-expression safflower determined by UPLC-MS analysis. The over-expression safflower line vs CK-group, error bar is mean ± SD, **P < 0.01 |
膨胀素和木葡聚糖转糖基/水解酶基因均属于多基因家族。据报道, 在拟南芥中有33个XTHs基因和36个EXPs基因[22, 29, 30], 在水稻中有29个XTHs基因和58个EXPs基因[31, 32], 并且在其他物种中也存在众多XTHs和EXPs。来自多基因家族的基因通常发挥不同的生理功能, 例如: XTHs和EXPs可以促进种子萌发[27, 33, 34], 并且在根系建成[35-37]、植株生长[38-40]以及花和果实的发育中[14, 41-43]发挥作用, 并且最近研究表明, XTHs可能参与非生物胁迫及寄生植物的寄生过程[44, 45]。因此, 认为红花中的某些XTHs或EXPs基因可能参与花冠发育, 其通过松弛和重构花冠细胞壁来影响花冠细胞的大小或数量, 从而促进花冠伸长。正如Shin在对BcXTH1基因功能的研究中发现, BcXTH1可以介导表皮和叶肉细胞的膨大进而引起叶片伸长[46]。本研究中, 基于红花花冠转录组数据库和表达谱芯片数据, 结合qRT-PCR验证, 通过PEARSON相关分析筛选出可能促进花冠伸长的4个CtXTHs和CtEXP1。基于生物信息学分析和表达模式分析, 认为CtXTH1可能是影响花冠伸长的关键基因。之后通过红花过表达遗传转化, 发现CtXTH1过表达能够显著促进花冠的伸长, 其小花数量及重量也均有显著提高, 并且其种子数及种子重也表现出增加的趋势。在对花冠显微结构的观察中发现, 花冠组织变得松散和不规则, 这种现象与DkXTH8过表达后的柿果实表皮细胞变化相似。因此推测CtXTH1与DkXTH8一样都能够引起细胞壁的松弛, 从而使得靶组织增大[47]。同时, 通过对过表达植株花冠主要黄酮类化合物的检测发现, CtXTH1的过表达对红花药材品质没有显著影响。
本研究首次揭示了红花CtXTHs和CtEXP1基因的序列特征和表达模式, 发现CtXTH1在红花花冠中特异性积累, 通过遗传转化实验, 证明了CtXTH1基因在促进红花花冠伸长进而提高花冠以及种子产量方面的重要价值, 为红花育种工作提供了重要线索。
| [1] | Asgarpanah J, Kazemivash N. Phytochemistry, pharmacology and medicinal properties of Carthamus tinctorius L.[J]. Chin J Integr Med, 2013, 19: 153–159. DOI:10.1007/s11655-013-1354-5 |
| [2] | Delshad E, Yousefi M, Sasannezhad P, et al. Medical uses of Carthamus tinctorius L. (safflower): a comprehensive review from traditional medicine to modern medicine[J]. Electron Physician, 2018, 10: 6672–6681. DOI:10.19082/6672 |
| [3] | Gecgel U, Demirci M, Esendal E, et al. Fatty acid composition of the oil from developing seeds of different varieties of safflower (Carthamus tinctorius L.)[J]. J Am Oil Chem Soc, 2007, 84: 47–54. DOI:10.1007/s11746-006-1007-3 |
| [4] | Tian ZM. The current situation, development advantages and countermeasures of Chinese safflower industry[J]. Yunnan Agric Sci Technol (云南农业科技), 2014(4): 57–59. |
| [5] | Lee J, Burns T, Light G, et al. Xyloglucan endotransglycosylase/hydrolase genes in cotton and their role in fiber elongation[J]. Planta, 2010, 232: 1191–1205. DOI:10.1007/s00425-010-1246-2 |
| [6] | Shao MY, Wang XD, Ni M, et al. Regulation of cotton fiber elongation by xyloglucan endotransglycosylase/hydrolase genes[J]. Gen Mol Res, 2011, 10: 3771–3782. DOI:10.4238/2011.October.27.1 |
| [7] | Li XB, Zhang JZ. Chemical structure and physiological function of hemicellulose[J]. Chin Bull Bot (植物学报), 1994, 11: 27–33. |
| [8] | Ferreira SDS, Hotta C, Poelking VGDC, et al. Co-expression network analysis reveals transcription factors associated to cell wall biosynthesis in sugarcane[J]. Plant Mol Biol, 2016, 91: 15–35. DOI:10.1007/s11103-016-0434-2 |
| [9] | Gruber M, Alahakoon A, Taheri A, et al. The biochemical composition and transcriptome of cotyledons from Brassica napus L. lines expressing the AtGL3 transcription factor and exhibiting reduced flea beetle feeding[J]. BMC Plant Biol, 2018, 18: 64–65. DOI:10.1186/s12870-018-1277-6 |
| [10] | Cosgrove DJ. New genes and new biological roles for expansins[J]. Curr Opin Plant Biol, 2000, 3: 73–78. DOI:10.1016/S1369-5266(99)00039-4 |
| [11] | Fry SC. Polysaccharide-modifying enzymes in the plant cell wall[J]. Ann Rev Plant Biol, 2003, 46: 497–520. |
| [12] | Zenoni S, Reale L, Tornielli GB, et al. Downregulation of the Petunia hybrida V. alpha-expansin gene PhEXP1 reduces the amount of crystalline cellulose in cell walls and leads to phenotypic changes in petal limbs[J]. Plant Cell, 2004, 16: 295–308. DOI:10.1105/tpc.018705 |
| [13] | Harada T, Torii Y, Morita S, et al. Cloning, characterization and expression of xyloglucan endotransglucosylase/hydrolase and expansin genes associated with petal growth and development during carnation flower opening[J]. J Exp Bot, 2011, 62: 815–823. DOI:10.1093/jxb/erq319 |
| [14] | Li XB, Xu WW, Chowdhury MR, et al. Comparative proteomic analysis of labellum and inner lateral petals in Cymbidium ensifolium flowers[J]. Int J Mol Sci, 2014, 15: 19877–19897. DOI:10.3390/ijms151119877 |
| [15] | Li Z, Xia WS, Hong R, et al. Cloning and functional verification of chrysanthemum CmXTHs genes related to petal elongation[J]. Acta Hort Sin (园艺学报), 2017, 11: 2150–2162. |
| [16] | Guo DD, Xue YR, Li DQ, et al. Overexpression of CtCHS1 increases accumulation of quinochalcone in safflower[J]. Front Plant Sci, 2017, 8: 1409. DOI:10.3389/fpls.2017.01409 |
| [17] | Liu F. Cloning and Functional Verification of Key Enzyme Genes in the Biosynthesis Pathway of Safflower Flavonoids (红花黄酮类化合物生物合成途径关键酶基因的克隆与功能验证) [D]. Shanghai: The Second Military Medical University (第二军医大学), 2014. http://med.wanfangdata.com.cn/Paper/Detail?id=DegreePaper_Y2630505 |
| [18] | Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731–2739. DOI:10.1093/molbev/msr121 |
| [19] | Kelley LA, Mezulis S, Yates CM, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nat Protoc, 2015, 10: 845–858. DOI:10.1038/nprot.2015.053 |
| [20] | He BX, Xue YR, Tu YH, et al. CtCHS4 induces the accumulation of safflower quinone chalcones in response to methyl jasmonate induction[J]. Acta Pharm Sin (药学学报), 2018, 53: 636–645. |
| [21] | Baumann MJ, Eklf JM, Michel G, et al. Structural evidence for the evolution of xyloglucanase activity from xyloglucan endo-transglycosylases: biological implications for cell wall metabolism[J]. Plant Cell, 2007, 19: 1947–1963. DOI:10.1105/tpc.107.051391 |
| [22] | Yokoyama R, Nishitani K. A comprehensive expression analysis of all members of a gene family encoding cell-wall enzymes allowed us to predict cis-regulatory regions involved in cell-wall construction in specific organs of arabidopsis[J]. Plant Cell Physiol, 2001, 42: 1025–1033. DOI:10.1093/pcp/pce154 |
| [23] | Becnel J, Natarajan M, Kipp A, et al. Developmental expression patterns of arabidopsis XTH genes reported by transgenes and genevestigator[J]. Plant Mol Biol, 2006, 61: 451–467. DOI:10.1007/s11103-006-0021-z |
| [24] | Keijzer CJ. The process of anther dehiscence and pollen dispersal. Part Ⅱ[J]. New Phytol, 1987, 3: 499–507. |
| [25] | Sanders PM, Bui AQ, Weterings K, et al. Anther developmental defects in Arabidopsis thaliana male-sterile mutants[J]. Sex Plant Reprod, 1999, 11: 297–322. DOI:10.1007/s004970050158 |
| [26] | Lee Y, Choi D, Kende H. Expansins: ever-expanding numbers and functions[J]. Curr Opin Plant Biol, 2001, 4: 527–532. DOI:10.1016/S1369-5266(00)00211-9 |
| [27] | Yan A, Wu MJ, Yan LM, et al. AtEXP2 is involved in seed germination and abiotic stress response in arabidopsis[J]. PLoS One, 2014, 9: e85208. DOI:10.1371/journal.pone.0085208 |
| [28] | Nardi CF, Villarreal N, Rossi F, et al. Overexpression of the carbohydrate binding module of strawberry expansin in Arabidopsis thaliana modifies plant growth and cell wall metabolism[J]. Plant Mol Biol, 2015, 88: 101–117. DOI:10.1007/s11103-015-0311-4 |
| [29] | Mayer K, Schller C, Wambutt R, et al. Sequence and analysis of chromosome 4 of the plant Arabidopsis thaliana[J]. Nature, 1999, 402: 769–777. DOI:10.1038/47134 |
| [30] | Carey RE, Hepler N, Cosgrove DJ. Selaginella moellendorffii has a reduced and highly conserved expansin superfamily with genes more closely related to angiosperms than to bryophytes[J]. BMC Plant Biol, 2013, 13: 4. DOI:10.1186/1471-2229-13-4 |
| [31] | Yokoyama R, Rose JKC, Nishitani K. A surprising diversity and abundance of xyloglucan endotransglucosylase/hydrolases in rice classification and expression analysis[J]. Plant Physiol, 2004, 134: 1088–1099. DOI:10.1104/pp.103.035261 |
| [32] | Sampedro J, Cosgrove DJ. The expansin superfamily[J]. Gen Biol, 2005, 6: 242–253. DOI:10.1186/gb-2005-6-12-242 |
| [33] | Kaku T, Tabuchi A, Wakabayashi K, et al. Xyloglucan oligosaccharides cause cell wall loosening by enhancing xyloglucan endotransglucosylase/hydrolase activity in azuki bean epicotyls[J]. Plant Cell Physiol, 2004, 45: 77–82. DOI:10.1093/pcp/pch007 |
| [34] | Miedes E, Zarra I, Hoson T, et al. Xyloglucan endotransglucosylase and cell wall extensibility[J]. J Plant Physiol, 2011, 168: 196–203. DOI:10.1016/j.jplph.2010.06.029 |
| [35] | Maris A, Suslov D, Fry SC, et al. Enzymic characterization of two recombinant xyloglucan endotransglucosylase/hydrolase (XTH) proteins of arabidopsis and their effect on root growth and cell wall extension[J]. J Exp Bot, 2009, 60: 3959–3972. DOI:10.1093/jxb/erp229 |
| [36] | Soltys-Kalina D, Rudzińska-Langwald A, Gniazdowska A, et al. Inhibition of tomato (Solanum lycopersicum L.) root growth by cyanamide is due to altered cell division, phytohormone balance and expansin gene expression[J]. Planta, 2012, 236: 1629–1638. DOI:10.1007/s00425-012-1722-y |
| [37] | Xu P, Cai XT, Wang Y, et al. HDG11 upregulates cell-wall-loosening protein genes to promote root elongation in arabidopsis[J]. J Exp Bot, 2014, 65: 4285–4295. DOI:10.1093/jxb/eru202 |
| [38] | Choi D, Lee Y, Cho HT, et al. Regulation of expansin gene expression affects growth and development in transgenic rice plants[J]. Plant Cell, 2003, 15: 1386–1398. DOI:10.1105/tpc.011965 |
| [39] | Matsui A, Yokoyama R, Seki M, et al. AtXTH27 plays an essential role in cell wall modification during the development of tracheary elements[J]. Plant J, 2005, 42: 525–534. DOI:10.1111/tpj.2005.42.issue-4 |
| [40] | Han Y, Han SK, Ban QY, et al. Overexpression of persimmon DkXTH1 enhanced tolerance to abiotic stress and delayed fruit softening in transgenic plants[J]. Plant Cell Rep, 2017, 36: 583–596. DOI:10.1007/s00299-017-2105-4 |
| [41] | Bae JM, Kwak MS, Noh SA, et al. Overexpression of sweetpotato expansin cDNA (IbEXP1) increases seed yield in arabidopsis[J]. Transgenic Res, 2014, 23: 657–667. DOI:10.1007/s11248-014-9804-1 |
| [42] | Li WC, Wu JY, Zhang HN, et al. De novo assembly and characterization of pericarp transcriptome and identification of candidate genes mediating fruit cracking in Litchi chinensis Sonn[J]. Int J Mol Sci, 2014, 15: 17667–17685. DOI:10.3390/ijms151017667 |
| [43] | Zhang ZY, Wang N, Jiang SH, et al. Analysis of the xyloglucan endotransglucosylase/hydrolase gene family during apple fruit ripening and softening[J]. J Agric Food Chem, 2017, 65: 429–434. DOI:10.1021/acs.jafc.6b04536 |
| [44] | Olsen S, Popper Z, Krause K. Two sides of the same coin: xyloglucan endotransglucosylases/hydrolases in host infection by the parasitic plant cuscuta[J]. Plant Signal Behav, 2016, 11: e1145336. DOI:10.1080/15592324.2016.1145336 |
| [45] | Olsen S, Striberny B, Hollmann J, et al. Getting ready for host invasion: elevated expression and action of xyloglucan endotransglucosylases/hydrolases in developing haustoria of the holoparasitic angiosperm cuscuta[J]. J Exp Bot, 2015, 258: 193–204. |
| [46] | Shin YK, Yum H, Kim ES, et al. BcXTH1, a brassica campestris homologue of arabidopsis XTH9, is associated with cell expansion[J]. Planta, 2005, 224: 32–41. |
| [47] | Han Y, Ban QY, Li H, et al. DkXTH8, a novel xyloglucan endotransglucosylase/hydrolase in persimmon, alters cell wall structure and promotes leaf senescence and fruit postharvest softening[J]. Sci Rep, 2016, 6: 39155–39170. DOI:10.1038/srep39155 |
 2019, Vol. 54
2019, Vol. 54


