肺癌是世界范围内发病率和死亡率排名首位的恶性肿瘤, 主要包括非小细胞肺癌(non-small cell lung cancer, NSCLC)和小细胞肺癌(small cell lung cancer, SCLC), 其中非小细胞肺癌所占比例约为80%~85%[1]。多数肺癌患者起病隐匿, 临床确诊时常常已处于中晚期, 失去了根治性切除的机会。尽管近年来肺癌的诊疗在早期筛查、微创手术及靶向治疗等方面已取得一定进展, 然而肺癌5年的相对存活率仍然只有16.6%, 其中远处转移患者5年存活率仅有3.9%[2]。
肺癌的发生发展涉及多种基因的改变, 特别是一些促生长因子的激活和肿瘤抑制子的抑制[3, 4]。如活化的转化生长因子-β1 (transforming growth factor-β1, TGF-β1)不仅促进肿瘤的血管生成, 而且对肺癌患者的肿瘤进展和转移具有重要的促进作用[5]; 肺癌肿瘤抑制物1 (tumor suppressor in lung cancer 1, TSLC1)是最近发现的一种抑癌基因, 它的缺失、低表达或抑制其表达等促进了肺癌的发生、侵袭和转移[6]。三结构域蛋白家族(tripartite motif, TRIM)是一类重要的E3泛素连接酶, 参与细胞增殖、天然免疫、细胞分化及细胞凋亡等多种生物学功能的调节。雌激素反应指蛋白(estrogen-responsive finger protein, EFP), 又名tripartite motif 25 (TRIM25), 是TRIM家族成员之一, 在早期筛选雌激素反应基因时被发现命名[7]。早期的研究主要集中于TRIM25作为E3泛素连接酶, 通过介导多种蛋白质如RIG-I[8]、KLF5[9]和ATBF1[10]的泛素化降解过程, 参与细胞生物功能的调节。近年来研究发现TRIM25不仅作为免疫细胞感应器, 参与先天性免疫调节, 同时也是一种促癌蛋白, 促进乳腺癌[11]、卵巢癌[12]、胃癌[13]和肺癌[14]等多种肿瘤的发生发展。尽管研究发现肺癌组织中高表达的TRIM25促进了肺癌的发展进程, 然而有关TRIM25调节肺癌恶性进展的分子机制尚不明确。
表皮生长因子受体(epidermal growth factor receptor, EGFR)是一种受体酪氨酸激酶, 与配体结合被激活后可以活化下游多种信号通路如MAPK/ERK信号通路、PI3K/Akt信号通路、STAT3信号通路等, 与肺癌增殖能力强、分化能力差、淋巴结转移率高、预后不良等密切相关[15-17]。大量研究发现40%~80%的NSCLC患者中具有EGFR异常高表达或信号过度活化的现象[18]。EGFR过表达不仅导致对靶向EGFR药物的耐受[19-21], 也导致肿瘤对多种化疗药物的耐受[22, 23]。在很多情况下, EGFR的表达量与EGFR的基因拷贝数并不直接相关, 表明EGFR的过表达可能是由于EGFR蛋白降解调节异常所致。阐明EGFR降解机制, 将有助于寻找新的靶向分子, 制定新的治疗策略, 为新型靶向药物的研发提供重要的理论依据。
通过基因集富集分析(gene set enrichment analysis, GSEA), 本课题组发现在TRIM25高表达的肺癌患者中, EGFR信号明显富集。基于此, 猜想在肺癌肿瘤组织中, TRIM25与促癌蛋白EGFR可能存在一定的联系, TRIM25可能通过调节EGFR信号通路, 发挥促肿瘤功能。迄今为止, 未有文献报道TRIM25与EGFR之间的关系。本研究拟探寻TRIM25、EGFR和肺癌恶性进展之间的联系, 阐明TRIM25促进肺癌发生发展的作用和机制。
材料与方法试剂和抗体 人表皮生长因子(epidermal growth factor, EGF)购于PeproTech公司; 放线菌酮(cycloheximide, CHX)购于Sigma Aldrich公司; 胰酶、胎牛血清、RPMI-1640培养基、IMDM培养基购于Gibco公司; RIPA细胞裂解液、BCA蛋白浓度测定试剂盒均购自北京索莱宝科技有限公司; 转染试剂VigoFect购于北京威格拉斯生物技术有限公司; Western blot超敏发光液购自北京普利莱基因技术有限公司。TransZol Up、EasyScript One-Step gDNA Removal和cDNA Synthesis Super Mix购自北京全式金生物技术有限公司。所用抗体: pERK (ERK1/2, Thr202/Tyr204)、ERK (ERK1/2)、pAkt (Ser473)、Akt、pEGFR (Tyr1068)、EGFR、pSTAT5 (Tyr694)、STAT5和TRIM25均购于Cell Signaling Technology公司; DDK、Myc、GFP抗体均购于北京博尔迈生物技术有限公司; GAPDH、HRP标记二抗购于中杉金桥生物技术有限公司。
细胞及细胞培养 肺癌细胞系A549、H1975、PC-9购自中国医学科学院基础医学研究所细胞中心; HEK 293T细胞由本实验室长期保存。A549、H1975和PC-9细胞培养于含10%胎牛血清(FBS)的RPMI-1640培养基中; HEK 293T细胞培养于含10% FBS的IMDM培养基中, 所有细胞置于37 ℃、5% CO2的孵箱中培养, 根据细胞生长状态进行传代。
质粒 pcDNA6A-EGFR-MYC质粒由本实验室前期保存; 表达质粒TRIM25-DDK、UB-GFP购于北京义翘神州科技有限公司。
人肺癌组织 人肺癌组织样本来源于中国医学科学院肿瘤医院的肺癌患者, 根据《赫尔辛基宣言》, 已取得所有肺癌患者的知情同意, 并得到了中国医学科学院肿瘤医院伦理委员会的批准。
主要仪器 电泳仪(北京六一仪器厂)、qPCR仪(美国BIO-RAD公司)、ImageQuant LAS 4000mini成像系统(美国通用电气公司)、激光扫描共聚焦显微镜(日本奥林巴斯公司)。
实时荧光定量PCR (real-time PCR)检测
细胞总RNA提取用PBS溶液洗涤细胞后, 将TransZol Up加入到细胞中, 裂解并收集细胞, 向裂解液中加入氯仿涡旋混匀, 静置5 min, 4 ℃、10 000 ×g离心15 min, 取上清液置于新的离心管中, 并加入一定比例的异丙醇, 颠倒混匀, 静置10 min后4 ℃、10 000 ×g离心10 min, 弃去上清, 加入一定量的75%乙醇, 4 ℃、7 500 ×g离心5 min, 弃上清室温晾干后加入RNA溶解酶。
RNA逆转录成cDNA按照逆转录试剂盒说明书将RNA逆转录成cDNA。引物由睿博兴科生物技术有限公司合成, 序列如下所示: EGFR F: 5'-GACAGG CCACCTCGTCG-3'; EGFR R: 5'-TGCGTGAGCTTGT TACTCGT-3'; TRIM25 F: 5'- AGAGCCTGACCAAGA GGGAT- 3'; TRIM25 R: 5' - GTGGATTTGTGTGTGGA CGC - 3'; Actin F: 5' - AGAGCTACGAGCTGCCTGAC - 3'; Actin R: 5'-AGCACTGTGTTGGCGTACAG-3'。
Q-PCR反应反应体系(20 μL)如下: SYBR PremixExTaqTM 10 μL, 去离子水6 μL, 上下游引物各1 μL, 模板cDNA 2 μL。两步法反应过程如下: 95 ℃预变性30 s, 进入循环, 95 ℃变性5 s, 60 ℃退火和延伸30 s, 循环次数40次。以Actin作为内参, 将所得Ct值按照2-ΔΔCt的方的方法进行处理。
Western blot (WB) 收集细胞, 加入裂解液, 置于冰上裂解30 min, 4 ℃、12 000 r · min-1离心30 min, 收集上清液, 测定其蛋白浓度并进行定量处理, 加入5×上样缓冲液, 98 ℃变性10 min。蛋白样品进行聚丙烯酰胺凝胶电泳, 湿法转膜, 5%脱脂牛奶室温封闭0. 5 h, 4 ℃一抗孵育过夜, 次日, 用TBST洗膜液洗涤3次, 每次10 min, 加入相应二抗室温孵育2 h, 洗膜3次后加入ECL solution进行显影。
RNAi实验 TRIM25 siRNA由广州锐博生物科技有限公司合成, 序列如下: si-h-TRIM25_001, GCAA ATGTTCCCAGCACAA; si-h-TRIM25_002, CCTGAGC TCCTGGAGTATT。将适量细胞接种于6孔板中, 待其密度合适时, 将TRIM25 siRNA稀释于100 μL的RPMI-1640中, 轻轻混匀; 另外, 将Lipofectamine RNAi MAX2 μL稀释于100 μL的RPMI-1640中, 室温放置5 min后, 将含有Lipofectamine RNAi MAX的培养基滴加到含有siRNA的培养基中, 轻轻混匀, 室温孵育5 min后滴加到细胞中。
免疫荧光染色 将细胞接种于预先铺有盖玻片的12孔板中, 待细胞密度合适时, 用PBS清洗细胞, 4%多聚甲醛固定细胞20 min, PBS清洗3次, 0.5% Triton X-100透化20 min, PBS清洗3次, 3% BSA室温封闭0.5 h, 加入一抗(1:100稀释), 4 ℃孵育过夜, PBS清洗细胞3次, 每次3 min, 加入对应的二抗(1:200稀释), 室温避光孵育2 h, PBS清洗细胞3次, 每次3 min, 用含有DAPI的封片剂进行封片, 荧光显微镜观察图像并拍照。
免疫组化染色 将病理石蜡切片放于60 ℃孵箱烘烤1 h, 冷却至室温后进行以下流程:二甲苯30 min, 二甲苯30 min, 100%乙醇3 min, 90%乙醇3 min, 80%乙醇3 min, 70%乙醇3 min, 一相水3 min; 将石蜡切片浸入枸椽酸抗原修复液中, 进行微波抗原修复; 之后冷却至室温后, 一相水5 min, PBS 5 min, 0.5% Triton透化20 min, 过氧化氢孵育15 min, 封闭液孵育30 min, 滴加一抗4℃孵育过夜; 次日, 用PBS清洗3次后滴加相应的二抗, 室温孵育2 h, PBS清洗3次。DAB显色, 一相水冲洗, 苏木素染色数分钟, 过1%盐酸酒精, 流水返蓝15 min后, 70%乙醇3 min, 80%乙醇3 min, 90%乙醇3 min, 二甲苯15 min, 二甲苯15 min进行脱水, 中性树胶封片。
免疫共沉淀实验(Co-immunoprecipitation, CO-IP) 待细胞生长到70%的密度时进行质粒转染, 质粒转染24~48 h后收集细胞, 加入细胞裂解液(含蛋白酶抑制剂), 冰上裂解30 min, 4 ℃、12 000 r·min-1离心30 min, 将上清转移至新的1.5 mL离心管中。吸取少量的裂解液用于Western blot分析, 剩余的裂解液加入3 μg的抗体和20 μL protein A琼脂糖珠, 4 ℃缓慢摇晃孵育过夜, 使抗体与protein A琼脂糖珠偶连。次日, 用缓冲洗液清洗4次, 每次3 000 r · min-1离心5 min。加入2×上样缓冲液, 98 ℃变性10 min, 样品进行Western blot分析。
CCK-8检测细胞活力 收集对数期生长的A549-Control siRNA和A549-TRIM25 siRNA细胞, 对其进行计数后按3×103个细胞/孔接种于96孔培养板中, 置于37 ℃、5% CO2的孵箱中培养72 h后, 弃去原培养基, 每孔加入混合有10 μL CCK-8和90 μL完全培养基的混合液, 置于培养箱中孵育1 h, 酶标仪检测450 nm波长下的吸光度值(A), 计算细胞活力。
Transwell细胞侵袭实验 用培养基稀释2μg·μL-1的Fibronectin母液, 均匀混合, 将30 μL Fibronectin工作液均匀地涂到小室外侧晾干; 用培养基稀释50 mg·L-1的Matrigel, 并将Matrigel工作液均匀地滴加至Transwell小室内, 小室晾干后加入无血清的培养基水化30 min; 用胰酶消化收集细胞后PBS清洗1~2次, 用无血清的培养基重悬细胞并进行计数; 将适量细胞接种于Transwell小室内, Transwell小室放置于含有完全培养基的24孔板中, 置于细胞培养箱中培养; 培养24 h后, 弃去Transwell小室内的培养基, 用PBS清洗小室外侧, 放入4%多聚甲醛中固定20 min, PBS清洗3次, 结晶紫染色30 min; 棉签擦去小室内部细胞, PBS清洗, 置于倒置显微镜下观察图像并拍照。
GSEA分析 从http://xena.ucsc.edu/网站下载肺腺癌TCGA数据LUAD HiSeqV2_PANCAN (n = 576)。按照TRIM25表达高低进行排序, 前20%患者(n = 116)和后20% (n = 116)分别被认为是TRIM25高表达组和TRIM25低表达组。从http://software.broadinstitute.org/gsea/index.jsp网站下载EGFR (BIOCARTA_EGF_ PATHWAY)、RAS (BIOCARTA_RAS_PATHWAY)和ALK (BIOCARTA_ALK_PATHWAY)基因集。运用GSEA软件分别对TRIM25高表达组和低表达组中EGFR、RAS、ALK信号集进行富集分析。其中FDR值(false discovery rate)小于0.25被视为具有显著性差异。
统计学分析 实验平均重复3次以上, 数据用mean ± SEM表示, 使用Graphpad Prism 5统计分析软件进行数据统计和计算, 用t检验各组间的差异, 组中的数据比较采用单因素方差分析(one-way ANOVA), 显著性结果以P < 0.01 或P < 0.001 表示。
结果 1 高表达的TRIM25与肺癌不良预后正相关为了探究TRIM25的表达水平与肺癌患者不良预后之间的关系, 本研究首先利用R2: Genomics Analysisand Visualization Platform (http://r2.amc.nl)进行生存率分析, 结果发现低表达TRIM25的肺癌患者生存期明显高于高表达TRIM25的患者(图 1A)。免疫印迹检测肺癌患者中肿瘤组织和癌旁组织中TRIM25蛋白表达水平, 结果显示肿瘤组织中TRIM25的表达水平显著高于癌旁组织(图 1B)。利用TRIM25特异性siRNA敲低肺癌细胞A549中的TRIM25, 结果发现, 与对照组相比, 敲低TRIM25不仅抑制肿瘤细胞增殖, 其抑制率为34% (图 1C); 而且降低了肿瘤细胞的侵袭能力, 其抑制率为42% (图 1D)。以上结果表明, 肺癌组织中高表达的TRIM25, 促进了肺癌细胞的增殖和侵袭, 并与肺癌不良预后正相关。
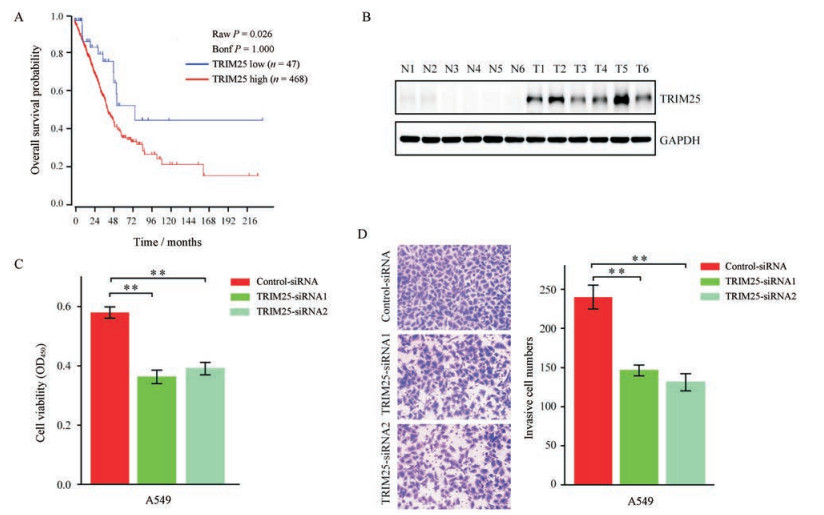
|
Figure 1 Tripartite motif 25 (TRIM25) positively correlates with poor survival of lung cancer patients and promotes tumor proliferationand invasion. A: Overall survival of patients with lung cancer stratified by R2: Genomics Analysis and Visualization Platform (http://r2.amc.nl) according to TRIM25 expression level. Patients were divided into 2 groups: high TRIM25 expression vs low TRIM25 expression; B: Western blot (WB) analyses of TRIM25 expression in human lung cancer tissues (T) or in the adjacent normal tissues (N); C: Cell proliferation was measured by CCK-8 assay in A549 cells transiently transfected with control-siRNA or TRIM25-siRNAs; D: The invasive capacities of A549 cells transfected with control-siRNA or TRIM25-siRNAs were evaluated with Transwell assays. n = 3, mean ± SEM. **P < 0.01 |
鼠类肉瘤病毒癌基因(kirsten rat sarcoma viral oncogene homologue, KRAS)突变、EGFR突变和棘皮动物微管相关蛋白4-间变性淋巴瘤激酶(echinoderm microtubule-associate protein-like 4 - anaplastic lymphoma kinase, EML4-ALK)融合是肺癌发病过程中最常见的几个驱动基因。基于TCGA肺癌患者数据库, 课题组利用GSEA方法分析了TRIM25与这几个驱动基因之间的关系, 结果显示高表达TRIM25时EGFR信号显著富集(图 2A); 但这种现象在RAS和ALK信号通路中并不存在(图 2B、C)。采用免疫印迹对肺癌患者肿瘤组织中TRIM25和EGFR表达情况进行检测, 结果表明高表达TRIM25的患者, 其肺癌组织中EGFR的表达量也高(图 2D)。免疫组化检测结果进一步印证肺癌组织中TRIM25与EGFR蛋白表达水平具有良好的正相关性(图 2E、F)。以上结果表明, 肺癌组织中TRIM25和EGFR呈正相关, 这提示TRIM25可能通过调节EGFR或EGFR信号通路发挥促肿瘤功能。
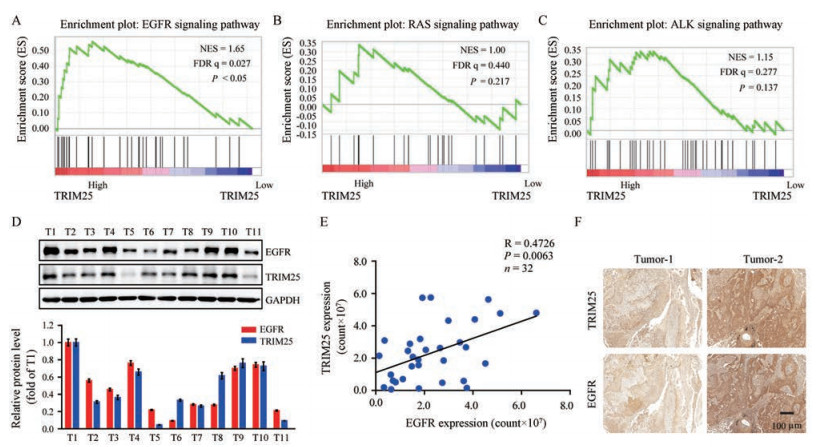
|
Figure 2 TRIM25 positively correlates with EGFR signaling and expression in lung cancer. A-C: GSEA enrichment plot for the " EGFRsignaling" , " RAS signaling" and " ALK signaling" gene modules in TRIM25-high or TRIM25-low groups; D: WB analyses of TRIM25 andEGFR expression in human lung cancer tissues; E: Correlation between TRIM25 and EGFR expression in lung cancer patients. The P valueis measured by Spearman's rank correlation test; F: Representatives of immunohistochemical staining of TRIM25 or EGFR expression intumor tissue sections from the same patients. EGFR: Epidermal growth factor receptor; RAS: Rat sarcoma; ALK: Anaplastic lymphomakinase; FDR q: False discovery rate q value; NES: Normalized enrichment score. n = 3, mean ± SEM |
为了验证TRIM25是否参与EGFR的调节, 本研究利用TRIM25特异性siRNA敲低肺癌细胞系A549中的TRIM25, 结果显示, 敲低TRIM25降低了EGFR表达量; 在H1975、PC-9等肺癌细胞系中也得到了一致的结果(图 3A)。此外, 与对照组相比, 敲低肺癌细胞中TRIM25后, EGF诱导的EGFR、ERK以及STAT5的磷酸化水平也受到抑制(图 3B)。以上结果表明, 敲低肺癌细胞中的TRIM25, 不仅下调了EGFR的表达水平, 而且也抑制了EGFR下游信号活性。
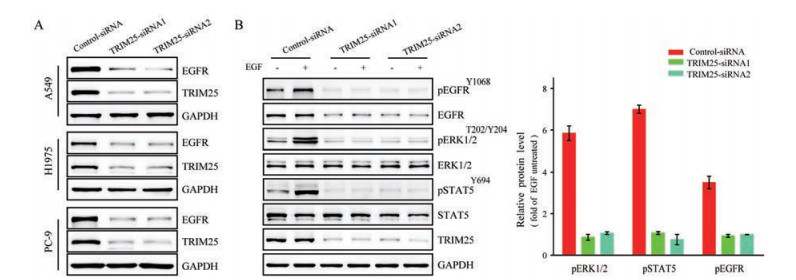
|
Figure 3 Knockdown of TRIM25 decreased EGFR expression and downstream signal activation. A: WB analyses of TRIM25 and EGFRexpression in the indicated lung cancer cells transiently transfected with control-siRNA or TRIM25-siRNAs; B: WB analyses of EGFR and its signaling proteins in A549 cells transiently expressed control-siRNA or TRIM25-siRNAs. n = 3, mean ± SEM |
TRIM25正向调节了EGFR蛋白表达, 为确认TRIM25对EGFR表达调控的机制, 本研究从EGFR转录和蛋白稳定性两个层次进行分析。实时定量PCR结果显示, 敲低A549细胞中的TRIM25并没有影响EGFR的转录水平(图 4A), 由此提示TRIM25可能调节EGFR的蛋白稳定性。随后, 在不同的时间点加入蛋白合成抑制剂放线菌酮(CHX), 利用CHX阻断EGFR从头合成途径, 进而观察EGFR的降解速度, 结果发现, 对照组A549细胞中EGFR的半衰期超过24 h, 而敲低细胞中的TRIM25后, EGFR的半衰期缩短至8 h以内(图 4B), 由此表明TRIM25延长了EGFR的半衰期, 增加了EGFR的蛋白稳定性。真核细胞内蛋白质的降解途径主要包括泛素-蛋白酶体途径和溶酶体途径[24], 为进一步探索TRIM25对EGFR降解途径的调节, 本研究利用蛋白酶体抑制剂MG132和自噬抑制剂chloroquine观察EGFR的降解过程, 结果发现蛋白酶体抑制剂MG132可以逆转EGFR的降解(图 4C), 但自噬抑制剂chloroquine则不能(图 4D), 表明TRIM25主要通过泛素-蛋白酶体途径抑制EGFR降解, 维持EGFR蛋白稳定性。EGFR与配体结合后内化首先进入早期内体, 进入早期内体的EGFR通常有两条去路: ①再次循环回到细胞膜; ②转运到溶酶体进行降解。利用EGF刺激对照组和敲低TRIM25的A549细胞, 分别观察了EGFR和Rab11 (再循环内体标记物), 或与LAMP1 (溶酶体标记物)的共定位情况。结果显示, 对照组中大量的EGFR和Rab11共定位; 而敲低TRIM25后EGFR与Rab11的共定位显著减少, 而主要和LAMP1共定位(图 4E)。该结果进一步表明, TRIM25抑制了EGFR降解。

|
Figure 4 TRIM25 maintains EGFR stability. A: The mRNA levels of TRIM25 and EGFR were detected by quantitative PCR (qPCR) inA549 cells transiently transfected with control-siRNA or TRIM25-siRNA; B: Protein stability of EGFR was determined by cycloheximide(CHX) assay in A549 cells transiently transfected with control-siRNA or TRIM25-siRNA; C, D: Effect of MG132 (C) or chloroquine (D) on EGFR stability regulated by TRIM25 overexpression. HEK 293T cells transfected with EGFR-MYC alone or with TRIM25-DDK were incubated with CHX plus MG132 (10 mmol · L-1) (C) or chloroquine (80 μmol · L-1) (D) for the indicated time. Indicated proteins were detected by WB analyses; E: Co-localization analysis of EGFR with Rab11 and LAMP1 under EGF treatment in control and TRIM25-silenced A549 cells. n = 3, mean ± SEM |
为进一步探究TRIM25维持EGFR蛋白稳定性的机制, 本研究利用Co-IP实验检测TRIM25和EGFR两者的相互作用。结果显示, TRIM25与EGFR存在相互作用(图 5A)。免疫荧光实验进一步显示这两种蛋白存在共定位(图 5B)。前期结果表明TRIM25主要通过泛素-蛋白酶体途径抑制EGFR降解, 维持EGFR蛋白稳定性(图 4C、D), TRIM25是一种E3泛素连接酶。基于此, 课题组猜想TRIM25可能是EGFR的E3泛素连接酶, 通过调节EGFR泛素化过程增加其蛋白稳定性。Co-IP结果显示, 敲低A549细胞中的TRIM25后, EGFR泛素化降低(图 5C), 提示TRIM25可增加EGFR的泛素化。多数研究发现K48位泛素化主要介导蛋白质降解, 而K63位泛素化主要调节蛋白质的功能和稳定性[25-28]。因此, 本研究进一步分析TRIM25对EGFR K48和K63位泛素化修饰的影响。结果显示, 在细胞中过表达TRIM25增加了EGFR的K63位泛素化修饰(图 5D), 而对K48位的泛素化没有影响(图 5E)。以上结果提示, TRIM25可能是EGFR的E3泛素连接酶, 通过促进EGFR的K63位泛素化修饰维持其蛋白稳定性。
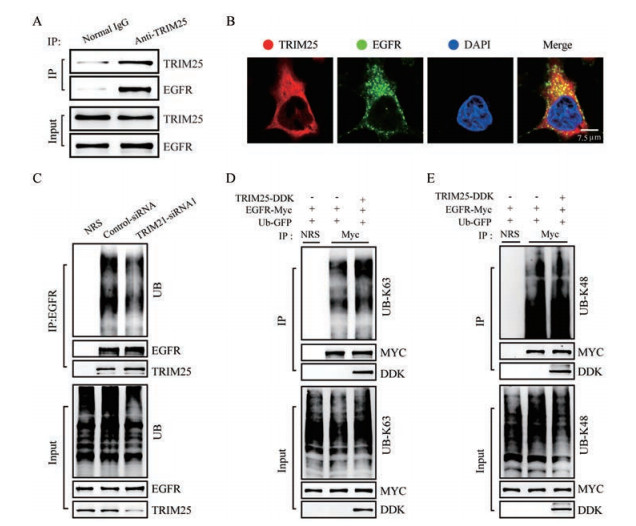
|
Figure 5 EGFR stability was maintained by K63-linked ubiquitination. A: Co-immunoprecipitation showed interaction between TRIM25and EGFR; B: Immunostaining images shows co-localization of TRIM25 and EGFR; C: The ubiquitination level of EGFR was detected incontrol or TRIM25-silenced A549 cells; D: The K63-linked ubiquitination of EGFR was detected by WB; E: The K48-linked ubiquitination of EGFR was detected by WB |
为进一步确认TRIM25是通过维持EGFR蛋白稳定性, 上调EGFR信号发挥促进肺癌细胞增殖和侵袭能力, 本研究利用EGFR表达质粒进行回复实验。结果发现与对照组相比, 敲低肺癌细胞A549中的TRIM25, 细胞的增殖和侵袭能力明显下降(图 6A、B); 而在此基础上, 恢复EGFR的蛋白表达后, 敲低TRIM25产生的抑制增殖和侵袭表型得到部分逆转:与敲低TRIM25为进一步确认TRIM25是通过维持EGFR蛋白稳相比, 其细胞增殖率增加了1.5倍, 侵袭率提高了1.6倍(图 6A、B); 同样, 在H1975肺癌细胞中得到了一致的结论:细胞增殖率增加了2倍, 侵袭率提高了1.7倍(图 6A、B)。此外, WB结果也表明在A549细胞中敲低TRIM25, 抑制了EGF诱导的ERK1/2、STAT5和EGFR的磷酸化水平, 而恢复EGFR蛋白表达后, 显著提高了EGF诱导的ERK1/2、STAT5和EGFR的磷酸化水平(图 6C)。以上结果表明, TRIM25的确通过维持EGFR及其下游信号, 发挥促肿瘤功能。
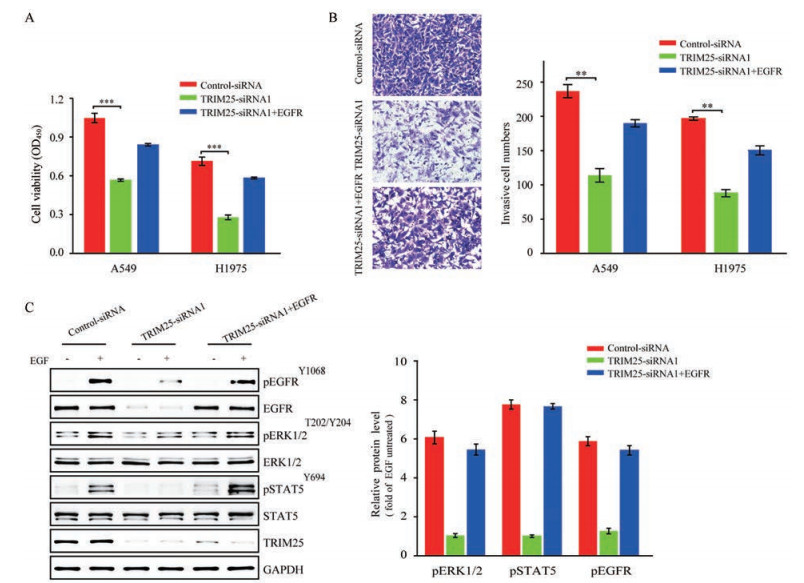
|
Figure 6 TRIM25 promotes lung cancer cell proliferation and invasion via maintaining EGFR signal. A-B: The proliferation and invasion of A549 cells and H1975 cells were evaluated through CCK-8 (A) and Transwell (B) assays, respectively; C: WB analyses of EGFR and its signaling proteins in A549 cells treated as indicated. n = 3, mean ± SEM. **P < 0.01, ***P < 0.001 |
肺癌由于发病率高、易转移以及缺乏有效的治疗策略, 成为严重影响人类健康的恶性肿瘤。大量研究发现, EGFR高表达或异常活化与肺癌的发生发展、复发转移以及耐药密切相关[29]。EGFR酪氨酸激酶抑制剂(EGFR-TKIs)作为一线药物, 已广泛应用于EGFR突变的非小细胞肺癌患者, 但其长期疗效却并不令人满意。主要原因在于, 有的肿瘤患者对EGFR靶向治疗不敏感; 而那些最初对EGFR靶向治疗敏感的患者往往也在一年内产生耐药。研究发现除了获得耐药性突变, EGFR信号的持续活化和传导不仅为癌细胞提供了生存优势, 而且也增加了肿瘤细胞的耐药性[30]。基于此, 研究者需要寻找靶向EGFR治疗肺癌的新策略。本研究发现在临床肺癌患者样本中, TRIM25与EGFR存在正相关性。敲低TRIM25减弱了其与EGFR的相互作用, 促进EGFR降解, 并抑制EGFR下游信号活化。因此, 靶向干扰TRIM25/EGFR相互作用、促进EGFR降解, 可能是肺癌治疗的新策略。
近年来研究发现TRIM家族与肿瘤的形成密切相关, 既可以作为癌基因也可作为抑癌基因。已有文献报道TRIM25可促进肺癌的发生发展, 但其具体的分子机制并未阐明。TRIM25作为重要的E3泛素连接酶, 主要通过对底物进行泛素化修饰发挥作用。泛素分子本身有7个赖氨酸残基, 这些赖氨酸残基都可以参与多聚泛素链的合成过程, 目前对K48位和K63位上的泛素化修饰研究的最为深入。K48位的泛素化通常介导蛋白质降解, K63位的泛素化则通常调节蛋白质功能以及维持蛋白稳定性[25-28]。已有文献报道, TRIM25可对RIG-1 (retinoic-acid-inducible gene I)进行K63位泛素化修饰, 增加其蛋白稳定性及其活性, 在抗病毒先天性免疫反应过程中发挥重要作用[31]。而且还能介导蛋白质发生K48位泛素化修饰进行降解[9, 32], 如Liu等[33]研究发现亲环素A (cyclophilin A, CypA)通过与TRIM25竞争性地结合线粒体抗病毒信号蛋白(mitochondria antiviral signaling protein, MAVS), 抑制TRIM25介导的MAVS K48位泛素化降解, 增强其蛋白稳定性。本研究结果提示, TRIM25可能通过介导EGFR K63位泛素化, 维持了EGFR的蛋白稳定性及其下游信号活性。在后续研究中, 课题组还需采用体内、外泛素化实验进一步验证TRIM25是否能促进EGFR K63位泛素化, 并寻找关键的泛素化位点; 同时, 还需要在整体动物水平进一步明确TRIM25对肺癌发生发展的作用和机制。
综上所述, 本研究结果表明, TRIM25通过介导EGFR K63位泛素化, 维持了EGFR的蛋白稳定性以及EGFR下游信号的持续活化, 增强了肺癌细胞的增殖和侵袭能力。本研究结果, 有利于深入了解TRIM25发挥促肿瘤功能的机制, 为今后相关新型靶向药物的研究提供了重要的理论基础。
| [1] | Zarogoulidis K, Zarogoulidis P, Darwiche K, et al. Treatment of non-small cell lung cancer (NSCLC)[J]. J Thorac Dis, 2013, 5: S389–S396. |
| [2] | American Cancer Society. Cancer Facts & Figures[R]. Atlanta (GA): American Cancer Society, 2013. |
| [3] | Miyazaki J, Hirota S, Abe T. Metastasis of lung cancer to the gastrointestinal tract, presenting with a volcano-like ulcerated mass[J]. Dig Endosc, 2015, 27: 397–398. |
| [4] | Littlefield P, Jura N. EGFR lung cancer mutants get specialized[J]. Proc Natl Acad Sci U S A, 2013, 110: 15169–15170. DOI:10.1073/pnas.1314719110 |
| [5] | Xue XY, Zhao S, Zhang ZN, et al. Transforming growth factor beta and lung cancer[J]. Int J Clin Exp Med, 2016, 9: 9766–9780. |
| [6] | Sussan TE, Pletcher MT, Murakami Y, et al. Tumor suppressor in lung cancer 1(TSLC1) alters tumorigenic growth properties and gene expression[J]. Mol Cancer, 2005, 4: 28–36. DOI:10.1186/1476-4598-4-28 |
| [7] | Inoue S, Orimo A, Hosoi T, et al. Genomic binding-site cloning reveals an estrogen-responsive gene that encodes a ring finger protein[J]. Proc Natl Acad Sci U S A, 1993, 90: 11117–11121. DOI:10.1073/pnas.90.23.11117 |
| [8] | Gack MU, Shin YC, Joo CH, et al. TRIM25 ring-finger E3 ubiquitin ligase is essential for RIG-I-mediated antiviral activity[J]. Nature, 2007, 446: 916–920. DOI:10.1038/nature05732 |
| [9] | Zhao KW, Sikriwal D, Dong X, et al. Oestrogen causes degradation of KLF5 by inducing the E3 ubiquitin ligase EFP in ER-positive breast cancer cells[J]. Biochem J, 2011, 437: 323–333. DOI:10.1042/BJ20101388 |
| [10] | Dong XY, Fu X, Fan S, et al. Oestrogen causes ATBF1 protein degradation through the oestrogen-responsive E3 ubiquitin ligase EFP[J]. Biochem J, 2012, 444: 581–590. DOI:10.1042/BJ20111890 |
| [11] | Ueyama K, Ikeda K, Sato W, et al. Knockdown of Efp by DNA-modified small interfering RNA inhibits breast cancer cell proliferation and in vivo tumor growth[J]. Cancer Gene Ther, 2010, 17: 624–632. DOI:10.1038/cgt.2010.19 |
| [12] | Sakuma M, Akahira J, Suzuki T, et al. Expression of estrogen-responsive finger protein (Efp) is associated with advanced disease in human epithelial ovarian cancer[J]. Gynecol Oncol, 2005, 99: 664–670. DOI:10.1016/j.ygyno.2005.07.103 |
| [13] | Zhu Z, Wang Y, Zhang C, et al. TRIM25 blockade by RNA interference inhibited migration and invasion of gastric cancer cells through TGF-β signaling[J]. Sci Rep, 2016, 6: 19070. DOI:10.1038/srep19070 |
| [14] | Qin Y, Cui H, Zhang H, et al. Overexpression of TRIM25 in lung cancer regulates tumor cell progression[J]. Technol Cancer Res Treat, 2016, 15: 707–715. DOI:10.1177/1533034615595903 |
| [15] | Zhou YC, Ma YH, Shi HT, et al. Epidermal growth factor receptor T790M mutations in non-small cell lung cancer (NSCLC) of Yunnan in southwestern China[J]. Sci Rep, 2018, 8: 15426. DOI:10.1038/s41598-018-33816-x |
| [16] | Jiang WX, Cai CQ, Hu PC, et al. Personalized medicine in non-small cell lung cancer:a review from a pharmacogenomics perspective[J]. Acta Pharm Sin B, 2018, 8: 530–538. DOI:10.1016/j.apsb.2018.04.005 |
| [17] | Volm M, Dring P, Wodrich W. Prognostic significance of the expression of c-fos, c-jun, and c-erbB1 oncogene products in human squamous cell lung carcinomas[J]. J Cancer Res Clin Oncol, 1993, 119: 507–510. DOI:10.1007/BF01686458 |
| [18] | Tebbutt N, Pedersen MW, Johns TG. Targeting the ERBB family in cancer:couples therapy[J]. Nat Rev Cancer, 2013, 13: 663–673. DOI:10.1038/nrc3559 |
| [19] | Lin AQ, Chen YQ, Chen XR, et al. Lorlatinib:a third generation ALK inhibitor for treatment of non-small cell lung carcinoma[J]. Acta Pharm Sin (药学学报), 2019, 54: 601–610. |
| [20] | Regales L, Gong Y, Shen R, et al. Dual targeting of EGFR can overcome a major drug resistance mutation in mouse models of EGFR mutant lung cancer[J]. J Clin Invest, 2009, 119: 3000–3010. |
| [21] | Wheeler DL, Dunn EF, Harari PM. Understanding resistance to EGFR inhibitors-impact on future treatment strategies[J]. Nat Rev Clin Oncol, 2010, 7: 493–507. DOI:10.1038/nrclinonc.2010.97 |
| [22] | Wei YJ, Zou ZJ, Becker N, et al. EGFR-mediated Beclin 1 phosphorylation in autophagy suppression, tumor progression, and tumor chemoresistance[J]. Cell, 2013, 12: 1269–1284. |
| [23] | Sette G, Salvati V, Memeo L, et al. EGFR inhibition abrogates leiomyosarcoma cell chemoresistance through inactivation of survival pathways and impairment of CSC potential[J]. PLoS One, 2012, 7: e46891. DOI:10.1371/journal.pone.0046891 |
| [24] | Jayhyuk M, Kyung BK, Craig MC. The ubiquitin-proteasome pathway and proteasome inhibitors[J]. Med Res Rev, 2001, 21: 245–273. DOI:10.1002/(ISSN)1098-1128 |
| [25] | Frédérick AM, Stéphane R. K48-linked ubiquitination and protein degradation regulate 53BP1 recruitment at DNA damage sites[J]. Cell Res, 2012, 22: 1221–1223. DOI:10.1038/cr.2012.58 |
| [26] | Zhang N, Wang Q, Ehlinger A, et al. Structure of the S5a:K48-linked diubiquitin complex and its interactions with Rpn13[J]. Mol Cell, 2009, 35: 280–290. DOI:10.1016/j.molcel.2009.06.010 |
| [27] | Wang G, Gao Y, Li L, et al. K63-linked ubiquitination in kinase activation and cancer[J]. Front Oncol, 2012, 2: 5. |
| [28] | Kulathu Y, Komander D. Atypical ubiquitylation-the unexplored world of polyubiquitin beyond Lys48 and Lys63 linkages[J]. Nat Rev Mol Cell Biol, 2012, 13: 508–523. DOI:10.1038/nrm3394 |
| [29] | Yewale C, Baradia D, Vhora I, et al. Epidermal growth factor receptor targeting in cancer:a review of trends and strategies[J]. Biomaterials, 2013, 34: 8690–8707. DOI:10.1016/j.biomaterials.2013.07.100 |
| [30] | Tan X, Lambert PF, Rapraeger AC, et al. Stress-induced EGFR trafficking:mechanisms, functions, and therapeutic implications[J]. Trends Cell Biol, 2016, 26: 352–366. DOI:10.1016/j.tcb.2015.12.006 |
| [31] | Zeng W, Sun L, Jiang X, et al. Reconstitution of the RIG-I pathway reveals a signaling role of unanchored polyubiquitin chains in innate immunity[J]. Cell, 2010, 141: 315–330. DOI:10.1016/j.cell.2010.03.029 |
| [32] | Urano T, Saito T, Tsukui T, et al. Efp targets 14-3-3σ for proteolysis and promotes breast tumour growth[J]. Nature, 2002, 417: 871–875. DOI:10.1038/nature00826 |
| [33] | Liu W, Li J, Zheng WN, et al. Cyclophilin A-regulated ubiquitination is critical for RIG-I-mediated antiviral immune responses[J]. Immunology, 2017, 6: e24425. |
 2019, Vol. 54
2019, Vol. 54


