2. 承德医学院附属医院, 河北 承德 067000;
3. 宁夏大学农学院, 宁夏 银川 750021;
4. 中国医学科学院、北京协和医学院药用植物研究所, 北京 100193
2. Affiliated Hospital of Chengde Medical College, Chengde 067000, China;
3. School of Agriculture, Ningxia University, Yinchuan 750021, China;
4. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China
银柴胡是石竹科繁缕属植物银柴胡(Stellaria dichotoma L. var. lanceolate)的干燥根, 为2015年版《中华人民共和国药典》收载中药[1], 味甘、性寒, 具有清虚热、除疳热的功效, 用于阴虚发热、骨蒸劳热、小儿疳热等症[2]。银柴胡在临床中主要用于感冒高热、过敏性疾病、抗肿瘤等方面的治疗。此外, 银柴胡常与其他药材配伍, 如在乌鸡白凤丸中与牡蛎、生地、天冬归类, 同起到滋阴清热之功能, 为乌鸡白凤丸中不可或缺的一味药材[3]; 与青蒿等药材配伍, 具有良好的抗菌消炎作用[4]。因此, 银柴胡的临床应用非常广泛。
银柴胡主要分布在宁夏、内蒙古、陕西省毗邻的干旱少雨沙生草原区, 极耐干旱[5]。目前银柴胡的野生资源相对匮乏, 商品药材绝大部分来源于人工种植, 市场上出现了的混伪品, 如有蝇子草、女娄菜、丝石竹、石竹等的干燥根[6, 7], 导致银柴胡临床用药的安全性和有效性得不到保证。银柴胡混伪品的混入不仅严重干扰了药材市场的正常运行, 而且对以此为原料的中成药生产产生极大干扰。针对银柴胡的鉴别方法, 国内学者开展了大量研究, 主要集中在性状、显微、理化及光谱鉴定[8]。但这些鉴定方法由于受鉴定人员经验、药材生长和采收加工的影响, 难以快速准确的鉴定银柴胡及其混伪品。
DNA条形码(DNA barcoding)技术作为中药材物种鉴定研究中的新方法[9], 是利用标准的、有足够变异的、易扩增且相对较短的DNA片段对物种进行快速、准确的鉴定[10, 11]。具有鉴定结果准确、重复性良好、方法通用性强等优点。DNA条形码鉴定技术已经在中药材冬虫夏草、枸杞、羌活、党参、天南星、锁阳等药材及其混伪品的鉴定中取得了良好的效果[12], 成为近年来中药鉴定研究的重点和亮点。同时国家药典委员会已将中药材DNA条形码分子鉴定指导原则收载于2015年版《中国药典》第四部[1]。其在中药材鉴定、药品流通、监督等诸多方面也得到了很好的应用。而有关银柴胡DNA条形码真伪鉴定研究亟待开展, 本文利用4条常用DNA候选条形码序列ITS、rbcL、psbA-trnH和matK对银柴胡及其混伪品进行鉴定研究, 分析比较不同候选序列对银柴胡的真伪鉴定能力, 为银柴胡及其混伪品的分子鉴定提供优选序列, 以期建立一种快速、准确高效的银柴胡真伪鉴定方法, 为银柴胡药材的准确鉴定提供科学依据。
材料与方法材料 本研究采集银柴胡及其混伪品的基原植物和药材样本共计60份, 分别来自宁夏、甘肃、江西、湖北等多个省份。基原植物样品采集后立刻装入含硅胶的密封袋中, 并勤换硅胶, 直至样品干燥完全。所有银柴胡及其混伪品的基原植物经宁夏大学农学院李小伟副教授或宁夏药品检验研究院丁锐副主任药师鉴定, 样品保存于宁夏中药材开发与利用工程技术研究中心储存室-70 ℃冰箱。所有药材样品均经原宁夏药品检验所韩义欣主任药师鉴定。样品采集信息见表 1。本研究同时从GenBanK数据库下载条形码序列, 序列筛选参照陈士林等[13]建立的筛选校对机制, 即BLAST分析、系统树分析、Barcoding gap检验, 共整理银柴胡及其混伪品的114条有效条形码序列(表 2)。
| Table 1 Sample information and GenBank accession numbers of Stellariae Radix and its adulterants |
| Table 2 Accession numbers downloaded from GenBank of different barcodes from various species |
试剂 DL2000 Marker、RNase酶、植物基因组DNA提取试剂盒(北京天根生化科技有限公司); psbA-trnH、ITS、matK、rbcL序列引物(上海生工生物工程有限公司); 琼脂糖(西班牙, Biowest Agarose); 2×Taq plus Master Mix (南京诺唯赞生物科技有限公司); GelRed染料(美国Biotium公司)。
仪器 MyCycler PCR仪、Gel Doc XR+凝胶成像系统、Powerpac Basic琼脂糖凝胶电泳仪(美国伯乐有限公司); MiniSpin plus高速离心机(德国Eppendorf公司); Geno 2010高通量组织研磨机(美国SPEX SamplePrep公司); ND2000核酸蛋白检测仪(美国NanoDrop公司); 自动高压灭菌锅(上海博讯仪器有限公司); Mini离心机(珠海黑马医学仪器有限公司); SZ-1涡旋混合器(常州国宇仪器制造有限公司); 恒温水浴锅(北京长源实验设备厂)。
DNA提取 取经鉴定的银柴胡及其混伪品基原植物样品30 mg, 干燥药材去除表皮取中间部分50 mg, 加入GP1缓冲液和钢珠2枚, 经高通量组织研磨机研磨4或6 min后, 采用DNA提取试剂盒提取总DNA, 详细操作参照试剂盒说明书[14]。
PCR扩增及测序 本研究选用国际通用序列ITS、rbcL、psbA-trnH及matK 4条DNA条形码候选序列。4对引物序列及PCR反应条件如表 3。PCR体系为25 μL, 包含2×Taq Plus Master Mix 12.5 μL, 正反向引物各1 μL, DNA模板1 μL, ddH2O 9.5 μL[12]。
| Table 3 Primers sequences and corresponding PCR reaction conditions[12] |
取4 μL PCR产物用1%的琼脂糖凝胶(含GelRed染料)电泳, 在凝胶成像仪中观察并拍照保存。并将扩增成功的PCR产物直接送至中国农业科学院作物科学研究所进行双向测序。
序列分析及评价 利用序列拼接软件CodonCode Aligner V7.01对测序峰图文件进行校对拼接, 去除低质量序列及引物区; ITS序列除去18S和28S序列, 保留ITS1、5.8S和ITS2[15]; 使用MEGA 7.0对拼接后序列进行比对分析, 比较不同序列的比对后长度、变异位点和遗传距离, 分析序列的碱基组成, 对各个候选序列进行种间种内变异分析, 做出Barcoding gap图[16], 并对数据进行1 000次自展, 以K2P模型采用NJ法建立系统进化树, 分析银柴胡及混伪品的亲缘关系。同时运用相似性搜索算法(BLAST)计算各序列的鉴定成功率[17]。
结果与分析 1 候选序列扩增及测序成功率PCR扩增和测序成功率是DNA条形码分子鉴定的两个重要指标。4条候选序列扩增及测序结果如表 4, 其中ITS、psbA-trnH和rbcL扩增成功率达100%, matK扩增成功率最小为84.1%。ITS的测序成功率最高, 为95.2%, psbA-trnH的测序成功率较低, 为90.3%。综合扩增和测序成功率, ITS序列最高(95.2%), matK最低(75.0%)。
| Table 4 The success rate of PCR amplification and bio-direction sequencing |
4条序列的特征参数如长度、GC含量、遗传距离、变异位点数、变异率及鉴定成功率等信息详见表 5。各序列的长度范围为211~797 bp, 其中psbA-trnH最短, 为211~388 bp, matK相对较长, 为714~797 bp; 其中平均遗传距离最大的为psbA-trnH, 其次是ITS。如表 5所示, matK和ITS的鉴定成功率都较高, 分别为92%和90%。综上分析, ITS序列变异率和平均遗传距离较大, 且鉴定成功率最高。
| Table 5 Identification efficiency and information of sequences |
对4种DNA条形码候选序列Barcoding gap分析(图 1), ITS序列有明显的间隔区, 种间变异范围与种内变异范围无重叠区域, 有利于银柴胡及其混伪品的区分。rbcL序列无明显间隔区, matK序列种间种内变异有部分重叠, psbA-trnH序列无重叠区, 但种内种间有部分差异不明显。

|
Figure 1 Barcoding gap plots of different candidate sequences. A: ITS sequence; B: matK sequence; C: psbA-trnH sequence; D: rbcL sequence |
利用MEGA 7.0软件构建NJ系统进化树。如图 2所示, ITS可将正品银柴胡聚为一支, 与其亲缘关系较近的同属混伪品二柱繁缕也能明显分为不同分支, 混伪品女娄菜和石竹聚为一支。而rbcL和matK不能把同属混伪品二柱繁缕分开, psbA-trnH序列所构建的进化树显示混伪品菊科植物山白菊和中华苦荬菜与银柴胡同科蝇子草属和石头花属植物聚在一支, 无法很好地反映不同种间的遗传变异及同种样品间的同源性。
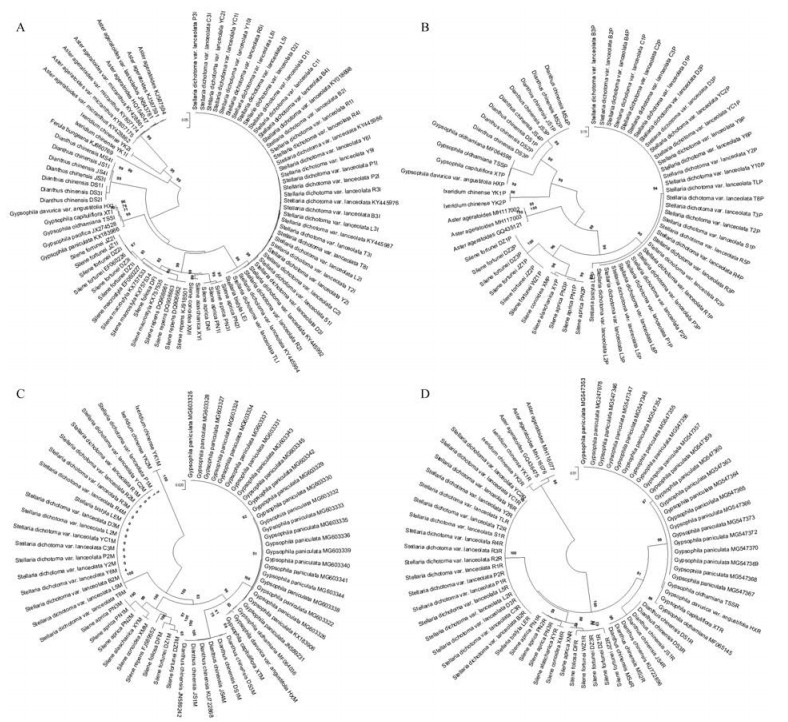
|
Figure 2 NJ trees based on four DNA barcoding sequences for Stellariae Radix and its adulterants. A: ITS; B: psbA-trnH; C: matK; D: rbcL. Bootstrap 1 000 repetitions, the value on the support only shows self-support support rate ≥ 70% |
银柴胡作为在临床及中成药生产中应用广泛的中药材, 部分混伪品流入市场, 直接影响着相关中药的质量及临床用药安全。由于受鉴定人员经验、种质资源、药材采收时期等因素影响, 利用传统生药鉴定的方法已经难以满足银柴胡真伪鉴定的需求。本研究选取4种候选条形码序列ITS、psbA-trnH、rbcL和matK, 以PCR扩增和测序成功率、遗传距离、变异率、Barcoding gap及进化树等为指标, 分析其对银柴胡及其混伪品的鉴别情况[18]。结果显示, 4条序列中, ITS的PCR扩增率、测序成功率、平均遗传距离和变异率都较高, 其鉴定成功率达到90%;在检验不同序列的Barcoding gap时, ITS序列有明显的间隔区, 宜用于银柴胡真伪鉴别; NJ系统进化树显示, ITS可将银柴胡聚在一起, 银柴胡同属植物混伪品二柱繁缕与银柴胡明显分为不同分支, 可对银柴胡及其混伪品进行准确鉴定。另外, ITS是最常用的系统发育序列[19], 本研究中基于ITS序列构建的进化树将样品聚为三支, 分别为石竹科、伞形科和菊科, 将各科同属的样本聚为一支, 并能将同属不同物种分为不同分支, 因此ITS序列也具有银柴胡分子进化研究的潜力。2018年, Meng等[20]基于ITS序列的银柴胡种质资源遗传多样性研究, 推测目前宁夏和甘肃陇西所有人工栽培的银柴胡种质资源来源相同, 是同一野生种的后代。
实验过程中4条序列的PCR扩增率都较高, 其中ITS、psbA-trnH和rbcL扩增成功率达100%, matK由于序列较长, 测序成功率略低。PCR扩增和测序未成功的样本均经多次重复试验。其中, ITS的测序成功率最高, 为95.2%[21]。在序列分析中, 鉴定成功率是考察不同序列对同一物种鉴别能力的重要指标之一。BLAST方法是将样本的DNA序列与全体序列进行碱基比对, 按碱基差异进行排名, 具有运行速度快、精度高的特点[22, 23]。结果显示, matK和ITS的鉴定成功率都较高, 分别为92%和90%。本实验采用通用的DNA条形码引物和PCR扩增条件, 并进行多次扩增和测序的重复试验以保证数据的可靠性, 采用了包括序列变异分析、遗传距离计算、Barcoding gap检验、物种鉴定效率比较等多种分析方法以保证结论的可靠性。
理想的DNA条形码序列, 种内遗传距离应该明显小于种间遗传距离, 且种内遗传距离和种间遗传距离存在明显差异, 两者内可形成一个半段的间隔区, 即“DNA barcoding gap”, 可用来判断种内不同个体之间的变异程度[11]。Zheng等[24]在百合属内不同物种分子鉴定中, 5条候选序列的Barcoding gap图表明, ITS序列种与种间变异明显分为两大部分, 其分布较高的遗传变异可以明显区分, ITS序列可作为鉴别百合属的较理想条形码序列。Yang等[25]对山矾属植物的最适DNA条形码片段筛选时, 从Barcoding gap图可看出, ITS序列种内和种间的遗传距离频率分布重叠区域较小, 满足DNA条形码候选序列要求。本实验中Barcoding gap图显示, ITS序列有明显的间隔区, 种间变异范围与种内变异范围无重叠区域, 作为优选序列有利于银柴胡药材的真伪鉴别。
ITS序列在扩增效率、鉴定成功率、种内与种间变异、Barcoding gap、系统发育树等方面均较其他序列表现良好。因此, ITS序列是准确鉴别银柴胡及其混伪品的理想DNA条形码鉴定序列。银柴胡DNA条形码的研究将为银柴胡市售药材的判定及混伪品鉴别提供可靠的分子水平依据。其也可作为《中国药典中药材DNA条形码标准序列》中银柴胡DNA条形码鉴定的补充, 为有效监管银柴胡药材流通领域的各个环节提供辅助手段, 有利于推动中药材市场的标准化和规范化, 对保障银柴胡药材的临床用药安全具有重要意义。
| [1] | Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China. Part 1(中华人民共和国药典)[S]. Beijing: China Medical Science Press, 2015: 22-23. |
| [2] | Ao L, Li J, LiuYH. Chemical constituents from Stellaria dichotoma and their anti-inflammatory activities[J]. Chin Tradit Pat Med (中成药), 2018, 40: 1106–1109. |
| [3] | Jiang LL, Liu CK. Study on the therapeutic effect and clinical application of Wuji Baifeng pills[J]. Clin J Chin Med (中医临床研究), 2012, 23: 31–33. |
| [4] | Gao XM. Chinese Pharmacy (中药学)[M]. Beijing: China Press of Traditional Chinese Medicine, 2007. |
| [5] | Shang BY. The quality analysis of cultivated Lanceolate Dichoto mous Starwort Root in Ningxia[J]. Ningxia Med J (宁夏医学杂志), 2012, 34: 451–452. |
| [6] | Cai SQ, Li J. Species Systematization and Quality Evaluation of Commonly Used Chinese Traditional Drugs (常用中药材品种整理和质量研究)[M]. Beijing: Beijing Medical University Press, 2005. |
| [7] | Yu KQ, Jiao LK, Ren SY, et al. Research progress of Chinese traditional medicinal Stellariae Radix[J]. Mod Chin Med (中国现代中药), 2015, 17: 1223–1229. |
| [8] | Chen F. The identification between Stellariae Radix and its adulterant Silene jenisseensis[J]. Guide China Med (中国医药指南), 2013, 11: 234–235. |
| [9] | Techen N, Parveen I, Pan Z, et al. DNA barcoding of medicinal plant material for identification[J]. Curr Opin Biotechnol, 2014, 25: 103–110. DOI:10.1016/j.copbio.2013.09.010 |
| [10] | Hebert PDN, Ratnasingham S, de Waard JR. Barcoding animal life:cytochrome coxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci, 2003, 270: S96–S99. |
| [11] | Li DZ, Liu JQ, Chen ZD, et al. Plant DNA barcoding in China[J]. J Syst Evolut, 2011, 49: 165. DOI:10.1111/jse.2011.49.issue-3 |
| [12] | Chen SL. DNA Barcoding Molecular Identification of Tradi tional Chinese Medicine (中药DNA条形码分子鉴定)[M]. Beijing: People's Medical Publishing House, 2012: 14. |
| [13] | Chen SL, Song JY, Yao H, et al. Strategy and key technique of identification of Chinese herbal medicine using DNA barcoding[J]. Chin J Nat Med (中国天然药物), 2009, 7: 322–327. |
| [14] | Li J, Hao CQ, Zhang R, et al. Screening of the total DNA extrac tion methods for different material of Radix Stellariae[J]. Jiangsu Agric Sci (江苏农业科学), 2018, 46: 43–46. |
| [15] | Zheng SH, Wei WL, Ren WG, et al. Molecular identification of the species of Pinellia and its adulterants by DNA barcoding[J]. Chin Bull Bot (植物学报), 2014, 49: 710–719. DOI:10.3724/SP.J.1259.2014.00710 |
| [16] | Wang J, Hou YF, Bai Z, et al. Screening the universal sequence of DNA barcodes in curcuma kwangsiensis[J]. China J Tradit Chin Med Pharm (中华中医药杂志), 2015, 30: 100–103. |
| [17] | Feng SS, Zheng SH, Li YK, et al. Identification of Radix et Rhizoma Clematidis and its adulterants using DNA barcoding[J]. Acta Pharm Sin (药学学报), 2014, 49: 260–266. |
| [18] | Lu JN, Cheng H, Fan JJ, et al. DNA molecular identification of Chinese herbal materials of Dysosma by ITS2 barcode sequence[J]. Chin Tradit Herb Drugs (中草药), 2018, 49: 3907–3911. |
| [19] | Wang DY, Wang Q, Wang YL, et al. Evaluation of DNA barcodes in Codonopsis (Campanul-aceae) and in some large angiosperm plant genera[J]. PLoS One, 2017, 12: e0170286. DOI:10.1371/journal.pone.0170286 |
| [20] | Meng XS, Dai XH, Liu P, et al. Study on genetic diversity of ITS sequence in Stellaria dichotoma var. lacceolata[J]. J Chin Med Mater (中药材), 2018, 41: 55–59. |
| [21] | Wang YD, Han XN, Zhao YD, et al. Identification of commercial Bupleuri Radix and its adulterants based on ITS2 barcode[J]. Chin Tradit Herb Drugs (中草药), 2017, 48: 3590–3596. |
| [22] | Lu QZ. The Study on DNA Molecular Identification of 12 Kinds of Common Medicinal Pteridophytes in Lushan Region (12种庐山地区常见药用蕨类植物的DNA分子鉴定研究)[D]. Nanchang: Nanchang University, 2018. |
| [23] | Zhou M, Fan LL, Gao HX, et al. The DNA barcoding research of Hedyotis diffusa[J]. Lishizhen Med Mater Med Res (时珍国医国药), 2018, 29: 103–106. |
| [24] | Zheng SH, Li YK, Ren WG, et al. Molecular identification in genus of Lilium based on DNA barcoding[J]. Acta Pharm Sin (药学学报), 2014, 49: 1730–1738. |
| [25] | Yang P, Zhou HY, Wu HE, et al. Screening research on DNA barcoding sequences of medicinal plants of Symplocos[J]. J Chin Med Mater (中药材), 2018, 41: 1331–1334. |
 2019, Vol. 54
2019, Vol. 54


