2. 西藏藏医学院, 西藏 拉萨 850000
 的基原,具有重要的药用价值。在课题组前期品种整理及遗传背景分析基础上,本文分别测定秦艽组10种23个居群69份样品以及外类群椭圆叶花锚Halenia elliptica 3份样品的线粒体nad1基因第2内含子区(nad 1/b-c)以及nad5基因第4内含子区(nad5/d-e)部分序列,对其在秦艽组植物的分子系统发育分析及物种鉴定中的意义进行评价。结果显示:nad1/b-c具一粗壮秦艽G.robusta King ex Hook.f.稳定变异位点;同时,对粗茎秦艽G.crassicaulis Duthie ex Burk.及西藏秦艽G.tibetica King ex Hook.f.有一定物种分辨率;nad5/d-e序列相似度高,仅大叶秦艽G.macrophylla Pall.、黄管秦艽G.officinalis H.Smith及管花秦艽G.siphonantha Maxim.ex Kusnez.存在种内不同基因型。进而基于粗壮秦艽G.robusta King ex Hook.f.nad 1/b-c物种分子标记,设计一特异鉴定引物,建立粗壮秦艽特异性引物-PCR鉴定方法。本工作可为协同进化不完全、遗传背景复杂多样的中藏药高山基原植物DNA条形码的构建提供新思路。
的基原,具有重要的药用价值。在课题组前期品种整理及遗传背景分析基础上,本文分别测定秦艽组10种23个居群69份样品以及外类群椭圆叶花锚Halenia elliptica 3份样品的线粒体nad1基因第2内含子区(nad 1/b-c)以及nad5基因第4内含子区(nad5/d-e)部分序列,对其在秦艽组植物的分子系统发育分析及物种鉴定中的意义进行评价。结果显示:nad1/b-c具一粗壮秦艽G.robusta King ex Hook.f.稳定变异位点;同时,对粗茎秦艽G.crassicaulis Duthie ex Burk.及西藏秦艽G.tibetica King ex Hook.f.有一定物种分辨率;nad5/d-e序列相似度高,仅大叶秦艽G.macrophylla Pall.、黄管秦艽G.officinalis H.Smith及管花秦艽G.siphonantha Maxim.ex Kusnez.存在种内不同基因型。进而基于粗壮秦艽G.robusta King ex Hook.f.nad 1/b-c物种分子标记,设计一特异鉴定引物,建立粗壮秦艽特异性引物-PCR鉴定方法。本工作可为协同进化不完全、遗传背景复杂多样的中藏药高山基原植物DNA条形码的构建提供新思路。2. Tibetan Traditional Medical College, Lhasa 850000, China
龙胆科龙胆属Gentiana秦艽组Sect. Cruciata植物具有重要的药用价值, 其中大叶秦艽Gentiana macrophylla Pall.、麻花秦艽G. straminea Maxim.、小秦艽G. dahurica Fisch.及粗茎秦艽G. crassicaulis Duthie ex Burk.作为中药秦艽的基原植物, 根入药, 祛风湿, 清湿热, 止痹痛, 退虚热[1]。传统藏药解吉
课题组前期开展了秦艽组药用植物的分类学鉴定等基础性工作。在野外考察中注意到, 该组植物种下变异幅度较大, 一些居群个体显现种间过渡形态情况; 一些近缘种的鉴定较难把握。确定了藏药解吉基原植物之一的粗壮秦艽Gentiana robusta King ex Hook. f.为一自然杂交物种, 其母系来源可能是中药秦艽的基原植物之一——麻花秦艽G. straminea Maxim.[5]。同时, 注意到一些物种ITS区在基因组内存在明显多样性(协同进化不完全)。国内学者的研究工作亦显示:同域分布的近缘种间存在自然杂交现象[6, 7]。因此, 深入开展该组植物的品种整理工作及其遗传背景分析, 准确把握其种间间断、种内形态变异幅度及遗传标记的多态性等, 进而构建高效的DNA条形码, 具有重要的科学意义。
植物DNA条形码序列常来自于核基因组及叶绿体基因组, 通常认为线粒体基因较为保守, 在中药DNA分子鉴定中的作用有限。事实上, 线粒体基因组在不同类群(如科、属、种)间的进化速率是不相同的, 对其系统学意义及其物种鉴定意义的评价应视具体情况而具体分析[8]。植物mtDNA nad1基因由5个外显子构成(nad1/A-E), 编码线粒体呼吸传递链复合体Ⅰ (NADH-泛醌氧化还原酶)的nad1亚基, 是一个结构较为稳定的线粒体基因。其第2内含子(nad 1/b-c)序列在植物系统发育研究及中药基原植物物种鉴定中, 显示出应有的价值[9, 10]; 而nad5基因编码该复合体的第5个亚基。
基于课题组前期工作[11-14], 本文分别测定了nad1基因外显子B和C之间的第2内含子区(intron 2)以及nad5基因外显子D和E之间的第4内含子区(intron 4)部分序列, 对其在秦艽组植物的分子系统发育分析及物种鉴定中的意义进行初步评价, 以期为致同进化不完全、遗传背景复杂多样的中藏药高山基原植物DNA条形码的构建提供新思路。
材料与方法材料 在西藏、云南、甘肃、四川、青海等省区广泛取样, 每个居群平行取3株样品。获得龙胆属秦艽组10种23个居群69份标本; 采集龙胆科花锚属植物椭圆叶花锚Halenia elliptica D. Don作为外类群(3份标本)。压制花果期标本, 同时取新鲜叶片经硅胶快速干燥, 备用。凭证标本经赵志礼教授鉴定, 存放于上海中医药大学中药学院药用植物标本室, 见表 1。
| Table 1 Samples and voucher specimens |
总DNA提取 取上述硅胶快速干燥的植物叶片, 在液氮中研磨成粉末, 采用改良的CTAB法[15]提取总DNA。
PCR扩增和测序 对11种24个居群所有72份样品的线粒体nad1/b-c及nad5/d-e片段分别进行扩增, 引物及PCR反应条件参考相关文献[9, 16]。扩增产物经电泳检测后, 送上海生工生物有限公司纯化并测序(ABI 3730XL型测序仪)。
序列数据分析 测得的序列经Clustal W软件对位排列, 并进行手动校正; 去除两端模糊不清的位点(所有序列均上传至GenBank)。应用MEGA 7软件[17]进行遗传距离及系统学等分析。
特异性引物-PCR鉴定方法 在nad1/b-c序列数据分析基础上, 发现一稳定的粗壮秦艽G. robusta King ex Hook. f.物种鉴定分子标记, 据此设计特异性上游引物PF: 5′-GCTTCTTATCTAGAGAGGGGGGAC-3′; 下游引物nad1/3[9]: 5′-GGAGCTCGATTAGTTTCTGC- 3′。步骤: ① nad1/b-c片段扩增; ②反应产物电泳, 割胶纯化; ③稀释纯化产物作为模板, 特异性引物-PCR鉴定, 实验条件: 94 ℃, 4 min; 35× (94 ℃, 45 s; 63.7 ℃, 45 s; 72 ℃, 2 min); 72 ℃, 10 min; ④电泳检测。
结果 1 序列的获得共获得144条目的片段序列, 包括72条nad1/b-c片段及72条nad5/d-e片段。全部序列上传至GenBank, 并获得对应的登录号(表 2)。
| Table 2 GenBank accession numbers |
获得的11种24个居群所有72份样品的线粒体nad1/b-c序列, 经对齐、手动校正后, 序列长度:秦艽组10种均为573 bp, 而外类群椭圆叶花锚长601 bp (有4个插入, 长度分别为: 8 bp、2 bp、13 bp、5 bp)。黄管秦艽Gentiana officinalis H. Smith及管花秦艽G. siphonantha Maxim. ex Kusnez.存在种内不同基因型, 即黄管秦艽383位点存在“A/T”2种基因型, 两者比例为14︰1。管花秦艽在388位点存在“A/G”2种基因型, 两者比例为2︰1。除此之外, 其他物种内各样品间序列完全一致。
进一步分析显示:粗壮秦艽G. robusta King ex Hook. f.所有6个居群18个样品的序列在第45位点均为“C”, 而秦艽组其余9个种均为“T”; 该位点有望作为粗壮秦艽的特异DNA分子标记。粗茎秦艽G. crassicaulis Duthie ex Burk.及西藏秦艽G. tibetica King ex Hook. f.的第352和353位点均为“GA”, 而秦艽组其余8个种均为“TC”; 该位点与其他片段相组合, 有望作为粗茎秦艽及西藏秦艽的特异DNA分子标记; 黄管秦艽383位点及管花秦艽388位点的种内变异, 可作为相应物种鉴定的候选分子标记(表 3)。
| Table 3 Variable sites in nad1/b-c and nad5/d-e within Sect. Cruciata |
获得的11种24个居群所有72份样品的线粒体nad5/d-e序列, 经对齐、手动校正后, 序列长度:秦艽组10种均为915 bp, 而外类群椭圆叶花锚长885 bp (有3个插入, 长度分别为: 28 bp、6 bp、1 bp; 有1个5 bp缺失)。就秦艽组10种植物而言, 序列相似度高, 仅大叶秦艽Gentiana macrophylla Pall.、黄管秦艽G. officinalis H. Smith及管花秦艽G. siphonantha Maxim. ex Kusnez.存在种内不同基因型, 即大叶秦艽229位点存在“T/G”2种基因型, 两者比例为5︰1;黄管秦艽229位点存在“T/G”2种基因型, 两者比例为1︰2;管花秦艽在258位点存在“T/C”2种基因型, 两者比例为2︰1。除此之外, 其他物种内各样品间及各物种间序列完全一致(表 3)。
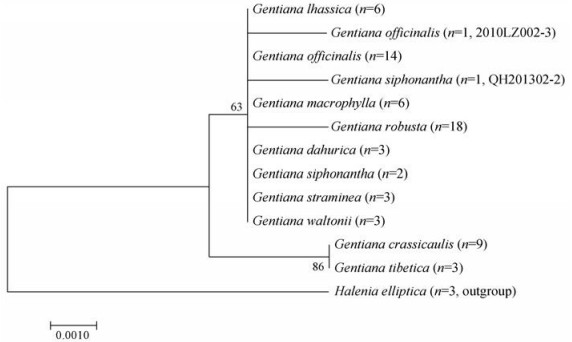
4 nad1/b-c片段物种鉴定意义的评价基于线粒体nad1/b-c序列, 以椭圆叶花锚Halenia elliptica D. Don为外类群, Kimura 2-parameter模型, 构建NJ系统树, bootstrap检验, 重复1 000次(MEGA7)。结果显示, 基因树的分子系统学关系与经典分类学观点相一致:外类群花锚属Halenia的椭圆叶花锚首先单独区分开; 在龙胆属Gentiana一大支中, 粗茎秦艽与西藏秦艽共聚为一支, 另一支中, 粗壮秦艽所有18个样品聚为次一小支, 而其他物种包括黄管秦艽及管花秦艽的不同基因型的物种分子间断不清晰(图 1)。
|
Figure 1 NJ phylogenetic tree based on mtDNA nad1/b-c within Sect. Cruciata. Halenia elliptica was set as an outgroup. Numbers above or below the nodes are bootstrap support values |
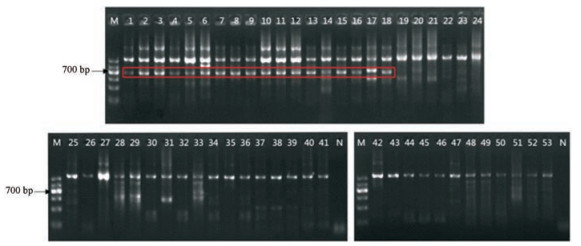
所有6个居群18个粗壮秦艽样品在700 bp处均出现一亮带, 其9个近缘种共35个样品则不显示条带。此处条带为粗壮秦艽特有, 割胶测序结果亦证实其序列正是特异性上游引物PF的目的条带。可依据该处条带的有无鉴定出粗壮秦艽及近缘物种。同时, 该特异性引物-PCR鉴定图谱中, 1 200 bp左右亦出现一非特异性条带, 初步推断该片段可能为nad1/b-c序列。
由于在构建粗壮秦艽特异性引物-PCR鉴定方法时, 注意到直接用样品总DNA作为模板, 特异性PCR结果不理想。因此, 先用一对通用引物nad1/2、nad1/3进行预扩增, 获得长度约为1 200 bp的nad1/b-c序列。将该产物割胶, 分离纯化。以此为模板, 应用特异性上游引物PF及下游通用引物nad1/3再次进行PCR扩增, 最终获得粗壮秦艽特异性引物-PCR鉴定图谱。第二步PCR时, 上游引物PF及下游通用引物nad1/3进行特异性PCR反应, 扩增出双链目的片段, 同时, 反应过程中, 下游引物nad1/3亦可能作为单一引物, 扩增出nad1/b-c单链产物, 因此出现1 200 bp左右的非特异性产物。为进一步验证推断, 将该产物割胶, 分离纯化。测序结果显示, 该片段确为nad1/b-c序列(图 2)。
|
Figure 2 PCR profile of different Sect. Cruciata species amplified using developed primer PF. Bands (c. 700 bp) in the box show the species-specific band amplified only in G. robusta. Lane M: GM333 DNA ladder. 1-18: G. robusta; 19-22: G. lhassica; 23-29: G. officinalis; 30-33: G. macrophylla; 34-36: G. dahurica; 37-39: G. straminea; 40-44: G. crassicaulis; 45-47: G. siphonantha; 48-50: G. tibetica; 51-53: G. waltonii. N: Negative control |
近年来, 核基因组的ITS区, 叶绿体基因组matK、rbcL、rpoC1、trnL (UAA) intron、psbA-trnH、atpB-rbcL、trnS (GCU)-trnG (UCC)、rpl20-rps12、trnL (UAA)-trnF (GAA)等片段及其组合越来越多地应用于植物的系统发育重建研究、中药基原植物遗传多样性分析及品种鉴定中[12, 14, 18, 19]; 一些药用植物如人参Panax ginseng C. A. Mey.等全基因组序列的测定, 为更加广泛筛选DNA分子标记, 探讨活性物质的生物合成途径等提供基础资料[20]。一般认为, 被子植物中线粒体基因的进化速率要比核基因慢5倍, 与哺乳动物线粒体基因比核基因进化至少快5倍形成巨大的反差[9]。就植物线粒体基因组片段而言, 尽管一些研究表明, 其中有些片段含有一定的系统发育信息, 而且较为稳定, 但相关研究报道仍然有限。目前, 《中国药典》已收载基于动物线粒体基因组片段的药材蕲蛇及乌梢蛇等DNA鉴定方法[1], 但植物来源中药材的DNA鉴定方法, 需要更深入的基础研究工作。
本文在野外考察基础上, 采集龙胆科龙胆属Gentiana秦艽组Sect. Cruciata 10种23个居群69份样品以及外类群椭圆叶花锚Halenia elliptica 3份样品, 分别测定线粒体nad1基因第2内含子区(nad1/b-c)以及nad5基因第4内含子区(nad5/d-e)部分序列。结果显示, 基于线粒体nad1/b-c序列构建的基因树, 其“属”一级的分子系统学关系与经典分类学观点相一致:同时, 对粗壮秦艽G. robusta King ex Hook. f.具有物种分辨率; 对粗茎秦艽G. crassicaulis Duthie ex Burk.及西藏秦艽G. tibetica King ex Hook. f.具有“类群”分辨率。而nad5/d-e分子系统学意义有限, 检测到的大叶秦艽Gentiana macrophylla Pall.、黄管秦艽G. officinalis H. Smith及管花秦艽G. siphonantha Maxim. ex Kusnez.存在的种内不同基因型, 有望作为相关物种的潜在分子标记。
粗壮秦艽作为一自然杂交物种, 协同进化(concerted evolution)不完全, 遗传背景复杂, 应用核基因组及叶绿体基因组片段构建DNA条形码有其局限性。而本文检测到的一nad1/b-c物种分子标记, 具有稳定性强等特点, 显示植物线粒体基因组片段可能携带特有的分子系统学信息。另外, nad1/b-c中的一分子标记, 可将粗茎秦艽及西藏秦艽与同组的其他8种植物区别开, 该片段可作为粗茎秦艽及西藏秦艽物种DNA条形码候选序列之一。
基于发现的粗壮秦艽G. robusta King ex Hook. f. nad1/b-c物种分子标记, 设计一特异鉴定引物PF, 优化PCR反应条件, 建立粗壮秦艽特异性引物-PCR鉴定方法。该反应退火温度63.7 ℃, 所有6个居群18个粗壮秦艽样品在700 bp处均出现一特异条带, 而9个近缘种共35个样品均不显示此条带。至于1 200 bp左右均出现的一非特异性条带, 经测序分析, 该片段为nad1/b-c序列。由于与目的片段长度差距较大, 两条带彼此有明显隔离, 不影响实际鉴定结果的判断。为进一步考察该方法的稳定性与专属性, 2014~2015年在西藏墨竹工卡县日多乡、西藏拉萨夺底沟及西藏山南错那县县城附近另采集粗壮秦艽3居群, 9份样品, 同时采集全萼秦艽2居群6份样品, 长梗秦艽2居群6份样品, 如法PCR扩增, 电泳检测。结果显示, 仅粗壮秦艽3居群9份样品在700 bp处均出现一特异条带。本文建立的粗壮秦艽特异引物鉴定方法可为相关药材的鉴定提供基础资料。
相较于核基因和叶绿体基因而言, 虽然线粒体基因在植物的系统发育重建研究、中药基原植物遗传多样性分析及品种鉴定中少有应用; 但只要筛选合适的序列片段, 线粒体基因仍能提供新的证据[9, 21]。
| [1] | Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China: Vol Ⅰ (中华人民共和国药典: 一部)[S]. Beijing: China Medical Science Press, 2015: 78-79, 270-271, 372-373. |
| [2] | Zhao ZL, Gaawe D, Wang ZT. Identification of medicinal plants used as Tibetan Traditional Medicine Jie-Ji[J]. J Ethnopharmacol, 2010, 132: 122–126. DOI:10.1016/j.jep.2010.07.051 |
| [3] | Yang YC. Tibetan Medicine (藏药志)[M]. Xining: Qinghai People Press, 1991: 9-12. |
| [4] | Ho TN. Flora Reipublicae Popularis Sinicae vol. 62(中国植物志, 62卷)[M]. Beijing: Science Press, 1988: 59-75. |
| [5] | Xiong B, Zhao ZL, Ni LH, et al. DNA-based identification of Gentiana robusta and related species[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 4680–4685. |
| [6] | Li XJ, Wang LY, Yang HL, et al. Confirmation of natural hybrids between Gentiana straminea and G. siphonantha (Gentianaceae) based on molecular evidence[J]. Acta Bot Yunnan (云南植物研究), 2007, 29: 91–97. |
| [7] | Hu QJ, Peng HC, Bi H, et al. Genetic homogenization of the nuclear ITS loci across two morphologically distinct gentians in their overlapping distributions in the Qinghai-Tibet Plateau[J]. Sci Rep, 2016, 6: 34244. DOI:10.1038/srep34244 |
| [8] | Lu JN, Zhao ZL, Ni LH, et al. Identification of medicinal plants based on mitochondrial DNA sequences[J]. Chin Tradit Herb Drugs (中草药), 2016, 47: 1791–1796. |
| [9] | Guo YL, Ge S. The utility of mitochondrial nad 1 intron in phylogenetic study of Oryzeae with reference to the systematic position of Porteresia[J]. Acta Phytotax Sin (植物分类学报), 2004, 42: 333–344. |
| [10] | Zhang T, Wang ZT, Xu LS, et al. Application of mitochondrial nad 1 intron 2 sequences to molecular identification of some species of Dendrobium Sw[J]. Chin Tradit Herb Drugs (中草药), 2005, 36: 1059–1062. |
| [11] | Ni LH, Zhao ZL. A morphometric comparison of three closely related species of Gentiana (Gentianaceae), endemic to the region of the Qinghai-Tibet Plateau[J]. Botany, 2018, 96: 209–215. DOI:10.1139/cjb-2017-0166 |
| [12] | Ni LH, Zhao ZL, Xu HX, et al. Chloroplast genome structures in Gentiana (Gentianaceae), based on three medicinal alpine plants used in Tibetan herbal medicine[J]. Curr Genet, 2017, 63: 241–252. DOI:10.1007/s00294-016-0631-1 |
| [13] | Ni LH, Zhao ZL, Xu HX, et al. The complete chloroplast genome of Gentiana straminea (Gentianaceae), an endemic species to the Sino-Himalayan subregion[J]. Gene, 2016, 577: 281–288. DOI:10.1016/j.gene.2015.12.005 |
| [14] | Ni LH, Zhao ZL, Xiong B, et al. A strategy for identifying six species of Sect. Cruciata (Gentiana) in Gansu using DNA barcode sequences[J]. Acta Pharm Sin (药学学报), 2016, 51: 821–827. |
| [15] | Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf material[J]. Phytochem Bull, 1987, 19: 11–15. |
| [16] | Duminil J, Pemonge MH, Petit RJ. A set of 35 consensus primer pairs amplifying genes and introns of plant mitochondrial DNA[J]. Mol Ecol Notes, 2002, 2: 428–430. DOI:10.1046/j.1471-8286.2002.00263.x |
| [17] | Kumar S, Stecher G, Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33: 1870–1874. DOI:10.1093/molbev/msw054 |
| [18] | Wei SJ, Zhao ZL, Ni LH, et al. Taxonomic identification of Tibetan herb Bawo Sebo and its chloroplast genome structure[J]. Acta Pharm Sin (药学学报), 2018, 53: 1009–1015. |
| [19] | Yao H, Yang P, Zhou H, et al. Identification of medicinal plant Dendrobium based on the chloroplast psbK-psbI intergenic spacer[J]. Acta Pharm Sin (药学学报), 2015, 50: 783–787. |
| [20] | Xu J, Chu Y, Liao BS, et al. Panax ginseng genome examination for ginsenoside biosynthesis[J]. GigaScience, 2017, 6: 1–15. |
| [21] | Geng LX, Zheng R, Ren J, et al. Application of new type combined fragments:nrDNA ITS+nad 1-intron 2 for identification of Dendrobium species of Fengdous[J]. Acta Pharm Sin (药学学报), 2015, 50: 1060–1067. |
 2019, Vol. 54
2019, Vol. 54


