2. 安徽工程大学生物与化学工程学院, 微生物发酵安徽省工程技术中心, 安徽 芜湖 241000;
3. 浙江海正药业股份有限公司, 浙江省抗真菌药物重点实验室, 浙江 台州 318000
2. Microorganism Fermentation Engineering and Technology Research Center of Anhui Province, Institute of Biologic and Chemical Engineering of Anhui Polytechnic University, Wuhu 241000, China;
3. Zhejiang Key Laboratory of Antifungal Drugs, Zhejiang Hisun Pharmaceutical Co., Ltd., Taizhou 318000, China
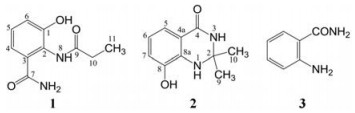
微生物次级代谢产物化学结构丰富、生物学功能多样, 在医药工业、食品工业、畜牧业和农业等领域具有重要的研究和开发价值[1, 2]。然而, 由于重复发现率不断升高, 使得采用传统手段从原有的微生物资源中发现新的次级代谢产物变得越来越困难[3-5]。因此, 开发新策略使微生物产生更多新的生物活性化合物的需求越来越迫切[6-8]。近些年来, 一些新的手段已经被尝试用于从已有的抗生素生产菌中寻找新的微生物次级代谢产物, 如:对全局性调控基因进行操作、多种菌株混合培养、抗生素抗性筛选等[9-11]。其中, 抗生素抗性筛选是利用一些作用于核糖体的特定抗生素进行耐药性诱变筛选, 得到的突变菌株的核糖体或RNA聚合酶会发生改变导致其基因表达发生变化, 因而可以提高次级代谢产物的产量或产生新的次级代谢产物[12-15]。在前期研究中, 本课题组从一株放线菌Streptomyces sp. HS-NF-1046中分离得到了新的环戊烯多取代衍生物hisunic acid [16]。为进一步挖掘该菌株的生物合成潜力, 本文采用利福霉素抗性筛选获得一株次级代谢产物的液相谱图发生变化的突变株, 对该菌株进行发酵, 然后通过提取分离与鉴定, 得到化合物1~3 (图 1), 其中化合物1和2为新化合物。
|
Figure 1 The chemical structures of compounds 1-3 |
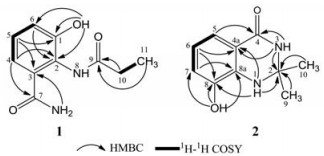
化合物1为白色粉末, HR-ESI-MS: m/z: 209.093 1 [M+H]+ (C10H13N2O3, 计算值为209.092 1), 提示化合物的分子式为C10H12N2O3, 计算其不饱和度为6。IR谱在3 443 cm-1和1 666 cm-1处的吸收峰提示分子中含有羟基和羰基。在1H NMR谱(表 1)中, δH 6.99 (1H, dd, J = 7.8, 1.6 Hz)、7.10 (1H, br d, J = 7.8 Hz)和7.13 (1H, t, J = 7.8 Hz)说明化合物1的结构中含有1个1, 2, 3-三取代的苯环; δH 1.09 (3H, t, J = 7.5 Hz)和2.37 (2H, m)提示化合物的结构中存在1个乙基; 4个化学位移为δH 10.29 (1H, br s)、9.70 (1H, s)、7.76 (1H, br s)、7.51 (1H, br s)的氢信号, 提示化合物中存在4个活泼氢。13C NMR和DEPT 135谱(表 1)显示, 化合物1中含有10个碳原子, 包括2个酰胺羰基碳原子(δC 174.4、170.3)、3个芳香族季碳原子(δC 151.8、130.9、124.8)、3个芳香族次甲基碳原子(δC 126.7、120.2、119.8)、1个脂肪族亚甲基碳原子(δC 29.6)和1个甲基碳原子(δC 10.0)。H3-11 (δH 1.09)和H2-10 (δH 2.37)的1H-1H COSY相关以及H3-11和H2-10与C-9 (δC 174.4)的HMBC相关, 说明化合物1中存在1个丙酰基。再结合化合物1的分子量, 4个活泼氢分别为1个氨基、1个亚氨基和1个羟基。通过比较化合物1和化合物3-hydroxy-2-N-iso-butyryl-anthranilamide的1H与13C NMR谱[17], 发现它们具有相似的化学结构, 其主要的区别在于化合物1中的丙酰基基团取代了3-hydroxy-2-N-iso-butyryl-anthranilamide上的异丁酰基团。同时, 在HMBC谱上(图 2), 羟基氢(δH 9.70, s)与C-1、C-2、C-6的相关关系表明该羟基位于1位, H-4与C-7、H-5与C-3的相关关系说明酰氨羰基C-7直接与C-3相连。在1H-1H COSY谱中(图 2), 两个活泼氢信号δH 7.76 (br s)和δH 7.51 (br s)存在相关信号, 能确定为CONH2基团中的两个氢信号。在HMBC谱中NH2与C-3的相关关系进一步证明了CONH2位于3位。因此, 结合化合物的分子式, 推导出丙酰基通过NH与苯环的2位相连。H-4、H-6与C-2, H-5与C-1的远程相关关系更进一步确证了1的化学结构。通过文献检索, 该化合物为新化合物, 名称为3-hydroxyl-2-N-propionyl-anthranilamide。
|
Figure 2 Key HMBC and 1H-1H COSY correlations of 1 and 2 |
| Table 1 1H and 13C NMR spectral data for 1 and 2 (DMSO-d6, J in Hz) |
化合物2为白色粉末, HR-ESI-MS: m/z: 193.096 8 [M+H]+ (C10H13N2O2, 计算值为193.097 2), 提示化合物的分子式为C10H12N2O2, 计算其不饱和度为6。在1H NMR谱中, 3个相互耦合的芳香质子信号δH 6.47 (1H, t, J = 7.8 Hz)、6.78 (1H, d, J =7.8 Hz)、7.10 (1H, d, J = 7.8 Hz), 提示化合物2中存在1个1, 2, 3-三取代的苯环。此外, 化合物2的高场区还存在两个甲基氢信号δH 1.39 (6H, s), 提示化合物2的结构中含有2个偕二甲基, 同时1H NMR谱中还显示有3个活泼氢信号δH 5.96 (1H, s)、7.95 (1H, s)、9.57 (1H, s)。化合物2的13C NMR, DEPT135 (表 1)及HMQC谱显示化合物2共有10个碳原子, 包括2个甲基碳原子δC 29.3, 1个脂肪族季碳原子δC 67.6, 1个酰胺羰基碳原子δC 163.7, 3个芳香族的次甲基碳原子δC 116.4、117.5、118.0和3个芳香族的季碳原子δC 115.0、136.9、144.4。通过比较, 化合物2和化合物2, 3-dihydro-5, 8-dimethoxy-2, 2-dimethyl quinazolin-4-(1H)-one的1H与13C NMR谱信号非常相似[18], 其不同点在于, 化合物2, 3-dihydro-5, 8-dimethoxy-2, 2-dimethyl quinazolin-4-(1H)-one结构中的5, 8-dimethoxy基团在化合物2中分别替换成了1个氢原子和1个羟基基团。化合物2中NH-1与C-2、C-4a、C-8, NH-3与C-2、C-4、C-4a, H-5与C-4, H-6与C-8、C-4a, H-7与C-8a, 8-OH与C-7、C-8和C-8a, 以及偕二甲基与C-2的HMBC相关更进一步确定了化合物2的结构。通过文献检索, 该化合物为新化合物, 名称为2, 3-dihydro-8-hydroxy-2, 2-dimethyl quinazolin-4-(1H)-one。
化合物3为淡黄色粉末, ESI-MS谱图给出相对分子质量为135, 结合化合物的1H NMR谱和13C NMR谱推出化合物的分子式为C7H8N2O。1H NMR谱中给出4组次甲基氢信号, 包括δH6.78 (1H, d, J = 8.2 Hz)、7.49 (1H, d, J = 7.88 Hz)、6.59 (1H, t, J = 6.96 Hz)、7.18 (1H, t, J = 7.16 Hz), 13C NMR谱中给出7个碳信号, 包括3个季碳信号δC 173.3、128.3、132.2, 4个次甲基碳信号δC 116.9、115.7、114.4、107.7。将上述数据与2-aminobenzamide的数据进行比较[19], 可以将化合物3确定为2-aminobenzamide。
实验部分仪器 Innova 5000摇床(New Brunswick Scientific, 美国); 50 L发酵罐(上海国强生化工程装备有限公司); Agilent 1100半制备HPLC, Zorbax SB-C18 (5 μm, 250 mm×9.4 mm)色谱柱(Agilent, 美国); Nicolet Magna FT-IR 750光谱仪(Nicolet, 日本); Varian CARY 300 BIO分光光度计(Varian, 美国); Bruker DRX-400核磁共振仪(Bruker, 日本); Q-TOF Micro LC-MS-MS质谱仪(Waters, 美国)。
试剂 丙酮、石油醚(国药集团化学试剂公司); 甲醇(临海市浙东特种试剂有限公司); 乙醇(安徽省安特食品有限公司)、乙酸乙酯(临安市青山化工试剂厂)、乙腈(Merck, 德国); 柱色谱硅胶、硅胶薄层色谱板(青岛海洋化工集团); Sephadex LH-20 (GE Healthcare, 英国); 氘代试剂(Cambridge Isotope Laboratory, 美国); 利福霉素(浙江海正药业股份有限公司); 麦芽抽提物(BD, 美国); 酵母抽提物(Oxoid, 英国); 琼脂(BD, 美国); 葡萄糖(国药集团化学试剂公司); 大豆粉(宁波北仑江南油脂公司); 甘露醇(青岛明月海藻集团有限公司)。
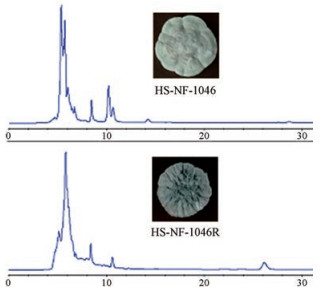
1 利福霉素抗性筛选原始菌株Streptomyces sp. HS-NF-1046在ISP2固体培养基上, 28 ℃生长7天。配制浓度分别为0.01、0.05、0.1、0.5、1、2.5、5和10 μg·mL-1的利福霉素平板, 将原始菌株的孢子悬液(1×108)分别涂布在不同浓度的抗生素平板上, 每个浓度设3个平行, 28 ℃生长4天后, 目测无菌落生长的最小浓度即为MIC (1 μg·mL-1)。再将原始菌株孢子液悬液(1×108)分别涂于1、2、4、8、16、32倍MIC浓度的利福霉素平板上, 28 ℃培养6~9天, 挑取生长出来的菌落, 共得到突变菌41株。将得到的突变菌株于对应利福霉素浓度的平板上划线, 28 ℃生长7天。然后刮取突变株孢子, 接种种子培养基(葡萄糖0.4%、酵母抽提物0.4%、麦芽抽提物1.0%、碳酸钙0.2%, pH 7.0~7.2; 250 mL摇瓶装量25 mL), 250 r·min-1摇床培养2天, 转接摇瓶发酵(大豆粉2.0%、甘露醇2.0%, pH 7.0~7.2; 250 mL摇瓶装量25 mL), 250 r·min-1摇床发酵7天。取摇瓶发酵液, 加入3倍体积甲醇浸泡, 超声30 min, 过滤取上清, 用氮吹浓缩后, 再用HPLC [色谱柱: Zorbax SB-C18 (5 μm, 250 mm×9.4 mm); 流动相: CH3CN/H2O (10:90, v/v); 流速: 1.5 mL·min-1; 检测波长: 220 nm]分析。最终发现一株菌落形态、孢子颜色发生明显变化的抗性突变株HS-NF-1046R, 其发酵产物HPLC分析图谱也发生了明显的变化(图 3)。
|
Figure 3 HPLC figure of the the strain HS-NF-1046 and the rifamycin resistant mutant strain HS-NF-1046R |
利福霉素抗性突变株HS-NF-1046R在ISP2固体培养基上, 28 ℃生长8天。刮取孢子进种子(葡萄糖0.4%、酵母抽提物0.4%、麦芽抽提物1.0%、碳酸钙0.2%, pH 7.0~7.2; 1000 mL摇瓶装量250 mL), 250 r·min-1摇床28 ℃培养2天。以5%接种量进50 L罐发酵(大豆粉2.0%、甘露醇2.0%, pH 7.0~7.2; 50 L罐装量30 L), 搅拌转速200 r·min-1, 通气量1 000 L·h-1, 罐压0.05 MPa, 28 ℃发酵7天, 最后得到发酵液30 L。
3 化合物分离提取发酵液经过滤得到菌丝体和上清液。其中, 菌丝体用3 L的纯化水冲洗, 冲洗液并入上清液, 然后将混合液用Diaion HP-20树脂吸附, 并用95%乙醇洗脱得到乙醇洗脱液; 另外, 菌丝体用3 L的甲醇浸泡过夜, 过滤得甲醇提取液。合并所得乙醇洗脱液和甲醇提取液, 在50 ℃减压将其浓缩至1 L, 然后用等体积的乙酸乙酯萃取3次得乙酸乙酯相。合并乙酸乙酯相, 并浓缩至干得到膏状物30 g。膏状物使用正相硅胶柱色谱(100~200目), 以石油醚/丙酮(10:0~60:40)梯度洗脱, 分别收集流分。各流分经薄层色谱检测, 合并得到4个组分(Fr.1~Fr.4)。组分Fr.3用半制备高压液相进行反相色谱分离[(洗脱剂: CH3CN-H2O, 10:90, v/v), 流速: 1.5 mL·min-1, 检测波长: 220 nm)], 得到化合物1 (tR 17 min, 6.4 mg)。组分Fr.4以上述相同的液相条件进行分离, 得到化合物2 (tR 27.8 min, 9.2 mg)和化合物3 (tR 26.1 min, 30.2 mg), 其中化合物3对应于HPLC谱图上约26 min处的新峰。
4 结构鉴定化合物1 白色粉末; UV (EtOH) λmax (log ε): 217 (4.36)、304 (3.50) nm; [α]D25 -136 (c 0.03, EtOH)。IR (KBr) vmax: 1 666、1 578、1 548、1 398、1 296、1 260、1 189、1 072、1 019、957 cm-1; HR-ESI-MS: m/z 209.093 1 [M+H]+ (计算值为209.092 1, C10H13N2O3)。1H和13C NMR数据见表 1。
化合物2 白色粉末; UV (EtOH) λmax (log ε): 220 (4.14)、348 (3.17) nm; [α]D25 -113 (c 0.05, EtOH)。IR (KBr) vmax: 3 356、1 644、1 574、1 517、1 419、1 265、1 204、1 073、1 011、950 cm-1; HR-ESI-MS: m/z 193.096 8 [M+H]+ (计算值为193.097 2, C10H13N2O2)。1H和13C NMR数据见表 1。
5 化合物抗肿瘤活性测定采用SRB法[20]评价新化合物对人肺癌细胞A549、人肝癌细胞HepG2、人结肠癌细胞HCT-116和白血病细胞K562的细胞毒活性, 结果显示, 化合物1和2对所有细胞的IC50值均大于100 μmol·L-1, 无明显的细胞毒性。
本研究表明通过利福霉素抗性筛选可以激活非常规的或处于沉默状态的次级代谢通路, 使突变株产生新的次级代谢产物, 有利于对已有的微生物资源进行深入挖掘。
| [1] | Katz L, Baltz RH. Natural product discovery:past, present, and future[J]. J Ind Microbiol Biotechnol, 2016, 43: 155–176. DOI:10.1007/s10295-015-1723-5 |
| [2] | Bérdy J. Bioactive microbial metabolites[J]. J Antibiot, 2005, 58: 1–26. DOI:10.1038/ja.2005.1 |
| [3] | Li JW, Vederas JC. Drug discovery and natural products:end of an era or an endless frontier?[J]. Science, 2009, 325: 161–165. DOI:10.1126/science.1168243 |
| [4] | Desbois AP. How might we increase success in marine-based drug discovery?[J]. Expert Opin Drug Discov, 2014, 9: 985–990. DOI:10.1517/17460441.2014.927863 |
| [5] | Peláez F. The historical delivery of antibiotics from microbial natural products-can history repeat?[J]. Biochem Pharmacol, 2006, 71: 981–990. DOI:10.1016/j.bcp.2005.10.010 |
| [6] | Chiang YM, Chang SL, Oakley BR, et al. Recent advances in awakening silent biosynthetic gene clusters and linking orphan clusters to natural products in microorganisms[J]. Curr Opin Chem Biol, 2011, 15: 137–143. DOI:10.1016/j.cbpa.2010.10.011 |
| [7] | Shi C, Sun Y, Xiao B, et al. Discovery of new antibiotics using genome data mining[J]. Acta Pharm Sin (药学学报), 2018, 53: 845–851. |
| [8] | Yan W, Ge HM, Wang G, et al. Pictet-spengler reaction-based biosynthetic machinery in fungi[J]. Proc Natl Acad Sci U S A, 2014, 111: 18138–18143. DOI:10.1073/pnas.1417304111 |
| [9] | Bok JW, Hoffmeister D, Maggio-Hall LA, et al. Genomic mining for aspergillus natural products[J]. Chem Biol, 2006, 13: 31–37. DOI:10.1016/j.chembiol.2005.10.008 |
| [10] | Hosaka T, Ohnishi-Kameyama M, Muramatsu H, et al. Antibacterial discovery in actinomycetes strains with mutations in RNA polymerase or ribosomal protein S12[J]. Nat Biotechnol, 2009, 27: 462–464. DOI:10.1038/nbt.1538 |
| [11] | Kurosawa K, Ghiviriga I, Sambandan TG, et al. Rhodostreptomycins, antibiotics biosynthesized following horizontal gene transfer from Streptomyces padanus to Rhodococcus fascians[J]. J Am Chem Soc, 2008, 130: 1126–1127. DOI:10.1021/ja077821p |
| [12] | Ochi K, Okamoto S, Tozawa Y, et al. Ribosome engineering and secondary metabolite production[J]. Adv Appl Microbiol, 2004, 56: 156–184. |
| [13] | Wu CJ, Yi L, Cui CB, et al. Activation of the silent secondary metabolite production by introducing neomycin-resistance in a marine-derived Pencicillium purpurogenum G59[J]. Mar Drugs, 2015, 13: 2465–2487. DOI:10.3390/md13042465 |
| [14] | Tanaka Y, Kasahara K, Hirose Y, et al. Activation and products of the cryptic secondary metabolite biosynthetic gene clusters by rifampin resistance (rpoB) mutations in actinomycetes[J]. J Bacteriol, 2013, 195: 2959–2970. DOI:10.1128/JB.00147-13 |
| [15] | Ochi K. From microbial differentiation to ribosome engineering[J]. Biosci Biotechnol Biochem, 2007, 7: 1373–1386. |
| [16] | Gao MY, Qi H, Li JS, et al. A new polysubstituted cyclopentene derivative from Streptomyces sp. HS-NF-1046[J]. J Antibiot, 2017, 70: 216–218. DOI:10.1038/ja.2016.111 |
| [17] | Chen GD, Gao H, Tang JS, et al. Benzamides and quinazolines from a mangrove actinomycetes Streptomyces sp. (No. 061316) and their inhibiting caspase-3 catalytic activity in vitro[J]. Chem Pharm Bull, 2011, 59: 447–451. DOI:10.1248/cpb.59.447 |
| [18] | Gould SJ, Eisenberg RL, Hillis LR, et al. Synthesis directed towards putative advanced intermediates in sarubicin A biosynthesis[J]. Tetrahedron, 1991, 47: 7209–7218. DOI:10.1016/S0040-4020(01)89723-4 |
| [19] | Roy AD, Subramanian A, Roy R. Auto-redox reaction:tin(Ⅱ) chloride-mediated one-step reductive cyclization leading to the synthesis of novel biheterocyclic 5, 6-dihydro-quinazolino[4, 3-b]quinazolin-8-ones with three-point diversity[J]. J Org Chem, 2010, 75: 3311–3316. DOI:10.1021/jo100345t |
| [20] | Skehan P, Storeng R, Scudiero D, et al. New colorimetric cytotoxicity assay for anticancer-drug screening[J]. J Natl Cancer Inst, 1990, 82: 1107–1112. DOI:10.1093/jnci/82.13.1107 |
 2019, Vol. 54
2019, Vol. 54


