2. 上海中医药大学, 上海 201203
2. Shanghai University of Traditional Chinese Medicine, Shanghai 201203, China
天然产物结构类型复杂且丰富, 具有广泛的生物活性, 是新药创新的重要来源。据统计, 近30年来美国食品药品监督管理局(FDA)在癌症治疗方面批准通过的药物中, 有近一半的药物直接或间接来自天然产物及其衍生物[1]。2015年, 源自天然产物的阿维菌素和青蒿素被授予诺贝尔医学奖或生理学奖, 预示着一个新的天然产物药物时代的到来[2]。
天然产物的靶点研究对于阐明药物作用机制和新药研发十分重要, 许多小分子化合物通过作用于多个靶点, 发挥相应的药理学作用[3, 4]。目前天然产物靶点识别的方法和技术取得了较大进展, Yue等[5]从基因组学、蛋白组学以及计算机辅助模拟等方面对天然产物靶点识别的传统方法、技术进行了详细的总结, 主要包括亲和色谱(affinity chromatography)[6]、活性蛋白质表达谱(activity based protein profiling, ABPP)[7-9]等。然而, 这些技术多需要对天然产物进行一定的化学结构修饰标记, 进而可能使天然产物丧失其功能活性。另外, 天然产物的化学结构修饰位点十分有限, 也成为该类方法识别天然产物靶点的干扰因素, 阻碍了天然产物的研究及其应用。鉴于需要标签修饰方法的不足, 近年来科学家们在天然产物上尝试了多种非标记方法(即无需对天然产物分子进行修饰的方法)并取得了一定的进展, 这些新方法主要包括直接法和间接法[10], 包含基因组学、蛋白组学、生物信息学等技术, 为天然产物靶点的识别和确证提供直接或者间接的证据。本文就对这些非标记方法进行综述, 为天然产物的靶点研究提供方法和思路。
1 直接法随着化学生物学[11-13]、基因组学[12]、蛋白组学[14, 15]、化学蛋白质组学[16]和生物信息学[17]等技术的发展成熟, 越来越多的新颖方法可以用于直接检测天然产物与靶蛋白的相互作用。这些技术主要通过采用药物与靶蛋白结合后使其稳定性发生改变的原理, 来识别天然产物可能作用的靶点。这些新颖的药物靶点检测技术包括药物亲和反应的靶点稳定性(drug affinity responsive target stability, DARTS)[18, 19]、氧化速率蛋白稳定性(stability of proteins from rates of oxidation, SPROX)[20, 21]、蛋白质热稳定性分析(cellular thermal shift assay, CETSA)[22, 23]、热蛋白质组分析(thermal proteome profiling, TPP)[24]等, 并且都得到了广泛的应用和认可(图 1)。

|
Figure 1 Direct methods for target identification of label-free natural products |
DARTS技术的概念由Lomenick等[18]于2009年首次提出并进行了相关研究。该研究组提出药物与靶蛋白结合后使蛋白结构更加稳定, 从而具有抗蛋白酶水解的特性。为了确证这一推测, 研究人员选取了与雷帕霉素和FK506抑制剂活性结合的FKBP12蛋白为研究对象, 采用枯草杆菌蛋白酶进行水解, 并通过SDS-PAGE观察到FKBP12与天然产物结合后的抗水解作用。该研究确证了药物与蛋白结合后更加稳定, 并建立了系统性靶点识别DARTS技术方法[18]。DARTS实验需要用特定的方法对酶解效果进行检测, 常用的方法有SDS-PAGE、胶染色技术(如考马斯亮蓝染色)等, 此外也可以运用蛋白二维电泳(2D-PAGE)、凝胶或非凝胶质谱(LC-MS/MS)等方法进行检测。通过比对, 识别药物组与对照组酶解的明显差异, 进而找到药物的候选靶点进行后续实验确证[19]。由于DARTS技术操作简单、耗时短, 得到了广泛的应用。
Kost等[25]利用DARTS技术证实了哌嗪类小分子RX-5902通过与磷酸化的p68RNA螺旋酶相结合, 进而下调c-Myc等基因的表达, 抑制癌细胞的生长, 揭示了其抗癌机制。Jung等[26]应用DARTS技术识别KRIBB53与OCT4结合并使之降解, 从而使OCT4-阳性的睾丸生殖细胞癌细胞发生凋亡。Tanabe等[27]利用DARTS技术发现苦参碱可直接激活细胞外的热休克蛋白90 (heat shock protein 90, HSP90), 进而增强脊髓损伤小鼠的轴突生长和功能恢复, 证实了DARTS的可行性。DARTS技术不需要对天然产物进行任何化学修饰, 为确定天然产物直接结合的靶点提供了便利, 但DARTS鉴定蛋白丰度较低的靶点能力十分有限[18]。DARTS实验中蛋白酶解所需的蛋白酶, 以及细胞裂解液的选择均会影响天然产物靶点识别[28], 干扰DARTS技术的准确性。
1.2 SPROX与DARTS类似, SPROX是另一种基于配体诱导的靶点稳定性的检测方法。不同于DARTS技术检测蛋白酶解变化, SPROX主要检测靶蛋白的甲硫氨酸的氧化水平, 利用的原理是药物与靶点结合后可以增加靶蛋白的抗氧化能力[21]。当蛋白复合物与天然产物孵育后, 在化学变性剂存在下使用过氧化氢等氧化剂氧化蛋白。然后采用不同类型的质谱技术(如LC-MS/MS)对选择性氧化的甲硫氨酸进行定量。这种技术利用甲硫氨酸残基的氧化速率分析蛋白质折叠或展开反应的热力学性质[21], 能够检测分析与药物相互作用的多个靶点蛋白, 亦能够对天然产物结合靶点蛋白的亲和力进行定量[29]。
DeArmond等[20]应用SPROX技术寻找中药活性成分白藜芦醇的靶点, 发现除了之前已经确定的胞质脱氢酶, 白藜芦醇还存在6个潜在的靶点蛋白。Wallace等[30]将SPROX技术与iTRAQ和质谱技术结合, 鉴定出天然产物Manassantin A的21个靶点蛋白, 为阐明其作用机制奠定了基础。Xu等[31]运用SPROX技术对具有良好抗肿瘤活性的天然产物格尔德霉素和其已知靶点HSP90的亲和力进行了研究, 证实了解离常数(Kd值)取决于在SPROX分析之前格尔德霉素与裂解物平衡的时间。虽然SPROX技术具有较好的应用潜力, 但也存在一些不足: SPROX需要甲硫氨酸残基的氧化来测量热力学变化, 但不同的甲硫氨酸残基可能表现出相同的氧化速率, 不足以为天然产物配体与靶点蛋白相互作用的确证提供充分的信息[28]。
1.3 CETSACETSA技术由Molina等[22]于2013年首次提出并使用, 这种方法可以用于活细胞、细胞裂解液及动物组织样本中药物与靶点结合的确证等研究。与上述方法相似, CETSA主要基于天然产物与靶蛋白结合后热力学稳定性发生变化的原理。将天然产物与样本孵育之后, 在不同的温度梯度下分别进行加热。天然产物结合的靶蛋白热稳定性较高, 未结合的蛋白稳定性较弱而较容易发生降解, 之后利用免疫印迹(Western-Blot)或者基于质谱的方法根据其熔解曲线分析可溶性蛋白的热稳定性, 从而确证天然产物与细胞内蛋白的结合[22, 23]。
Wang等[32]利用CETSA结合分子对接和质谱等技术, 鉴定到从中药莪术中提取得到的具有多种生物活性的天然产物莪术醇, 能与鼻咽癌(NPC)细胞中的NCL蛋白结合并使之降解, 从而发挥抗癌作用, 证实了NCL是莪术醇治疗鼻咽癌的靶点之一。Jin等[33]将CETSA技术和DARTS技术结合, 对天然产物geranylnaringenin (CG902)与STAT3之间的关系进行了确证, 发现CG902通过激活SHP-2来抑制STAT3活性, 进而在癌症治疗中发挥作用。CETSA亦可以用于动物组织中, Ishii等[34]在小鼠的脾脏和脑中利用CETSA成功地评估了小鼠RIPK1与药物的靶点结合(target engagement), 表明CETSA能够有效地应用到临床前和临床药物开发中并为之提供有效的工具。但CETSA需要的药物浓度高, 且常用于靶点的确定, 对于发现新靶点仍存在通量不足的缺点。
1.4 TPP为了克服CETSA灵敏度不高和通量不足的缺点和挑战, 增加其在靶点和脱靶识别以及生物标志物识别上的应用, Savitski等[24]于2014年在CETSA的基础上开发了TPP技术。将天然产物和溶剂对照分别与活细胞或细胞裂解液孵育之后, 在10个温度下分别进行加热, 对未变性的可溶性蛋白样品加以TMT10 (isobaric tandem mass tag 10-plex)孵育, 进行LC-MS/MS检测分析, 经过数据标准化之后绘制熔解曲线, 从而找到稳定性差异的蛋白。Franken等[35]发现TPP在37~67 ℃的温度范围内能够无偏地分析天然产物与靶点蛋白的直接或间接结合, 用细胞提取液与药物孵育可检测与天然产物的直接结合, 在完整细胞上进行的TPP通过与细胞提取液水平上进行的TPP结果比对可获知天然产物的下游靶点[24]。TPP方法的关键是对样品进行TMT10孵育以及生物信息学分析[35]。
Savitski等[24]最初将TPP应用到从细菌中提取到的天然产物星苞菌素的靶点确证, 并识别了超过50个的靶点蛋白。Miettinen等[36]利用TPP技术发现帕博西尼(palbociclib)可增加20S蛋白酶体的热稳定性并使ECM29减少, 从而使细胞发生衰老, 解释了这种CDK4/6抑制剂治疗乳腺癌导致细胞衰老的机制, 为天然产物的靶点研究提供了思路。TPP技术具有稳定性好, 鉴定蛋白数量多的优点, 而且不需要用抗体进行孵育检测, 是一种广谱的蛋白鉴定技术。但TPP存在耗时长[35]、花费高、对膜蛋白的检测有限[24, 37]等问题。
2 间接法近年来用于间接识别和确证非标记天然产物与靶点结合的方法也逐渐发展起来。如降解标签(degradation tag, dTAG)[38]等蛋白降解技术, RNA干扰(RNA interface, RNAi)[39-42]、成簇的规则间隔短回文重复序列(clustered regularly interspaced short palindrome repeats, CRISPR)/Cas9基因编辑技术[43-47]等基因组学的方法, 以及Connectivity Map (CMAP)[48]等基于生物信息学分析的方法(图 2)。这些技术主要从生理反应或生化特征的改变推断药物靶点。下文主要对这几种方法进行阐述。
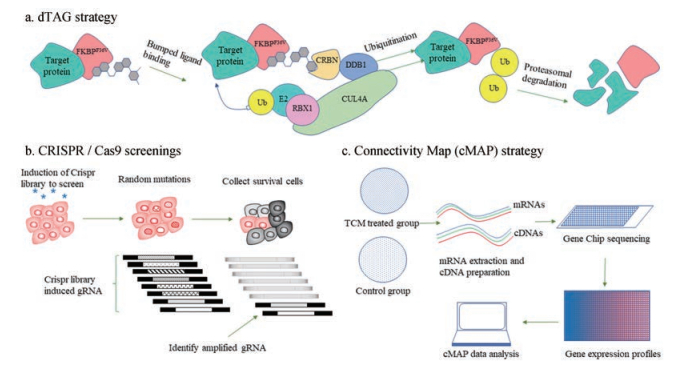
|
Figure 2 Indirect methods for target identification of label-free natural products |
之前曾报道过基于邻苯二甲酰亚胺(phthalimide)结合的蛋白降解技术[49]以及靶向嵌合体的蛋白水解(proteolysis targeting chimeras, PROTACs)等蛋白降解技术[50], 但这两种技术需要确定目标蛋白的特定降解配体, 不利于天然产物的靶点识别和确证。You等[51]对之前开发的多种靶向诱导蛋白降解技术进行了总结, 发现越来越多的蛋白降解技术已经应用到天然产物靶点的识别和确证中。Nabet等[38]最近发明了一项名为dTAG的蛋白降解技术, 可以作为药物靶点识别和确证的重要工具。该技术利用CRISPR把一个外源变异非天然蛋白FKBP12F36V与需降解的目标蛋白融合, 然后用过膜性较好、可以与FKBP12F36V和E3链接酶(cereblon, CRBN)选择性结合的dTAG化合物降解这个融合蛋白并进行生物学研究。dTAG技术是一种广谱的蛋白降解技术, 可使用一种dTAG分子对广谱蛋白进行精准地降解。Nabet等[38]证明dTAG-13是一个选择性高、快速高效的dTAG分子。对于天然产物的候选靶点, 可以用dTAG分子进行降解以进行分析确证。dTAG可以应用于细胞、组织和生物体, 为天然产物靶点的识别和确证提供了十分强大的工具。
2.2 基因组学方法近年来, 基于RNA的技术在药物靶点确证中取得了较大进展。RNAi包括shRNA (short-hairpin RNA)和siRNA (small-interfering RNA), 被用作基因敲除工具以确证天然产物与靶点的相互作用及药靶基因的筛选[40-42]。通过基因敲除的方法操纵靶点蛋白的表达水平, 从而确证天然产物与该靶蛋白的相互作用。近来这些技术与高通量的方法结合扩大了这些基因组学技术的应用范围, 使得单次实验便可沉默成百上千个基因, 成为了间接识别天然产物靶点的有力工具。尽管RNAi在天然产物靶点识别中有非常广阔的应用前景, 其存在的脱靶效应和假阳性结果等缺点仍需要克服[52]。
CRISPR/Cas9是近年来发展起来的一种基因编辑技术, 包括CRISPRa和CRISPRi。与RNAi技术相比, CRISPR/Cas9技术能够有效地克服脱靶效应[53]。2014年Shalem[43]和Wang[44]两个课题组分别报道了可以用于天然产物靶点识别的CRISPR文库, 其在天然产物的靶点筛选中具有更高的效率。目前, 科学家们至少建立了13个可用于人的CRISPR敲除文库, 3个CRISPRa文库和2个CRISPRi文库。这些文库不仅可以用于药物靶点的筛选, 也可以用于临床研究[46]。
用CRISPR文库随机诱变后, 将天然产物或药物如抗癌药与细胞孵育, 收集耐药的细胞, 分析其向导RNA (guide RNA, gRNA)序列并扩增富集, 富集得到的gRNA即被预测为药物的靶基因。为使筛选得到的候选靶基因有意义, 维持gRNA的多样性及Cas9的活性十分重要[46]。Kasap等[54]将CRISPR/Cas9与高通量测序、计算突变识别(computational mutation discovery)结合开发了一种被命名为DrugTargetSeqR的新技术, 并对一种已经进入临床试验的抗癌药ispinesib进行了实验。实验将kinesin-5鉴定为ispinesib的候选靶基因之一, 与前期文献报道一致[55]。Wu等[56]用CRISPR/Cas9技术发现来源于中药补骨脂的活性分子isobavachalcone, 可以通过抑制二氢乳清酸脱氢酶(DHODH)调节和抑制急性髓性白血病细胞的凋亡和分化, 证实了该方法的可行性。
2.3 Connectivity Map (CMAP)等基于生物信息学分析方法联通图谱CMAP是由博德研究所于2006年建立的基于基因表达谱数据的生物应用数据库(http://www.broadinstitute.org)[48]。CMAP数据库基于图形匹配的原理, 通过一个大型的特征基因表达图谱数据库, 建立了药物、基因与疾病之间的相互关联。该数据库于2017年进行了更新[57], 现有约5 000个生物活性小分子处理人细胞后的1 500 000多种全基因组转录数据。CMAP数据库利用基因芯片技术, 获取不同小分子药物的基因表达图谱, 通过对比分析不同样本的表达谱数据, 可以广泛应用于老药新用、新药发现、药物作用机制推测等领域[58, 59]。
近几年CMAP数据库也逐渐应用到中药及天然产物领域, 主要应用于天然产物小分子靶点识别、揭示中药复方作用机制等[60]。2017年, 本课题组Lv等[61]在CMAP数据库的基础上, 建立了国内首个天然产物小分子基因表达谱数据库平台, 通过与CMAP结合使用可以用于预测天然产物小分子的药理学活性、天然产物分子靶点及通路识别和天然产物新药创制等领域。该数据库平台初步选取常见中药的102个小分子处理标准化细胞, 获得基因表达谱与CMAP数据库相结合对比分析, 并成功应用于天然产物氯化两面针碱, 发现其具有阻断α肾上腺素受体降血压的新功能[61]。此外, Lv等[62]还应用该数据库平台与CMAP数据库结合分析, 发现丹参酮IIA可以选择性作用于PKCζ和PKCε靶点, 为理解丹参酮IIA的抗肿瘤作用机制提供了新的思路。
3 总结与展望中药和天然产物是新药研究中的重要宝库, 但由于对天然产物的靶点研究尚不明确, 严重阻碍了中药的发展和国际化。阐明天然产物-靶点的相互作用对于理解天然产物的作用机制和疗效十分重要, 寻找新的靶点识别和确证方法刻不容缓。相对于标记天然产物的方法, 非标记天然产物的方法可以最大程度地保留天然产物的结构和活性, 扩大了其应用范围。
在现代药物研发领域中, 无化学修饰的靶点识别技术目前已得到广泛应用, 但仍存在一些不足。在靶点信息不明确的情况下, 首先需要前期大量实验对药物的孵育浓度、孵育时间和温度等条件进行摸索确定, 过程较为繁琐。其次, 靶点对非标记天然产物的敏感性问题也是重要的限制因素。例如在DARTS实验中, 某些蛋白与天然产物结合后, 也有可能会被蛋白酶水解, 使实验结果假阴性率较高[63]。在CETSA和TPP技术中, 也存在天然产物结合靶点使靶点蛋白热稳定性变差的情况, 使许多靶点未被检出, 造成实验的误差。此外, 从组学产生的大量数据中找到相应天然产物的靶点信息并非易事[64]。
一种较好的靶点识别和确证的方法, 应该满足以下条件:其一是能最大程度地保持天然产物的结构和活性, 不会导致活性的降低或者丧失; 其二是具有广谱的适用性, 不仅适用于高丰度蛋白, 也适用于含量较低的蛋白, 并适用于不同的组织、细胞、有机体等。随着蛋白质组学、基因组学和生物信息学以及质谱技术的发展和更新, 更多的新技术将会整合不同技术的优点, 降低检测的花费和时间。这些新技术除了应用于天然产物的靶点识别外, 也将更加明确地阐明药物的作用机制及毒性, 为解决新药研发、药物机制研究、人类疾病标志物识别等难题提供重要的方法基础。
| [1] | Newman DJ, Cragg GM. Natural products as sources of new drugs from 1981 to 2014[J]. J Nat Prod, 2016, 79: 629–661. DOI:10.1021/acs.jnatprod.5b01055 |
| [2] | Shen B. A new golden age of natural products drug discovery[J]. Cell, 2015, 163: 1297–1300. DOI:10.1016/j.cell.2015.11.031 |
| [3] | Paolini GV, Shapland RH, van Hoorn WP, et al. Global mapping of pharmacological space[J]. Nat Biotechnol, 2006, 24: 805–815. DOI:10.1038/nbt1228 |
| [4] | Mohamad SZ, Mohd F, Liggi S, et al. Global mapping of traditional Chinese medicine into bioactivity space and pathways annotation improves mechanistic understanding and discovers relationships between therapeutic action (sub) classes[J]. Evid Based Complement Alternat Med, 2016, 2016: 2106465. |
| [5] | Yue R, Shan L, Yang X, et al. Approaches to target profiling of natural products[J]. Curr Med Chem, 2012, 19: 3841–3855. DOI:10.2174/092986712801661068 |
| [6] | Koshelev IuA. Affinity chromatography and proteomic screening as the effective method for S100A4 new protein targets discovery[J]. Mol Biol (Mosk), 2014, 48: 868–872. |
| [7] | Niphakis MJ, Cravatt BF. Enzyme inhibitor discovery by activity-based protein profiling[J]. Annu Rev Biochem, 2014, 83: 341–377. DOI:10.1146/annurev-biochem-060713-035708 |
| [8] | Zweerink S, Pollmann T, Ninck S, et al. Activity-based protein profiling with natural product-derived chemical probes in human cell lysates[J]. Methods Mol Biol, 2017, 1491: 23–46. DOI:10.1007/978-1-4939-6439-0 |
| [9] | Chen X, Wong YK, Wang J, et al. Target identification with quantitative activity based protein profiling (ABPP)[J]. Proteomics, 2017, 17: 1600212. DOI:10.1002/pmic.v17.3-4 |
| [10] | Chang J, Kim Y, Kwon HJ. Advances in identification and validation of protein targets of natural products without chemical modification[J]. Nat Prod Rep, 2016, 33: 719–730. DOI:10.1039/C5NP00107B |
| [11] | Schenone M, Dancik V, Wagner BK, et al. Target identification and mechanism of action in chemical biology and drug discovery[J]. Nat Chem Biol, 2013, 9: 232–240. DOI:10.1038/nchembio.1199 |
| [12] | Lee H, Lee JW. Target identification for biologically active small molecules using chemical biology approaches[J]. Arch Pharm Res, 2016, 39: 1193–1201. DOI:10.1007/s12272-016-0791-z |
| [13] | Long MJ, Poganik JR, Ghosh S, et al. Subcellular redox targeting:bridging in vitro and in vivo chemical biology[J]. ACS Chem Biol, 2017, 12: 586–600. DOI:10.1021/acschembio.6b01148 |
| [14] | Sleno L, Emili A. Proteomic methods for drug target discovery[J]. Curr Opin Chem Biol, 2008, 12: 46–54. DOI:10.1016/j.cbpa.2008.01.022 |
| [15] | Chernobrovkin A, Marin-Vicente C, Visa N, et al. Functional Identification of Target by Expression Proteomics (FITExP) reveals protein targets and highlights mechanisms of action of small molecule drugs[J]. Sci Rep, 2015, 5: 11176. DOI:10.1038/srep11176 |
| [16] | Yang HQ, Li XJ. Chemical proteomics and discovery of drug targets[J]. Acta Pharm Sin (药学学报), 2011, 46: 877–882. |
| [17] | Haupt VJ, Schroeder M. Old friends in new guise:repositioning of known drugs with structural bioinformatics[J]. Brief Bioinform, 2011, 12: 312–326. DOI:10.1093/bib/bbr011 |
| [18] | Lomenick B, Hao R, Jonai N, et al. Target identification using drug affinity responsive target stability (DARTS)[J]. Proc Natl Acad Sci U S A, 2009, 106: 21984–21989. DOI:10.1073/pnas.0910040106 |
| [19] | Lomenick B, Jung G, Wohlschlegel JA, et al. Target identification using drug affinity responsive target stability (DARTS)[J]. Curr Protoc Chem Biol, 2011, 3: 163–180. |
| [20] | DeArmond PD, Xu Y, Strickland EC, et al. Thermodynamic analysis of protein-ligand interactions in complex biological mixtures using a shotgun proteomics approach[J]. J Proteome Res, 2011, 10: 4948–4958. DOI:10.1021/pr200403c |
| [21] | Strickland EC, Geer MA, Tran DT, et al. Thermodynamic analysis of protein-ligand binding interactions in complex biological mixtures using the stability of proteins from rates of oxidation[J]. Nat Protoc, 2013, 8: 148–161. DOI:10.1038/nprot.2012.146 |
| [22] | Molina MD, Jafari R, Ignatushchenko M, et al. Monitoring drug target engagement in cells and tissues using the cellular thermal shift assay[J]. Science, 2013, 341: 84–87. DOI:10.1126/science.1233606 |
| [23] | Jafari R, Almqvist H, Axelsson H, et al. The cellular thermal shift assay for evaluating drug target interactions in cells[J]. Nat Protoc, 2014, 9: 2100–2122. DOI:10.1038/nprot.2014.138 |
| [24] | Savitski MM, Reinhard FB, Franken H, et al. Tracking cancer drugs in living cells by thermal profiling of the proteome[J]. Science, 2014, 346: 1255784. DOI:10.1126/science.1255784 |
| [25] | Kost GC, Yang MY, Li L, et al. A novel anti-cancer agent, 1-(3, 5-dimethoxyphenyl)-4-[J]. J Cell Biochem, 2015, 116: 1595–1601. DOI:10.1002/jcb.v116.8 |
| [26] | Jung J, Kim Y, Song J, et al. KRIBB53 binds to OCT4 and enhances its degradation through the proteasome, causing apoptotic cell death of OCT4-positive testicular germ cell tumors[J]. Carcinogenesis, 2018, 39: 838–849. DOI:10.1093/carcin/bgy054 |
| [27] | Tanabe N, Kuboyama T, Tohda C. Matrine directly activates extracellular heat shock protein 90, resulting in axonal growth and functional recovery in spinal cord injured-mice[J]. Front Pharmacol, 2018, 9: 446. DOI:10.3389/fphar.2018.00446 |
| [28] | Lomenick B, Olsen RW, Huang J. Identification of direct protein targets of small molecules[J]. ACS Chem Biol, 2011, 6: 34–46. DOI:10.1021/cb100294v |
| [29] | West GM, Tucker CL, Xu T, et al. Quantitative proteomics approach for identifying protein-drug interactions in complex mixtures using protein stability measurements[J]. Proc Natl Acad Sci U S A, 2010, 107: 9078–9082. DOI:10.1073/pnas.1000148107 |
| [30] | Wallace MA, Kwon DY, Weitzel DH, et al. Discovery of manassantin A protein targets using large-scale protein folding and stability measurements[J]. J Proteome Res, 2016, 15: 2688–2696. DOI:10.1021/acs.jproteome.6b00237 |
| [31] | Xu Y, Wallace MA, Fitzgerald MC. Thermodynamic analysis of the geldanamycin-Hsp90 interaction in a whole cell lysate using a mass spectrometry-based proteomics approach[J]. J Am Soc Mass Spectrom, 2016, 27: 1670–1676. DOI:10.1007/s13361-016-1457-2 |
| [32] | Wang J, Wu J, Li X, et al. Identification and validation nucleolin as a target of curcumol in nasopharyngeal carcinoma cells[J]. J Proteomics, 2018, 182: 1–11. DOI:10.1016/j.jprot.2018.04.025 |
| [33] | Jin Y, Yoon YJ, Jeon YJ, et al. Geranylnaringenin (CG902) inhibits constitutive and inducible STAT3 activation through the activation of SHP-2 tyrosine phosphatase[J]. Biochem Pharmacol, 2017, 142: 46–57. DOI:10.1016/j.bcp.2017.06.131 |
| [34] | Ishii T, Okai T, Iwatani-Yoshihara M, et al. CETSA quantitatively verifies in vivo target engagement of novel RIPK1 inhibitors in various biospecimens[J]. Sci Rep, 2017, 7: 13000. DOI:10.1038/s41598-017-12513-1 |
| [35] | Franken H, Mathieson T, Childs D, et al. Thermal proteome profiling for unbiased identification of direct and indirect drug targets using multiplexed quantitative mass spectrometry[J]. Nat Protoc, 2015, 10: 1567–1593. DOI:10.1038/nprot.2015.101 |
| [36] | Miettinen TP, Peltier J, Hartlova A, et al. Thermal proteome profiling of breast cancer cells reveals proteasomal activation by CDK4/6 inhibitor palbociclib[J]. EMBO J, 2018, 37: e98359. DOI:10.15252/embj.201798359 |
| [37] | Reinhard FB, Eberhard D, Werner T, et al. Thermal proteome profiling monitors ligand interactions with cellular membrane proteins[J]. Nat Methods, 2015, 12: 1129–1131. DOI:10.1038/nmeth.3652 |
| [38] | Nabet B, Roberts JM, Buckley DL, et al. The dTAG system for immediate and target-specific protein degradation[J]. Nat Chem Biol, 2018, 14: 431–441. DOI:10.1038/s41589-018-0021-8 |
| [39] | Ngo VN, Davis RE, Lamy L, et al. A loss-of-function RNA interference screen for molecular targets in cancer[J]. Nature, 2006, 441: 106–110. DOI:10.1038/nature04687 |
| [40] | Wittrup A, Ai A, Liu X, et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown[J]. Nat Biotechnol, 2015, 33: 870–876. DOI:10.1038/nbt.3298 |
| [41] | Henderson MC, Azorsa DO. High-throughput RNAi screening for the identification of novel targets[J]. Methods Mol Biol, 2013, 986: 89–95. DOI:10.1007/978-1-62703-311-4 |
| [42] | Ngo VN. Identification of pathogenetically relevant genes in lymphomagenesis by shRNA library screens[J]. Methods Mol Biol, 2013, 971: 245–263. DOI:10.1007/978-1-62703-269-8 |
| [43] | Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343: 84–87. DOI:10.1126/science.1247005 |
| [44] | Wang T, Wei JJ, Sabatini DM, et al. Genetic screens in human cells using the CRISPR-Cas9 system[J]. Science, 2014, 343: 80–84. DOI:10.1126/science.1246981 |
| [45] | Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8: 2281–2308. DOI:10.1038/nprot.2013.143 |
| [46] | Kurata M, Yamamoto K, Moriarity BS, et al. CRISPR/Cas9 library screening for drug target discovery[J]. J Hum Genet, 2018, 63: 179–186. DOI:10.1038/s10038-017-0376-9 |
| [47] | Lu D, Li L, Deng XM. CRISPR/Cas9 and its application in drugs development[J]. Acta Pharm Sin (药学学报), 2018, 53: 11–20. |
| [48] | Lamb J, Crawford ED, Peck D, et al. The connectivity map:using gene-expression signatures to connect small molecules, genes, and disease[J]. Science, 2006, 313: 1929–1935. DOI:10.1126/science.1132939 |
| [49] | Winter GE, Buckley DL, Paulk J, et al. Drug development[J]. Science, 348: 1376–1381. |
| [50] | Bondeson DP, Mares A, Smith IE, et al. Catalytic in vivo protein knockdown by small-molecule PROTACs[J]. Nat Chem Biol, 2015, 11: 611–617. DOI:10.1038/nchembio.1858 |
| [51] | You QD, Lu MC, Jiang ZY. Protein degradation as an innovative strategy in drug discovery[J]. Acta Pharm Sin (药学学报), 2017, 52: 1777–1782. |
| [52] | Marine S, Bahl A, Ferrer M, et al. Common seed analysis to identify off-target effects in siRNA screens[J]. J Biomol Screen, 2012, 17: 370–378. DOI:10.1177/1087057111427348 |
| [53] | Cho SW, Kim S, Kim Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Res, 2014, 24: 132–141. DOI:10.1101/gr.162339.113 |
| [54] | Kasap C, Elemento O, Kapoor TM. DrugTargetSeqR:a genomics-and CRISPR-Cas9-based method to analyze drug targets[J]. Nat Chem Biol, 2014, 10: 626–628. DOI:10.1038/nchembio.1551 |
| [55] | Luo L, Carson JD, Molnar KS, et al. Conformation-dependent ligand regulation of ATP hydrolysis by human KSP:activation of basal hydrolysis and inhibition of microtubule-stimulated hydrolysis by a single, small molecule modulator[J]. J Am Chem Soc, 2008, 130: 7584–7591. DOI:10.1021/ja710889h |
| [56] | Wu D, Wang W, Chen W, et al. Pharmacologic inhibition of dihydroorotate dehydrogenase induces apoptosis and differentiation in acute myeloid leukemia cells[J]. Haematologica, 2018: 188185. DOI:10.3324/haematol.2018.188185 |
| [57] | Subramanian A, Narayan R, Corsello SM, et al. A next generation connectivity map:L1000 platform and the first 1000000 profiles[J]. Cell, 2017, 171: 1437–1452. DOI:10.1016/j.cell.2017.10.049 |
| [58] | Zhang XF, Kang YB, Su JH, et al. Application of connectivity map technology in traditional Chinese medicines[J]. J Zhejiang Univ (Agric Life Sci) (浙江大学学报·农业与生命科学版), 2016, 42: 543–550. |
| [59] | Qu XA, Rajpal DK. Applications of connectivity map in drug discovery and development[J]. Drug Discov Today, 2012, 17: 1289–1298. DOI:10.1016/j.drudis.2012.07.017 |
| [60] | Lv C, Wang YC, Liu RH, et al. Application of connectivity map database to research on Chinese materia medica[J]. Chin Herb Med, 2016, 8: 117–120. DOI:10.1016/S1674-6384(16)60019-9 |
| [61] | Lv C, Wu X, Wang X, et al. The gene expression profiles in response to 102 traditional Chinese medicine (TCM) components:a general template for research on TCMs[J]. Sci Rep, 2017, 7: 352. DOI:10.1038/s41598-017-00535-8 |
| [62] | Lv C, Zeng HW, Wang JX, et al. The antitumor natural product tanshinone ⅡA inhibits protein kinase C and acts synergistically with 17-AAG[J]. Cell Death Dis, 2018, 9: 165. DOI:10.1038/s41419-017-0247-5 |
| [63] | Wang L, Xiao ZY, Zhou WX, et al. Exploration of the experimental conditions of drug affinity responsive target stability[J]. Int J Pharm Res (国际药学研究杂志), 2016, 43: 485–490. |
| [64] | Saxena C. Identification of protein binding partners of small molecules using label-free methods[J]. Expert Opin Drug Discov, 2016, 11: 1017–1025. DOI:10.1080/17460441.2016.1227316 |
 2019, Vol. 54
2019, Vol. 54


