2. 江苏理工学院电气信息工程学院生物信息与医药工程研究所, 江苏 常州 213001
2. Institute of Bioinformatics and Medical Engineering, School of Electrical and Information Engineering, Jiangsu University of Technology, Changzhou 213001, China
掌叶大黄Rheum palmatum L.、唐古特大黄R. tanguticum Maxim. ex Balf.和药用大黄R. offcinale Baill.为蓼科大黄属多年生高大草本, 为2015版《中华人民共和国药典》收录的大黄三基源, 其干燥根及根茎药用, 始载于《神农本草经》, 性寒、味苦, 具有泻下攻积、清热泻火、凉血解毒、逐瘀通经、利湿退黄之功效, 常用于实热积滞便秘、血热吐衄、目赤咽肿等症的临床治疗[1]。现代研究揭示大黄主要含有蒽醌类、酚类、萘苷类、酰基糖苷类、二苯乙烯类及鞣质等多种成分, 蒽醌类化合物为其主要活性成分, 具有抗肿瘤[2]、抗炎[3]及保肝[4]等药理活性。以大黄药材或蒽醌类成分为原料生产的药物就有数百种, 大黄的临床应用非常广泛。
作为正品大黄的一个重要来源, 掌叶大黄资源丰富, 质量最佳, 产量、市场份额显著高于其他两种。当前, 大量研究已在掌叶大黄分子鉴别、质量评价、活性成分等方面取得重要进展。叶绿体基因matK序列的差异可在分子水平鉴别掌叶大黄真伪[5]。掌叶大黄质量与产地、生长年限、气压、湿度或温度等生态因素密切相关[6, 7]。在生长发育进程中, 蒽醌类在掌叶大黄根中的累积含量大于叶柄和叶片[8]。这些研究说明植物生理生态调控作用在大黄活性成分合成与积累方面起重要作用。然而, 目前尚不清楚大黄蒽醌类代谢通路及调控机制, 就无法科学阐明大黄药材品质形成的内在生物学规律。蒽醌类主要成分又是大黄药材质量控制的关键指标, 利用现代分子生物学技术解析大黄蒽醌类生物合成通路及表达调控就显得更为迫切。
转录组测序技术是基因组与蛋白组的桥梁, 能够从整体水平研究特定生物样本在某一生理状态下所有基因转录本全局信息, 揭示特定条件下生物体生长发育、次生代谢及生理适应等方面的分子机制[9]。随着本草基因组学的迅速发展, 基于高通量测序技术的转录组分析策略广泛用于药用植物功能基因组研究, 现已解析柴胡[10]、人参[11]和商陆[12]等众多大宗药材的转录组特征, 为阐明中药种质资源遗传基础奠定了重要基础。本研究在明确掌叶大黄幼苗活性成分含量的基础上, 利用二代高通量测序平台Illumina HiSeqTM 2000 150PE进行幼苗转录组测序分析, 挖掘蒽醌类的生物合成通路及调控方面的遗传信息, 为掌叶大黄品质形成机制及大黄质量控制研究提供科学依据。
材料与方法植物材料 掌叶大黄种子系课题组2017年11月采自甘肃省宕昌县阿坞乡麻界村, 东经104°10'6.5"、西经34°16'51.98", 海拔2 377 m, 经陕西中医药大学张岗教授鉴定为掌叶大黄R. palmatum L.。将大黄种子用沙土(1:4)栽种至花盆中, 置于20 ± 2 ℃的温室, 光照16 h, 黑暗8 h, 培养2个月取幼苗备用。
仪器和试药 Waters 2695高效液相色谱仪, 包括四元超高压溶剂系统、自动进样恒温样品管理器, Waters 2998 PAD检测器, Empower 3色谱工作站(Waters, USA); GB204型电子分析天平(北京赛多利斯); KQ-200KED超声波清洗机(江苏昆山); GZX-9140MBE电热鼓风干燥箱(上海博迅)。
对照品没食子酸(批号122811)、儿茶素(11c15)、番泻苷B (11z15)购自天津西玛科技有限公司; 大黄素(110795-200505)、大黄酸(0757-200206)、大黄素甲醚(110758-200610)、大黄酚-8-O-葡萄糖苷(110796-200615)、大黄素-8-O-葡萄糖苷(10756-200110)购自中国食品药品检定研究院。大黄酚(B2038)和芦荟大黄素(B20772)购自上海源叶生物科技有限公司。色谱甲醇购自上海泰坦科技有限公司。娃哈哈纯净水购自杭州娃哈哈集团有限公司。其他试剂均为国产分析纯。
掌叶大黄HPLC (high performance liquid chromatography)含量测定 取掌叶大黄幼苗若干株, 随机选择3株混合, 3次平行取样。常规方法烘干, 参照课题组前期构建大黄HPLC含量测定方法[13]进行。色谱柱为武本C18 (5 μm, 4.6 mm × 250 mm)色谱柱; 流动相由甲醇(A)和0.2%磷酸水(B)组成, 梯度洗脱(0~5 min, 5%~15% A; 5~15 min, 15%~30% A; 15~25 min, 30%~35% A; 25~31 min, 35%~42% A; 31~46 min, 42%~53% A; 46~66 min, 53%~68% A; 66~75 min, 68%~100% A; 75~85 min, 100% A), 检测波长260 nm, 柱温30 ℃, 体积流量1.0 mL·min−1。进样量为10 μL。在上述色谱条件下分析, 理论板数按各个成分计算均不低于5 000, 与相邻组分峰的分离度均大于1.5, 色谱峰对称因子均在0.95~1.05。
对照品制备:精密称取没食子酸、儿茶素、番泻苷B、大黄酚-8-O-葡萄糖苷、大黄素-8-O-葡萄糖苷、芦荟大黄素、大黄酸、大黄素、大黄酚和大黄素甲醚等10种对照品适量, 分别置于10 mL容量瓶中, 用甲醇溶解并稀释至刻度, 摇匀, 质量浓度分别为0.224、0.71、0.45、0.172、0.234、0.079、0.077、0.028、0.048、0.027 mg·mL−1的对照品储备液。分别精密量取各对照品储备液1 mL, 甲醇稀释10倍, 得到相应质量浓度的混合对照品储备液。4 ℃保存备用。
供试品制备:取掌叶大黄幼苗烘干, 称重, 分别研磨粉碎混匀, 取0.1 g, 精密称定, 置于50 mL锥形瓶中, 精密加入甲醇4.5 mL, 称重。超声处理30 min (功率500 W, 频率40 kHz), 放至室温, 补足失量, 10 500 r·min−1离心12 min, 取上清液, 过0.22 μm微孔滤膜, 待测。
转录组测序分析 委托北京诺禾致源科技股份有限公司利用Illumina HiSeqTM 2000 150PE进行掌叶大黄幼苗转录组高通量测序分析。
取掌叶大黄新鲜幼苗全株, 采用EASYspin植物RNA快速提取试剂盒(Aidlab, China)制备总RNA, NanoDropTM 2000分光光度计(Thermo Fisher, USA)检测完整性。用带有Oligo (dT)的磁珠富集mRNA, 加入fragmentation buffer将mRNA打断成短片段, 用六碱基随机引物(random hexamers)合成cDNA第一链; 然后加入缓冲液、dNTPs、RNase H和DNA polymerase I合成cDNA第二链; 再经过QiaQuick PCR试剂盒(QIAGEN, Germany)纯化并加EB缓冲液洗脱之后做末端修复、加poly (A)并连接测序接头, 然后用琼脂糖凝胶电泳进行片段大小选择, 最后进行PCR扩增构建测序文库。
测序原始图像数据经base calling转化为序列数据raw reads, 经数据评估、过滤除杂和冗余处理等质控得到clean reads, 再利用Trinity做转录组de novo组装分析。Trinity首先将具有一定长度overlap的reads连成更长的片段, 这些通过reads overlap关系得到的不含N的组装片段作为组装出来的unigenes。
利用BLAST将unigenes序列与蛋白数据库NR、NT、Swiss-port、PFAM、KOG (Cluster of Orthologous Groups of proteins、蛋白相邻类的聚簇)和KEGG (京都基因与基因组百科全书)进行比对(E值< 1×10−5), 得到与相应unigenes具有最高序列相似性的蛋白, 进而得到unigenes注释信息。根据NR注释信息, 使用Blast2GO软件得到unigenes的GO (gene ontology)注释, 用WEGO软件对所有unigenes做GO功能分类统计, 从宏观上认识该物种的基因功能分布特征。使用MISA检测掌叶大黄转录组unigenes, 搜索SSRs (simple sequence repeats)并进行统计分析。
蒽醌生物合成基因筛选 蒽醌类生物合成主要由甲羟戊酸(MVA)、2-C-甲基-D-赤藓醇4-磷酸(MEP)、莽草酸(shikimate)和聚酮(polyketide)途径介导的骨架基因, 与衍生化修饰基因经细胞色素(cytochrome P450, CYP450)、尿苷二磷酸−糖基转移酶(UDP-glycosyltransferases, UGT)等共同参与完成[14, 15]。对这些基因的筛选主要根据unigenes在Nr数据库中的注释结果手动检索, 以FPKM (expected number of Fragments Per Kilobase of transcript sequence per Millions base pairs sequenced)计算基因表达量。
全长ungienes基因的RT-PCR验证 选择蒽醌及黄酮类生物合成7个全长候选unigenes, 包括乙酰-CoA乙酰转移酶(acetyl-CoA C-acetyltransferase, AACT)、MVA激酶(mevalonate kinase, MK)、1-脱氧-D-木酮糖5-磷酸合成酶(1-deoxy-D-xylulose-5-phosphate synthase, DXS)、2-甲基-D-赤藓糖醇-4-磷酸胞苷酰基转移(2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase, MCT)、苯丙氨酸解氨酶(phenylalanine ammonialyase, PAL)、肉桂酸-4-羟基化酶(cinnamate 4-hydroxylase, C4H)、黄烷酮-3-羟化酶(flavanone-3-hydroxylase, F3H)等, 利用Primer 3分别设计扩增ORF的特异引物(表 1), 采用RT-PCR验证。标准PCR体系包含以下组分: 10× Ex Taq Buffer (20 mmol·L−1) 2.0 μL, dNTP Mixture (各2.5 mmol·L−1) 1.6 μL, cDNA Template 0.5 μL, 正反向引物各0.5 μL, TaKaRa Ex Taq (5 U·μL−1) 0.1 μL, 补ddH2O至25 μL。PCR程序为: 95 ℃ 3 min, 95 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 2.0 min, 36个循环; 72 ℃ 10 min, 12 ℃保温。PCR产物经电泳分析, 送武汉金开瑞生物工程有限公司测序。
| Table 1 Seven candidate genes and ORF amplification primers |
掌叶大黄幼苗HPLC色谱分析结果(图 1)表明, 10种标准品在掌叶大黄幼苗中均能检测到, 各标准品含量存在明显差异。全株植物中各标品含量依次为:儿茶素 > 大黄素-8-O-葡萄糖苷 > 大黄酚-8-O-葡萄糖苷 > 大黄素 > 大黄酚 > 番泻苷B > 大黄素甲醚 > 没食子酸 > 芦荟大黄素 > 大黄酸。其中, 大黄特征性成分大黄素、大黄酚、大黄素甲醚、芦荟大黄素、大黄酸的含量分别为0.021%、0.016%、0.014%、0.002%和0.001%, 说明掌叶大黄生长发育过程中次生代谢途径比较活跃。

|
Figure 1 HPLC analyses of mixed reference substances (A) and R. palmatum L. (B) seedlings. 1: Gallic acid; 2: Catechin; 3: Senna glycoside B; 4: Chrysophanol-8-O-glucoside; 5: Emodin-8-O-glucoside; 6: Aloe-emodin; 7: Rhein; 8: Emodin; 9: Chrysophanol; 10: Physcion |
掌叶大黄幼苗转录组测序得到74 638 394条raw reads, 过滤产生了73 630 974条高质量clean reads, 包含11.04 Gb个核苷酸信息, 将原始数据提交SRA数据库获得注册号SRP160030。Q20和Q30分别为96.43%、90.85%, GC量为49.12%, 说明测序质控良好, clean reads质量合格。Trinity无参组装获得93 646个unigenes, 平均长度1 108 nt, 最长达到14 605 nt, 最短序列为201 bp, N50为1 794 nt。Unigenes长度分布显示, 51 280条unigenes长度超过1 000 nt, 205 86条序列大于2 000 nt。
3 转录组unigenes的功能注释使用BLAST将所有unigenes与NR、Swiss-port、KOG、KEGG等数据库进行一致性比对分析, 对各数据库注释的unigenes数目进行统计, 进而获得掌叶大黄转录组unigenes的功能注释信息。结果表明, 67 076条unigenes (71.62%)在NR数据库比对成功得到注释, 在KEGG中注释28 158个(30.06%), 在GO、KOG等数据库获得注释的unigenes数目依次为49 885 (53.26%)、20 362 (21.74%)。10 444条unigenes同时在所有数据库中注释, 至少有一种数据库注释成功的unigenes共71 304条(76.14%)。
以NR数据库为例进行分析, unigenes注释同源基因的物种分布如图 2所示, 在相似序列匹配度较高的物种中, 甜菜Beta vulgaris L.所占比例最高, 18 390条(27.6%); 其次为葡萄Vitis vinifera L., 9 256条(13.9%), 可可豆Theobroma cacao L. 2 899条(3.6%), 麻风树Jatropha curcas L. 2 397条(3.6%), 莲Nelumbo nucifera Gaertn. 2 395条(3.6%), 其余匹配物种比例在4.3%以下, unigenes小于1 000条比例小于3.2%, 16 616条占27.4%。

|
Figure 2 Species distribution of transcriptomic unigenes against NR database |
根据NR注释信息得到GO功能分类(图 3), 49 885个unigenes被注释到生物过程、细胞组分和分子功能3个GO类别的57个小组。细胞组分中细胞(cell)和细胞部分(cell part)相关基因丰度最高, 达15 483和15 480条; 其次是细胞器(organelle), 有10 348条; 共质体(symplast)基因较少, 有11条。生物过程主要聚集在代谢过程(metabolic process)和细胞过程(cellular process), 涉及的基因分别有26 898条和29 119条; 单组织过程(single-organism process)和生物调节(biological regulation)基因数量分别为21 056、9 917条。分子功能中具有催化活性(catalytic activity)和结合功能的基因(binding)数量较高, 分别为22 942和29 184条, 其他类别基因数目普遍较少。

|
Figure 3 GO classification of transcriptomic unigenes |
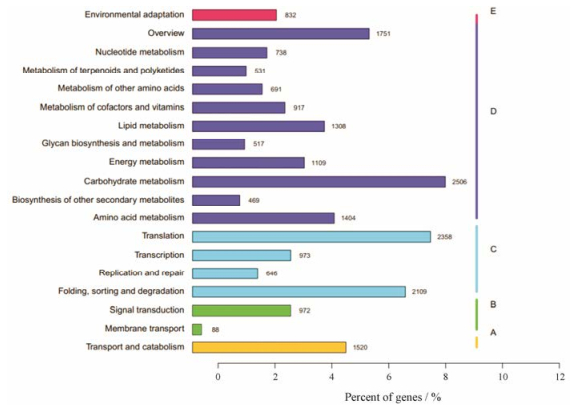
进一步进行KOG功能分类(图 4), 共得到25个不同的KOG功能类群种类比较全面, 包括大多数的生命活动; 翻译后修饰, 蛋白反转、伴侣最多, 有2 708条; 一般功能预测的基因数量次之, 为2 585条; 翻译、核糖体结构和生源unigene数目1 704条; 其他种类基因丰度不尽相同。掌叶大黄转录组unigenes参与KEGG代谢通路(图 5)分为五大分支:细胞过程A (cellular processes) 1 520条、环境信息处理B (environmental information processing) 1 060条、遗传信息处理C (genetic information processing) 6 086条、代谢D (metabolism) 11 941条和有机系统E (organismal systems) 832条; 19条子通路, 其中碳水化合物代谢最多1 097条, 单萜生物合成最少为1条。

|
Figure 4 KOG annotation distribution of transcriptomic unigenes |
|
Figure 5 KEGG classification of assembled unigenes |
KEGG代谢通路分析还发现1 107条unigenes参与类胡萝卜素、苯丙素类、玉米素、生物碱、黄酮类、花青素等生物合成相关的19个次生代谢标准通路(表 2)。其中, 苯丙素的生物合成(ko00940)基因数量最多, 为233个; 萜类化合物骨架生物合成(ko00900)基因数量次之, 为201条; 与类胡萝卜素的生物合成代谢通路(ko00906)有关的基因有183条; 有77个unigenes与玉米素的生物合成(ko00908)相关; 类黄酮的生物合成(ko00941)基因70条; 异喹啉生物碱(ko00950)基因有55条; 花青素生物合成、咖啡因的代谢、黄酮和黄酮醇的生物合成、甜菜红色素、吲哚生物碱的生物合成、异黄酮类的生物合成通路基因数较少。
| Table 2 Secondary metabolism KEGG pathway analysis of transcriptomic unigenes |
参与大黄蒽醌类骨架生物合成MVA、MEP、莽草酸及聚酮途径候选基因见表 3。从表 3可以看出, 这4个途径关键酶基因表达数量和表达量存在差异。MVA途径关键酶有乙酰-CoA乙酰转移酶(AACT)、HMG-CoA合酶(HMGS)、HMG-CoA还原酶(HMGR)、MVA激酶(MK)、MVP激酶(PMK)、MVPP脱羧酶(MPD)和IPP异构酶(IPPs), 共57条unigenes。其中, 编码MK unigenes数量最多, 为16条, 但平均表达量不高, 仅1.91; 3条unigenes编码IPPs, 平均表达量最高, 达到29.15。
| Table 3 Unigenes involved in anthraquinone biosynthesis |
33条unigenes编码MEP途径关键酶, 包括1-脱氧-D-葡萄糖-5-磷酶合成酶(DXS)、1-脱氧-D-葡萄糖-5-磷酶还原异构酶(DXR)、2-甲基-D-赤藓糖醇-4-磷酸胞苷酰基转移酶(MCT/ispD)、4-二磷酸胞苷-2-甲基-D-赤藓糖醇激酶(CMK/ispE)、4-羟基-3-甲基丁烯-2-烯基-1-二磷酸合酶(ispG/HDS)、2-C-甲基-D-赤藓糖醇-1, 4-环磷酸二磷酸合成酶(MDS/ispF)和4-羟基-3-甲基丁烯-2-烯基-1-二磷酸还原酶(HDR/ ispH)。HDR/ispH序列5条, 平均表达量最高, 为87.91; MDS/ispF平均表达量次之, 为48.85;其余基因表达水平较低, MCT/ispD平均表达量仅0.09。
61条unigenes编码莽草酸途径关键酶, 包括3-脱氧-D-阿拉伯庚酮糖-7-磷酸合成酶(DAHPS)、3-脱氧奎尼酸合成酶(DHQS)、莽草酸脱氧酶(SDH)、莽草酸激酶(SMK)、EPSP合成酶(EPSPs)、分支酸合成酶(CS)、异分支酸合成酶(IS/MenF)。其中, CS表达量为34.24, IS/MenF平均表达量仅1.07。2-琥珀酰-5-烯醇丙酮酸-6-羟基-3-环己烯-1-羧酸合酶(MenD)、2-琥珀酰-6-羟基-2, 4-环己二烯-1-羧酸合酶(MenH)、2-琥珀酰苯甲酸合酶(MenC)没有被注释。
聚酮途径unigenes共21条, 分别编码聚酮合成酶(PKS)、Ⅲ型聚酮合成酶(PKSⅢ)、聚酮环化酶(PKC)及查尔酮合酶(CHS)。PKC平均表达量最高, 为16.61, PKSⅢ平均表达量最低仅1.71, CHS unigenes数量多达12条, 仅1条PKS基因序列。
4.2 蒽醌修饰相关基因根据NR注释结果, 共找到125条CYP450基因, 隶属于22个CYP450家族。属于CYP71家族的unigenes最多, 有25.40%;其次是CYP94、CYP87和CYP76, 分别为15.08%、10.32%和7.2%。而CYP89、CYP81家族成员最少, 仅各有1个unigene。共找到属于13个UGT亚家族的73个UGTs, 其中包括21个UGT80、2个UGTPg19、10个UGT71、3个UGT70、15个UGT92、2个UGT84、3个UGT7、2个UGT89、4个UGT72、1个UGTPg26、4个UGT88、1个UGTPg35、4个UGT85。
5 SSRs分析利用MISA软件对转录组unigenes进行SSRs分析(表 4), 93 646个unigenes中共计18 885个SSRs。其中, 单碱基重复SSR数量最丰富, 有7 542个(39.94%), 其中A重复最多。二碱基重复SSRs数量次之, 有5 714个, 占SSRs总量的30.26%, 其中AG/CT重复类型数量最多。四碱基和五碱基重复分别为249和82个, 各占1.32%、0.43%;六碱基重复相对较少, 仅占0.41%。此外, 还发现SSRs重复单元数量存在一定变化。
| Table 4 SSRs analysis of transcriptomic unigene |
利用表 1中的7对ORF扩增引物进行RT-PCR分析, 图 6结果表明, 7个基因特异引物均产生目标条带。进一步PCR产物直接测序分析表明, 这7个基因与原unigenes ORF一致, 说明这些候选unigenes为全长基因。

|
Figure 6 Agarose gel electrophoresis of seven candidate genes amplified by RT-PCR |
随着高通量测序技术的不断进步, 其广泛应用在药用植物转录组分析中, 并取得重大进展[16]。本研究在明确掌叶大黄幼苗主要有效成分的基础上, 采用Illumina HiSeqTM 2000 100PE测序平台, 首次进行掌叶大黄转录组测序分析, 填补了大黄转录组信息的空白。高通量测序数据约11.04 G, 利用Trinity组装共得到93 646个unigenes, 测序质量良好、质控严格, 序列长度与reads分布区域对应合理。转录组序列信息量庞大, 数据基本涵盖全转录组信息, 能够清晰反映传统中药材大黄的基因表达特征, 为深入研究大黄生长发育、次生代谢、转录调控等生物学过程功能基因的批量发掘提供支持。
基于高通量测序的转录组数据通常采用生物信息学分析策略进行基因注释和功能分类。本研究利用多种生物信息软件, 对掌叶大黄转录组进行注释和功能分类。基于BLAST分析, 将所有unigenes与NR、Swiss-port、PFAM、KOG等数据库比对, 这与已报道的柴胡[10]、人参[11]和商陆[12]等物种转录组测序注释比例类似, 说明掌叶大黄转录组中存在大量序列特征及功能尚未知的unigenes。GO分类揭示掌叶大黄转录组特性与生物过程、细胞组分和分子功能相关; KOG功能分析从基因组水平寻找直系同源体, 预测未知ORF的生物学功能, 可大大提高基因功能注释的准确性。本研究共得到25个不同的KOG类群, 说明掌叶大黄转录组KOG种类比较全面。130个KEGG标准代谢通路基因可能参与掌叶大黄水分吸收、矿质营养、光合作用和呼吸作用等生命代谢活动; 此外, 还发现大量unigenes参与萜类、芪类、生物碱、黄酮类、花青素等生物合成相关的19个次生代谢标准通路, 有助于研究大黄次生代谢物生物合成途径。
目前, 蒽醌类成分主要分为大黄素型和茜草素型, 通常认为前者由聚酮途径合成, 后者由MVA/ MEP偶联莽草酸途径介导, 但是植物中蒽醌生物合成通路仍不清楚。茜草科短小蛇根草Ophiorrhiza pumila[14]和豆科决明Cassia obtusifolia[15]转录组研究发现大量MVA、MEP、莽草酸及聚酮途径基因, 提示蒽醌生物合成与这4个途径相关。本研究以掌叶大黄为对象, 在明确蒽醌类特征性成分含量的基础上, 利用转录组高通量测序技术系统筛选, 获得57、32、61、21条unigenes分别编码MVA、MEP、莽草酸及聚酮途径的28个关键酶, 其中RT-PCR验证7个unigenes为全长基因, 为下一步研究大黄素型蒽醌生物合成的基因分子功能提供基础数据。拟南芥MenD/MenH/MenC基因融合为一个基因[17], 该基因在短小蛇根草和决明中均有转录[14, 15], 本研究未检测到MenD/MenH/MenC的表达, 也未发现聚酮途径polyketide hydrolase转录本, 可能与样品的生长发育状态有关。此外, 基于FPKM统计这些关键酶基因的表达特征存在一定差异, 推测它们通过不同的表达调控机制参与了蒽醌类生物合成。
在萜类、黄酮等次生代谢物的衍生化修饰过程中, CYP450和UGT主要起催化氧化/羟基化和糖基化的重要作用[10]。微生物中有这两类基因修饰蒽醌成分的研究。黄枝孢霉Cladosporium fulvum CYP450能够催化蒽醌类nataloe-emodin二聚化[18], 芽孢杆菌Bacillus licheniformis DSM13 UGT能够在体内外糖基化大黄素和芦荟大黄素而不影响抗癌活性, 稳定性显著提高[19]。本研究发现转录组中大量CYP450、UGTs序列, 可能在大黄蒽醌类衍生修饰中起作用, 哪些unigenes如何参与蒽醌类衍生化修饰仍需深入研究。
SSR标记包括EST-SSR、基因组SSR。基于cDNA文库和转录组测序的EST-SSR在植物遗传多样性、分子标记等研究方面应用广泛[20]。本研究MISA发现SSR从单核苷酸类型到六核苷酸类型均具备, 表明掌叶大黄基因组内具有较高丰度的SSR。重复类型以三核苷酸为主, 双核苷酸所占比例次之。这与以三核苷酸重复类型为主的主要作物水稻、大麦等的研究结果相同[21]。在双核苷酸重复SSRs中AG/CT最多, 三核苷酸重复中AAG/CTT最多, 与人参[22]等的情况一致, 说明SSRs重复类型可能存在一定的保守性。
本研究首次运用二代高通量测序技术开展了掌叶大黄幼苗转录组测序研究, 获得了大量掌叶大黄遗传信息和基因表达特征, 发掘了蒽醌类次生代谢物的生物合成途径关键基因, 为后期进一步研究蒽醌类生物合成基因克隆及功能分析提供基础资料, 有助于深入解析大黄蒽醌生物合成通路及调控机制, 为科学阐释大黄生长发育、生理适应及品质形成提供理论基础。
| [1] | Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Part Ⅰ. 2015 ed. Beijing: China Medical Science Press, 2015. |
| [2] | Yi J, Yang JR, Gao F, et al. Emodin enhances arsenic triox-ide-induced apoptosis via generation of reactive oxygen species and inhibition of survival signaling[J]. Cancer Res, 2015, 64: 108–116. |
| [3] | He ZH, Zhou R, He M F, et al. Anti-angiogenic effect and mechanism of rhein from Rhizoma Rhei[J]. Phytomedicine, 2011, 18: 470–478. DOI:10.1016/j.phymed.2010.10.006 |
| [4] | Yang F, Xu Y, Xiong A, et al. Evaluation of the protective effect of Rhei Radix et Rhizoma against α-naphthylisothiocyanate induced liver injury based on metabolic profile of bile acids[J]. J Ethnopharmacol, 2012, 144: 599–604. DOI:10.1016/j.jep.2012.09.049 |
| [5] | Zhang XQ, Liu CS, Yan XL, et al. Sequence analysis and identification of a chloroplast matK gene in Rhei Rhizoma from different botanical origins[J]. Acta Pharm Sin (药学学报), 2013, 48: 1722–1728. |
| [6] | Lu GD, Li CY, Wang HZ, et al. Analysis on quality of Rheum palmatum L. from Gansu province based on multicriteria method[J]. Chin J Inf TCM (中国中医药信息杂志), 2017, 24: 57–63. |
| [7] | Wei WL, Zeng R, Huang LF. Correlation analysis between quality of Rheum palmatum L. and ecological factors[J]. World Sci Technol-Mod of Tradit Chin Med (世界科学技术-中医药现代化), 2015, 17: 1849–1854. |
| [8] | Liu J, Liu P, Duan JA, et al. Main components analysis in different parts of Rheum palmatum[J]. Chin Tradit Herb Drugs (中草药), 2017, 48: 567–572. |
| [9] | Qi YX, Liu YB, Rong WH. RNA-Seq and its applications:a new technology for transcriptomics[J]. Hereditas (遗传), 2011, 33: 1191–1202. |
| [10] | Sui C, Zhang J, Wei J, et al. Transcriptome analysis of Bupleurum chinense focusing on genes involved in the biosynthesis of saikosaponins[J]. BMC Genomics, 2011, 12: 539. DOI:10.1186/1471-2164-12-539 |
| [11] | Jung I, Kang H, Kim JU, et al. The mRNA and miRNA transcriptomic landscape of Panax ginseng under the high ambient temperature[J]. BMC Syst Biol, 2018, 12(Suppl 2): 27. |
| [12] | Zhao L, Zhu YH, Zhang L, et al. Transcriptome analysis reveals candidate genes involved in esculentoside A biosynthesis in Phytolacca americana[J]. Acta Pharm Sin (药学学报), 2017, 52: 1471–1480. |
| [13] | Yan YG, Yin LM, Wang HY, et al. HPLC method for simultaneous determination of nine components from leaves of Rheum officinale by HPLC[J]. Chin Tradit Herb Drugs (中草药), 2016, 47: 2360–2364. |
| [14] | Yamazaki M, Mochida K, Asano T, et al. Coupling deep transcriptome analysis with untargeted metabolic profiling in Ophiorrhiza pumila to further the understanding of the biosynthesis of the anti-cancer alkaloid camptothecin and anthraquinones[J]. Plant Cell Physiol, 2013, 54: 686–696. DOI:10.1093/pcp/pct040 |
| [15] | Rama Reddy NR, Mehta RH, Soni PH, et al. Next generation sequencing and transcriptome analysis predicts biosynthetic pathway of sennosides from senna (Cassia angustifolia Vahl.), a non-model plant with potent laxative properties[J]. PLoS One, 2015, 10: e0129422. DOI:10.1371/journal.pone.0129422 |
| [16] | Zhang ZB, Hou L, Pan Q, et al. Advances in high-throughput transcriptome research of traditional Chinese medicines[J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 1553–1558. |
| [17] | Gross J, Cho WK, Lezhneva L, et al. A plant locus essential for phylloquinone (vitamin K1) biosynthesis originated from a fusion of four eubacterial genes[J]. J Biol Chem, 2016, 281: 17189–17196. |
| [18] | Griffiths S, Mesarich CH, Saccomanno B, et al. Elucidation of cladofulvin biosynthesis reveals a cytochrome P450 monooxygenase required for anthraquinone dimerization[J]. Proc Natl Acad Sci U S A, 2016, 113: 6851–6856. DOI:10.1073/pnas.1603528113 |
| [19] | Ghimire GP, Koirala N, Pandey RP, et al. Modification of emodin and aloe-emodin by glycosylation in engineered Escherihia coli[J]. World J Microbiol Biotechnol, 2015, 31: 611–619. DOI:10.1007/s11274-015-1815-4 |
| [20] | Varshney RK, Graner A, Sorrells ME. Genic microsatellite markers in plants:features and applications[J]. Trends Biotechnol, 2005, 23: 48–55. DOI:10.1016/j.tibtech.2004.11.005 |
| [21] | Cardle L, Ramsay L, Milbourne D, et al. Computational and experimental characterization of physically clustered simple sequence repeats in plants[J]. Genetics, 2000, 156: 847–854. |
| [22] | Li C, Zhu Y, Guo X, et al. Transcriptome analysis reveals ginsenosides biosynthetic genes, microRNAs and simple sequence repeats in Panax ginseng C. A. Meyer[J]. BMC Genomics, 2013, 14: 245. DOI:10.1186/1471-2164-14-245 |
 2018, Vol. 53
2018, Vol. 53


