非核糖体肽(nonribosomal peptide, NRP)是指不依赖于核糖体而由非核糖体肽合成酶(nonribosomal peptide synthetase, NRPS)催化合成的肽类化合物, 具有广泛的药理活性[1]。许多非核糖体肽是常见的抗生素[2], 例如, 短杆菌肽A (gramicidin A)、短杆菌肽S (gramicidin S)、万古霉素(vancomycin)、恩镰孢菌素B (enniatin B)、短杆菌素(tyrothricin)、多粘菌素B (polymyxin B)和替考拉宁(teicoplanin)被用作抗菌药物; 放线菌素D (actinomycin D)、博来霉素(bleomycin)和罗米地辛(romidepsin)被用作抗肿瘤药物; 其他非核糖体肽如环孢素A (cyclosporine A)是重要的免疫抑制剂而被用作器官移植; 麦角新碱(ergometrine)被用于妇科产后止血; 麦角胺(ergotamine)被用于治疗偏头痛[3]。此外, 临床上应用的许多非核糖体肽类药物实际上是天然非核糖体肽经过结构优化后的衍生物。从来源上讲, 目前发现的非核糖体肽中超过80%来自于真菌和放线菌, 其次是粘细菌和蓝藻等单细胞原核生物。基因组序列分析结果表明, 古生菌、后生动物和甲藻也有合成非核糖体肽的潜力[4]。
有些NRPS还与其他酶系如聚酮类(polyshy; ketide)[5, 6]或萜类(terpenoid)合成酶系共同完成杂合分子的合成。典型的NRPS由多个模块(module)组成, 这些模块按特定的空间顺序排列[7, 8], 每个模块由多个功能结构域(domain)或催化单元组成[9], 结构多样的非核糖体肽在这些模块及其结构域上有序地进行合成[1]。本文重点对近年来在NRPS结构域及其作用机制研究方面取得的进展、以及新发现的几种非常规装配进行综述, 为寻找新的非核糖体肽类化合物及其新药研发提供理论依据。
1 NRPS的基本结构域及其作用机制典型的NRPS模块具有腺苷化(adenylation, A)结构域、硫酯化(thiolation, T)结构域、缩合(condensation, C)结构域[2, 7]和硫酯酶(thioesterase, Te)结构域, 分别负责氨基酸的活化、肽链的延伸、酰胺键的形成, 以及肽链的释放[3, 9, 10]。概括来说(图 1), A结构域在ATP的作用下结合氨基酸底物形成相应的氨酰-AMP[2], 氨酰-AMP与T结构域的磷酸泛酰巯基乙胺的巯基结合, 形成氨酰-S-载体复合物, 最后, 分别携带有氨酰基和肽酰基的载体与C结构域上的特定区域结合, 氨酰-S-载体复合物上的氨基向肽酰-S-载体复合物上的酰基进行亲核攻击[2], 形成新的肽键, 经过多个模块的作用最终形成完整的肽链[8], 其中肽链中的氨基酸与NRPS中的模块一一对应[7]。在某些NRPS的模块中还含有差向异构化(epimerization, E)结构域、甲酰化(formylation, F)结构域[1]、甲基化(methylation, M)结构域、杂环化(heterocyclization, Cy)结构域[6, 8]、还原(reduction, R)结构域和氧化(oxidation, Ox)结构域等, 这些结构域参与肽链的结构修饰[1, 8]。最终, 在Te结构域的作用下, 成熟的肽链从NRPS装配线上释放, 在释放的过程中Te结构域还会介导产物的环化[3]。
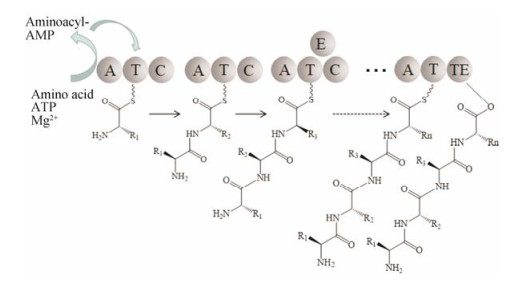
|
Figure 1 Simplified scheme of conventional nonribosomal peptide synthetase (NRPS) |
A结构域的大小约60 kDa, 属于腺苷化酶家族(adenylating enzymes, ANL)[11]。ANL催化两步反应, 第一步反应是利用ATP催化羧酸底物腺苷化, 释放PPi并生成酰基-AMP中间产物, 第二步反应是催化硫酯键的形成并释放AMP[3, 12]。氨基酸与A结构域结合的位置由保守的天冬氨酸和赖氨酸残基确定, 它们分别可以稳定氨基酸的氨基部分和羧酸盐部分。保守的天冬氨酸残基位于底物结合口袋的入口处, 可被进一步修饰以更好地适应氨基酸底物的侧链。A结构域中包括天冬氨酸和赖氨酸残基在内目前已有8个氨基酸残基被确认参与到底物侧链的识别过程中, 从而组成A结构域的特异性代码或称为非核糖体密码[3]。近年来, 研究者们通过异源表达A结构域, 并结合体外的酶促反应鉴定其结合的氨基酸底物[13-16]。Zwittermicin A是苏云金芽胞杆菌(Bacillus thuringiensis) YBT- 1520产生的一种氨基多元醇类抗生素, 属于聚酮-非核糖体肽杂合化合物, 其生物合成需要NRPS的参与。通过在大肠杆菌中超量表达zwittermicin A合成基因簇中的A结构域及依赖于ATP的焦磷酸交换实验, 证实了该A结构域可以特异性识别并活化2, 3-二氨基丙酸[13]。Xia等[14]在挖掘海洋放线菌Salinispora arenicola CNS-205中的相关NRPS沉默基因簇时, 发现并扩增出一个假定的氨基酸腺苷化结构域基因编码序列Sare0718。生物信息学分析显示Sare0718特异性识别的氨基酸底物可能为L-Val, 然而实验结果表明, Sare0718结合的特异性底物为L-Ala。A结构域的特异性底物与生物信息学预测的结果并不相符, 可能是因为目前已建立的A结构域底物特异性密码子数据库的实验数据几乎全部来源于陆生微生物, 利用该数据库预测如Salinispora arenicola CNS-205等的海洋放线菌A结构域的特异性底物并不准确[15]。因此, 在研究A结构域结合的特异性底物时, 仍需结合实验进行验证。
1.2 硫酯化(thiolation, T)结构域T结构域, 又被称为PCP (peptide carrier protein)结构域, 由大约80个氨基酸构成, 分子量约为10 kDa[17], 在结构上含有4个α螺旋, 第2个α螺旋的N末端含有高度保守的丝氨酸残基(GxxS核心motif), 其被磷酸泛酰巯基乙胺转移酶(phosphopantetheine transferase, PPTase)修饰[3, 18, 19]。PPTase将CoA上的4'-磷酸泛酰巯基乙胺残基转移到前酶(apo-T domain)的保守丝氨酸残基上, 这时前酶就变成了全酶(holo-T domain)[20, 21]。全酶可以被看成NRPS装配线上灵活的机械臂, 它可转移氨酰硫酯中间体到其他催化中心, 在非核糖体肽的生物合成中发挥着极其重要的作用[3, 10, 22]。
1.3 缩合(condensation, C)结构域C结构域的大小约50 kDa, 主要负责非核糖体肽合成过程中酰胺键的形成, 催化n-1模块的氨基酸或新生肽链连接到n模块的氨基酸上[3, 23]。根据对氨基酸底物的立体选择性, C结构域可以分为LCL和DCL结构域, LCL结构域主要介导两个L-氨基酸的缩合, DCL结构域则催化上游D-氨基酸和下游L-氨基酸的缩合[24]。C结构域是呈V字型的伪二聚体, 由N末端亚结构域CNTD和C末端亚结构域CCTD构成。作为氯霉素乙酰转移酶超家族的成员, CNTD和CCTD在交界处形成一个裂缝, 使Tn-1的摆动臂和Tn的摆动臂在传递氨基酸的过程中分别可以从相反的方向穿过裂缝到达保守区(HHxxxDG)[14]。该保守区的第2个组氨酸促进α氨基基团的亲核进攻, 并稳定四面体过渡态, 在C结构域发挥缩合作用的过程中起十分重要的作用[3]。诺卡菌素A (nocardicin A)的生物合成需要NRPS成员NocB的参与, 将其C结构域(位于module 5中)中的第2个保守His突变为Ala, 突变体不能再产生诺卡菌素A生物合成途径的中间体, 进一步证实了保守区中His的重要性[25]。C结构域在非核糖体肽合成过程中还发挥着第二选择性过滤器的作用, 在A结构域错误地进行底物选择的情况下, C结构域的识别作用能减少NRPS合成的错误率。当肽链逐渐延伸时, 控制其序列的准确性会变得愈加复杂, 这时C结构域可以被看作是主要的选择性过滤器[3]。除此之外, C结构域也会影响相邻A结构域的底物特异性[23]。在铜绿微囊藻(Microcystis aeruginosa) PCC 7806中, NRPS成员McyBB和McyC参与微囊藻毒素(microcystin)的合成。Meyer等[26]探究了McyBB和McyC的底物特异性, 实验表明: McyBB和McyC的C-A-PCP模块完全切除C结构域后, 腺苷化的氨基酸种类明显增加, 特异性下降, 而A-PCP模块恢复C结构域的子域CCTD后, McyBB腺苷化的氨基酸种类有一定程度的减少, 特异性有所增强, 验证了C结构域确实会影响A结构域的底物特异性。
1.4 硫酯酶(thioesterase, Te)结构域Te结构域的分子质量约30 kDa, 负责从NRPS装配线上释放肽链[3]。该过程主要分为两步:首先, Te结构域将T结构域结合的中间体转移到其活性位点上, 该活性位点一般为保守的丝氨酸残基, 少数情况下为半胱氨酸。然后, 上述中间体被亲核试剂(nucleophile)攻击, 根据亲核试剂的不同, 肽链会发生不同的反应。当亲核试剂为水时, 肽链会发生水解; 当亲核试剂为分子内的氨基(来自肽链的N末端或氨基酸残基的侧链)或羟基(来自丝氨酸或苏氨酸的侧链)时, 肽链会发生环化形成环肽[27]。Te结构域还可以对将要结合的中间体进行校对, 以确保肽链在释放之前的准确性和完整性[3]。如在绿脓杆菌螯铁蛋白(pyochelin)的生物合成过程中, Te结构域只选择结构中含有噻唑啉的肽链进行水解或环化, 形成结构正确的产物[28, 29]。参与替考拉宁(teicoplanin)和万古霉素(vancomycin)等糖肽类抗生素生物合成的Te结构域也在肽链的释放步骤中充当选择性过滤器的作用, 肽链只有在发生氧化修饰后, 才能在Te结构域的作用下释放产生活性肽[27]。
1.5 其他功能结构域除了A结构域、T结构域、C结构域和Te结构域, 在某些NRPS的模块中还包含其他结构域, 如在参与短杆菌酪肽(tyrocidine A)合成的NRPS中包含的立体异构化(epimerization, E)结构域[30]; 在参与环孢菌素(cyclosporin)合成的NRPS中包含的甲基化(methylation, M)结构域[31]; 在参与达托霉素(daptomycin)合成的NRPS中包含的杂环化(heterocyclization, Cy)结构域[31]; 在参与博来霉素(bleomycin)合成的NRPS中包含的氧化(oxidation, Ox)结构域[3]; 在参与索拉霉素(soframycin)合成的NRPS中包含的还原(reduction, R)结构域[3]等。其中, 立体异构化结构域的功能是在A结构域识别与活化L-氨基酸、且L-氨基酸结合至T结构域的机械臂上后, E结构域可将L-氨基酸催化成D-氨基酸[30] (图 2)。甲基化结构域可以催化在装配过程中的肽链发生甲基化反应, 其甲基供体一般为S-腺苷甲硫氨酸(S-adenosyl methionine, SAM)。在NRP中甲基化一般发生在氮原子上, 肽链中氮原子的甲基化通常发生在缩合反应之前; 而在一些PK/NRP杂合分子中甲基化通常发生在碳原子上[31]。杂环化结构域可将半胱氨酸和丝氨酸/苏氨酸残基分别杂环化为噻唑啉(thiazoline)和噁唑啉(oxazoline)五元杂环, Cy结构域在这个过程中不仅催化环化反应, 还催化肽键的形成和脱水反应。研究表明, Cy结构域首先催化肽键的形成, 然后环化半胱氨酸的巯基侧链或丝氨酸/苏氨酸的羟基侧链至刚形成的肽键上, 最后脱水形成噻唑啉和噁唑啉中的C=N键[31]。氧化结构域催化的氧化反应依赖于黄素单核苷酸(flavin mononucleotide, FMN)进行, Ox结构域一般位于A结构域亚基(Asub)的保守模体A8和A9之间, 比如参与埃博霉素(epothilone)生物合成的NRPS成员EpoB和参与粘噻唑(myxothiazol)生物合成的NRPS成员MtaD; 或位于T结构域的C末端。不同Ox结构域在NRPS中的位置不仅不同, 而且它们之间的序列相似性仅约为40%[3]。在一些NRP和PK-NRP杂合体的C端具有醛或醇官能团, 这是由于参与合成这些化合物的NRPS中包含还原(reduction, R)结构域。R结构域代替常规NRPS末端的Te结构域, NADPH依赖的R结构域能催化与硫酯结合的肽基发生双电子或四电子的还原, 分别形成醛或伯醇类官能团[3]。上述结构域或通过提供蛋白酶抗性、或通过与其他结构域相互作用, 或通过提高蛋白的亲和力, 或改变无活性的NRP构象使其恢复活性等不同的方式行使功能, 对NRP的生物合成发挥重要作用[1]。

|
Figure 2 NRPS of tyrocidine A biosynthesis and mechanism of the epimerization reaction. a: Domains and module architecture of the NRPS including the epimerization domain; b: Reaction mechanism of the epimerization domains |
NRPS除了上述的装配方式之外, 近年来也发现了一些非常规的装配模式, 主要包括:重复使用模块, 即至少有一个以上的模块被重复使用, 产生两个以上的相同缩合单元; 模块跳跃, 即功能完整的模块在肽链延伸过程中被跳跃; 反式上载, 即某一模块对应的氨基酸底物由外在的A结构域负责活化[32]。
2.1 重复使用模块有些非核糖体肽的合成是在重复型NRPS (iterative NRPS)的作用下完成的, 其合成的主要特点在于重复使用一套C-A-T模块, 形成多个最小合成单体, 最终在Te结构域的作用下形成多聚体。津岛链霉菌(Streptomyces tsusimaensis) 15141来源的缬氨霉素(valinomycin, VLM)的生物合成过程就运用上述策略(图 3)。VLM是环十二肽, 包含3个四缩酚酸肽重复单元(D-Hiv-D-Val-L-Lac-L-Val)。缬氨霉素由VLM1和VLM2两个NRPS负责合成, NRPS中每个模块负责选择激活对应的氨基酸底物并加入到上游已形成的肽链中, 最后形成四缩酚酸肽并以共价连接的形式转移到C末端Te结构域的活性位点丝氨酸残基上。利用VLM1和VLM2再次进行两轮上述的过程, 就形成了线性十二肽, 最后, Te结构域释放线性十二肽, 并催化其环化形成VLM[33]。

|
Figure 3 The model for the biosynthesis of valinomycin (VLM) in S. tsusimaensis ATCC 15141. TA and DH2 domains are hypothetical |
有些NRPS存在的模块并未发挥功能, 这种现象被称为模块跳跃(module skipping)。如myxochromide S是黏细菌产生的带有不饱和聚酮侧链的环肽化合物, 介导其生物合成的NRPS成员(MchB和MchC)共含有6个模块, 但合成的产物(myxochromide S)却只包含5个氨基酸残基。通过比较A结构域的特异性识别底物, 推测module 4被跳跃而未发挥作用(图 4)[32]。研究结果还表明, 不仅NRPS中的整个模块可以被跳跃, 模块中的个别结构域也可以被跳跃。如图 4所示, AptA、AptB、AptC和AptD参与cyanobacterium Nostoc sp. CENA543中anabaenopeptins和namalides的生物合成。作为anabaenopeptins的同源类似物, namalides比anabaenopeptins的结构更为简短。当这些NRPS中的所有模块和结构域正常发挥作用时, 则合成anabaenopeptins; 当AptC的module 5和AptD的C结构域及A结构域均被跳跃时, 则只能生成namalides (图 5)[34]。

|
Figure 4 Simplified scheme of module skipping within myxochromide S biosynthesis |

|
Figure 5 Simplified scheme of module skipping within the anabaenopeptins and namalides biosynthesis |
常规NRPS中A结构域负责将氨基酸活化并上载到同一模块的T结构域, 称为顺式(In cis)上载, 这种NRPS称为线性NRPS。而某一模块对应的氨基酸由外在的A结构域负责活化, 则被称为反式(In trans)上载, 这种NRPS称为非线性NRPS[32]。在耶尔森杆菌素(yersiniabactin)的生物合成中涉及两个主要的NRPS: HMWP1和HMWP2, 但仅HMWP2中含有可活化氨基酸底物的A结构域。HMWP2能特异性识别并活化L-Cys, 活化的L-Cys不但能分别结合至HMWP2的T1和T2中, 还能结合至HMWP1的T3中, 完成yersiniabactin的生物合成(图 6)[35]。

|
Figure 6 Domain organization of HMWP1 and HMWP2. KS: β-Ketoacyl synthase; AT: Acyltransferase; MT: Methyltransferase; KR: Ketoreductase; ACP: Acyl carrier protein; Cy: Cyclization; T: Thiolation; TE: Thioesterase; ArCP: Aryl carrier protein; A: Adenylation |
NRPS的模块中还存在A结构域串联(tandem adenylation domains)的现象。在Streptomyces pyridomyceticus NRRL B-2517中, 吡啶霉素(pyridomycin)的生物合成是在NRPS成员PyrG的作用下完成的, PyrG包含两个串联的A结构域和PKS型酮还原酶结构域(图 7)。研究结果表明只有第二个A结构域(A2)能特异性识别并激活底物(α-酮基-β-甲基戊酸), 而第一个A结构域(A1)只在维持蛋白结构稳定方面发挥作用, 并不参与氨基酸底物的识别, 这可能是由于在A1结构域中两个重要氨基酸的突变(K517G, E327A)及保守模体motif A3的缺乏造成的[36]。

|
Figure 7 PyrG module including two tandem adenylation domains within the pyridomycin biosynthesis |
近年来的研究表明, C结构域不仅能催化n-1模块的氨基酸或新生肽链连接到n模块的氨基酸上, 还可以催化一些前体连接到游离的其他非氨基酸底物上。发光杆菌(Photorhabdus luminescens)产生的pepteridine是在NRPS-蝶啶杂合的生物合成基因簇的作用下合成的。该基因簇中含有8个功能基因, 其中plu2793、plu2794、plu2797、plu2798和plu2799为蝶啶生物合成单元, plu2795为丙酮酸脱氢酶合成单元, plu2796与丙酮酸脱氢酶具有同源性, 同时又包括NRPS的T结构域和C结构域。C结构域在这个过程中催化丙酰基缩合到游离的四氢蝶呤核心上, 形成pepteridines A; 催化乙酰基缩合到游离的四氢蝶呤核心上, 形成pepteridines B (图 8)[37]。
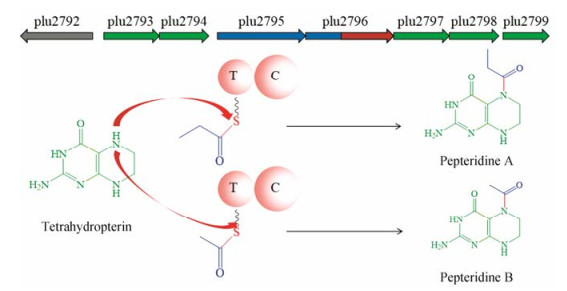
|
Figure 8 The pepteridine biosynthetic gene cluster and key NRPS-pteridine coupling reaction |
在NRPS的功能域组成中通常包含差向异构化(epimerization, E)结构域, 又可称为立体异构化结构域, 可使L-氨基酸异构化为D-氨基酸。来源于短芽孢杆菌(Bacillus brevis)的短杆菌酪肽(tyrocidine)由TycA、TycB和TycC负责合成, 其中TycA和TycB的C末端均含有E结构域, 使得产物短杆菌酪肽中的苯丙氨酸均为D构型(图 2a)[30]。Te结构域的典型功能是介导肽链的释放及环化, 但最近研究表明, Te结构域还可以代替E结构域发挥差向异构化的功能。在单环的β-内酰胺类抗生素诺卡菌素A (nocardicin A)的生物合成中涉及NocA和NocB, 其中NocB中module 5的Te结构域催化C末端的L-对羟基苯基甘氨酸[L-(p-hydroxyphenyl) glycine, L-pHPG]异构化为D-对羟基苯基甘氨酸, 发挥差向异构化功能(图 9)[38]。

|
Figure 9 Domain and module architecture of NocA and NocB during the nocardicin A biosynthesis with thioesterase domain playing the role of epimerization |
细菌中非核糖体肽的合成主要利用Te结构域进行释放和环化作用, 而近年来在真菌中发现了不依赖Te结构域的环化机制。如参与apicidin合成的NRPS中含有CT (condensation- like)结构域(图 10), 序列分析结果表明, CT结构域与C结构域拥有相同的保守区(HXXXDXXS), 该保守区中His仍是起关键催化作用的氨基酸, 但系统进化分析表明CT结构域与C结构域并不在一个分支中, 这也就暗示了CT结构域与C结构域功能的差异性。研究表明CT结构域可代替Te结构域作为末端结构域产生环化形式的肽[39]。2012年唐奕团队通过在酿酒酵母中体外重构Penicillium aethiopicum中包含3个模块的TqaA, 验证了TqaA可将线性的邻氨基苯甲酸-D-色氨酸-L-丙氨酰三肽环化为fumiquinazoline F (图 11), 并发现在体外重构的TqaA-ΔCT菌株无法产生目的产物fumiquinazoline F, 证明了TqaA的CT结构域确实催化了环化反应[40]。为进一步了解真菌大环化的策略, 2016年唐奕团队又鉴定了TqaA中CT结构域的晶体结构, 发现CT结构域的活性位点含有芳香基团, α2螺旋与β12~β13的loop区接触广泛, CT结构域中的α1螺旋代替了C结构域的N末端loop区, 以及loop 4充当活性口袋之上的盖子等。上述CT结构域晶体结构的特征都为解释TqaA中CT结构域催化环化反应形成fumiquinazoline F提供了依据, 同时为真菌NRPS末端模块的结构解析奠定了基础, 也为理性设计新的大环化多肽类产物提供了依据[39]。
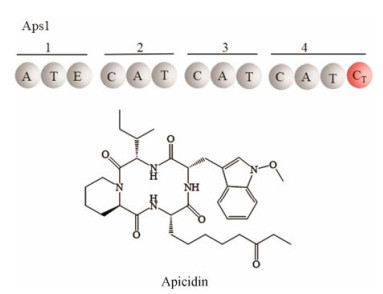
|
Figure 10 The NRPS domains including condensation-like domain within the apicidin biosynthesis |

|
Figure 11 Cyclization by a terminal condensation-like domain within the fumiquinazoline F biosynthesis |
综上所述, 在非核糖体肽合成酶各个功能结构域之间的相互作用下, 通过常规或非常规的装配方式产生结构多样的非核糖体肽, 正是这样的相互作用使得NRPS的装配得以有序高效地进行。在有些NRPS-PKS杂合的装配线中还存在模块间的交会对接(docking)结构域, 它为模块间底物的顺利呈递发挥了纽带作用。近年来发现了许多结构新颖的非核糖体肽, 通过生物信息学、基因工程和合成生物学等技术对这些新化合物的合成相关基因簇进行了挖掘[41], 并对其中关键NRPSs功能域的新功能和新的装配方式有了更为清晰的了解[10]。这些研究成果丰富了对非核糖体肽类化合物生物合成途径的认识, 为今后生物合成有活性的NRP, 继而寻找具有临床应用前景的新药或其先导物奠定了基础。然而, 对于基因簇较庞大的NRPSs的基因操作仍存在困难, 如存在NRPSs表达效率低及功能特异性改变等问题, 有些宿主菌株还缺乏相关的修饰酶基因, 因此还需构建能稳定并高效表达NRPSs的宿主, 甚至需要将一些外源的修饰酶基因整合到宿主基因组上, 并对NRPSs的蛋白结构、组成模块和作用机制等方面进行更深入具体的研究。目前已有研究表明, 在普伐他汀(plipastatin)的生物合成中, 通过移动Te结构域, 即改变Te结构域在模块中的位置, 可控制环肽的大小, 继而获得氨基酸数量与普伐他汀不同的多肽[42]。今后可以通过对NRPSs的不同功能结构域进行重组[43], 人工设计新型NRPSs[44], 并利用其合成与已知非核糖体肽药物相比性能更佳、不良反应更小的同源类似物, 甚至产生具有全新结构的活性化合物促进非核糖体肽类新药的开发和临床应用。
| [1] | Reimer JM, Aloise MN, Harrison PM, et al. Synthetic cycle of the initiation module of a formylating nonribosomal peptide synthetase[J]. Nature, 2016, 529: 239–242. DOI:10.1038/nature16503 |
| [2] | Takeda K, Kemmoku K, Satoh Y, et al. N-Phenylacetylation and nonribosomal peptide synthetases with substrate promiscuity for biosynthesis of heptapeptide variants, JBIR-78 and JBIR-95[J]. ACS Chem Biol, 2017, 12: 1813–1819. DOI:10.1021/acschembio.7b00314 |
| [3] | Süssmuth RD, Mainz A. Nonribosomal peptide synthesis-principles and prospects[J]. Angew Chem Int Ed Engl, 2017, 56: 3770–3821. DOI:10.1002/anie.201609079 |
| [4] | Wang H, Fewer DP, Holm L, et al. Atlas of nonribosomal peptide and polyketide biosynthetic pathways reveals common occurrence of nonmodular enzymes[J]. Proc Acad Natl Sci USA, 2014, 111: 9259–9264. DOI:10.1073/pnas.1401734111 |
| [5] | Greunke C, Glöckle A, Antosch J, et al. Biocatalytic total synthesis of ikarugamycin[J]. Angew Chem Int Ed Engl, 2017, 56: 4351–4355. DOI:10.1002/anie.201611063 |
| [6] | Dowling DP, Kung Y, Croft AK, et al. Structural elements of an NRPS cyclization domain and its intermodule docking domain[J]. Proc Acad Natl Sci USA, 2016, 113: 12432–12437. DOI:10.1073/pnas.1608615113 |
| [7] | Wang SY. Advances in the study of the mechanism and application of nonribosomal peptide synthetases[J]. Acta Microbiol Sin (微生物学报), 2007, 47: 734–737. |
| [8] | Bloudoff K, Fage CD, Marahiel MA, et al. Structural and mutational analysis of the nonribosomal peptide synthetase heterocyclization domain provides insight into catalysis[J]. Proc Acad Natl Sci USA, 2017, 114: 95–100. DOI:10.1073/pnas.1614191114 |
| [9] | Sung CT, Chang SL, Entwistle R, et al. Overexpression of a three-gene conidial pigment biosynthetic pathway in Aspergillus nidulans reveals the first NRPS known to acetylate tryptophan[J]. Fungal Genet Biol, 2017, 101: 1–6. DOI:10.1016/j.fgb.2017.01.006 |
| [10] | Payne JAE, Schoppet M, Hansen MH, et al. Diversity of nature's assembly lines-recent discoveries in non-ribosomal peptide synthesis[J]. Mol BioSyst, 2017, 13: 9–22. DOI:10.1039/C6MB00675B |
| [11] | Gulick AM. Conformational dynamics in the acyl-CoA synthetases, adenylation domains of nonribosomal peptide synthetases, and firefly luciferase[J]. ACS Chem Biol, 2009, 4: 811–827. DOI:10.1021/cb900156h |
| [12] | Li LL, Lin SJ. Recent advances in ANL andenylating enzymes[J]. Biotechnol Bull (生物技术通报), 2013(4): 14–20. |
| [13] | Song CX, Zhao CM, Yu ZN, et al. Prediction, overexpression and activity confirmation of adenylation domain in zwittermicin A biosynthesis gene cluster[J]. Acta Microbiol Sin (微生物学报), 2008, 48: 1260–1265. |
| [14] | Xia SS, Ma YL, Zhang W, et al. Identification of Sare0718 as an alanine-activating adenylation domain in marine actionmycete Salinispora arenicola CNS-205[J]. PLoS One, 2012, 7: e37487. DOI:10.1371/journal.pone.0037487 |
| [15] | Lu SL, Qi C. Determination of enzyme activity of Sare0357, a gene of adenylation domain from marine actinomycete S. arenicola CNS-205[J]. J Anhui Agric Sci (安徽农业科学), 2014, 42: 11632–11635. |
| [16] | Hou J, Robbel L, Marahiel MA. Identification and characterization of the lysobactin biosynthetic gene cluster reveals mechanistic insights into an unusual termination module architecture[J]. Chem Biol, 2011, 18: 655–664. DOI:10.1016/j.chembiol.2011.02.012 |
| [17] | Haslinger K, Redfield C, Cryle MJ. Structure of the terminal PCP domain of the non-ribosomal peptide synthetase in teicoplanin biosynthesis[J]. Proteins, 2015, 83: 711–721. DOI:10.1002/prot.v83.4 |
| [18] | Tufar P, Rahighi S, Kraas FI, et al. Crystal structure of a PCP/Sfp complex reveals the structural basis for carrier protein posttranslational modification[J]. Chem Biol, 2014, 21: 552–562. DOI:10.1016/j.chembiol.2014.02.014 |
| [19] | Liu Y, Zheng TF, Bruner SD. Structural basis for phosphorpantetheinyl carrier domain interactions in the terminal module of nonribosomal peptide synthetases[J]. Chem Biol, 2011, 18: 1482–1488. DOI:10.1016/j.chembiol.2011.09.018 |
| [20] | La Clair JJ, Foley TL, Schegg TR, et al. Manipulation of carrier proteins in antibiotic biosynthesis[J]. Chem Biol, 2004, 11: 195–201. DOI:10.1016/j.chembiol.2004.02.010 |
| [21] | Zheng ZM, Gu XB, Yu HQ, et al. Advances in main domains of nonribosomal peptide synthetases[J]. Chin J Antibio (中国抗生素杂志), 2005, 30: 120–124. |
| [22] | Kittilä T, Mollo A, Charkoudian LK, et al. New structural data reveal the motion of carrier proteins in nonribosomal peptide synthesis[J]. Angew Chem Int Ed Engl, 2016, 55: 9834–9840. DOI:10.1002/anie.201602614 |
| [23] | Bloudoff K, Schmeing TM. Structural and functional aspects of the nonribosomal peptide synthetase condensation domain superfamily:discovery, dissection and diversity[J]. Biochim Biophys Acta (BBA)-Proteins Proteomics, 2017, 1865: 1587–1604. DOI:10.1016/j.bbapap.2017.05.010 |
| [24] | Clugston SL, Sieber SA, Marahiel MA, et al. Chirality of peptide bond-forming condensation domains in nonribosomal peptide synthetases:the C5 domain of tyrocidine synthetase is a DCL catalyst[J]. Biochemistry, 2003, 42: 12095–12104. DOI:10.1021/bi035090+ |
| [25] | Gaudelli NM, Long DH, Townsend CA. β-Lactam formation by a non-ribosomal peptide synthetase during antibiotic biosynthesis[J]. Nature, 2015, 520: 383–387. DOI:10.1038/nature14100 |
| [26] | Meyer S, Kehr JC, Mainz A, et al. Biochemical dissection of the natural diversification of microcystin provides lessons for synthetic biology of NRPS[J]. Cell Chem Biol, 2016, 23: 462–471. DOI:10.1016/j.chembiol.2016.03.011 |
| [27] | Peschke M, Brieke C, Heimes M, et al. The thioesterase domain in glycopeptide antibiotic biosynthesis is selective for cross-linked aglycones[J]. ACS Chem Biol, 2018, 13: 110–120. DOI:10.1021/acschembio.7b00943 |
| [28] | Patel HM, Walsh CT. In vitro reconstitution of the Pseudomonas aeruginosa nonribosomal peptide synthesis of pyochelin:characterization of backbone tailoring thiazoline reductase and N-methyltransferase activities[J]. Biochemistry, 2001, 40: 9023–9031. DOI:10.1021/bi010519n |
| [29] | Quadri LEN, Keating TA, Patel HM, et al. Assembly of the Pseudomonas aeruginosa nonribosomal peptide siderophore pyochelin:in vitro reconstitution of aryl-4, 2-bisthiazoline synthetase activity from PchD, PchE, and PchF[J]. Biochemistry, 1999, 38: 14941–14954. DOI:10.1021/bi991787c |
| [30] | Samel SA, Czodrowski P, Essena LO. Structure of the epimerization domain of tyrocidine synthetase A[J]. Acta Cryst, 2014, D70: 1442–1452. |
| [31] | Walsh CT, Chen HW, Keating TA, et al. Tailoring enzymes that modify nonribosomal peptides during and after chain elongation on NRPS assembly lines[J]. Curr Opin Chem Biol, 2001, 5: 525–534. DOI:10.1016/S1367-5931(00)00235-0 |
| [32] | Pan HX, Tang GL. Unconventional assembling ways catalyzed by nonribosomal peptide synthetases[J]. Microbiology (微生物学通报), 2013, 40: 1783–1795. |
| [33] | Cheng YQ. Deciphering the biosynthetic codes for the potent anti-SARS-CoV cyclodepsipeptide valinomycin in Streptomyces tsusimaensis ATCC 15141[J]. ChemBioChem, 2006, 7: 471–477. DOI:10.1002/cbic.v7:3 |
| [34] | Shishido TK, Jokela J, Fewer DP, et al. Simultaneous production of anabaenopeptins and namalides by the cyanobacterium Nostoc sp. CENA543[J]. ACS Chem Biol, 2017, 12: 2746–2755. DOI:10.1021/acschembio.7b00570 |
| [35] | Keating TA, Suo ZC, Ehmann DE, et al. Selectivity of the yersiniabactin synthetase adenylation domain in the two-step process of amino acid activation and transfer to a holo-carrier protein domain[J]. Biochemistry, 2000, 39: 2297–2306. DOI:10.1021/bi992341z |
| [36] | Huang TT, Li LL, Brock NL, et al. Functional characterization of PyrG, an unusual nonribosomal peptide synthetase module from the pyridomycin biosynthetic pathway[J]. ChemBioChem, 2016, 17: 1421–1425. DOI:10.1002/cbic.v17.15 |
| [37] | Perez CE, Park HB, Crawford JM. Functional characterization of a condensation domain that links nonribosomal peptide and pteridine biosynthetic machineries in Photorhabdus luminescens[J]. Biochemistry, 2018, 57: 354–361. DOI:10.1021/acs.biochem.7b00863 |
| [38] | Gaudelli NM, Townsend CA. Epimerization and substrate gating by a TE domain in β-lactam antibiotic biosynthesis[J]. Nat Chem Biol, 2014, 10: 251–258. DOI:10.1038/nchembio.1456 |
| [39] | Zhang JR, Liu Ni, Cacho RA, et al. Structural basis of nonribosomal peptide macrocyclization in fungi[J]. Nat Chem Biol, 2016, 12: 1001–1003. DOI:10.1038/nchembio.2202 |
| [40] | Gao X, Haynes SW, Ames BD, et al. Cyclization of fungal nonribosomal peptides by a terminal condensation-like domain[J]. Nat Chem Biol, 2012, 8: 823–830. DOI:10.1038/nchembio.1047 |
| [41] | Nikolouli K, Mossialos D. Bioactive compounds synthesized by non-ribosomal peptide synthetases and type-Ⅰ polyketide synthases discovered through genome-mining and metagenomics[J]. Biotechnol Lett, 2012, 34: 1393–1403. DOI:10.1007/s10529-012-0919-2 |
| [42] | Gao L, Liu HX, Ma Z, et al. Translocation of the thioesterase domain for the redesign of plipastatin synthetase[J]. Sci Rep, 2016, 6: 38467. DOI:10.1038/srep38467 |
| [43] | Xie XM, Wang F, Chen JY, et al. Advances in daptomycin biosynthesis[J]. Microbiology (微生物学通报), 2013, 40: 1754–1764. |
| [44] | Muchiri R, Walker KD. Paclitaxel biosynthesis:adenylation and thiolation domains of an NRPS TycA PheAT module produce various arylisoserine CoA thioesters[J]. Biochemistry, 2017, 56: 1415–1425. DOI:10.1021/acs.biochem.6b01188 |
 2018, Vol. 53
2018, Vol. 53


