2. 西藏藏医学院, 西藏 拉萨 850000
2. Tibetan Traditional Medical College, Lhasa 850000, China
藏医药学是祖国民族医药学的重要组成部分, 具有悠久的发展历史及丰富的科学与藏族文化内涵。藏药基原植物多生长于青藏高原, 生存环境严酷。这些高山植物物种经历了不断发展进化的过程, 具备丰富多样的形态、基因特征及化学成分。因此, 深入开展藏药品种整理, 探讨高山植物物种遗传多样性及藏药材品质与具体产地的相关性等问题, 可为藏药材客观、准确的品种鉴定、活性物质基础研究、质量标准的完善、濒危藏药植物的就地保护及栽培学研究等工作打下坚实基础, 具有重要的科学意义。但由于种种原因, 藏药品种整理及质量标准研究等工作尚不能适应藏医药学发展的需要。目前, 2015年版《中华人民共和国药典》收载的藏药品种有限, 系统的藏药材质量标准工作尚在积累之中; 当务之急是开展相关深入的基础性研究工作。
常见藏药品种之一“巴莴色保” (
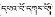

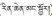


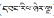
叶绿体是绿色植物细胞内具有自主遗传信息的细胞器, 拥有自身完整的一套基因组[2]。典型的叶绿体基因组结构由两个反向重复区(inverted repeats, IRs)、大单拷贝区(large single copy, LSC)和小单拷贝区(small single copy, SSC)组成。近年来, 随着大规模测序技术的快速发展, 植物叶绿体基因组的研究工作得到极大推动。叶绿体基因组的结构、大小和基因种类一般较为保守, 同时其进化速率仅次于核基因组, 基因组序列含有大量的遗传信息, 现已成为药用植物物种鉴定及研究其系统进化的有力工具[3-5]。
龙胆科(Gentianaceae)植物全世界约700种, 第一大属龙胆属Gentiana约400种, 而獐牙菜属Swertia近170种, 两者占龙胆科总数的81%[6]; 同时, 常见中藏药材多来源于此, 如秦艽、龙胆、当药、青叶胆、解吉(


民族植物学考察及分类学鉴定 与西藏藏医学院藏族同行合作开展藏药“巴莴色保”的考察工作。凭证标本及实验样品采自西藏错那县, 采集号:高娜娜等2015XZ004A, 地理坐标N 27°55.407', E 091°51.594', 海拔4 240 m。同时, 采集新鲜叶片经硅胶快速干燥, 保存备用。标本各器官的观察, 花部解剖, 尤其注意叶序类型与腺窝的形态学特征等, 并参考相关文献[6, 11], 进行植物分类学鉴定。凭证标本存放于上海中医药大学中药学院药用植物标本室。
DNA提取 以单株植物为叶绿体全基因组测序样本, 参考Doyle的CTAB法[12], 提取样品总DNA。
叶绿体全基因组序列的获得及图谱绘制 参考被子植物叶绿体全基因组测序通用引物[13], 并用Sanger法测序[14]。对样品叶绿体基因组片段进行PCR扩增, 其中通用引物无法扩出的片段以及gaps, 参考相关序列[15]进行引物的优化调整, 应用PrimerSelect (Lasergene 7.0)软件, 重新设计引物。扩增获得的叶绿体基因组各片段序列, 采用Lasergene SeqMan软件进行拼接, 最终得到轮叶獐牙菜叶绿体全基因组序列。
采用叶绿体基因组在线注释软件DOGMA[16]对样品叶绿体全基因组进行初步注释; 参考近缘种序列[15, 17, 18], 结合编码蛋白基因起始密码子、终止密码子, 对序列进行手工校正; tRNA基因应用在线软件tRNAscan-SE 2.0[19]进行验证。
将注释好的基因组序列通过Sequin软件提交到NCBI, 并以GenBank格式文件输出, 提交到在线软件OGDRAW[20], 导出后获得样品叶绿体基因组物理图谱。
基因组序列结构的初步分析 应用在线分析软件REPuter[21]对样品基因组正向重复序列(F: Forward repeat sequence)、反向重复序列(R: Reverse repeat sequence)、正向互补序列(C: Complement sequence)和回文序列(P: Plaindromic sequence) 4种类型的重复序列进行分析, 参数设置为: n≥30 (重复序列的长度), 90% (序列的一致度), 3 (Hamming distance)。
应用MISA软件[22]查找基因组简单重复序列(SSR)位点, 设置参数为:单核苷酸(mono-)≥10, 二核苷酸(di-)≥5, 三核苷酸(tri-)≥4, 四核苷酸(tetra-)≥3, 五核苷酸(penta-)≥3, 六核苷酸(hexa-) ≥3。
轮叶獐牙菜叶绿体基因组序列系统学意义的初步评价 基于龙胆科已报道的叶绿体基因组序列, 以玄参科齿叶玄参Scrophularia dentata Royle ex Benth.为外类群, ML法构建系统进化树(MEGA5.0[23]), 参数设置为Bootstrap: 1000, Model: Kimura 2-parameter。
结果与分析 1 “巴莴色保”基原植物形态特征及分类学位置多年生草本。茎粗壮。基生叶莲座状; 茎生叶轮生, 叶片椭圆形或椭圆状卵形。塔形复聚伞花序, 多花; 花4数, 下垂; 花萼宿存; 花冠黄绿色, 有深紫色脉纹, 钟状, 裂片倒卵形, 先端截形, 具不规则细波状齿, 基部腺窝1, 黄色, 卵状三角形, 裸露; 子房卵状椭圆形。蒴果。种子两端具狭翅[6]。我国高山特有物种, 分布于西藏南部; 生于灌丛中, 海拔3 800~4 200 m (图 1)。

|
Figure 1 Swertia verticillifolia. A: Habit; B: Flower, front view; C: Corolla lobe with a nectary, ruler: 1 mm scale; D: Flower, lateral view; E: Ovary ovoid-ellipsoid; F: Capsule; G: Seed both ends winged |
经分类学鉴定, 其基原植物为龙胆科獐牙菜属大花组(Sect. Macranthos)轮叶獐牙菜Swertia verticillifolia T. N. Ho et S. W. Liu。物种鉴定要点:基生叶发达, 茎生叶轮生; 花冠钟状, 黄绿色, 4裂, 每裂片具裸露腺窝1。该组植物国产4种, 列一检索表区别之。
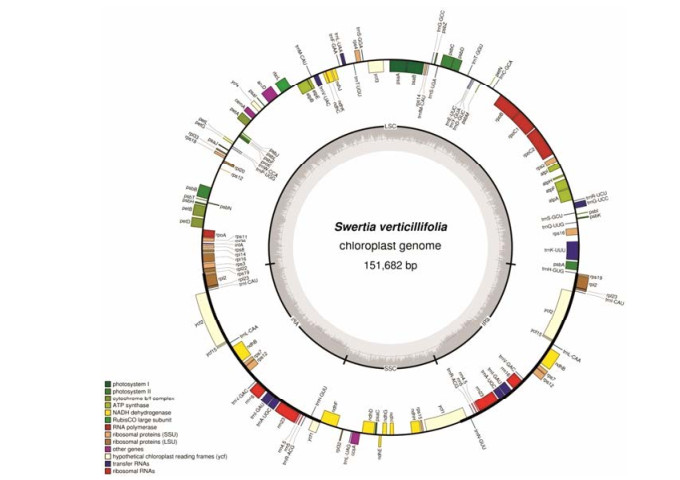
2 轮叶獐牙菜叶绿体基因组基本结构轮叶獐牙菜叶绿体基因组序列总长151 682 bp, 包括两个反向重复区域(IRa、IRb:各25 362 bp)、一个大单拷贝区(LSC: 82 623 bp)和一个小单拷贝区(SSC: 18 335 bp), 表 1。基因组基因分布情况: 84个编码蛋白基因、37个tRNA基因和8个rRNA基因, 共129个基因。完整序列已上传GenBank, 获得登录号MF795137 (图 2、表 2)。
| Table 1 Base composition in Swertia verticillifolia chloroplast genome |
|
Figure 2 Chloroplast genome map of Swertia verticillifolia. Genes outside of the map transcripted in the clockwise direction and genes inside of the map transcripted in the counter-clockwise direction. Each color represents the same class of genes |
| Table 2 Gene composition in Swertia verticillifolia chloroplast genome. ×2: Repeat genes (In IR regions) |
应用在线分析软件REPuter对叶绿体基因组进行F、P、R、C4种类型重复序列分析, 得到F重复10个、P重复8个, 没有得到C重复和R重复。所有重复序列长度在30~39 bp之间, 多数重复都位于基因间区以及基因内含子, 主要位于大单拷贝区(LSC)。
4 基因组简单重复序列分析应用MISA软件搜索简单重复序列(SSR)位点, 设置一定参数, 共得到45个SSR, 其中29个单核苷酸、4个二核苷酸、4个三核苷酸和8个四核苷酸。未检测到五、六核苷酸SSR。大部分SSR (64.44%)位于LSC区, 31.11%位于SSC区, 仅有2个(4.44%)位于IR区。所有SSR中, 68.89%位于基因间隔区(IGS), 17.78%位于基因编码区(CDS), 11.11%位于内含子区域(intron), 另有2.22%横跨基因编码区与间隔区(CDS-IGS)。在29个单核苷酸SSR中, 主要以A/T重复为主, 仅有1个C重复; 二核苷酸SSR均为AT/TA重复。三核苷酸SSR多为4次重复, 四核苷酸SSR多为3次重复。
5 獐牙菜属Swertia与龙胆属Gentiana植物基因组结构及其系统学意义本课题组已测定的龙胆属植物叶绿体全基因组序列:粗茎秦艽Gentiana crassicaulis Duthie ex Burk.、西藏秦艽G. tibetica King ex Hook. f.、麻花艽G. straminea Maxim.及粗壮秦艽G. robusta King ex Hook. f. (登录号: KJ676538、KU975374、KJ657732、KT159969); GenBank下载相关序列:龙胆属大叶秦艽G. macrophylla Pall.及线叶龙胆G. lawrencei Burk. var. farreri (Balf. f.) T. N. Ho, 獐牙菜属川西獐牙菜Swertia mussotii Franch. (NC_035719、KX096882、NC_031155)。
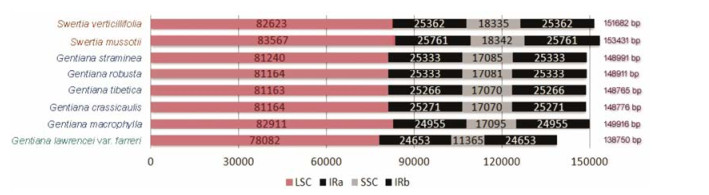
就现有资料来看, 獐牙菜属植物基因组序列长度在151~154 kb之间, 龙胆属植物长度在138~150 kb之间(图 3)。两者长度有近1 700 bp的差距。另外, 除线叶龙胆缺少6个ndh基因外[24], 两属其他植物基因组结构基本相同。
|
Figure 3 Comparison of the plastid genome sequence of Swertia and Gentiana chloroplast genomes. The genomes within Swertia are longer (151-154 kb), and shorter within Gentiana (138-150 kb) |
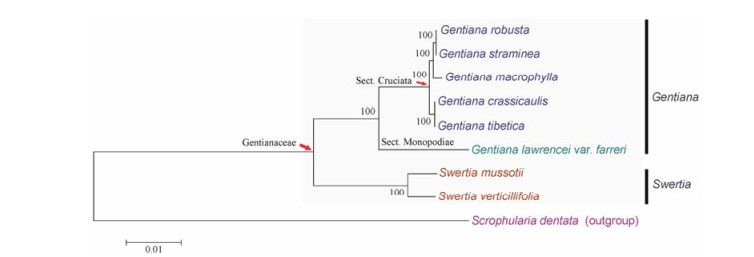
基于上述两属8种植物全基因组序列, 以玄参科齿叶玄参Scrophularia dentata Royle ex Benth.为外类群, 构建ML系统进化树(图 4), 结果显示:首先分出外类群, 进而分为两大支, 一大支为“獐牙菜属”支, 另一大支为“龙胆属”支。“龙胆属”大支再分出两支: “秦艽组Sect. Cruciata”支及“多枝组Sect. Monopodiae”支; 树中显示的分子系统发育关系与经典分类学观点高度一致。
|
Figure 4 Maximum likelihood (ML) trees based on Swertia and Gentiana chloroplast genomes. Scrophularia dentata was set up as an outgroup |
我国青藏高原是世界龙胆科植物分布中心之一, 近60%的物种分布于此。深入探讨各类群的生物学特性, 物种形成机制及系统发育关系等问题, 具有重要的科学意义。同时, 该科植物具有重要的药用价值, 如何可持续利用有限的高山药用植物资源?当务之急是开展种质资源调查、物种遗传多样性分析等基础性工作。
獐牙菜属Swertia为龙胆科第二大属, 约170种, 国产7组79种[6]。目前, 獐牙菜属植物相关遗传背景研究工作有限。就叶绿体全基因组资料来看, 仅本文报道的大花组(Sect. Macranthos)轮叶獐牙菜S. verticillifolia T. N. Ho et S. W. Liu以及GenBank下载的多枝组(Sect. Ophelia)川西獐牙菜S. mussotii Franch. 2条完整基因组序列。尚未见其他5组植物相关序列资料, 与该属具有的物种多样性而言, 基础工作明显不足。同时, 基于龙胆科有限序列资料构建的系统发育树, 显示叶绿体基因组蕴含的分子系统发育信息, 在该科属间系统关系的探讨、物种DNA条形码候选序列的筛选等方面具有重要意义。
比较轮叶獐牙菜与川西獐牙菜叶绿体全基因组结构特点:两者序列长度相差约1 800 bp。编码区有多段的插入/缺失型变异位点, 多数位于基因编码区起始子与终止子前后。其中ycf1与ycf2基因上的变异位点位于基因编码区中段, ycf1基因编码区上有较多插入/缺失片段。可见ycf1, ycf2基因变异速率较快, 适合物种鉴定及近缘种亲缘关系梳理[25]。基因间区位置, 变异幅度最大的是位于反向重复区的IGS (ycf15, trnL-CAA), 两者存在269 bp连续的长度多态性差异, 可作为两物种鉴定的候选DNA分子标记。有研究显示, ycf15基因独特的序列结构及其显现的丰富多样的遗传信息不仅具有一定的物种鉴定意义, 而且在植物系统进化分析研究中有较大的潜力[26]。
简单重复序列(SSR), 也称微卫星DNA, 广泛分布于各种真核生物的基因组中, 在植物基因组中的含量非常丰富。因其重复单位的数目存在高度变异, 造成序列多个位点的多态性, 故而SSR标记是目前常用的微卫星标记之一, 已广泛应用于种群遗传学、系统发育研究中[27-31]。同时, 在药用植物的相关研究中亦有报道。如黄芩Scutellaria baicalensis Georgi的基因组SSR分子标记的开发[32]; 对党参Codonopsis pilosula (Franch.) Nannf.转录组中SSR位点信息分析[33]。有关獐牙菜属药用植物SSR分子标记研究工作, 如川西獐牙菜Swertia mussotii Franch.转录组SSR的探讨[34]。本文共得到轮叶獐牙菜叶绿体基因组45个SSR, 其中29个单核苷酸、4个二核苷酸、4个三核苷酸和8个四核苷酸。下一步将开展轮叶獐牙菜转录组SSR分析, 为该物种的种质资源开发、物种多样性研究及分子育种研究提供科学资料。
叶绿体基因组序列含有大量遗传信息, 特定片段psbA-trnH等与核基因组ITS区等序列组合, 在构建药用植物物种DNA条形码工作中显示了独特的作用[35]。本文比较獐牙菜属与龙胆属属间叶绿体全基因组结构特点:两属序列长度范围明显不同, 属间长度多态性变异幅度较大区域集中在小单拷贝区。初步筛选得到IGS (rrn5, rrn4.5)、IGS (rps15, ycf1)、IGS (ndhF, rpl32)、IGS (ndhG, ndhI)及ndhA 5个片段, 可作为两属属间候选DNA分子标记。以齿叶玄参为外类群, 构建系统进化树, 支持初筛片段结果。
藏药“巴莴色保”基原植物——轮叶獐牙菜Swertia verticillifolia T. N. Ho et S. W. Liu为我国高山特有植物, 仅分布于西藏南部, 生长在海拔3 800 m以上的高山草甸, 资源量有限。目前, 尚未见深入的生物学特性及药学研究工作。在开展“巴莴色保”藏药材相关的性状、显微及理化鉴定及质量标准制订等工作的同时, 物种传粉生物学、种子生物学等物种就地保护相关基础性研究工作亦刻不容缓。
| [1] | Ga wu. Jingjing Bencao (藏药晶镜本草, 藏文版)[M]. Beijing: The Ethnic Publishing House, 1995: 231. |
| [2] | McFadden GI. Primary and secondary endosymbiosis and the origin of plastids[J]. J Phycol, 2001, 37: 951–959. DOI:10.1111/jpy.2001.37.issue-6 |
| [3] | Yap JYS, Rohner T, Greenfield A, et al. Complete chloroplast genome of the Wollemi pine (Wollemia nobilis): structure and evolution[J]. PLoS One, 2015, 10. |
| [4] | Li XW, Hu ZG, Lin XH, et al. High-throughput pyrose-quencing of the complete chloroplast genome of Magnolia officinalis and its application in species identification[J]. Acta Pharm Sin (药学学报), 2012, 47: 124–130. |
| [5] | Yao H, Yang P, Zhou H, et al. Identification of medicinal plant Dendrobium based on the chloroplast psbK-psbI intergenic spacer[J]. Acta Pharm Sin (药学学报), 2015, 50: 783–787. |
| [6] | Ho TN. Flora Reipublicae Popularis Sinicae vol. 62 (中国植物志, 62卷)[J]. Beijing: Science Press, 1988: 1–376. |
| [7] | Ni LH, Zhao ZL, Xu HX, et al. Chloroplast genome structures in Gentiana (Gentianaceae), based on three medicinal alpine plants used in Tibetan herbal medicine[J]. Curr Genet, 2017, 63: 241–252. DOI:10.1007/s00294-016-0631-1 |
| [8] | Ni LH, Zhao ZL, Xu HX, et al. The complete chloroplast genome of Gentiana straminea, (Gentianaceae), an endemic species to the Sino-Himalayan subregion[J]. Gene, 2016, 577: 281–288. DOI:10.1016/j.gene.2015.12.005 |
| [9] | Ni LH, Zhao ZL, Xiong B, et al. A strategy for identifying six species of Sect. Cruciata (Gentiana) in Gansu using DNA barcode sequences[J]. Acta Pharm Sin (药学学报), 2016, 51: 821–827. |
| [10] | Zhao ZL, Gaawe D, Wang ZT. Identification of medicinal plants used as Tibetan Traditional Medicine Jie-Ji[J]. J Ethnopharmacol, 2010, 132: 122–126. DOI:10.1016/j.jep.2010.07.051 |
| [11] | Ho TN, Liu SW. New taxa of Swertia L. from China[J]. Acta Phytotax Sin (植物分类学报), 1980, 18: 75–85. |
| [12] | Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf material[J]. Phyochem Bull, 1987, 19: 11–15. |
| [13] | Qian J, Song J, Gao H, et al. The complete chloroplast genome sequence of the medicinal plant Salvia miltiorrhiza[J]. PLoS One, 2013, 8: e57607. DOI:10.1371/journal.pone.0057607 |
| [14] | Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors[J]. Proc Natl Acad Sci U S A, 1977, 74: 5463–5467. DOI:10.1073/pnas.74.12.5463 |
| [15] | Xiang B, Li X, Qian J, et al. The complete chloroplast genome sequence of the medicinal plant Swertia mussotii using the PacBio RS Ⅱ platform[J]. Molecules, 2016, 21: 1029. DOI:10.3390/molecules21081029 |
| [16] | Ku C, Chung WC, Chen LL, et al. The complete plastid genome sequence of Madagascar Periwinkle Catharanthus roseus (L.) G. Don: plastid genome evolution, molecular marker identification, and phylogenetic implications in Asterids[J]. PLoS One, 2013, 8: e68518. DOI:10.1371/journal.pone.0068518 |
| [17] | Nie XJ, Lv SZ, Zhang YX, et al. Complete chloroplast genome sequence of a major invasive species, crofton weed (Ageratina adenophora)[J]. PLoS One, 2012, 7: e36869. DOI:10.1371/journal.pone.0036869 |
| [18] | Choi KS, Park SJ. The complete chloroplast genome sequence of Aster spathulifolius (Asteraceae); genomic features and relationship with Asteraceae[J]. Gene, 2015, 572: 214–221. DOI:10.1016/j.gene.2015.07.020 |
| [19] | Goremykin VV, Holland B, Hirsch-Ernst KI, et al. Analysis of Acorus calamus chloroplast genome and its phylogenetic implications[J]. Mol Biol Evol, 2005, 22: 1813–1822. DOI:10.1093/molbev/msi173 |
| [20] | Lohse M, Drechsel O, Bock R. OrganellarGenomeDRAW (OGDRAW): a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J]. Curr Genet, 2007, 52: 267–274. DOI:10.1007/s00294-007-0161-y |
| [21] | Kurtz S, Choudhuri JV, Ohlebusch E, et al. REPuter: the manifold applications of repeat analysis on a genomic scale[J]. Nucleic Acids Res, 2001, 29: 4633–4642. DOI:10.1093/nar/29.22.4633 |
| [22] | Thiel T, Michalek W, Varshney RK, et al. Exploiting EST databases for the development and characterization of gene- derived SSR-markers in barley (Hordeum vulgare L.)[J]. Theo Appl Gen, 2003, 106: 411–422. DOI:10.1007/s00122-002-1031-0 |
| [23] | Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731–2739. DOI:10.1093/molbev/msr121 |
| [24] | Fu PC, Zhang YZ, Geng HM, et al. The complete chloroplast genome sequence of Gentiana lawrencei var. farreri (Gentianaceae) and comparative analysis with its congeneric species[J]. Peer J, 2016, 4: e2540. DOI:10.7717/peerj.2540 |
| [25] | Huang JL, Sun GL, Zhang D. Molecular evolution and phylogeny of the angiosperm ycf2 gene[J]. J Syst Evol, 2010, 48: 240–248. DOI:10.1111/jse.2010.48.issue-4 |
| [26] | Gao NN, Zhao ZL, Ni LH. Prospect: identification of medicinal plant based on chloroplast gene ycf15[J]. Chin Tradit Herb Drugs (中草药), 2017, 48: 3210–3217. |
| [27] | Powell W, Morgante M, McDevitt R, et al. Polymorphic simple sequence repeat regions in chloroplast genomes: applications to the population genetics of pines[J]. Proc Natl Acad Sci U S A, 1995, 92: 7759–7763. DOI:10.1073/pnas.92.17.7759 |
| [28] | Provan J, Corbett G, McNicol JW, et al. Chloroplast DNA variability in wild and cultivated rice (Oryza spp.) revealed by polymorphic chloroplast simple sequence repeats[J]. Genome, 1997, 40: 104–110. DOI:10.1139/g97-014 |
| [29] | Pauwels M, Vekemans X, Gode C, et al. Nuclear and chloroplast DNA phylogeography reveals vicariance among European populations of the model species for the study of metal tolerance, Arabidopsis halleri (Brassicaceae)[J]. New Phytol, 2012, 193: 916–928. DOI:10.1111/j.1469-8137.2011.04003.x |
| [30] | Xue J, Wang S, Zhou SL. Polymorphic chloroplast microsatellite loci in Nelumbo (Nelumbonaceae)[J]. Am J Bot, 2012, 99: e240–e244. DOI:10.3732/ajb.1100547 |
| [31] | Jiao Y, Jia HM, Li XW, et al. Development of simple sequence repeat (SSR) markers from a genome survey of Chinese bayberry (Myrica rubra)[J]. BMC Genomics, 2012, 13: 151–154. DOI:10.1186/1471-2164-13-151 |
| [32] | Qi LJ, Long P, Jiang C, et al. Development of microsatellites and genetic diversity analysis of Scutellaria baicalensis Georgi using genomic-SSR markers[J]. Acta Pharm Sin (药学学报), 2015, 50: 500–505. |
| [33] | Wang D, Cao LY, Gao JP. Data mining of simple sequence repeats in Codonopsis pilosula transcriptome[J]. Chin Tradit Herb Drugs (中草药), 2014, 45: 2390–2394. |
| [34] | Liu Y, Yue CJ, Wang Y, et al. Data mining of simple sequence repeats in transcriptome sequences of Tibetan medicinal plant Zangyinchen Swertia mussotii[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 2068–2076. |
| [35] | Xiang L, Zhang W, Chen SL. Literature study and DNA barcoding of traditional Chinese medicine Qinghao (Artemisia annua L.)[J]. Acta Pharm Sin (药学学报), 2016, 51: 486–495. |
 2018, Vol. 53
2018, Vol. 53


