2. 山西大学 化学生物学与分子工程教育部重点实验室, 山西 太原 030006;
3. 山西大学 生物医学研究院, 山西 太原 030006
2. Key Laboratory of Chemical Biology and Molecular Engineering of Ministry of Education, Shanxi University, Taiyuan 030006, China;
3. Institutes of Biomedical Sciences, Shanxi University, Taiyuan 030006, China
代谢组学从整体角度测定机体对病理生理条件变化所产生的代谢应答, 从代谢物层面反映生物学事件, 研究生命活动规律, 因而成为病理生理、药理毒理等的重要研究手段[1]。核磁共振(NMR)、液相色谱-质谱联用(LC-MS)和气相色谱-质谱联用(GC- MS)是代谢组学研究的主要技术[2]。与NMR相比, 质谱技术具有较高的灵敏度和分辨率, 其中GC-MS因具有较为成熟的代谢物标准图库, 方便对代谢组分的定性鉴别, 且检测谱可覆盖更多低极性成分, 在代谢组学研究中应用广泛[3, 4]。
应用代谢组学手段研究抑郁症病理和药物抗抑郁药理, 加深了对抑郁症的认识[5-7]。对双相抑郁患者尸检脑组织样品的代谢组学研究显示[5], 谷氨酸盐、N-乙酰天冬氨酸等代谢物丰度与健康对照显著不同, 且随药物治疗相应回调; 对抑郁症患者粪便代谢组学研究[6], 发现了“脂质、氨基酸代谢紊乱”和“小分子生物化学紊乱”, 为提高对抑郁症病理生理特征的认识, 深入研究抑郁症发病机制和开发生物标志物提供了重要参考[5-7]。
代谢组学数据分析运用多元统计(PCA、PLS-DA等)手段从海量代谢物获得差异信息, 不仅可以对病理生理状态进行有效分类, 还可给出病理生理指标相关的特征性代谢物, 与形态学、行为学和有限生化指标检测等方法相比具有较高灵敏性和广谱性[1]。但传统静态代谢组学研究复杂疾病病理仅从横断面数据入手研究, 未充分考虑疾病病理过程的复杂性, 因而易得到相对片面的结论, 不能全面反映生理病理演变规律[8, 9]。
动态代谢组学将生理病理动态演变过程纳入分析范畴, 区分随时间演变的群体差异和不随时间变化的个体差异, 更准确全面反映生理病理特点[9, 10]。其中方差同步主成分分析[ANOVA (analysis of variance)-simultaneous component analysis, ASCA]充分考虑生理病理过程, 按时间因素、表型因素、时间因素和表型因素的交互效应三方面将代谢组学数据分解为效应矩阵, 分别对此三组矩阵进行PCA分析, 得到各效应方面随时间改变的得分变化趋势, 从而对生理病理过程给出更为准确的生物学解释, 已成功应用于药物毒理[8]和营养干预[9]等研究。
基于上述GC-MS代谢组学的优势, 和静态代谢组学数据分析的不足, 本研究应用GC-MS测定大鼠CUMS造模过程血浆代谢组数据, 联合应用静态和动态代谢组数据分析手段, 分析抑郁大鼠血浆代谢物变化规律, 为深入了解抑郁症病理提供参考。
材料与方法实验动物 SPF级雄性SD大鼠, 体重(200 ± 20 g), 购自北京维通利华实验动物技术有限公司, 动物许可证号: SCXK (京) 2014-0001。昼夜节律光照下饲养, 环境适应一周后开始实验。
试剂与仪器 N-甲基-N-(三甲基硅烷)三氟乙酰胺(MSTFA)、1%三甲基氯硅烷、甲氧胺盐酸盐(methoxyamine hydrochloride)购自美国Sigma-Aldrich公司; 色谱纯乙腈、甲醇购自赛默飞世尔科技(中国)有限公司。丙酸、丁酸、D-阿洛糖、肌醇、亚麻酸(9, 12, 15- octadecatrienoic acid)购自中国食品药品检定研究院。
Trace GC-PolarisQ Mass气相色谱-质谱联用仪(美国Thermo-Fisher); 5810R台式高速冷冻离心机(德国Eppendorf); 代谢笼(苏州实验动物); YSD-4药理生理实验多用仪(北京和同创业)。
CUMS大鼠模型复制 按照Li等[11]的方法复制CUMS抑郁大鼠模型。SD大鼠在室温(24 ± 1) ℃、湿度(60 ± 5) %及昼夜节律光照环境适应7天后, 随机分为对照组和模型组各8只。对照组SD大鼠群养, 不给予刺激; 模型组大鼠给予总计10种实验刺激, 持续28天, 为保证刺激因素的随机不可预知性, 各种刺激方式随机排列, 且每种刺激不连续出现, 出现总次数不超过4次。
行为学测试与样品采集 CUMS模型复制过程第0、7、14、21和28天分别进行体重和糖水偏爱率测定。为降低误差, 固定在测量日9时测量体重。糖水偏爱率测试按以下方案:给实验大鼠提供双瓶水, 一瓶为普通饮用水, 另一瓶为含1%蔗糖的饮用水。测试前整晚禁食禁水, 后同时给予上述双瓶水, 自由饮水1 h后称取饮水瓶重量计算消耗量。按以下公式计算:糖水偏爱率(%) =糖水摄入量(g) /总饮水摄入量(g) × 100%。所有实验大鼠于造模过程第0、7、14、21和28天眼眶毛细管取血, 置于EDTA抗凝管。收集的血液样品常温静置30 min, 3 000 r·min-1离心15 min, 吸取上清, 转移至1 mL洁净EP管, 置-80 ℃保存。
GC-MS分析测试
样品制备 将冻存的血浆样品于冰水混合物中解冻后, 取200 μL加入乙腈400 μL, 充分振荡均匀, 4 ℃, 13 000 r·min-1离心20 min, 取上清液400 μL, 30 ℃下真空干燥12 h。加入甲氧胺吡啶溶液(15 mg·mL-1) 30 μL, 70 ℃下作用1 h, 反应结束后加入MSTFA 50 μL, 40 ℃作用90 min进行衍生化反应, 加入内标二十四烷-正庚烷100 μL, 混匀待测。
色谱质谱条件 DB-5MS色谱柱(5%二苯基- 95%二甲基聚硅氧烷, 30 m × 0.25 mm × 0.25 μm, Agilent J & W Scientific, Folsom, 美国); 进样口温度280 ℃; 1/10进样分流比分流进样; EI离子源温度和传输线温度分别为200 ℃和280 ℃; 电子能量为70 eV; 质谱扫描采用全扫描模式, 扫描范围m/z 50~650。程序升温条件为:起始80 ℃, 保持3 min; 以7 ℃·min-1速度升温至140 ℃, 保持4 min; 以5 ℃·min-1速度升温至180 ℃, 保持6 min, 再以5 ℃·min-1速度升温至280 ℃, 保持2 min。
GC-MS图谱处理 首先在Xcalibur工作站中将图谱由raw格式转化为NetCdf格式。使用R软件导入XCMS数据包进行滤噪、谱峰对齐及提取等操作, 将生成的csv文件导入Excel中, 以每个样品总峰面积分别进行归一化。
静态多元统计分析 将CUMS模型复制第28天代谢组数据归一化后导入SIMCA-P 14.0 (Umetric, Sweden)软件, 经帕莱托标度化后进行PCA、PLS-DA和OPLS-DA分析。利用载荷图S-Plot确定组间差异变量, 采用SPSS 22.0进行独立样本t检验, 以变量在项目中的重要性(VIP值> 1)以及t检验显著性(P <0.05)确定差异代谢物。
方差同步主成分分析(ANOVA-simultaneous component analysis, ASCA) 将CUMS造模过程中5个时间点(0、7、14、21和28天)代谢组数据归一化后载入MetaboAnalyst网站(metaboanalyst.ca)在线分析[12], 数据使用常和标准化(constant sum)、对数变换(log transformation)和帕莱托标度化。使用Leverage评价代谢物对模型的贡献, SPE评价模型对特定代谢物的适配程度。具有低SPE值和高Leverage值的代谢物对模型贡献值较大, 作为ASCA分析差异代谢物。
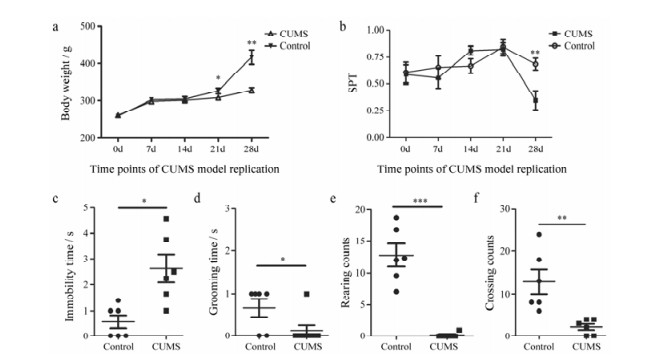
结果 1 CUMS模型复制成功为检验CUMS模型复制效果, 分别测定模型组和对照组各时间点的体重和糖水偏爱率, 结果如图 1a和1b所示。模型组体重在第21天时显著低于对照组(P < 0.05), 在第28天时极显著低于对照组(P < 0.01)。模型组糖水偏爱率在第28天时极显著低于对照组(P < 0.01)。为进一步评价CUMS模型复制效果, 在造模第28天对大鼠进行了旷场实验, 结果如图 1c~1f所示。模型组在静止时间、理毛时间、直立次数、穿越格数等旷场实验指标方面与对照组相比均发生显著变化。上述结果提示, 第28天时CUMS模型复制成功。
|
Figure 1 Dynamic variations of body weight (a) and sugar preference rate (b) of rats under CUMS (chronic unpredictable mild stress) model replication, and abundance of plasma neurotransmitters (c-f) and open-field test results at the 28 d of CUMS model replication. Body weight was weighed at 9 a.m. of each time point. SPT represents sugar water preference rate, calculated with formula: SPT = sucrose consumption / (sucrose consumption + water consumption). Open field tests were performed on the 28th day of CUMS model replication |
使用实验大鼠血浆GC-MS总离子流色谱图, 通过NIST 05质谱库检索、对照品比对、文献参考、HMDB数据库(http://www.hmdb.ca/)比对等方法共指认出34种内源性代谢产物, 其中包括氨基酸、糖类、脂肪酸等, 结果见表 1。
| Table 1 Plasma metabolites of rats based on GC-MS detection |
使用PCA方法对CUMS复制过程所有时间点的血浆GC-MS结果进行分析, 结果如图 2a所示。血浆主成分在CUMS模型复制的第0天和第7天未见明显差异, 而在第14天产生了较大范围波动, 到第21天时整体与第0天明显分开, 第28天继续保持这种分开趋势。结果提示, 大鼠CUMS模型复制第14天时对外界持续刺激产生剧烈反应, 第21天时整体代谢轮廓与模型复制起点相比已发生显著变化。

|
Figure 2 PCA scatter plot of plasma metabolites of rats under CUMS model replication (a), and OPLS-DA scores plot (b) and S-plot (c) of rats at the 28th day of CUMS model replication. PCA scatter plot is consisted of total plasma metabolites from 5 time points (0, 7, 14, 21, and 28 d) of CUMS model replication. OPLS-DA and S-Plot were constructed based on the total plasma metabolites of CUMS and Control groups on the 28th day of CUMS model replication |
为寻找与CUMS模型复制相关的血浆代谢物, 对模型组与对照组进行OPLS-DA分析, 并作代谢物载荷图(图 2b、2c)。由图 2b可见, 对照组和模型组可以明显分离, 说明两组血浆代谢物发生明显改变。按照代谢物载荷图(图 2c)中距原点越远对分组差异贡献(VIP)越大原则, 筛选得到VIP > 1的代谢物, 结合代谢物峰面积的独立样本t检验, 确定峰面积具有显著差异的血浆代谢物(表 2)。
| Table 2 Typical plasma metabolites of rats with successful model replication of CUMS. *VIP: Variable importance for the projection. Metabolites with VIP > 1 in the OPLS-DA model and P < 0.05 in the independent samples t-test were considered to be typical metabolites contributing to the separation of the study groups |
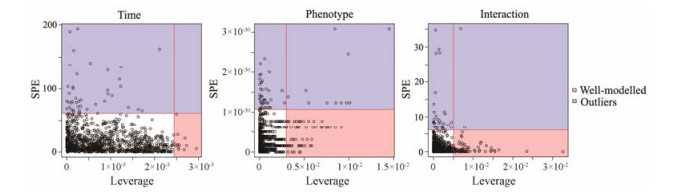
为深入考察CUMS模型复制过程中代谢物的动态变化, 使用ASCA动态代谢组学手段对各时间点的血浆GC-MS数据进行分析, 结果如图 3所示。按照Leverage值越高且SPE值越低的代谢物对模型贡献值越大的原则, 获得随时间、表型及二者交互作用显著变化的代谢物(表 3)。其中D-阿洛糖、肌醇和1, 5-脱水-D-山梨醇是随CUMS模型复制时间显著变化的代谢物; 丙酸、丁酸和D-阿洛糖是与CUMS表型相关的差异代谢物; 甲胺、乙烷、丙酸、丁酸和D-阿洛糖是与时间和表型交互作用显著相关的代谢物。
|
Figure 3 Leverage/SPE scatter plots of the ASCA variables submodels. The metabolomics dataset from all five time points of CUMS model replication were introduced to MetaboAnalyst web portal (www.metaboanalyst.ca) to construct these scatter plots. ANOVA (analysis of variance)-simultaneous component analysis (ASCA) was applied to split the original dataset into subsets describing the variations of phenotypes, the variations of time, and their interactions. Leverage was employed to evaluate the importance of a metabolite to the model, and SPE (special purpose entity) was used to test the fitness of a model for the metabolite. Variables with high Leverage value and low SPE value were considered to have significant contributions to the model |
| Table 3 Typical plasma metabolites of rats under CUMS model replication inferred from ASCA analysis. #SPE: Special purpose entity. SPE was used to test the fitness of a model for the metabolite |
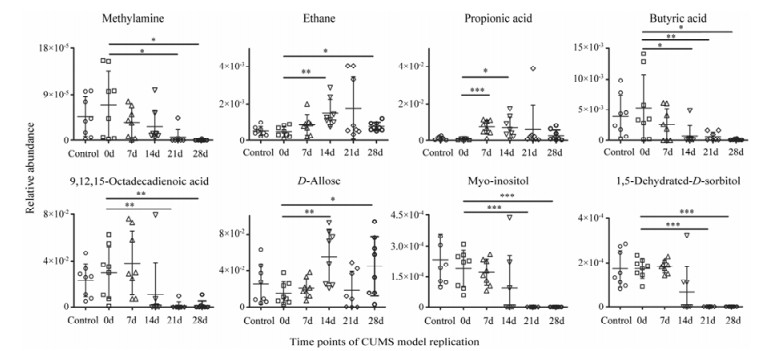
为进一步确证本研究静态和动态代谢组学分析获得的大鼠抑郁相关血浆代谢物(表 2、3), 对各代谢物在CUMS模型复制过程中的动态变化进行分析, 结果如图 4所示。静态和动态代谢组学共同发现的差异代谢物丙酸, 在CUMS模型复制第28天时, 与第0天相比显著降低(t检验, P < 0.01), 这与大鼠体重和糖水偏爱率及旷场实验指标变化趋势相符(图 1a和1b); 共同发现的差异代谢物D-阿洛糖在CUMS模型复制第14和28天时, 与CUMS模型复制起点相比显著升高。
|
Figure 4 Dynamic variations of typical plasma metabolites of rats under CUMS replication. Relative abundances of typical metabolites were represented by their relative peak area. Student t-test was performed to infer the inter-group difference. Differences supported by statistic test with *P < 0.05, **P < 0.01, ***P < 0.001 were shown |
动态代谢组学发现的差异代谢物丰度在CUMS模型复制的第28天时, 与第0天相比均发生显著变化; 且除乙烷外, 其他差异代谢物丰度在第21天时, 已经发生了上述显著变化, 这与大鼠体重变化趋势相符(图 1a); 静态代谢组学发现的差异代谢物亚麻酸丰度仅在CUMS模型复制第7天和第14天时, 与第0天相比发生显著变化。上述结果说明, 动态代谢组学数据分析可发现更多血浆差异代谢物, 且差异代谢物的丰度变化与CUMS模型的行为学指标变化相符程度更高。
讨论代谢组学近年来迅速发展成为疾病病理和药物药理毒理研究的重要方法, 为深入认识疾病病理发挥了重要作用[13, 14]。随着认识的不断深入, 人们发现复杂疾病病理发展是多重病理机制相互作用的过程, 并伴随机体代谢状态的不断变化[15]。而基于横断面数据的传统代谢组学分析方法不能全面反映复杂疾病的病理发展过程, 包含时间因素的动态代谢组学数据有助于更全面准确反映机体代谢状态[16, 17]。传统静态代谢组学分析方法只是将原始数据的最大变异进行降维, 把与时间相关的变异作为混杂因素处理, 忽略了随时间变化因素对整体的重要作用[18]。为更真实反映代谢组学数据的内部结构, 在数据降维的同时捕捉随时间趋势产生的变异, 人们研发了多种动态代谢组学数据分析方法[9]。其中方差同步主成分分析可用于检验不同来源变异随时间的变化趋势, 已被成功应用于化学计量学[19]、蛋白质组学[20]、心理测量学[21]等研究。
本研究通过比较静态代谢组和动态代谢组数据分析方法, 研究抑郁大鼠血浆代谢物变化规律。在两种方法均发现丙酸和D-阿洛糖作为差异代谢物的同时, 动态代谢组学数据分析发现了更多血浆差异代谢物, 且差异代谢物丰度与抑郁指标变化趋势符合程度更高。本研究为联用动态和静态代谢组学数据分析方法、深入研究抑郁症病理提供了新思路。
| [1] | Weng R, Shen S, Tian Y, et al. Metabolomics approach reveals integrated metabolic network associated with serotonin deficiency[J]. Sci Rep, 2015, 5: 11864. DOI:10.1038/srep11864 |
| [2] | Xia XT, Sun N, Liu CC, et al. Discovering potential biomarkers of depression and drug intervention of paroxetine base on 1H NMR metabolomics[J]. Acta Pharm Sin (药学学报), 2016, 51: 595–599. |
| [3] | Peng GJ, Shi BY, Tian JS, et al. 1H NMR based metabolomics study on the anti-depressant effect of genipin in rat hippocampus[J]. Acta Pharm Sin (药学学报), 2014, 49: 209–216. |
| [4] | Tian JS, Liu CC, Xiang H, et al. Investigation on the antidepressant effect of sea buckthorn seed oil through the GC- MS-based metabolomics approach coupled with multivariate analysis[J]. Food Function, 2015, 6: 3585–3592. DOI:10.1039/C5FO00695C |
| [5] | Lan M, Mcloughlin G, Griffin J, et al. Metabonomic analysis identifies molecular changes associated with the pathophysiology and drug treatment of bipolar disorder[J]. Mol Psychiatry, 2009, 14: 269–279. DOI:10.1038/sj.mp.4002130 |
| [6] | Wu D. Metabonomics Based Research on Fecal of De-pressed Patients (基于代谢组学的抑郁患者粪便研究)[D]. Chongqing: Chongqing Medical University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10631-1016783363.htm |
| [7] | Huang T, Lin C. Advances in biomarkers of major depressive disorder[J]. Adv Clin Chem, 2015, 68: 177–204. DOI:10.1016/bs.acc.2014.11.003 |
| [8] | Dai D, Gao Y, Chen J, et al. Time-resolved metabolomics analysis of individual differences during the early stage of lipopolysaccharide-treated rats[J]. Sci Rep, 2016, 6: 34136. DOI:10.1038/srep34136 |
| [9] | Wang WJ, Zhang QJ, Qu SY, et al. Introduction to data analysis method of dynamic metabolomics[J]. Chin J Health Stat (中国卫生统计), 2016, 33: 1075–1078. |
| [10] | Velzen E, Westerhuis J, Duynhoven J, et al. Multilevel data analysis of a crossover designed human nutritional intervention study[J]. J Proteome Res, 2008, 7: 4483–4491. DOI:10.1021/pr800145j |
| [11] | Li Z, Zheng X, Gao X, et al. Study of plasma metabolic profiling and biomarkers of chronic unpredictable mild stress rats based on gas chromatography/mass spectrometry[J]. Rapid Commun Mass Spectrom, 2010, 24: 3539–3546. DOI:10.1002/rcm.4809 |
| [12] | Xia J, Wishart D. Using MetaboAnalyst 3.0 for comprehensive metabolomics data analysis[J]. Curr Protoc Bioinform, 2016, 55: 14.10.1–14.10.91. DOI:10.1002/0471250953.2016.55.issue-1 |
| [13] | Clish C. Metabolomics:an emerging but powerful tool for precision medicine[J]. Cold Spring Harb Mol Case Stud, 2015, 1: a000588. DOI:10.1101/mcs.a000588 |
| [14] | Hoffman J, Lyu Y, Pletcher S, et al. Proteomics and metabolomics in ageing research:from biomarkers to systems biology[J]. Essays Biochem, 2017, 61: 379–388. DOI:10.1042/EBC20160083 |
| [15] | Trushina E, Nemutlu E, Zhang S, et al. Defects in mito-chondrial dynamics and metabolomic signatures of evolving energetic stress in mouse models of familial Alzheimer's disease[J]. PLoS One, 2012, 7: e32737. DOI:10.1371/journal.pone.0032737 |
| [16] | Montana G, Berk M, Ebbels T. Modelling short time series in metabolomics:a func- tional data analysis approach[J]. Adv Exp Med Biol, 2011, 696: 307–315. DOI:10.1007/978-1-4419-7046-6 |
| [17] | Huang X, Zeng J, Zhou L, et al. A new strategy for analyzing time-series data using dynamic networks:identifying prospective biomarkers of hepatocellular carcinoma[J]. Sci Rep, 2016, 6: 32448. DOI:10.1038/srep32448 |
| [18] | Smilde A, Westerhuis J, Hoefsloot H, et al. Dynamic me-tabolomic data analysis:a tutorial review[J]. Me-tabolomics, 2010, 6: 3–17. |
| [19] | de Noord O, Theobald E. Multilevel component analysis and multilevel PLS of chemical process data[J]. J Chemom, 2005, 19: 301–307. DOI:10.1002/(ISSN)1099-128X |
| [20] | Harrington P, Vieira N, Espinoza J, et al. Analysis of vari-ance-principal component analysis:a soft tool for proteomic discovery[J]. Anal Chim Acta, 2005, 544: 118–127. DOI:10.1016/j.aca.2005.02.042 |
| [21] | Timmerman M, Kiers H. Four simultaneous component models for the analysis of multivariate time series from more than one subject to model intra-individual and inter-individual differences[J]. Psychometrika, 2003, 68: 105–121. DOI:10.1007/BF02296656 |
 2018, Vol. 53
2018, Vol. 53


