尿苷肽类抗生素主要包括pacidamycins、mureidomycins、napsamycins和sansanmycins (SSs) (图 1), 主要表现为对结核分枝杆菌和铜绿假单胞菌的抑制活性, 其作用机制为抑制细菌细胞壁生物合成关键酶MraY (UDP-N-乙酰胞壁酸-五肽转位酶)的活性[1]。由于以MraY为靶点的药物还未用于临床, 可以有效避免与现用药物交叉耐药的情况。尿苷肽类抗生素化学结构中包含一个独特的母核结构: 4', 5'-烯胺-3'-脱氧尿苷通过肽键与假四肽肽链中的N-甲基-2, 3-二氨基丁酰基(N-methyl-2, 3-diamino butyryl, DABA)相连。SSs是2007年本所首次分离得到的新型尿苷肽类抗生素, 由Streptomyces sp. SS产生[2]。迄今为止, 文献报道的SS类化合物结构差异较小[3], 主要体现在组成假四肽肽链中氨基酸的种类不同: AA1以间位酪氨酸(m-Tyr)、酪氨酸(Tyr)、苯丙氨酸(Phe)为主, AA3通常为脂肪族氨基酸[如甲硫氨酸(Met)和丙氨酸(Ala)], 而AA4在绝大多数情况下为色氨酸(Trp)。由于靶点的新颖性以及结构的独特性, SS类化合物成为寻找新型抗耐药结核药物的先导化合物之一。

|
Figure 1 The chemical structures of uridyl peptide antibiotics. AA: Amino acid; m-Tyr: Meta-tyrosine; Met: Methionine; Trp: Tryptophan; Ala: Alanine; Phe: Phenylalanine |
近年来利用合成生物学手段实现了青蒿素[4]和紫杉醇[5]前体在微生物工程菌株中的合成, 展现了合成生物学技术在天然药物生产中的应用前景。合成生物学通过在微生物中人为设计和组装遗传元器件, 形成新的生物学系统, 在发现和创造新结构化合物中也将发挥重要作用[6]。选择一个合适的宿主是应用该技术的基础之一。目前所发现的具有抗感染、抗肿瘤、免疫抑制等作用的抗生素约有三分之二来源于链霉菌[7], 因此链霉菌属的工程菌株是利用该技术进行抗生素相关研究的良好宿主。天蓝色链霉菌是第一个完成基因组测定的模式菌株, 2011年Gomez-Escribano等[8]对S. coelicolor M145进行基因工程改造, 敲除了4个内源性抗生素生物合成基因簇, 并在rpoB和rpsL中依次引入点突变, 分别得到了其衍生菌株M1146、M1152和M1154, 由于其遗传背景清楚, 抗生素产量较M145有不同程度的提高, 成为异源表达的良好宿主。目前多种抗生素生物合成基因簇已在这几个菌株中成功表达, 例如Wang等[9]将nikkomycin生物合成基因簇sanG、sanF的启动子进行替换, 而后在S. coelicolor M1146中实现异源表达, 为利用合成生物学手段获得新结构核苷肽类化合物的研究奠定了基础。
为克服S. sp. SS遗传操作困难及菌株退化等问题, 同时也为后续合成生物学操作奠定基础, 本文利用PCR-Targeting构建了完整的SS生物合成基因簇, 并在S. coelicolor M1146、M1152和M1154中进行异源表达。借助于HPLC、HPLC-HRMS/MS等技术, 对异源表达菌株和对照菌株的次级代谢产物进行了系统对比分析, 结果表明, S. sp. SS中的主产物sansanmycin A (SS-A)在三株异源表达菌株中均有产生, 表明SS生物合成基因簇在S. coelicolor中实现了异源表达, 其中在S. coelicolor M1154中SS-A的产量与未退化的S. sp. SS相当, 同时, 在S. coelicolor M1154的发酵液中发现了一个可能为新结构的SS类似物, 通过与已知SSs化合物的MS/MS谱进行比较, 初步推测了该化合物的结构。
材料与方法 菌株、培养基和培养条件S. sp. SS[2]是SS产生菌, 采用S5培养基[10]进行固体培养以用于传代, 培养温度为28 ℃。S. coelicolor M1146、M1152和M1154[8]及其衍生菌株用MS培养基[11]进行固体培养以用于传代和接合转移, 所有链霉菌菌株均采用φ培养基[12]进行液体培养用于提取基因组DNA。SS种子和发酵培养基组成成分参见文献2。Escherichia coli DH5α[13]用于常规的基因克隆; E. coli ET12567/ pUZ8002[14]用于接合转移, 将DNA由大肠杆菌导入链霉菌; E. coli BW25113/pIJ790[15]用于λRed/ET介导的同源重组。大肠杆菌均用LB培养基培养, 前两种培养温度为37 ℃, E. coli BW25113/pIJ790中含有温敏型质粒pIJ790, 培养温度为30 ℃, 质粒丢失温度为37 ℃。根据需要分别加入50 μg·mL-1阿普霉素、100 μg·mL-1氨苄青霉素、50 μg·mL-1卡那霉素或25 μg·mL-1氯霉素对菌株进行抗性筛选。
引物本文所用的PCR扩增引物见表 1。
| Table 1 Primers for PCR |
限制性内切酶购自日本TaKaRa公司; 2×Q5高保真聚合酶和T4 DNA连接酶购自美国New England Biolabs公司; pGEM-T质粒购自美国Promega公司; 色谱甲醇购自上海泰坦公司; 色谱纯水为屈臣氏蒸馏水经Millipore纯化仪纯化而得; 固相萃取柱(Spe-Pack C18)和色谱柱(Waters X Bridge C18, 4.6 mm × 150 mm, 5.0 μm)购自美国Waters公司。
DNA基本操作分子生物学常规操作参照分子克隆实验指南[13], 链霉菌基因组的提取按照链霉菌操作手册进行[12]。
构建用于完整SS基因簇拼接的质粒pSET-HF黏粒10R-1[16]覆盖了SS基因簇上游的部分序列及ssaH-F, 但是不包含SS生物合成必需的ssaF下游序列, 为此本文通过设计与黏粒10R-1上下游两端序列的同源双交换来完成基因簇的拼接, 构建了以pSET152[17]为载体含有两个同源臂的重组质粒pSET-HF。构建过程如下:以pGEM-T质粒为模板, 用引物对Amp-F/Amp-R扩增氨苄青霉素抗性基因片段, 加入XbaⅠ和NdeⅠ酶切位点。以S. sp. SS的基因组DNA为模板, 用引物对UP-F/UP-R扩增orf-2-ssaH片段, 命名为前臂U, 加入NdeⅠ和KpnⅠ酶切位点, 约1 kb; 用引物对F-orf1-F/F-orf1-R扩增ssaF-orf2片段, 命名为后臂D, 加入KpnⅠ和EcoRⅠ酶切位点, 约7.2 kb。上述3个片段测序正确后连入XbaⅠ和EcoRⅠ酶切过的pSET152得到重组质粒pSET-HF, 其包含氨苄霉素抗性基因以及生物合成基因orf-2-ssaH和ssaF-orf2。
相关菌株的发酵与代谢产物的分析将固体培养基上培养7天后的相关菌株先接种于SS种子培养基进行培养, 28 ℃、200 r·min-1, 48 h后, 以5% (V/V)的接种量转接到发酵培养基, 于28 ℃、200 r·min-1条件下进行发酵。取第5天的发酵液, 经5 000 r·min-1离心10 min, 除去菌体和不溶物。取上清液1 mL经Spe-Pack C18处理, 以1 mL 60%甲醇水洗脱, 洗脱液经0.22 μm滤膜过滤, 滤液用于HPLC、HPLC-MS等检测。
HPLC在Waters 2695高效液相色谱仪(美国Waters公司)上测试。采用两相梯度洗脱的方法, 其中A相为甲醇, B相为0.05% (V/V)醋酸铵水溶液。HPLC检测时, A相在40 min内由20% (V/V)逐渐递增至80% (V/V), 流速为0.7 mL·min-1, 柱温箱设定为40 ℃, 检测波长为254 nm。采用外标法对主产物SS-A进行定量, 操作如下:用不同浓度的SS-A标准品进行进样, 以浓度为变量与峰面积做线性回归, 当线性R2 > 0.99时, 通过峰面积比值计算重组工程菌株发酵液中SS-A浓度。
HPLC-MS检测, 采用液相色谱仪Agilent 1100和离子阱质谱仪Agilent 5630 (美国安捷伦公司)。柱温箱设定为25 ℃, 其他条件同HPLC。洗脱液经过色谱柱后, 使用三通等分至二极管阵列检测器(diode array detector, DAD)和质谱检测器中。离子阱质谱检测条件:喷雾电压: 4.5伏(V); 载气:氮气(N2), 载气流速: 10 L·min-1; 离子源温度: 325 ℃; 碰撞裂解气体:高纯氦气(He); 一级质谱(1D MS)使用全扫描模式, 范围为m/z 300~2 000。数据使用仪器自带的Data Analysis, version 3.3进行处理。
高分辨质谱(HPLC-HRMS/MS)在LTQ Orbitrap XL组合式高分辨质谱仪(美国Thermo Fisher Scientific公司)上测试。流动相同HPLC, 条件调整为A相在35 min内由5% (V/V)逐渐递增至80% (V/V), 柱温箱设定为40 ℃, 数据使用软件Xcalibur 2.0.7进行处理。
结果 1 异源表达 1.1 质粒pSSA的构建与验证前期本课题组构建了S. sp. SS的大片段基因组文库, 并成功解析了SS生物合成基因簇(约33.1 kb), 但所筛选到的阳性黏粒都没有包含完整的SS生物合成基因簇[16]。本文中利用基于λ Red/ET重组产生的PCR-Targeting方法[18]将完整的基因簇拼接到整合型载体pSET152[17]上(图 2)。重组质粒pSET-HF以pSET152为载体, 包含了氨苄青霉素抗性基因筛选标记和与黏粒10R-1的上游及下游同源的两个同源臂orf-2-ssaH及ssaF-orf2, 具备了应用λ Red/ET重组系统的条件。pSET-HF经KpnⅠ酶切和脱磷酸化处理获得线性片段, 将线性化pSET-HF经电转化导入E. coli BW25113/pIJ790/10R-1中应用λ Red/ET重组系统进行同源双交换, 用阿普霉素和氨苄青霉素筛选, 获得完整的SS生物合成基因簇, 用多组酶切和PCR进行验证, 正确的质粒命名为pSSA (图 2)。

|
Figure 2 Construction and verification of pSSA. A: Diagram of sansanmycin biosynthesis cluster and cosmid 10R-1; B: Schematic diagram of the construction of pSSA. Amr stands for apramycin resistance gene; Ampr stands for ampicillin resistance gene; int(φC31) stands for φC31 integrase gene; oriT stands for origin of transfer from RT2; ori stands for replication origin of colE1. C: Restriction analysis of pSSA. Digestion of 6 colonies of pSSA with BamHⅠ, EcoRⅠ, and BglⅡ. M: λ-HindⅢ digest DNA Marker. D: PCR analysis of pSSA with 10R-1 and pSET-HF as controls. Primers orf-1-F/R, ssaB-F/R, ssaG-F/R were used to amplify a 978 bp, 1 180 bp and 831 bp DNA fragment of orf-1, ssaBand ssaG using 10R-1, pSET152-HF and pSSAas template. M: 1 kb plus DNA marker. Lane 1: 10R-1; Lane 2: pSET152-HF; Lane 3 and 4: pSSA; Lane 5: Negative control |
将重组质粒pSSA和pSET152通过接合转移分别导入S. coelicolor M1146、M1152和M1154, 获得异源表达菌株M1146/pSSA、M1152/ pSSA和M1154/pSSA以及对照菌株M1146/pSET152、M1152/pSET152和M1154/pSET152。
2 代谢产物分析 2.1 HPLC结果分析对野生型菌株S. sp. SS、异源表达菌株和对照菌株的发酵产物分别进行HPLC检测(图 3A), 以SS-A标准品(20 μg·mL-1)为对照, 野生型菌株和异源表达菌株在SS-A相同的保留时间(tR = 19.5 min)均出现一个明显的色谱峰, 且它们具有相同的在线紫外图谱(图 3B), 而对照菌株在该位置没有特征峰出现, 表明SS生物合成基因簇可能成功在天蓝色链霉菌中进行了表达, 主产物可能为SS-A。

|
Figure 3 Elucidation of SS-A. A: Comparison of the secondary metabolites in different strains by HPLC. SS-A stands as a control for structure identification of the main product of M1146/pSSA, M1152/pSSA and M1154/pSSA. Sansanmycin producing strain S. sp. SS also stands as a control to compare sansanmycin production with other heterologous expression strains. B: Characteristic ultraviolet absorption chromatography of SS-A and the main product of M1154/pSSA. C: Analyzing the compound SS-A in the fermentation broth of M1146/pSET152 (b), M1146/pSSA (c), M1152/pSSA (d) and M1154/pSSA (e) strains using extracted ion chromatography (EIC) of m/z 864.2 with SS-A (a) standing as a control |
为了进一步确定三株异源表达菌株产生的新峰是SSs类似物, 将其发酵液经Spe-Pack C18处理后分别进行HPLC-MS检测。结果如图 3C所示, 分别提取SS-A分子离子峰(m/z 864.2 [M+H]+), 对照菌株无响应(图 3C-b), 而三株异源表达菌株在保留时间为tR = 24.0 min出现响应(图 3C-c~e), 且与SS-A标准品(图 3C-a)一致, 进一步说明三株异源表达菌株的代谢产物中可能存在SS-A。
2.3 HPLC-HRMS/MS结果分析为进一步证明三株异源表达菌株表达的化合物是SS-A, 本实验选择产量较高的M1154/pSSA及其对照菌株M1154/pSET152的次级代谢产物分别进行HPLC-HRMS/MS分析。结果发现在异源表达发酵液样品中, 在保留时间为tR = 31.05 min (图 4A)峰中的化合物分子离子峰为m/z 864.331 5, 与SS-A的计算值(Calcd. 864.334 5 Da)之间的误差为3.47 ppm, 且其二级质谱与文献报道的SS-A二级质谱[2]一致(图 4B), 进一步确定异源表达菌株确实能产生SS-A, 证明SS生物合成基因簇在三株天蓝色链霉菌中成功实现异源表达。
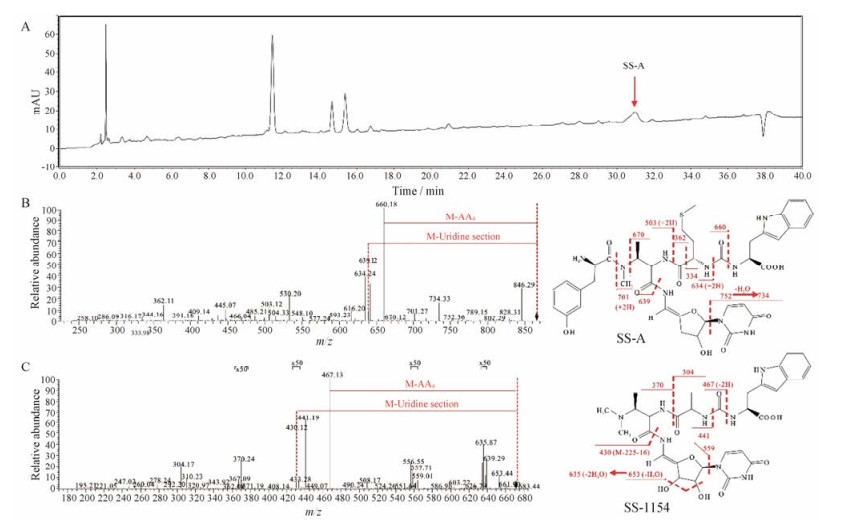
|
Figure 4 Analysis of the secondary metabolites in M1154/pSSA by HPLC-HRMS/MS. A: The HPLC-DAD spectrum; B: The MS/MS spectrum of SS-A; C: The MS/MS spectrum of SS-1154. The red lines are the auxiliary lines, while the fragment ions and their lost way are in red |
与此同时, 通过Xcalibur 2.0.7软件分析两株菌中的差异离子峰(分子量范围在600~1 000之间), 发现在异源表达菌株M1154/pSSA和对照菌株M1154/ pSET152之间有5个母离子差异峰, 除了SS-A, 分别对其他4个母离子的二级质谱进行分析, 结果显示其中一个分子离子峰可能为新结构的尿苷肽类化合物。该分子离子峰对应的色谱峰保留时间为34.42 min, 一级高分辨质谱显示其分子离子m/z 671.284 7 [M+H]+, 计算分子式为C30H39N8O10 (Calcd. 671.278 4 Da, 误差9.5 ppm)。由于产量低, 尚无法确定其紫外特征吸收情况。由其碎片离子分析(图 4C), 该化合物有可能为N-末端氨基酸AA1缺失的SSs类化合物。首先其基峰离子来源于一个204 Da的丢失, 这与其他C-末端为色氨酸的SSs类化合物二级质谱情况一致; 碎片离子m/z 430.12 (M-225-16)可能对应化合物丢失尿苷的肽基部分, 其与分子离子之间的差值比正常的SSs类化合物的尿苷部分大16 Da, 提示该化合物对应的尿苷部分比SS-A多一个羟基, 且由化合物丢失尿嘧啶的碎片离子m/z 559.01 (中性丢失为112 Da, 对应为尿嘧啶)以及两个脱水后的碎片离子m/z 653.44和m/z 635.87, 推测羟基应位于4', 5'-烯胺-尿苷的C-3';如图 4所示, SS-A的二级质谱中存在m/z 362特征离子碎片对应于AA3 (Met)至C-末端部分的结构, 而该片段在m/z 671.2847的二级碎片离子中也存在, 只是其相应由SS-A中m/z 362变化为m/z 304, 说明该化合物的AA3与SS-A不同, 可能为丙氨酸(Ala); 该化合物的DABA酰基(m/z 430~304)较文献[19]报道其他类SSs的相应结构要大14 Da, 推测其结构中可能还存在一个N-甲基。综合以上分析及其他相关的碎片离子, 推测该化合物的可能结构为N-甲基-3'-羟基-去间酪氨酰基pacidamycin 4, 命名为SS-1154 (图 4C)。
2.4 SS-A产量分析从图 3中可看出各菌株的化合物产量不同, 因此对野生型菌株及各异源表达菌株的主产物SS-A进行HPLC定量分析, 以SS-A标准品进行外标法定量, 绘制标准曲线, 线性R2 = 0.999 6, 表明该方法可行。将培养状态一致的菌株以相同体积的接种量进行发酵, 在生长曲线(图 5A)一致的情况下, 发酵产物经Spe-Pack C18处理后进行HPLC检测。M1146/pSSA中主产物的出峰位置有杂峰, 无法利用峰面积定量, 但从提取离子m/z 864.3的EIC图(图 3C)来看, SS-A在M1146/pSSA的产量明显低于其他菌株。野生型菌株S. sp. SS出现菌株退化现象, 经自然分离后, 未能获得产量恢复的菌株。由图 5B结果表明, 异源表达菌株M1152/pSSA的SS-A产量较野生型略低, 而M1154/pSSA的SS-A产量与未退化的S. sp. SS相当[19], 发酵水平达到了32 mg·L-1。多次传代后发酵结果一致, 说明SS生物合成基因簇的异源表达可以避开野生型菌株退化的问题, S. coelicolor M1154是一个良好的异源表达宿主。
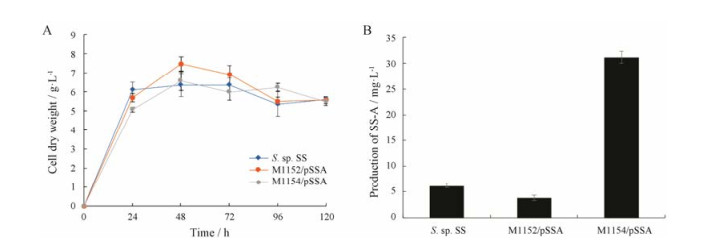
|
Figure 5 Comparison of the production level of SS-A in different strains. A: Time courses of cell growth; B: Sansanmycin A production level of wild type strain and heterologous expression strains. Bars indicate average values of 3 biological replicates and error bars represent standard deviation |
为了实现次级代谢产物的异源表达, 首先需要获得完整的生物合成基因簇。Liao等[20]使用λRed/ET重组技术获得了完整的nikkomycin生物合成基因簇并将其组装到整合型质粒pSET152上。本研究采用相同的技术手段, 以黏粒10R-1为基础, 利用PCR-Targeting的方法实现了片段拼接, 获得了完整的SS生物合成基因簇, 并将其导入到Gomez-Escribano等[8]构建的S. coelicolor M1146、M1152和M1154异源表达宿主中, 对异源表达菌株产物以及对照菌株产物进行检测, 以SS-A为标准品, 通过比较紫外特征吸收图谱、二级质谱图谱等, 表明异源表达菌株中有SS-A产生, 而对照菌株中没有SS-A产生, SS生物合成基因簇可以在天蓝色链霉菌中异源表达。Gomez-Escribano等[8]研究发现, 生物合成基因簇在S. coelicolor M1154中异源表达通常有更高的产量。本研究以HPLC方法对各异源表达菌株的SS-A产量进行分析, 结果表明与野生型菌株相比, SS-A在M1146/pSSA菌株中产量很低, 在M1152/pSSA菌株中产量次之, 在M1154/pSSA菌株产量达到最高。说明S. coelicolor M1154可能是SS生物合成基因簇异源表达的良好宿主。
有文献报道其他尿苷肽类抗生素, 如pacidamycin[21]、mureidomycin/napsamycin[22]的生物合成基因簇分别在S. lividans TK24和S. coelicolor M1154中实现了异源表达, 结果表明都可以产生1或2个已知结构化合物(图 1), 并且发现了新结构的类似物。此外, S. coeruleorubidus产生的主产物pacidamycin 4等的AA1均为m-Tyr, 而pacidamycin异源表达后仅产生AA1为Ala的主产物pacidamycin D和新衍生物pacidamycin S, 推测可能与异源表达的pacidamycin基因簇内缺少两个编码基因有关(在产生菌S. coeruleorubidus中存在于基因组其他位置[23]), 这两个基因编码苯丙氨酸羟化酶和相对应的NRPS的A domain。SS异源表达菌株与野生型菌株的主产物均为SS-A, 说明本研究克隆的SS生物合成基因簇是完整的。本研究利用HPLC-HRMS/MS方法对异源表达菌株M1154/pSSA的产物进行进一步分析, 发现了一个可能为新结构的SS类似物SS-1154。根据文献报道的尿苷肽类化合物生物合成途径[24]推测该结构中AA3替换为丙氨酸可能是由于负责肽链合成的非核糖体肽合成酶具有较大程度的底物宽容性[25], 加上异源宿主的环境不同于原产生菌从而出现这一改变; DABA中新增的N-甲基可能是由于异源宿主中存在其他能够使氨基甲基化的酶, 或者参与DABA生物合成的SsaV可能具有底物宽容性[23]; 而尿苷部分可能在3'位置新增-OH取代, 推测在合成尿苷结构的基础上被氧化, 可能与异源宿主中的氧化还原酶有关。目前尚未发现其他尿苷部分为该结构的SSs类似物, 因此该化合物具有一定的研究价值, 但由于其产量很低, 尚难获得足够量进行结构的进一步解析和生物活性的评价。
目前, 高通量测序技术发展迅猛, 微生物基因组序列越来越容易获得, 通过基因组挖掘技术有可能获得新的尿苷肽类化合物生物合成基因簇和新的生物合成元件。天蓝色链霉菌遗传背景清楚, 为进一步应用合成生物学方法获得新结构尿苷肽类化合物提供了便利, 有利于在异源宿主中进行生物合成元件的替换与组合, 有目的地合成新化合物, 为进一步提高尿苷肽类化合物的结构多样性奠定了基础。
| [1] | Winn M, Goss RJ, Kimurab K, et al. Antimicrobial nucleoside antibiotics targeting cell wall assembly: recent advances in structure-function studies and nucleoside biosynthesis[J]. Nat Prod Rep, 2010, 27: 279–304. DOI:10.1039/B816215H |
| [2] | Xie Y, Chen R, Si S, et al. A new nucleosidyl-peptide antibiotic, sansanmycin[J]. J Antibiot, 2007, 60: 158–161. DOI:10.1038/ja.2007.16 |
| [3] | Jiang ZB, Shi YY, Ren WC, et al. Identification of two novel sansanmycin analogues from ssaX knockout strain by HPLC-MS/MS[J]. Chin Med Biotechnol (中国医药生物技术), 2018, 13: 5–12. |
| [4] | Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440: 940–943. DOI:10.1038/nature04640 |
| [5] | Ajikumar PK, Xiao WH, Tyo KE, et al. Isoprenoid pathway optimization for taxol precursor overproduction in Escherichia coli[J]. Science, 2010, 330: 70–74. DOI:10.1126/science.1191652 |
| [6] | Wu LZ, Hong B. Synthetic biology toward microbial secondary metabolites and pharmaceuticals[J]. Acta Pharm Sin (药学学报), 2013, 48: 155–160. |
| [7] | Lucas X, Senger C, Erxleben A, et al. StreptomeDB: a resource for natural compounds isolated from Streptomyces species[J]. Nucleic Acids Res, 2013, 41: 1130–1136. DOI:10.1093/nar/gks1253 |
| [8] | Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters[J]. Microb Biotechnol, 2011, 4: 207–215. DOI:10.1111/j.1751-7915.2010.00219.x |
| [9] | Wang L, Du DY, Li JE, et al. Engineering and heterologous expression of a nikkomycin biosynthetic gene cluster[J]. Acta Microbiol Sin (微生物学报), 2015, 55: 707–718. |
| [10] | Wang L, Hu Y, Zhang Y, et al. Role of sgcR3 in positive regulation of enediyne antibiotic C-1027 production of Streptomyces globisporus C-1027[J]. BMC Microbiol, 2009, 9: 14. DOI:10.1186/1471-2180-9-14 |
| [11] | Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces Genetics[M]. Norwich, UK: John Innes Foundation, 2000. |
| [12] | Korn F, Weingartner B, Kutzner HJ. A study of twenty actinophages: morphology, serological relationship and host range[M]//Freerksen E, Tarnok I, Thumin H. Genetics of the Actinomycetales. New York: Gustav Fisher Verlag, 1978: 251-270. |
| [13] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual[M]. 3rd Edn. Cold Spring Harbor: Cold Spring Harbor Laboratory, 2001. |
| [14] | Paget MS, Chamberlin L, Atrih A, et al. Evidence that the extracytoplasmic function sigma factor σe is required for normal cell wall structure in Streptomyces coelicolor A3(2)[J]. J Bacteriol, 1999, 181: 204–211. |
| [15] | Gust B, Challis GL, Fowler K, et al. PCR-targeted Streptomycesgene replacement identifies a protein domain needed for biosynthesis of the sesquiterpene soil odor geosmin[J]. Proc Natl Acad Sci U S A, 2003, 100: 1541–1546. DOI:10.1073/pnas.0337542100 |
| [16] | Li Q, Wang L, Xie Y, et al. SsaA, a member of a novel class of transcriptional regulators, controls sansanmycin production in Streptomyces sp. strain SS through a feedback mechanism[J]. J Bacteriol, 2013, 195: 2232–2243. DOI:10.1128/JB.00054-13 |
| [17] | Bierman M, Logan R, O'Brien K, et al. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp[J]. Gene, 1992, 116: 43–49. DOI:10.1016/0378-1119(92)90627-2 |
| [18] | Zhang Y, Muyrers JP, Testa G, et al. DNA cloning by homologous recombination in Escherichia coli[J]. Nat Biotechnol, 2000, 18: 1314–1317. DOI:10.1038/82449 |
| [19] | Shi YY, Jiang ZB, Lei X, et al. Improving the N-terminal diversity of sansanmycin through mutasynthesis[J]. Microb Cell Fact, 2016, 15: 77. DOI:10.1186/s12934-016-0471-1 |
| [20] | Liao GJ, Li JE, Li L, et al. Cloning, reassembling and integration of the entire nikkomycin biosynthetic gene cluster into Streptomyces ansochromogenes lead to an improved nikkomycin production[J]. Microb Cell Fact, 2010, 9: 6. DOI:10.1186/1475-2859-9-6 |
| [21] | Emma JR, Sabine G, Amany ER, et al. Pacidamycin biosynthesis: identification and heterologous expression of the first uridyl peptide antibiotic gene cluster[J]. ChemBioChem, 2010, 11: 1700–1709. DOI:10.1002/cbic.v11:12 |
| [22] | Tang XY, Marcel G, Xie YY, et al. Identification of mureidomycin analogues and functional analysis of an N-acetyltransferase in napsamycin biosynthesis[J]. ChemBioChem, 2013, 14: 2248–2255. DOI:10.1002/cbic.v14.17 |
| [23] | Li QL, Xie YY, Wang LF, et al. Biosynthesis of uridyl peptide antibiotics: research advances[J]. J Int Pharm Res (国际药学研究杂志), 2012, 39: 17–25. |
| [24] | Gruschow S, Rackham EJ, Elkins B, et al. New pacidamycin antibiotics through precursor-directed biosynthesis[J]. Chembiochem, 2009, 10: 355–360. DOI:10.1002/cbic.v10:2 |
| [25] | Zhang W, Ntai I, Bolla ML, et al. Nine enzymes are required for assembly of the pacidamycin group of peptidyl nucleoside antibiotics[J]. J Am Chem Soc, 2011, 133: 5240–5243. DOI:10.1021/ja2011109 |
 2018, Vol. 53
2018, Vol. 53


