结核病(tuberculosis, TB)是由结核分枝杆菌(Mycobacterium tuberculosis, MTB)引起的一种慢性致死性疾病, 目前是全世界十大致死疾病之一[1]。近些年, 由于抗结核药物的大量使用、抗结核作用位点的突变、艾滋病和结核耐药菌株的出现等原因, 抗结核治疗面临严峻挑战。据世界卫生组织估算, 2016年全球有1 040万人患有结核病, 170万人因该病死亡(包括40万艾滋病毒感染者), 有60万利福平耐药新发病例, 其中有49万耐多药结核病患者[1]。耐多药结核病(MDR-TB)和广泛耐药结核病(XDR-TB)是控制和治疗结核病的重大挑战, 是结核病相关研究者的关注重点[2-4]。然而, 近几十年来仅有强生公司的新型结核分枝杆菌ATP合成酶抑制剂贝达喹啉(bedaquiline)和日本大冢公司的德拉马尼(delamanid)两种抗结核药物被批准上市, 而且目前已有耐药菌株出现及引起不良反应的报道[5]。因此, 寻找新的抗结核药物作用靶点, 开发新型抗结核药物迫在眉睫。
莽草酸途径广泛存在于自然界中, 是细菌、真菌、藻类和高等植物合成芳香族氨基酸、叶酸、泛醌、分枝菌酸等必需物质的一条重要代谢途径。共有7种酶参与了该途径催化反应, 分别是: 3-脱氧-D-阿拉伯-庚酮糖酸-7-磷酸合酶、3-脱氢奎尼酸合酶、脱氢奎尼酸脱氢酶、莽草酸脱氢酶、莽草酸激酶、5-烯醇酮莽草酸-3-磷酸合酶和分支酸合酶[6]。实验证明, 在细菌不补充外源芳香化合物的条件下, 该通路被阻断的突变株生长受到抑制, 证明该通路在维持细菌生命活动中起到至关重要的作用[7, 8]。由于人体内不存在莽草酸途径, 所以该通路中的相关蛋白是有潜在吸引力的抗菌靶标[7]。
结核分枝杆菌莽草酸激酶(MtSK)蛋白分子质量为18.58 kD, 由aroK基因(Rv2539c)编码, 该蛋白催化莽草酸途径的第五步反应, 使磷酰基团从ATP转移至莽草酸, 生成3-磷酸莽草酸并释放出1个ADP分子(图 1)。实验发现, aroK基因缺失导致MTB无法生长, 当回补外源性的aroK基因时, 突变株得以存活, 证明MtSK是维持MTB生长的必需蛋白[9]。本研究以MtSK为靶点, 克隆和表达MtSK蛋白并优化其测活方法, 建立了MtSK抑制剂筛选模型, 并对国家新药(微生物)筛选实验室化合物库进行筛选, 寻找结构新颖的抗结核先导化合物, 为抗结核药物的研发奠定基础。
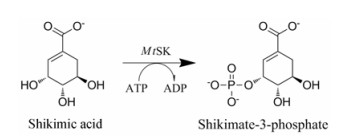
|
Figure 1 Mycobacterium tuberculosis shikimate kinase (MtSK) catalyzes the formation of shikimate-3-phosphate from shikimic acid using ATP as co-substrate |
大肠埃希菌E. coli DH5α和E. coli BL21 (DE3)为本室保存; 耻垢分枝杆菌Mycobacterium smegmatis mc2155 (ATCC 700084)由本室保存; 结核分枝杆菌H37Rv (ATCC 27294)和临床耐药菌株XDR-FJ05195由中国疾控中心结核病室提供。pET30a质粒由本实验室保存; T1 simple质粒购买自北京全式金生物技术有限公司。人癌细胞HeLa和人胚胎肾细胞HEK 293为本室保存。
试剂与试剂盒氨苄西林、Tris-HCl、四甲基二乙酸(TEMED)和异丙基硫代半乳糖苷(isopropyl thiogalactoside, IPTG)购自Amresco公司; 莽草酸、咪唑(imidazole)购自美国Sigma公司; 蛋白Marker购自BIO-RAD公司; 30%丙烯酰胺-双丙烯酰胺溶液、4×分离胶缓冲液、4×浓缩胶缓冲液、R-250考马斯亮兰染色液购自北京普利莱公司; 7H9液体培养基和ADC增菌液购自BD公司。DNA Marker、胶回收试剂盒EasyPure® Quick Gel Extraction Kit、质粒小提试剂盒EasyPure® Plasmid MiniPrep Kit、突变试剂盒Fast Mutagenesis System、蛋白质浓度测定试剂盒BCATM Protein Assay Kit购自Thermo公司; ATP酶检测试剂盒Kinase-Glo®Plus Luminescent Kinase Assay购自Promega公司。
化合物库筛选所用化合物库来自国家新药(微生物)筛选实验室, 纯度 > 95%, 可商业购买。
仪器ÄKTA蛋白层析系统、BIACORE T200、His trapTM HP亲和层析柱为美国GE Health产品; 酶标仪为美国Perkin Elmer公司的EnVision2014型。
MtSK蛋白的克隆、表达与纯化以结核分枝杆菌H37Rv基因组DNA为模板, PCR扩增基因MtSK。引物使用Primer 5.0软件设计, 上游引物: 5'-GTTAC CATATGGCACCCAAAGCGGTTC-3' (Nde Ⅰ), 下游引物: 5'-CGATACTCGAGTGTGGCCGCCTCGCT-3' (Xho Ⅰ)。扩增体系50 μL, 扩增的条件为: 95 ℃预处理5 min, 95 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸2 min, 共30个循环, 72 ℃充分延伸10 min。经Nde Ⅰ和Xho Ⅰ双酶切后, 连接到pET30a载体, 再转化DH5α感受态细胞。经PCR和酶切法验证后, 进行测序。选取序列完全正确的质粒转化大肠埃希菌BL21 (DE3)感受态细胞。挑取单克隆, 37 ℃、180 r·min-1过夜培养。第二天以1:100比例转接于含100 μg·mL-1卡那霉素的LB液体培养基, 培养至OD600约为0.8时, 加入终浓度为0.5 mmol·L-1 IPTG, 20 ℃、150 r·min-1诱导表达10 h。
离心收集菌体, 用裂解上样缓冲液(25 mmol·L-1 Tris-HCl, 500 mmol·L-1 NaCl, 20 mmol·L-1咪唑, pH 7.8)重悬, 采用压力破碎法裂解菌体, 20 Kpsi压力下破碎3个循环, 4 ℃、10 000 ×g将菌体离心30 min, 上清用0.45 μm滤膜过滤。由于表达的重组MtSK蛋白带有His-tag标签, 所以将所得滤液用Ni2+螯合的HisTrapTM HP亲和层析柱进行亲和层析。用洗脱缓冲液(25 mmol·L-1 Tris-HCl, 500 mmol·L-1 NaCl, 500 mmol·L-1咪唑, pH 7.8)洗脱, 收集洗脱峰, 对纯化所得蛋白进行蛋白定量。
MtSK蛋白活性的测定根据MtSK催化莽草酸途径第5步反应的反应机制, 本研究中使用了Promega公司开发的Kinase-Glo®Plus Luminescent Kinase Assay试剂盒, 利用荧光法检测反应体系中ATP的消耗量, 对体外表达蛋白的活性进行了测定。该试剂盒通过检测反应体系中剩余的ATP含量来测定MtSK蛋白活性。底物中的昆虫萤光素酶能够消耗ATP催化相应的底物产生发光物质。如果激酶蛋白活性越强, 则酶促反应结束后剩余的ATP量越少, 萤光素酶催化生成的发光物质也就越少。当ATP浓度在100 μmol·L-1内时, 发光强度与体系中ATP浓度存在线性关系, 与激酶活性呈负相关。
根据说明书及查阅MtSK活性测定相关文献[10, 11], 本研究设定酶活性检测体系如下: 100 mmol·L-1 Tris-HCl pH 7.5, 50 mmol·L-1 KCl, 5 mmol·L-1 MgCl2, 50 μmol·L-1 ATP, 50 μg·mL-1莽草酸, 1 μg·mL-1MtSK蛋白。荧光测活体系反应体积为50 μL, 反应在96孔板中进行。37 ℃条件下反应15 min后, 加入等体积的Kinase-Glo®Plus检测试剂, 室温孵育10 min, 使发光信号稳定, 将96孔板放入酶标仪中检测。同时设立不加MtSK组作为空白对照。
耻垢分枝杆菌mc2155对化合物库进行初筛由于MTB生长速度过慢, 无法满足大量的体外筛选要求, 因此本研究选择与MTB高度同源的耻垢分枝杆菌Mycobacterium smegmatis mc2155作为体外筛选的模式菌, 对化合物库中12万个样品进行初步筛选。筛选方法如下:在96孔板中进行, 耻垢分枝杆菌用7H9培养基(含10% ADC)培养, 接菌量为5×105 CFU·mL-1, 每孔200 μL。化合物的筛选终质量浓度为50 μg·mL-1, 37 ℃培养48 h观察结果。将抑制耻垢分枝杆菌生长的化合物建立初筛阳性库。
MtSK蛋白抑制剂高通量筛选模型的建立与筛选运用上述酶活测定方法, 对建立的初筛阳性化合物库中样品以终质量浓度10 μg·mL-1进行筛选。同时设立空白对照:以不加MtSK为阳性对照(即酶活性完全被抑制), 以加入与化合物等体积的DMSO组为阴性对照(即未加抑制剂)。抑制率计算公式:酶抑制率IP (%) = [1-(ΔSP-ΔSS)/(ΔSP-ΔSN)]×100%。其中, ΔSP是不加MtSK对照孔的发光值, ΔSN是加入等体积DMSO对照孔的发光值, △SS是样品实验孔的发光值。
根据上述条件, 对建立的初筛阳性化合物库中样品进行筛选, 并按照相同的反应条件和方法进行复筛。经排除假阳性后, 得到抑制效果最好的活性化合物IMB-T5297。
化合物IMB-T5297在耻垢分枝杆菌的MIC测定在96微孔板中进行, 耻垢分枝杆菌用7H9培养基(含10% ADC)培养, 接菌量为5×105 CFU·mL-1, 每孔200 μL。化合物的终质量浓度为0、0.78、1.56、3.12、6.25、12.5、25和50 μg·mL-1。37 ℃培养24 h观察结果, 以在小孔内完全抑制细菌生长的最低药物浓度为最低抑菌浓度(minimum inhibitory concentration, MIC)。
化合物在MtSK蛋白抑制剂模型上的IC50测定将筛选得到的活性化合物IMB-T5297溶于DMSO中, 配置成10 mg·mL-1母液, 将化合物倍比稀释, 分别获得不同浓度条件下的抑制率, 以抑制剂浓度的对数为横坐标, 抑制率为纵坐标, 利用Graphpad prism 5软件计算半数抑菌浓度(half maximal inhibitory concentration, IC50)。
HPLC方法验证化合物IMB-T5297对MtSK蛋白的抑制作用MtSK催化莽草酸途径第5步反应时会生成3-磷酸莽草酸并释放出1个ADP分子。根据此反应机制, 参考Tan等[12]建立的HPLC法测定术后疲劳综合征大鼠骨骼肌ATP、ADP和AMP含量的方法, 利用HPLC法测定反应中生成的ADP含量, 验证化合物IMB-T5297对MtSK蛋白的抑制作用。
HPLC法采用色谱柱Agilent ZORBAX SB-Aq (4.6 mm × 150 mm, 5 μm), 柱温25 ℃; 流动相为100 mmol·L-1磷酸盐缓冲液(含12 mmol·L-1磷酸氢二钠和88 mmol·L-1磷酸二氢钠, pH 6.5)-甲醇(体积比99.9:0.1);流速1 mL·min-1; 紫外检测波长为254 nm; 进样量为10 μL。
IMB-T5297对MtSK蛋白的抑制作用的酶动力学性质研究筛选条件下, 分别将底物莽草酸的浓度、化合物IMB-T5297的浓度进行倍比稀释, 分别获得不同莽草酸浓度条件下不同化合物浓度的抑制率。采用Lineweaver-Burk作图法以莽草酸浓度的倒数为横坐标, 反应速度的倒数为纵坐标作图, 采用多个抑制剂浓度, 得出不同抑制剂浓度的直线, 由这些直线的交点位置推测抑制剂的类型。
SPR实验检测IMB-T5297与MtSK蛋白的结合情况表面等离子共振(surface plasmon resonance, SPR)实验是利用表面等离子体共振现象和SPR谱峰对金属表面上电介质变化敏感的特点, 通过将受体蛋白固定在金属膜上, 检测受体蛋白与液相中配体小分子的特异性结合。由于MtSK蛋白在酸性条件下可以保持稳定, 因此可以使用CM5芯片进行SPR实验。实验缓冲溶液系统为PBS-P+缓冲液, 由Biacore T200 System (GE Healthcare)仪器完成。
MtSK蛋白突变采用Discovery Studio 4.0软件对化合物IMB-T5297与MtSK (PDB ID: 2IYQ)进行分子对接, 根据打分最高的对接模型分析化合物作用机制。本研究通过对接模型推测了一些影响化合物和蛋白结合的关键氨基酸位点, 并选取其中5个关键氨基酸进行突变: Asp34→Ala34、Arg58→Ala58、Gly81→Ala81、Arg117→Ala117、Arg136→Ala136。按照突变试剂盒说明书对PET30a-MtSK质粒进行定点突变, 突变后的质粒分别命名为PET30a-MtSK-Asp34、PET30a-MtSK-Arg58、PET30a-MtSK-Gly81、PET30a-MtSK-Arg117、PET30a-MtSK-Arg136。把突变后的质粒转入E.coli BL21 (DE3)菌株, 将阳性菌株送到生物公司进行测序, 使用测序正确的菌株分别进行蛋白纯化, 并对突变蛋白活性进行测定。
MTT比色法检测化合物IMB-T5297的细胞毒性根据活细胞线粒体中的琥珀酸脱氢酶能使外源性3-(4, 5-二甲基噻唑-2)-2, 5-二苯基四氮唑溴盐(3-(4, 5-dimethyl-2-thiazolyl)-2, 5-diphenyl-2-H-tetrazolium bromide, MTT)还原为水不溶性的蓝紫色结晶甲瓒并沉积在细胞中, 而死细胞无此功能。采用MTT法来检测阳性化合物IMB-T5297对人癌细胞HeLa和人胚胎肾细胞HEK293存活的影响, 从而初步判断IMB-T5297对人体细胞是否有毒性。MTT检测步骤见文献[13]。
化合物IMB-T5297抗结核活性测定应用Lu等[14]建立的Alamar Blue法在无菌96微孔板中测定化合物IMB-T5297抗结核活性。接种菌株为结核杆菌标准株H37Rv和临床分离耐药株XDR-FJ05195, 接菌量为1×106 CFU·mL-1, 每孔100 μL。化合物IMB-T5297的终质量浓度为64、32、16、8、4、2、1、0.5、0.25 μg·mL-1。同时设置阳性对照孔(不加化合物的培养基)和阴性对照孔(以蒸馏水取代培养基)。按Alamar Blue法进行培养, 观察各孔颜色的变化, MIC定义为从蓝色变为粉红色的最低药物浓度。
结果 1 重组MtSK质粒pET30a-MtSK的构建与表达MtSK PCR产物琼脂糖凝胶电泳检测的结果与预期片段大小(531 bp)一致, 重组质粒pET30a-MtSK经Nde Ⅰ和Xho Ⅰ双酶切后得到两条DNA片段, 大小分别与MtSK基因(531 bp)和pET30a载体(5 422 bp)一致。经DNA测序结果证明, 重组质粒pET30a-MtSK构建成功。转入重组质粒pET30a-MtSK的大肠杆菌经IPTG诱导表达蛋白, 经HisTrapTM HP亲和层析柱纯化, 得到较高纯度的MtSK蛋白(图 2)。
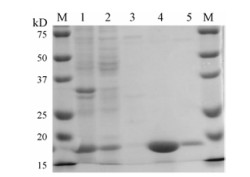
|
Figure 2 MtSK SDS-PAGE. M: Protein marker; 1: The proteins of whole bacteria; 2: The proteins of supernatant; 3: 20% elution fraction; 4: 60% elution fraction; 5: 100% elution fraction |
使用商业化的荧光检测试剂盒Kinase-Glo®Plus Luminescent Kinase Assay测定MtSK酶活性, 确认表达纯化的MtSK蛋白是否具有活性。实验测定了MtSK浓度分别为0、0.25、0.5、1、2、4和8 μg·mL-1, 37 ℃反应15 min时的发光值, 测定结果如图 3, 结果显示纯化得到的MtSK蛋白具有良好的催化活性。

|
Figure 3 Change of luminescence value (Lum) of MtSK reaction. The luminescence value was measured when the reaction was carried out for 15 min with MtSK (0, 0.25, 0.5, 1, 2, 4 or 8 μg·mL-1). RLU: Relative light units |
用耻垢分枝杆菌对化合物库中12万个样品进行筛选, 若化合物抑制耻垢分枝杆菌生长, 则认为是初筛阳性化合物。经过筛选, 共得到267个初筛阳性化合物, 建立初筛阳性化合物库。
4 MtSK蛋白抑制剂高通量筛选模型的的评价以酶活测定反应为基础建立高通量筛选模型, 经评价, 其Z '因子为0.81, 信号噪音比、信号背景比均符合高通量筛选的要求。设定抑制率大于50%为初筛阳性化合物, 得到5个阳性化合物, 阳性率为2%。通过进一步复筛, 选取抑制作用最强的化合物IMB-T5297进行深入研究。化合物IMB-T5297的结构如图 4A所示。
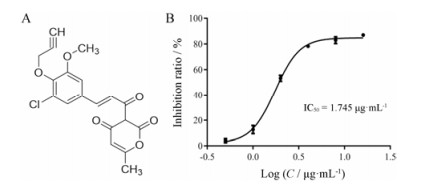
|
Figure 4 Characteristics of compound IMB-T5297. A: Chemical structure of compound IMB-T5297 [(E)-3-(3-(3-chloro-5-methoxy-4-(prop-2-yn-1-yloxy)phenyl)acryloyl)-6-methyl-2H-pyran-2, 4(3H)-dione]. B: Dose-response curve for the inhibition of MtSK by compound IMB-T5297. Different concentration of compound IMB-T5297 (16, 8, 4, 2, 1, 0.5 μg·mL-1) was added in the system, and the IC50 value was plotted as the ratio of the radioactivity signal over the concentration of compound (log plot) that fitted to a variable-slope dose-response equation. The experiment was repeated three times |
化合物IMB-T5297对MtSK活性抑制作用如图 4B所示, 随着化合物IMB-T5297浓度的增高, 对MtSK活性抑制作用越来越强, 表现出明显的剂量依赖性。通过Graphpad prism 5软件拟合曲线得知, 化合物IMB-T5297对MtSK酶活性IC50约为1.745 μg·mL-1。
6 HPLC方法验证化合物IMB-T5297对MtSK蛋白的抑制作用为了验证HPLC方法的可行性, 首先配制了5 mmol·L-1 ADP对照品储备液, 用超纯水将对照品储备液分别稀释成2 560、1 280、640、320、160、80、40、20、10和5 μmol·L-1, 在与样品相同条件下进行测定。结果表明ADP在5~100 μmol·L-1内线性关系良好, 样本的检测浓度在线性范围内(图 5A)。
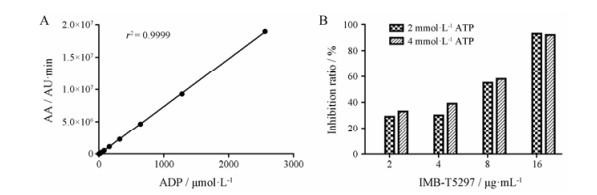
|
Figure 5 Detecting interaction between compound IMB-T5297 and MtSK by HPLC. A: The standard curve of ADP. B: Inhibition rate of compound IMB-T5297 under different ATP concentration to MtSK (1 μg·mL-1). AA is the peak area of ADP. AU: Absorbance unit |
HPLC分别检测了在2和4 mmol·L-1 ATP浓度下, 浓度分别为0、2、4、8和16 μg·mL-1化合物IMB-T5297对MtSK的抑制作用, 再次证明IMB-T5297对MtSK的抑制作用具有浓度依赖性, 同时说明ATP浓度影响较小(图 5B)。
7 化合物IMB-T5297对MtSK蛋白抑制作用的酶动力学性质研究对MtSK蛋白具有抑制作用的阳性化合物酶动力学性质进行研究, 在不同浓度抑制剂的作用下, 反应速度随底物浓度的变化, 采用Lineweaver-Burk作图法以莽草酸浓度的倒数为横坐标, 反应速度的倒数为纵坐标作图。结果显示, 不同浓度化合物抑制率的直线大致交于纵轴正半轴, 推测化合物IMB-T5297可能为莽草酸的竞争性抑制剂(图 6)。
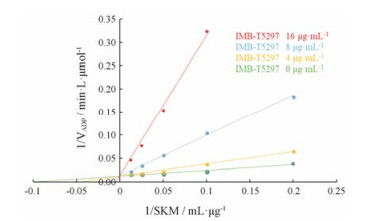
|
Figure 6 In vitro mode of inhibition graphs of compound IMB-T5297 against substrate of shikimic acid (SKM). Lineweaver-Burk plot of 1/VADP (min·L·μmol-1) against [SKM-1] (mL·μg-1) at 0, 4, 8, 16 μg·mL-1 of IMB-T5297 |
经反复实验发现, MtSK蛋白在pH 5酸性条件下可以保持稳定, 所以选用CM5芯片进行SPR实验, 缓冲溶液系统为PBS-P+缓冲液, 实验由BIACORE T200仪器完成。首先通过CM5芯片表面与MtSK蛋白His标签的螯合作用捕获目标蛋白, 控制蛋白结合信号在7 500 RU左右。化合物IMB-T5297溶解在运行缓冲液中, 配制成浓度梯度。在运行缓冲液PBS-P+缓冲液中加入2% DMSO, 增加化合物溶解性。IMB-T5297浓度从低到高分别为1.56、3.12、6.25、12.5、25、50、100和200 μmol·L-1。实验结果显示, 化合物IMB-T5297能够以剂量依赖性方式与MtSK蛋白发生结合(图 7A)。拟合曲线表明, 该实验可信度较高, 化合物IMB-T5297与MtSK蛋白结合的KD值为2.151×10-5 mol·L-1, 亲和力较高(图 7B)。
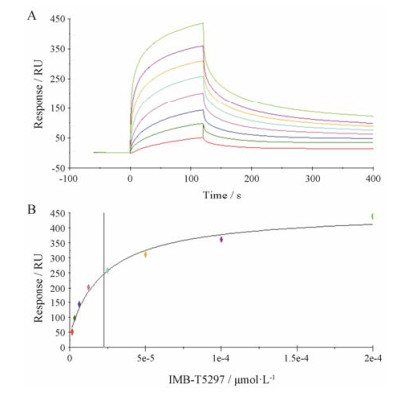
|
Figure 7 Detecting interaction between compound IMB-T5297 and MtSK by surface plasmon resonance (SPR). A: Compound IMB-T5297 binding towards MtSK determined by Biacore surface plasmon resonance. A sensor chip coated with purified MtSK was exposed to various concentrations of IMB-T5297. Sensorgrams generated by gradient concentrations of compound IMB-T5297 using the SPR aptasensor. The concentration from bottom to top was 1.56, 3.12, 6.25, 12.5, 25, 50, 100, 200 μmol·L-1. B: Fitting curve of compound IMB-T5297 and MtSK. SPR responses generated from gradient concentrations of compound IMB-T5297 using the SPR aptasensor. The concentration of compound IMB-T5297 from left to right was 1.56, 3.12, 6.25, 12.5, 25, 50, 100, 200 μmol·L-1. RU: Response unit |
化合物IMB-T5297与MtSK蛋白的对接结果如图 8所示, 化合物与MtSK蛋白58、81、117、136位氨基酸之间存在结合力较强的氢键, 与34位氨基酸之间形成Pi-anion相互作用, 推测这5个位点的氨基酸可能是化合物IMB-T5297与MtSK蛋白结合的关键氨基酸。
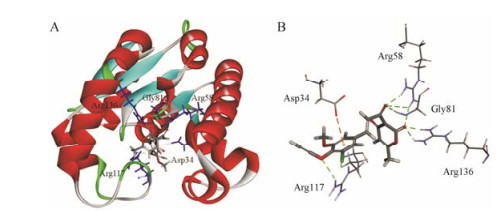
|
Figure 8 Molecular docking of MtSK (PDB ID: 2IYQ) and compound IMB-T5297 by Discovery Studio 4.0. A: Overview of the active pocket of MtSK bound to IMB-T5297. IMB-T5297 is represented by a stick model. The blue sticks represent the amino acid residues interacting with IMB-T5297; B: The hydrogen bond formed between amino acid residues (Arg58, Gly81, Arg117, Arg136) and compound IMB-T5297 is marked green color line. The Pi-anion interaction formed between amino acid residue (Asp34) is marked orange color line |
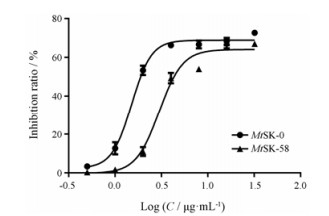
本研究使用点突变试剂盒把以上5个位点的氨基酸分别突变为丙氨酸, 突变完成的质粒分别标记为PET30a-MtSK-Asp34、PET30a-MtSK-Arg58、PET30a-MtSK-Gly81、PET30a-MtSK-Arg117、PET30a-MtSK-Arg136。将突变完成的质粒转入表达菌株E. coli BL21 (DE3)中进行表达, 经HisTrapTM HP亲和层析柱纯化蛋白, 得到较高纯度的MtSK突变蛋白(图 9A)。用上述MtSK酶活测定方法对纯化得到的5个突变蛋白进行活性测定, 结果表明仅有Arg58突变对酶活性影响较小(图 9B)。分别测定化合物IMB-T5297对MtSK原蛋白及MtSK-Arg58突变蛋白的抑制率(图 10), 结果显示化合物IMB-T5297对MtSK-Arg58突变蛋白的IC50 (2.992 μg·mL-1)高于对MtSK原蛋白IC50 (1.507 μg·mL-1), 说明相同浓度的化合物对MtSK原蛋白的抑制作用更强。因此推测, Arg58很有可能是MtSK与化合物IMB-T5297结合的关键位点。
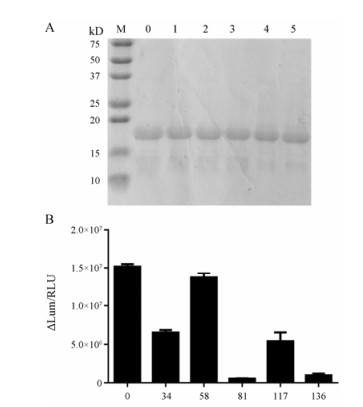
|
Figure 9 Characteristics of MtSK protein and mutant proteins. A: SDS-PAGE of MtSK protein and mutant proteins. M: Protein marker; 0: MtSK; 1: MtSK-Asp34; 2: MtSK-Arg58; 3: MtSK-Gly81; 4: MtSK-Arg117; 5: MtSK-Arg136. B: Comparison of activity of MtSK protein and mutant proteins. 0, 34, 58, 81, 117, 136 on the axis of abscissa represented MtSK protein, MtSK-Asp34 protein, MtSK-Arg58 protein, MtSK-Gly81 protein, MtSK-Arg117 protein, MtSK-Arg136 protein, respectively |
|
Figure 10 Dose-response curve for the inhibition of MtSK-0 and MtSK-58 by compound IMB-T5297. Different concentration of compound IMB-T5297 (32, 16, 8, 4, 2, 1, 0.5 μg·mL-1) was added in the system, and the IC50 value was plotted as the ratio of the radioactivity signal over the concentration of compound (log plot) that fitted to a variable-slope dose–response equation. The IC50 of compound IMB-T5297 to MtSK-0 and MtSK-58 are 1.507 and 2.992 μg·mL-1, respectively |
采用MTT法检测化合物IMB-T5297对人宫颈癌细胞HeLa和人胚胎肾细胞HEK 293的毒性影响。结果显示, 化合物IMB-T5297在质量浓度为100 μg·mL-1时, 对HeLa细胞和HEK293细胞的生长均无明显影响, 说明化合物IMB-T5297对细胞毒性较小或无毒。
11 化合物IMB-T5297抗菌活性化合物IMB-T5297对耻垢分枝杆菌(Mycobacterium smegmatis mc2155)、结核分枝杆菌标准株H37Rv (Mycobacterium tuberculosis H37Rv)和临床分离耐药株XDR-FJ05195的MIC分别为20、49.723和47.754 μg·mL-1 (表 1)。
| Table 1 Anti-tuberculosis activity of compound IMB-T5297 |
根据结核分枝杆菌MtSK晶体结构数据(PDB: 2IYQ)与相关文献[15]报道, MtSK蛋白拥有一个盖子结构域(Gly112-Asp124)和一个NMP结合域(Thr33-Glu61)。后者是该蛋白的主要活性区域, 也是莽草酸分子的结合口袋。莽草酸分子的羧基与Arg58和Arg136作用, 3号位的羟基与Asp34、Gly80残基作用, 同时2号位的羟基与Asp34也有相互作用[16]。本文通过对MtSK原蛋白及5个突变蛋白进行酶活性分析, 发现Asp34、Arg117、Gly81、Arg136对维持MtSK的蛋白活性具有重要作用, 但由于突变蛋白活性较低, 无法测定这些氨基酸是否对化合物与蛋白的结合起到关键作用。而Arg58突变蛋白仍保留大部分活性, 所以本文比较了化合物IMB-T5297对MtSK原蛋白及MtSK-Arg58突变蛋白的抑制率, 结果显示化合物对MtSK-Arg58突变蛋白的抑制率明显降低, 说明化合物很可能与Arg58形成氢键并与莽草酸竞争性结合蛋白的活性位点, 进而抑制了蛋白的催化活性。
过去大多数对MtSK抑制剂的研究停留在通过计算机辅助药物设计, 合成天然底物类似物阶段[17-19]。近年来, 由于MtSK具有良好的成靶潜力, 人们对其抑制剂进行了更深入研究。Mulabagal和Calderón[10]首次使用超滤液相色谱/质谱(UF-LC/MS)和液质联用(LC/MS)方法筛选MtSK抑制剂, 评估了4个化合物对MtSK蛋白的抑制作用, 其中3个化合物是吡唑啉酮类似物, 一个是星形孢菌素。Masoko等[20]首次从传统草药Sutherlandia frutescens中分离得到MtSK抑制剂——α-亚麻酸, 该化合物是一种含有3个双键的多元不饱和脂肪酸。Mehra等[21]首次报道了MtSK变构位点的抑制剂——苯并噻唑类化合物5489375, 该化合物可能主要与莽草酸结构域的残基结合, 与残基Arg43、Ile45和Phe57形成重要的相互作用, 从而阻止了Arg117的胍基基团与3-磷酸莽草酸的磷酸基团之间形成强的盐桥相互作用, 影响了产物的释放。Simithy等[22]使用LC/MS方法检测了来自美国国家过敏症和传染病研究所(NIAID)的404个抗结核化合物对MtSK蛋白的抑制作用, 得到14个对MtSK有抑制作用的化合物, 这些化合物的骨架结构均为噁二唑-酰胺或2-氨基苯并噻唑。但在最新研究中, Alturki等[23]利用液质联用、氢谱、动态光散射和透射电子显微镜等实验技术对Simithy等[22]得到的14个化合物进行研究, 发现这14个化合物是通过形成聚集体而抑制MtSK活性, 是MtSK的非特异性抑制剂。以上结果提示, 在研究以噁二唑-酰胺或2-氨基苯并噻唑为骨架的化合物作为MtSK抑制剂时, 要注意排除假阳性。本文筛选得到的化合物IMB-T5297是一个含有苯环的二酮类化合物, 与上述所有研究者得到的MtSK抑制剂结构不同, 具有较强的新颖性, 为MtSK抑制剂的结构优化提供了参考。
本研究获得了一个MtSK的选择性抑制剂IMB-T5297。该化合物表现出一定的体外抗结核活性, 与MtSK亲和力较好, 抑制作用较强, 且没有明显的细胞毒性, 通过结构改造有可能成为抗结核先导化合物, 为发现新机制的抗结核药物提供了有价值的新思路。
| [1] | World Health Oranization. Global Tuberculosis Report[R]. Geneva: WHO Press, 2017: 1-10. |
| [2] | Lu Y, Qiao F, You XF, et al. Research progresses of Mycobacterium tuberculosis cytochrome P450s as a potential drug target[J]. Acta Pharm Sin (药学学报), 2014, 49: 427–434. |
| [3] | Yang X, Zang X. Recent advances in study of mycobacterial membrane protein large 3 inhibitors[J]. Acta Pharm Sin (药学学报), 2017, 52: 1379–1386. |
| [4] | Gunther G. Multidrug-resistant and extensively drug-resistant tuberculosis: a review of current concepts and future challenges[J]. Clin Med (Lond), 2014, 14: 279–285. DOI:10.7861/clinmedicine.14-3-279 |
| [5] | Hoffmann H, Kohl TA, Hofmann-Thiel S, et al. Delamanid and bedaquiline resistance in Mycobacterium tuberculosis ancestral Beijing genotype causing extensively drug-resistant tuberculosis in a Tibetan refugee[J]. Am J Respir Crit Care Med, 2016, 193: 337–340. DOI:10.1164/rccm.201502-0372LE |
| [6] | Kapnick SM, Zhang Y. New tuberculosis drug development: targeting the shikimate pathway[J]. Expert Opin Drug Discov, 2008, 3: 565–577. DOI:10.1517/17460441.3.5.565 |
| [7] | Ducati RG, Basso LA, Santos DS. Mycobacterial shikimate pathway enzymes as targets for drug design[J]. Curr Drug Targets, 2007, 8: 423–435. DOI:10.2174/138945007780059004 |
| [8] | Starcevic A, Akthar S, Dunlap WC, et al. Enzymes of the shikimic acid pathway encoded in the genome of a basal metazoan, Nematostella vectensis, have microbial origins[J]. Proc Natl Acad Sci U S A, 2008, 105: 2533–2537. DOI:10.1073/pnas.0707388105 |
| [9] | Parish T, Stoker NG. The common aromatic amino acid biosynthesis pathway is essential in Mycobacterium tuberculosis[J]. Microbiology, 2002, 148: 3069–3077. DOI:10.1099/00221287-148-10-3069 |
| [10] | Mulabagal V, Calderón AI. Development of an ultrafiltration-liquid chromatography/mass spectrometry (UF-LC/MS) based ligand-binding assay and an LC/MS based functional assay for Mycobacterium tuberculosis shikimate kinase[J]. Anal Chem, 2010, 82: 3616–3621. DOI:10.1021/ac902849g |
| [11] | Simithy J, Gill G, Wang Y, et al. Development of an ESI-LC-MS-based assay for kinetic evaluation of Mycobacterium tuberculosis shikimate kinase activity and inhibition[J]. Anal Chem, 2015, 87: 2129–2136. DOI:10.1021/ac503210n |
| [12] | Tan SJ, Yu Z, Du L, et al. HPLC determination of ATP, ADP and AMP in skeletal muscle of rats of postoperative fatigue syndrome[J]. Chin J Pharm Anal (药物分析杂志), 2011, 31: 2025–2029. |
| [13] | Kanamaru T, Nakano Y, Toyoda Y, et al. In vitro and in vivo antibacterial activities of TAK-083, an agent for treatment of Helicobacter pylori infection[J]. Antimicrob Agents Chemother, 2001, 45: 2455–2459. DOI:10.1128/AAC.45.9.2455-2459.2001 |
| [14] | Lu Y, Wang B, Zheng M, et al. Study of Alamar blue and MTT assays for determining the MIC of anti-tuberculosis drugs[J]. Chin J Antituberc (中国防痨杂志), 2007, 29: 499–501. |
| [15] | Gan J, Gu Y, Li Y, et al. Crystal structure of Mycobacterium tuberculosis shikimate kinase in complex with shikimic acid and an ATP analogue[J]. Biochemistry, 2006, 45: 8539–8545. DOI:10.1021/bi0606290 |
| [16] | Pereira JH, de Oliveira JS, Canduri F, et al. Structure of shikimate kinase from Mycobacterium tuberculosis reveals the binding of shikimic acid[J]. Acta Crystallogr D Biol Crystallogr, 2004, 60: 2310–2319. DOI:10.1107/S090744490402517X |
| [17] | Kumar M, Verma S, Sharma S, et al. Structure-based in silico design of a high-affinity dipeptide inhibitor for novel protein drug target shikimate kinase of Mycobacterium tuberculosis[J]. Chem Biol Drug Des, 2010, 76: 277–284. |
| [18] | Vianna CP, de Azevedo WF Jr. Identification of new potential Mycobacterium tuberculosis shikimate kinase inhibitors through molecular docking simulations[J]. J Mol Model, 2012, 18: 755–764. DOI:10.1007/s00894-011-1113-5 |
| [19] | Blanco B, Prado V, Lence E, et al. Mycobacterium tuberculosis shikimate kinase inhibitors: design and simulation studies of the catalytic turnover[J]. J Am Chem Soc, 2013, 135: 12366–12376. DOI:10.1021/ja405853p |
| [20] | Masoko P, Mabusa IH, Howard RL. Isolation of alpha-linolenic acid from Sutherlandia frutescens and its inhibition of Mycobacterium tuberculosis' shikimate kinase enzyme[J]. BMC Complement Altern Med, 2016, 16: 366. DOI:10.1186/s12906-016-1344-1 |
| [21] | Mehra R, Rajput VS, Gupta M, et al. Benzothiazole derivative as a novel Mycobacterium tuberculosis shikimate kinase inhibitor: identification and elucidation of its allosteric mode of inhibition[J]. J Chem Inf Model, 2016, 56: 930–940. DOI:10.1021/acs.jcim.6b00056 |
| [22] | Simithy J, Reeve N, Hobrath JV, et al. Identification of shikimate kinase inhibitors among anti-Mycobacterium tuberculosis compounds by LC-MS[J]. Tuberculosis (Edinb), 2014, 94: 152–158. DOI:10.1016/j.tube.2013.12.004 |
| [23] | Alturki MS, Fuanta NR, Jarrard MA, et al. A multifaceted approach to identify non-specific enzyme inhibition: application to Mycobacterium tuberculosis shikimate kinase[J]. Bioorg Med Chem Lett, 2018, 28: 802–808. DOI:10.1016/j.bmcl.2017.12.002 |
 2018, Vol. 53
2018, Vol. 53


