多重耐药菌株的出现, 已经对人类健康造成巨大威胁, 然而目前正在开发和/或临床试验的新抗生素药物却并不多[1]。运用传统随机筛选方法发现具有新颖作用机制的抗生素数量逐年减少[2], 而对已知靶标抗生素的化学修饰不会产生全新作用机制的新化合物实体, 细菌对一种抗生素产生抗性, 往往会降低对该类中各个抗生素的敏感度[3]。随着DNA测序成本的降低和分析速度的提高, 研究者能够获得大量病原体基因组以及土壤、海洋和人类相关宏基因组的数据信息。利用基因组序列数据与生物信息学方法, 可深度剖析细菌耐药机制, 确定新药靶点, 寻找新的抗生素[4]。本文将介绍现有的耐药性相关数据库资源, 并简要概述基因挖掘在新抗生素发现中的应用。
1 抗生素耐药性与数据库抗生素耐药性是由于基因、移动DNA元件及其细菌宿主之间复杂的相互作用, 以及人类为控制细菌生长所施加的强烈选择压力而产生的[5], 细菌通过基因突变来获得对抗生素的抗性[6], 这些突变使抗生素作用靶位发生改变, 使得抗生素不能再对其靶标施加作用。与此同时, 抗性基因还可从环境有机体的基因水平转移获得, 且这种基因的水平转移具有很大的随机性[7], 难以识别或预测抗性基因的来源, 但是通过建立数据库并发展相关生物信息学分析工具, 人们可以记录可能的抗性基因, 以及引起基因变异的因素。从而确定引起耐药性的主要因素, 分析抗性产生的历史, 以期预测未来抗性发展的可能趋势[8]。例如, Wang等[9]对耐多药的鲍曼不动杆菌菌株MDR-SHH02的抗性基因进行鉴定的过程中, 利用致病岛数据库(PAIDB)预测了MDR-SHH02的基因组序列中的致病岛, 并通过与抗生素抗性基因数据库(ARDB)比对, 成功地发现了MDR-SHH02中潜在的抗生素抗性基因。此外, 运用专门的数据库进行筛选也是较为简便的鉴定基因组中毒力基因的方法[10], 检测这种毒力或抗性标记对于理解细菌与宿主的相互作用以及开发新药、疫苗和分子诊断工具至关重要。一个典型的实例是Kwok等[11]对猪丹毒病原菌爆发株SY1027的研究, 通过使用VFDB数据库中BLASTp工具搜索鉴定了37个潜在毒力基因, 其中包括7个可能与抗生素耐药性相关并且高度保守的基因。
现在已经有许多与抗生素耐药性相关数据库(表 1)[12-25], 这里简要介绍几个常用数据库。2009年, Liu和Pop[12]创建了ARDB, 这是一个人工录入的数据库, 旨在集中所有关于抗生素耐药性的信息, 用于识别和表征新基因, 并促进测序数据中抗性信息的一致注释。其中已经确定了53个与一线和二线抗生素耐药有关的基因[26]。2012年, Zankari等[14]开发了ResFinder, 这是一个基于网络的应用程序, 使用BLAST程序鉴定从全基因组数据中得到的抗生素耐药基因, 该软件可以从短读序片段装配成的部分或完整基因组中检测出1 800种不同抗性基因。2013年, McArthur课题组[13]开发了CARD, 该工具整合了含4 221个抗性基因的数据库, 使用CARD对鲍曼不动杆菌菌株TCDC-AB0715的全基因组进行分析, 可鉴定出64个与许多抗生素抗性相关的开放阅读框。
| Table 1 List of computing resources developed for new antibiotic discovery |
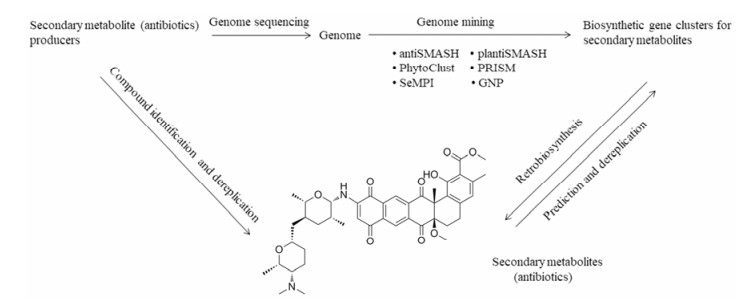
微生物天然产物是由专门的生物合成基因簇产生的。相应的生物合成酶用简单的构建模块逐步组装成复杂的次级代谢产物。筛选与天然产物生物合成相对应的基因位点序列数据的过程称为基因组挖掘。基因组测序技术和生物信息学的发展为发现新抗生素提供了新的技术手段[27], 当前已有的丰富的微生物基因组序列, 为通过基因组挖掘寻找新的生物活性小分子及开发抗生素奠定了基础。通过预测细菌在感染宿主过程中的必需基因, 可表征细菌的生理学功能, 为抗生素研发提供新的靶点, 如细菌毒力因子[28]。展现基因组挖掘在抗生素发现过程中重要性的一个例子是抗生素taromycin A (1, 图 1)的发现[29]。此项研究中运用了转化介导的重组克隆方法(transformation-associated recombination, TAR)。通过基因挖掘发现tar基因簇在抗生素生物合成中的作用, 为激活此沉默基因簇, 对包含该基因簇的基因组区域进行TAR克隆和遗传重构, 从而成功地从海洋放线菌Saccharomonospora sp. CNQ-490中表达了一个非核糖体肽合成酶生物合成基因簇, 最终得到taromycin A。这种可有效获得隐蔽生物合成途径的“即插即用”方法, 将有利于发现和开发天然产物候选药物。

|
Figure 1 Chemical structures of taromycin A (1), retimycin A (2), cinerubin B (3), arenimycin B (4) |
随着近年来丰富的遗传数据的出现, 次生代谢产物基因簇数据也日益增多, 已有多个挖掘数据工具被开发出, 并可将检测到的生物合成基因簇(BGC)与大量已知的次生代谢产物分子相关联, 从而可从基因簇预测最终产生的化合物(图 2), 一些基因组挖掘工具资源可参见表 2[30-34]。目前比较成功的基因组挖掘的例子包括:利用分子网络[基于串联质谱(MS/MS)的计算方法]和肽基因组学从35个Salinispora菌株中发现retimycin A (2, 图 1)[35], 以及利用糖原基因组学从链霉菌属Streptomyces sp. SPB74菌株分离得到抗生素cinerubin B (3, 图 1)和利用肽基因组学从海洋放线菌Salinispora aenicola CNB-527中发现了arenimycin B[36] (4, 图 1)。
|
Figure 2 A schematic procedure of new antibiotics discovery |
| Table 2 List of genomic mining tools resource |
经典的基因组挖掘方法是从基因组序列中寻找编码次级代谢物生物合成的酶的基因, 尽管次级代谢产物的种类很多, 但是这些化合物中的许多生物合成原理或合成途径具有很强的保守性。许多核心生物合成酶具有高度的氨基酸序列相似性。这些核心途径中的保守基因可用作Southern杂交实验中的探针。或者用于设计引物, 进行PCR扩增和筛选, 目前研究最深入的是聚酮化合物和非核糖体肽的生物合成[37], 其中聚酮化合物合酶(PKS)和非核糖体肽合成酶(NRPS)是主要的酶, 它们使用相对简单的结构单元, 例如丙二酸和氨基酸来构建高度复杂的天然产物。
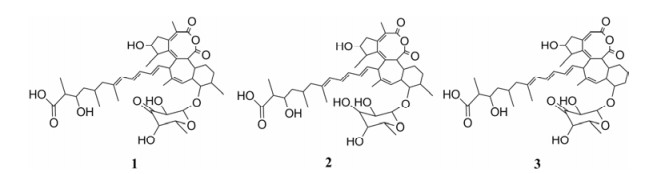
Yang等[38]从堆肥中分离出一种枯草芽孢杆菌fmb60, 其能产生具有广泛抗菌活性的代谢物, 基因组信息表明其能够合成新的PKS型抗生素。通过研究菌株fmb60的基因组序列, 使用antiSMASH筛选潜在的次级代谢物簇, 预测出枯草芽孢杆菌中NRPS/ PKS产物的化学结构, 最终得到3种具有抗菌活性的代谢产物aurantinins B、C和D (图 3)。
|
Figure 3 Structures of the aurantinins B (1), C (2) and D (3), respectively |
Son等[39]分析链霉菌KCB13F003基因组序列过程中, 发现了编码黄素依赖性卤化酶和非核糖体肽合成酶的隐蔽基因簇, 通过改变菌株的生长环境激活隐蔽基因簇的方法, 然后利用LC-MS进行筛选, 最终得到了新的环状缩酚酸肽ulleungamides A和B, 其对革兰阳性致病菌包括耐甲氧西林和耐喹诺酮金黄色葡萄球菌表现出了中等强度的抗菌活性。
2.2 比较基因组数据挖掘在经典的基因组挖掘基础上还可以进一步改进, 不仅关注单个基因, 同时还关注部分或完整的基因簇(BGC)。例如, 将已有基因组中的BGC与其他微生物BGC的测序数据, 以及MIBiG数据库进行比较[40], 从而发现在其他生物体中是否有类似的合成途径, antiSMASH也具有这种功能[41], 同时提供这种信息的MultiGeneBlast算法也可用来识别任何给定序列的相似基因簇/操纵子, Tao等[42]使用这种方法揭示了thiotetronate抗生素的生物合成途径, 同时深入了解了迭代聚酮化合物合酶-非核糖体肽合成酶的作用, 从而提出一种新的硫插入机制。
另一种用来评估已知BGC新颖性的方法是使用基因组邻域网络(GNN), 它在扩展序列相似性网络的同时, 也考虑了“目标”基因的邻域, 能够大规模预测蛋白质家族中未表征的酶的体外酶活性和体内生理功能(代谢途径)。最近进行一项研究表明[43], 通过构建烯二炔基因组邻域网络(GNN), 可分析次级代谢物基因簇。这种烯二炔GNN的分析促进了快速基因簇注释, 同时揭示烯二炔生物合成基因簇的遗传趋势。虽然这种有针对性的基因组挖掘方法非常强大, 并且能够促进新化合物的识别以及生物合成途径和分子之间的关联, 但它们有一个很大的缺陷。这种方法需要已知的同源酶序列或已经确定的BGC, 且结果仅限于已知的生物合成类型, 因此往往需要考虑将化合物的新结构类别连接到它们各自的BGC。一旦确定了BGC和合成途径, 也可以在其他生物体中检测同源簇和相应的化合物[44]。
3 宏基因组挖掘在过去的10年中, 宏基因组学的出现极大地扩展了研究者对微生物学的认识, 一般而言, 宏基因组学, 即来自异质群体的DNA分析, 是研究特定环境中不可培养微生物生物合成潜力的有力工具。经典方法包括将宏基因组DNA克隆到容易培养的宿主如大肠杆菌中, 根据所用载体的不同, 产生cosmid、fosmid或细菌人工染色体(BAC)文库。这些宏基因组文库可以进行功能性筛选从而发现新的微生物种群和新的基因或基因簇, 获得新的生理活性物质, 包括抗生素、酶和新的药物。几项基于宏基因组学的研究主要集中于使用功能性或基于序列的方法从自然环境中表征抗生素耐药基因(ARGs), 基于序列的方法和功能性宏基因组学研究之间的一个重要区别在于, 基于序列的方法可以使用ARG数据库如抗生素抗性数据库(ARDB)和综合抗生素抗性数据库(CARD)中的宏基因组信息, 通过计算机分析来识别先前已知的ARG, 而功能性宏基因组学方法的优势在于不限于先前已知的序列, 它可探索在不同的环境中发生的不可预见的ARGs, 从而能够发现新的ARG和新型的耐药机制[45, 46], Gonzalez-Plaza等[47]就应用功能性宏基因组学在克罗地亚抗生素生产废水排放点上游沉积物中检测到7种潜在的新型ARGs, 包括编码胸苷酸合成酶、二氢叶酸还原酶和D类β-内酰胺酶的基因。另外, dos Santos等[48]在研究β-内酰胺类抗生素耐药性时, 筛选了一个巴西Cerrado土壤宏基因组文库以期了解β-内酰胺类抗生素的抗性, 此过程中他们鉴定了在大肠杆菌中能使羧苄青霉素产生抗性的双加氧酶的开放阅读框, 其揭示了双加氧酶在芳香族化合物代谢上的关键作用。以上研究表明, ARG研究可以揭示新的抗菌药物耐药机制以及ARGs在自然环境中的功能, 不过, 值得注意的是, ARG在自然环境中的多样性比以前想象的要大得多, 因此必须努力建立更多的策略来确定环境中的新ARG。
4 展望自20多年前对第一个细菌基因组进行测序以来, 人们期望通过基因组数据阐明高价值细菌靶点来发现抗生素, 然而目前主要基于比较基因组学方法的初步研究并没有达到最初的预期, 因为很少有基于此方法发现的新药进入市场, 尽管如此, 细菌基因组序列依然能对抗生素的发现产生巨大的贡献, 基于新一代测序方法的新技术发展可以加速基因组和宏基因组研究, 从而为比较基因组学补充大量数据以完善鉴定抗生素靶点的方法。基因组序列信息也可以在不同的环境条件下鉴定对于细菌生长和存活至关重要的基因, 此外, 生物信息学工具也被开发出来, 用于鉴定抗菌化合物和肽(如细菌素)生物合成途径中的功能性细菌基因组。综上所述, 面对目前的抗生素耐药性危机, 需要多种策略来开发新的抗生素。尽管基于基因组测序数据的方法尚未产生显著的结果, 但是它们对抗生素发现的贡献可能仅仅只是开始。
| [1] | Aminov R. History of antimicrobial drug discovery: major classes and health impact[J]. Biochem Pharmacol, 2017, 133: 4–19. DOI:10.1016/j.bcp.2016.10.001 |
| [2] | Xu ZQ, Xu ZY. Recent progress in development of antibiotics against Gram-negative bacteria[J]. Acta Pharm Sin (药学学报), 2013, 48: 993–1004. |
| [3] | Rossiter SE, Fletcher MH, Wuest WM. Natural products as platforms to overcome antibiotic resistance[J]. Chem Rev, 2017, 117: 12415–12474. DOI:10.1021/acs.chemrev.7b00283 |
| [4] | Fields FR, Lee SW, Mcconnell MJ. Using bacterial genomes and essential genes for the development of new antibiotics[J]. Biocheml Pharmacol, 2017, 134: 74–86. DOI:10.1016/j.bcp.2016.12.002 |
| [5] | Gillings MR, Paulsen IT, Tetu SG. Genomics and the evolution of antibiotic resistance[J]. Ann N Y Acad Sci, 2017, 1388: 92–107. DOI:10.1111/nyas.2017.1388.issue-1 |
| [6] | Santos-Beneit F, Ordonez-Robles M, Martin JF. Glycopeptide resistance: links with inorganic phosphate metabolism and cell envelope stress[J]. Biochem Pharmacol, 2017, 133: 74–85. |
| [7] | Poole K. At the nexus of antibiotics and metals: the impact of Cu and Zn on antibiotic activity and resistance[J]. Trends Microbiol, 2017, 25: 820–832. DOI:10.1016/j.tim.2017.04.010 |
| [8] | Sandiford SK. Genome database mining for the discovery of novel lantibiotics[J]. Expert Opin Drug Discov, 2017, 12: 489–495. DOI:10.1080/17460441.2017.1303475 |
| [9] | Wang H, Wang J, Yu P, et al. Identification of antibiotic resistance genes in the multidrug-resistant Acinetobacter baumannii strain, MDR-SHH02, using whole-genome sequencing[J]. Int J Mol Med, 2017, 39: 364–372. DOI:10.3892/ijmm.2016.2844 |
| [10] | Bakour S, Sankar SA, Rathored J, et al. Identification of virulence factors and antibiotic resistance markers using bacterial genomics[J]. Future Microbiol, 2016, 11: 455–466. DOI:10.2217/fmb.15.149 |
| [11] | Kwok AH, Li Y, Jiang J, et al. Complete genome assembly and characterization of an outbreak strain of the causative agent of swine erysipelas--Erysipelothrix rhusiopathiae SY1027[J]. BMC Microbiol, 2014, 14: 176. DOI:10.1186/1471-2180-14-176 |
| [12] | Liu B, Pop M. ARDB-antibiotic resistance genes database[J]. Nucleic Acids Res, 2009, 37: D443–D447. DOI:10.1093/nar/gkn656 |
| [13] | Jia BF, Raphenya AR, Alcock B, et al. CARD 2017: expansion and model-centric curation of the comprehensive antibiotic resistance database[J]. Nucleic Acids Res, 2017, 45: D566–D573. DOI:10.1093/nar/gkw1004 |
| [14] | Zankari E, Hasman H, Cosentino S, et al. Identification of acquired antimicrobial resistance genes[J]. J Antimicrob Chemother, 2012, 67: 2640–2644. DOI:10.1093/jac/dks261 |
| [15] | Gupta SK, Padmanabhan BR, Diene SM, et al. ARG-ANNOT, a new bioinformatic tool to discover antibiotic resistance genes in bacterial genomes[J]. Antimicrob Agents Chemother, 2014, 58: 212–220. DOI:10.1128/AAC.01310-13 |
| [16] | Tsafnat G, Copty J, Partridge SR. RAC: repository of antibiotic resistance cassettes[J]. Database (Oxford), 2011, 2011: bar054. |
| [17] | Thai QK, Boes F, Pleiss J. The Lactamase Engineering Database: a critical survey of TEM sequences in public databases[J]. BMC Genomics, 2009, 10: 390. DOI:10.1186/1471-2164-10-390 |
| [18] | Yoon SH, Park YK, Kim JF. PAIDB v2.0: exploration and analysis of pathogenicity and resistance islands[J]. Nucleic Acids Res, 2015, 43: D624–D630. DOI:10.1093/nar/gku985 |
| [19] | Cosentino S, Larsen MV, Aarestrup FM, et al. Pathogen Finder-distinguishing friend from foe using bacterial whole genome sequence data[J]. PLoS One, 2013, 8: e77302. DOI:10.1371/journal.pone.0077302 |
| [20] | Zhou CE, Smith J, Lam M, et al. MvirDB-a microbial database of protein toxins, virulence factors and antibiotic resistance genes for bio-defence applications[J]. Nucleic Acids Res, 2007, 35: D391–D394. DOI:10.1093/nar/gkl791 |
| [21] | Chen L, Zheng D, Liu B, et al. VFDB 2016: hierarchical and refined dataset for big data analysis-10 years on[J]. Nucleic Acids Res, 2016, 44: D694–D697. DOI:10.1093/nar/gkv1239 |
| [22] | Cruz-Morales P, Kopp JF, Martinez-Guerrero C, et al. Phylogenomic analysis of natural products biosynthetic gene clusters allows discovery of arseno-organic metabolites in model streptomycetes[J]. Genome Biol Evol, 2016, 8: 1906–1916. DOI:10.1093/gbe/evw125 |
| [23] | Blin K, Medema MH, Kottmann R, et al. The antiSMASH database, a comprehensive database of microbial secondary metabolite biosynthetic gene clusters[J]. Nucleic Acids Res, 2017, 45: D555–D559. DOI:10.1093/nar/gkw960 |
| [24] | Dejong CA, Chen GM, Li H, et al. Polyketide and nonribosomal peptide retro-biosynthesis and global gene cluster matching[J]. Nat Chem Biol, 2016, 12: 1007–1014. DOI:10.1038/nchembio.2188 |
| [25] | Flissi A, Dufresne Y, Michalik J, et al. Norine, the knowledgebase dedicated to non-ribosomal peptides, is now open to crowdsourcing[J]. Nucleic Acids Res, 2016, 44: D1113–D1118. DOI:10.1093/nar/gkv1143 |
| [26] | Joshi RS, Jamdhade MD, Sonawane MS, et al. Resistome analysis of Mycobacterium tuberculosis: identification of aminoglycoside 2'-N-acetyltransferase (AAC) as co-target for drug desigining[J]. Bioinformation, 2013, 9: 174–181. DOI:10.6026/bioinformation |
| [27] | Okada BK, Seyedsayamdost MR. Antibiotic dialogues: induction of silent biosynthetic gene clusters by exogenous small molecules[J]. FEMS Microbiol Rev, 2017, 41: 19–33. DOI:10.1093/femsre/fuw035 |
| [28] | Maura D, Ballok AE, Rahme LG. Considerations and caveats in anti-virulence drug development[J]. Curr Opin Microbiol, 2016, 33: 41–46. DOI:10.1016/j.mib.2016.06.001 |
| [29] | Yamanaka K, Reynolds KA, Kersten RD, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J]. Proc Natl Acad Sci U S A, 2014, 111: 1957–1962. DOI:10.1073/pnas.1319584111 |
| [30] | Blin K, Wolf T, Chevrette MG, et al. AntiSMASH 4.0-improvements in chemistry prediction and gene cluster boundary identification[J]. Nucleic Acids Res, 2017, 45: W36–W41. DOI:10.1093/nar/gkx319 |
| [31] | Zierep PF, Padilla N, Yonchev DG, et al. SeMPI: a genome-based secondary metabolite prediction and identification web server[J]. Nucleic Acids Res, 2017, 45: W64–W71. DOI:10.1093/nar/gkx289 |
| [32] | Topfer N, Fuchs LM, Aharoni A. The PhytoClust tool for metabolic gene clusters discovery in plant genomes[J]. Nucleic Acids Res, 2017, 45: 7049–7063. DOI:10.1093/nar/gkx404 |
| [33] | Johnston CW, Skinnider MA, Wyatt MA, et al. An automated Genomes-to-Natural Products platform (GNP) for the discovery of modular natural products[J]. Nat Commun, 2015, 6: 8421. DOI:10.1038/ncomms9421 |
| [34] | Alanjary M, Kronmiller B, Adamek M, et al. The Antibiotic Resistant Target Seeker (ARTS), an exploration engine for antibiotic clusterprioritization and novel drug target discovery[J]. Nucleic Acids Res, 2017, 45: W42–W48. DOI:10.1093/nar/gkx360 |
| [35] | Duncan KR, Cruesemann M, Lechner A, et al. Molecular networking and pattern-based genome mining improves discovery of biosynthetic gene clusters and their products from salinispora species[J]. Chem Biol, 2015, 22: 460–471. DOI:10.1016/j.chembiol.2015.03.010 |
| [36] | Kersten RD, Ziemert N, Gonzalez DJ, et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules[J]. Proc Natl Acad Sci U S A, 2013, 110: E4407–E4416. DOI:10.1073/pnas.1315492110 |
| [37] | Fisch KM, Schaeberle TF. Toolbox for antibiotics discovery from microorganisms[J]. Archiv Pharm, 2016, 349: 683–691. DOI:10.1002/ardp.v349.9 |
| [38] | Yang J, Zhu X, Cao M, et al. Genomics-inspired discovery of three antibacterial active metabolites, aurantinins B, C, and D from compost-associated Bacillus subtilis fmb60[J]. J Agric Food Chem, 2016, 64: 8811–8820. DOI:10.1021/acs.jafc.6b04455 |
| [39] | Son S, Hong YS, Jang M, et al. Genomics-driven discovery of chlorinated cyclic hexapeptides ulleungmycins A and B from a streptomyces species[J]. J Nat Prod, 2017, 80: 3025–3031. DOI:10.1021/acs.jnatprod.7b00660 |
| [40] | Medema MH, Kottmann R, Yilmaz P, et al. Minimum information about a biosynthetic gene cluster[J]. Nat Chem Biol, 2015, 11: 625–631. DOI:10.1038/nchembio.1890 |
| [41] | Weber T, Blin K, Duddela S, et al. AntiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters[J]. Nucleic Acids Res, 2015, 43: W237–W243. DOI:10.1093/nar/gkv437 |
| [42] | Tao W, Yurkovich ME, Wen S, et al. A genomics-led approach to deciphering the mechanism of thiotetronate antibiotic biosynthesis[J]. Chem Sci, 2016, 7: 376–385. DOI:10.1039/C5SC03059E |
| [43] | Rudolf JD, Yan X, Shen B. Genome neighborhood network reveals insights into enediyne biosynthesis and facilitates prediction and prioritization for discovery[J]. J Ind Microbiol Biotechnol, 2016, 43: 261–276. DOI:10.1007/s10295-015-1671-0 |
| [44] | Ziemert N, Alanjary M, Weber T. The evolution of genome mining in microbes -a review[J]. Nat Prod Rep, 2016, 33: 988–1005. DOI:10.1039/C6NP00025H |
| [45] | Su JQ, Cui L, Chen QL, et al. Application of genomic technologies to measure and monitor antibiotic resistance in animals[J]. Ann N Y Acad Sci, 2017, 1388: 121–135. DOI:10.1111/nyas.2017.1388.issue-1 |
| [46] | dos Santos DF, Istvan P, Quirino BF, et al. Functional metagenomics as a tool for identification of new antibiotic resistance genes from natural environments[J]. Microb Ecol, 2017, 73: 479–491. DOI:10.1007/s00248-016-0866-x |
| [47] | Gonzalez-Plaza JJ, Simatovic A, Milakovic M, et al. Functional repertoire of antibiotic resistance genes in antibiotic manufacturing effluents and receiving freshwater sediments[J]. Front Microbiol, 2018, 8: 2675. DOI:10.3389/fmicb.2017.02675 |
| [48] | dos Santos DFK, Istvan P, Noronha EF, et al. New dioxygenase from metagenomic library from Brazilian soil: insights into antibiotic resistance and bioremediation[J]. Biotechnol Lett, 2015, 37: 1809–1817. DOI:10.1007/s10529-015-1861-x |
 2018, Vol. 53
2018, Vol. 53


