2. 西南大学-西藏农牧学院药用植物联合研发中心, 重庆 400715
2. TAAHC-SWU Medicinal Plant Joint R & D Centre, Chongqing 400715, China
青蒿素是从我国传统中药青蒿(Artemisia annua) (分类学为黄花蒿)中提取出来的一种具有独特过氧桥结构的倍半萜内酯, 国际卫生组织将基于青蒿素的联合疗法推荐为治疗疟疾的首选方案[1]。随着对青蒿素研究的深入, 研究人员发现除了治疗疟疾之外, 还对红斑狼疮和糖尿病等疾病具有良好疗效。但是青蒿中青蒿素含量较低(约占干重的0.01%至0.8%)[2], 研究人员通过多种策略尝试来提高青蒿素含量, 包括传统育种、分子育种和使用外源诱导子等。现在已有多种诱导子被报道能影响青蒿素生物合成, 例如茉莉酸甲酯(MeJA)、脱落酸(ABA)、赤霉素(GA)和氯化钠等[3-7]。特别是MeJA能显著提高青蒿素生物合成途径关键酶基因的表达, 从而提高青蒿素含量。
JA是一种十分重要的植物激素, 参与多种植物对生物胁迫或非生物胁迫的响应, 也能够诱导萜类、生物碱和黄酮类等次生代谢产物的生物合成[8]。JAZ (jasmonate ZIM-domain)蛋白是JA信号转导过程中重要的负调控蛋白, 该家族蛋白具有2个保守的功能结构域, 即ZIM (又叫TIFY)结构域和Jas (又叫CCT_2)结构域, ZIM结构域一般位于JAZ中间, 在其近N末端侧含有TIFY (TIF[F/Y]XG) motif, 属于TIFY转录因子超家族成员之一[9, 10]。JAZ形成同源或异源二聚体依赖于TIFY, JAZ的ZIM结构域及其TIFY基序介导JAZ与共抑制子NOVELINTERACTOROFJAZ (NINJA)相互结合进而共同抑制JA信号转导[11, 12]; Jas结构域靠近JAZ的C端, JAZ通过Jas结构域与JA信号受体COI1 (CORONATINE INSENSITIVE1)或其他下游转录因子相互作用。JAZ通过以下几个步骤调控JA信号转导: ①在低JA浓度时, JAZ结合如MYC2等JA信号下游转录因子并抑制其活性; ②植物受到胁迫时大量合成JA, 然后转化为茉莉酸异亮氨酸(JA-Ile)从而介导COI1与JAZ的相互作用; ③ JAZ随后被SCFCOI1类型的E3泛素连接酶介导泛素化修饰从而被26S蛋白酶体降解, 与JAZ结合的转录因子被释放; ④转录因子激活下游JA响应基因的转录[10, 13]。
虽然JAZ在JA信号转导途径中的重要地位已经被证明, 但是在植物中JAZ属于由多个成员组成的小基因家族, 例如在拟南芥中有12个JAZ家族成员, 其功能冗余又有差异[12]。在丹参(Salvia miltiorrhiza)的研究表明JAZ在调控次生代谢的生物合成中也存在着冗余和功能差异, 例如SmJAZ8负调控丹参酚酸B的生物合成, 而SmJAZ3和SmJAZ9负调控丹参酮的生物合成[14]。在青蒿中已报道了4条JAZ经酵母双杂交结果显示均能与MYC2相互作用[15]。然而在青蒿中是否还存在能与AaMYC2和AaCOI1都具有相互作用的其他JAZ家族成员还需要进一步的研究。本研究采用RACE (rapid amplification of cDNA ends)技术从青蒿中获得新的JAZ家族基因, 进行生物信息学分析, 研究新基因在不同组织、MeJA诱导及伤害处理条件下的表达水平; 荧光双分子互补实验(bimolecular fluorescence complementation, BiFC)揭示其蛋白与AaCOI1和AaMYC2相互作用关系, 为进一步深入研究青蒿JAZ家族成员在JA信号转导机制和培育高含量青蒿奠定基础。
材料与方法材料 青蒿(Artemisia annua)和本氏烟草(Nicotiana benthamiana)种子由本实验室保存。大肠杆菌(Escherichia coli) JM107和农杆菌(Agrobacterium tumefaciens) GV3101由本实验室保存。质粒pJET Vector从Fermentas公司购买, BiFC载体pXY104和pXY106由本实验室保存。
RNA的提取与cDNA的合成 植物总RNA提取根据天根RNA提取试剂盒(DP419)的使用说明进行操作, 利用NanoDrop2000C检测RNA浓度, 同时利用1.2%琼脂糖凝胶电泳检测RNA的完整性和质量。按照RNA PCR Kit (AMV) Ver.3.0 (TaKaRa)试剂盒说明书反转录获得第一链cDNA用于核心片段PCR扩增和3'RACE, 按照BD SMARTTM RACE cDNA Amplification Kit和BD AdvantageTM 2 PCR Kit (CLONTECH)试剂盒说明书反转录获得cDNA用于5'RACE。
基因的克隆 根据青蒿的基因组序列通过Vector NTI11.5设计RACE引物, 用青蒿cDNA链作为模板进行AaJAZ 5'端和3'端的扩增, 胶回收产物与PMD19-T载体连接, 转化JM107感受态细胞并送测序, 将测序结果用VectorNTI11.5软件与基因组序列进行电子拼接并设计基因全长引物。以青蒿cDNA为模板, 进行PCR扩增, 回收相应条带, 连接T载体后转化大肠杆菌DH5α, 挑取阳性菌送测序。将测序结果在NCBI上进行BLAST比对, 判断其序列为JAZ5和JAZ6的电子全长序列, 获得两条基因的物理全长序列。以上步骤所用引物序列见表 1。
| Table 1 Primers used in the study |
生物信息学分析 用Vector NTI Suite 11.5软件和BlastP进行序列分析; 在Vector NTI Suite 11.5软件查找ORF和翻译核苷酸; 使用ExPASy提供的Proteomics Server预测AaJAZ5和AaJAZ6蛋白的理化性质, 使用NCBI预测AaJAZ5和AaJAZ6的保守结构域, 使用PredictProtein预测蛋白质的二级结构, 用CLUXTALX进行多重序列比对, 并借助于MEGA7软件采用邻位相联法(neighbor-joining, NJ)构建进化树, bootstrap检验的重复次数为1 000次。
组织表达谱分析 提取青蒿的根、茎、叶、花的总RNA, 取质量合格的RNA, 用Promega Reverse Transcription System进行cDNA第一链的合成, 得到cDNA第一链作为qPCR模板, 使用BIO-RAD iQTM5 Multicolor Real-Time PCR仪, 参照iTaqTMUniversal SYBR试剂盒说明书进行qPCR反应。qPCR反应体系和程序, 参照本实验室之前的报道[16, 17]。β-actin作为内参, 根据2-△△Ct方法计算各基因的相对表达量[18]。组织表达图谱采用SPSS 16.0软件中的单向方差分析(ANOVA)统计方法, 分析根、茎、叶、花之间的差异。其中, 小写字母代表在0.05水平下的比较, 差异显著; 大写字母代表在0.01水平下的比较, 差异极显著; 相同字母表示无显著性差异, 不同字母表示有显著差异。所用引物见(表 1)。
诱导表达谱分析 配制浓度为100 μmol·L-1 MeJA作为实验组, 0.8%乙醇作为对照组, 对30天苗龄的青蒿实生苗进行喷洒处理, 青蒿分别取上面的第三叶和第四叶, 青蒿样品在0、0.5、1、3和6 h分别取样, 样品放于液氮中速冻, 用于后续实验。30天苗龄的青蒿叶片用镊子夹叶片3次, 进行伤害处理, 取样方法同MeJA处理。MeJA诱导和机械伤害诱导图谱采用t检测统计方法分析0.5、1、3和6 h的基因表达量与0 h对照组之间的差异。
AaJAZ与AaMYC2、AaCOI1的BiFC分析 将AaJAZ基因编码区连接到BiFC载体pXY106上, 与黄色荧光蛋白的C端(YFPC)融合。青蒿AaMYC2编码区连接到BiFC载体pXY104上, 与黄色荧光蛋白的N端(YFPN)融合。采用农杆菌注射法瞬时转化烟草[19], 并在激光共聚焦显微镜Olympus FV1000 (Olympus Kyoto, Japan)下观察荧光信号并拍照。AaJAZ与AaCOI1基因的相互作用方法同上。
结果 1 AaJAZ5和AaJAZ6编码蛋白质的生物信息学分析 1.1 理化性质分析使用ExPASy Proteomics Server在线软件ProtParam tool预测, 结果为, AaJAZ5蛋白的分子质量为20.57 kD, 等电点是9.52, 分子式为C897H1458N256O286S5, 带负电荷的残基(Asp+Glu): 19, 带正电荷的残基(Arg+Lys): 27, 不稳定系数为61.59, 说明该蛋白属于不稳定蛋白; AaJAZ6蛋白的分子质量为33.48 kD, 等电点是8.94, 分子式为C1460H2315N411O465S13, 带负电荷的残基(Asp+Glu): 24, 带正电荷的残基(Arg+Lys): 28, 不稳定系数为45.58, 说明该蛋白也属于不稳定蛋白。
1.2 保守结构预测使用NCBI预测的AaJAZ5蛋白的保守结构域为位于C端154~178之间的24个氨基酸的Jas (CCT_2)结构域(pfam09425)和位于N端94~120之间的26个氨基酸的ZIM (TIFY)结构域(pfam06200); AaJAZ6蛋白的保守结构域为位于C端268~292之间的24个氨基酸的Jas (CCT_2)结构域(pfam09425)和位于N端144~175之间的31个氨基酸的ZIM (TIFY)结构域(pfam06200)。
1.3 氨基酸序列比对分析将AaJAZ5和AaJAZ6氨基酸序列与已报道的4条青蒿JAZ、12条拟南芥JAZ、3条烟草JAZ、以及一条单子叶植物水稻JAZ1和一条药用植物丹参JAZ3序列进行氨基酸序列比对分析。结果显示AaJAZ5和AaJAZ6均含有与其他物种中鉴定出的JAZ氨基酸序列中的保守结构域ZIM和Jas (图 1)。其中位于N端的ZIM结构域包含比较保守的TIFY序列, 在JAZ5/6的保守结构域的预测中ZIM (TIFY)基序位于其N端的26个氨基酸的位置相吻合, 其主要功能是结合JAZ形成同源或异源二聚体[20]; 而位于C端的Jas (CCT_2)结构域的位置也相吻合, 其主要功能是结合JA受体COI1或JA信号通路中的转录因子MYC2等[21]。
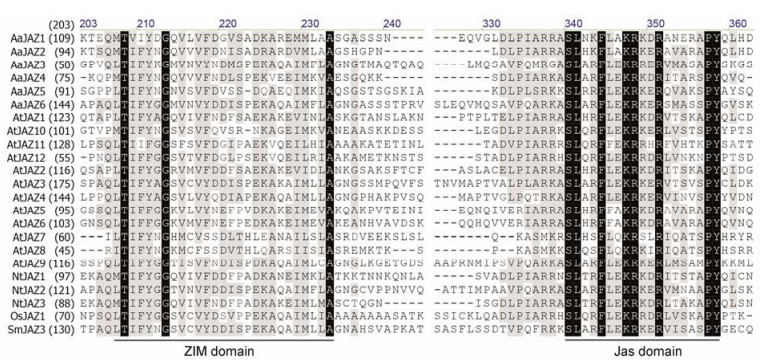
|
Figure 1 Multiple alignments of JAZ proteins from different plants. Aa: Artemisia annua; At: Arabidopsis thaliana; Nt: Nicotiana tobaccum; Os: Oryza sativa; Sm: Salvia miltiorrhiza |
在Predict Protein上预测两个蛋白的二级结构。其结果显示AaJAZ5蛋白中α-螺旋和无规卷曲是其主要结构元件, α-螺旋占33.51%, 无规卷曲占53.19%, 延伸链13.3%; AaJAZ6蛋白中α-螺旋和无规卷曲也是其主要结构元件, α-螺旋占22.96%, 无规卷曲占60.38%, 延伸链16.67%。
1.5 系统进化树分析从NCBI blastp的比对结果中选取来源于其他植物的26条JAZ氨基酸序列构建系统进化树(图 2)。从图 2中可以看出AaJAZ5与拟南芥JAZ10同源性较高, 位于同一分支上; 而AaJAZ6与青蒿JAZ1~JAZ4明显位于另外一个分支上, 暗示AaJAZ5可能与青蒿其他5个JAZ在功能上有分化。
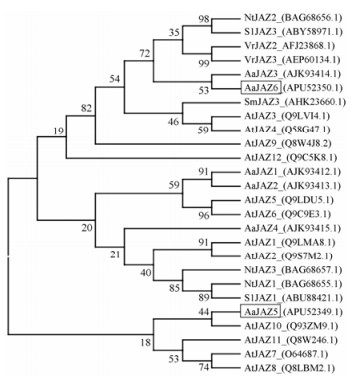
|
Figure 2 Phylogenetic tree analysis of AaJAZ5/AaJAZ6 proteins with other plant JAZ proteins. Aa: Artemisia annua; At: Arabidopsis thaliana; Nt: Nicotiana tobaccum; Sl: Solanum lycopersicum; Sm: Salvia miltiorrhiza; Vr: Vitis rupestris |
采用实时荧光定量PCR分析了AaJAZ5和AaJAZ6的组织表达和诱导表达模式。AaJAZ5在青蒿根、茎、叶和花中均有表达(图 3A)。AaJAZ5在青蒿叶中表达量最高, 约为其他组织中表达量的2.5倍; 在青蒿根、茎、花中, 该基因表达量没有显著性差异(图 3A)。在未经MeJA处理的青蒿中, AaJAZ5表达水平较低; 经MeJA处理0.5 h后, 其表达量急剧上升; 1 h后, AaJAZ5表达量达到最高, 提高了约140倍; 3 h后, AaJAZ5表达量开始回调, 但仍然显著性高于处理前(图 3B)。AaJZA5在机械伤害处理0.5和1 h后, 其表达量没有显著性变化; 机械伤害3~6 h后, 其表达量得到显著性提高, 提高7.5倍(图 3C)。这些结果表明AaJAZ5快速响应MeJA, 较慢响应机械伤害; MeJA极大幅度诱导AaJAZ5表达, 机械伤害中等程度诱导AaJAZ5表达。
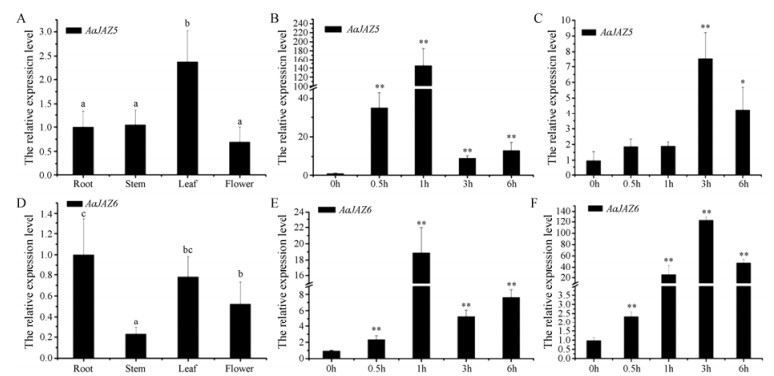
|
Figure 3 Relative expression levels of AaJAZs of Artemisia annua. A: AaJAZ5 gene relative expression in different tissues; B: The relative expression level of AaJAZ5 after treatment with MeJA in Artemisia annua; C: The relative expression level of AaJAZ5 after treatment with wounding in Artemisia annua; D: AaJAZ6 gene relative expression in different tissues; E: The relative expression level of AaJAZ6 after treatment with MeJA in Artemisia annua; F: The relative expression level of AaJAZ6 after treatment with wounding in Artemisia annua. The same letters represent the difference was not significant, and different letters represent the significant difference at the level of P < 0.05 given by one-way analysis variance (ANOVA) using SPSS 16.0 software; *Represents the significant difference at the level of P < 0.05 and **Indicates the significant difference at the level of P < 0.01, given by t-test |
AaJAZ6在青蒿根、茎、叶和花中均有表达(图 3D)。AaJAZ6在青蒿茎中表达量最低, 在根、叶、花中该基因表达量没有显著性差异(图 3D)。在未经MeJA处理的青蒿中, AaJAZ6表达水平较低; 经MeJA处理0.5 h后, 其表达量出现上升; 1 h后, AaJAZ6表达量达到最高, 提高了19倍以上; 3 h后, AaJAZ6表达量开始回调, 但仍然显著性高于处理前(图 3E)。AaJZA6在机械伤害处理0.5 h后, 其表达量显著性提高; 1 h后, 其表达量大幅度提高; 3 h后, 其表达量达到最高, 提高了120倍; 随后其表达量有所降低, 但仍远高于伤害前(图 3F)。这些结果表明AaJAZ6较慢响应MeJA, 快速响应机械伤害; MeJA能中等程度诱导AaJAZ6表达, 机械伤害能大幅度诱导AaJAZ6表达。
3 AaJAZ5/AaJAZ6与AaMYC2/AaCOI1的相互作用采用BiFC在烟草细胞中研究了AaJAZ5/AaJAZ6与AaMYC2/AaCOI1的相互作用。在白光条件下, 没有检测到任何荧光信号, 但是能够呈现出清晰的细胞形态和边界。在激发光条件下, 同时表达AaJAZ5-YFPC+AaMYC2-YFPN的烟草细胞中检测到强烈的绿色荧光; 同时表达AaJAZ6-YFPC+AaMYC2-YFPN的烟草细胞中也检测到强烈的绿色荧光, 表明AaJAZ5和AaJAZ6都能够与AaMYC2相互作用(图 4A)。在白光和激发光融合成像中, 荧光信号符合定位于细胞核的特征, 表明AaJAZ5和AaJAZ6与AaMYC2的相互作用发生在细胞核。
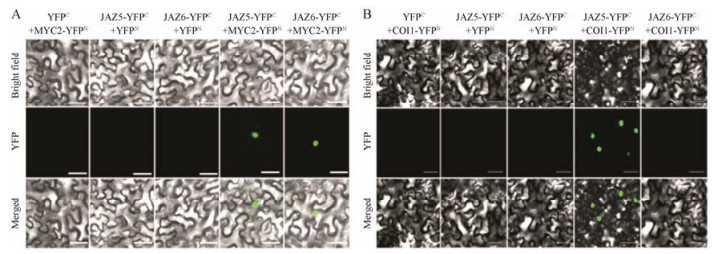
|
Figure 4 Either AaJAZ5 or AaJAZ6 interact with transcription factors of AaMYC2 and AaCOI1, respectively. A: AaJAZ5 or AaJAZ6 interact with transcription factors of AaMYC2; B: AaJAZ5 or AaJAZ6 interact with transcription factors of AaCOI1; Scale bar = 20 μm |
在激发光条件下, 同时表达AaJAZ5-YFPC+ AaCOI1-YFPN的烟草细胞中检测到强烈的绿色荧光; 在同时表达AaJAZ6-YFPC+AaCOI1-YFPN的烟草细胞中没有检测绿色荧光, 表明只有AaJAZ5能够与AaCOI1相互作用(图 4B)。在白光和激发光融合成像中, 荧光信号符合定位于细胞核的特征, 表明AaJAZ5与AaCOI1的相互作用发生在细胞核。BiFC结果表明, AaJAZ5和AaJAZ6两者都能够与正调控青蒿素生物合成的AaMYC2相互作用, 而只有AaJAZ5与JA受体AaCOI1有相互作用。
讨论JAZ作为JA信号途径中的抑制因子, 与SCFCOI1复合体结合激活MYC2调控JA信号通路中下游响应基因的表达[13, 14]。喷施MeJA能够大幅度提高青蒿素含量, 这是由于青蒿中的MeJA响应转录因子如MYC2、ORA和ERF等经MeJA诱导后进一步上调青蒿素生物合成途径基因的表达所致。其中青蒿MYC2 (AaMYC2)是JA信号介导调控青蒿素超量积累最为重要的转录因子[15]。随着对JA信号转导机制的解析, 发现JAZ、COI1和MYC2共同组成JA信号转导的核心元件。本研究得到的AaJAZ5和AaJAZ6的氨基酸序列中均具有保守的ZIM结构域和Jas结构域, 与已经报道的青蒿和拟南芥的JAZ家族成员的基本特征相符合[22, 23]。但是进化树分析表明AaJAZ5与青蒿其他几个JAZ位于不同的分支上, 暗示青蒿的JAZ家族基因也存在着功能分化。随后的组织表达谱分析发现AaJAZ5和AaJAZ6具有不同的组织表达模式。而在诱导表达模式上, AaJAZ5和AaJAZ6具有一定相似性:外源MeJA和机械伤害处理, AaJAZ5和AaJAZ6基因表达水平均显著提高, 这与拟南芥JAZ响应外源MeJA和机械伤害的报道一致[22, 24]。然而在MeJA和机械伤害处理后, AaJAZ5和AaJAZ6的表达存在明显差异, 出现了功能的分化: AaJAZ5是快速响应MeJA信号的JAZ基因; 而AaJAZ6是快速响应机械损伤的JAZ基因; 两者在对JA和机械损伤信号响应中具有一定程度的功能互补。JAZ基因家族不同成员的功能分化和冗余在拟南芥中也有相关报告[12]。
基于BiFC发现AaJAZ5和AaJAZ6都能够与AaMYC2相互作用, 与已报道的其他4个青蒿JAZ与AaMYC2均有相互作用的结果一致[15], 意味着AaJAZ5和AaJAZ6通过与AaMYC2相互作用影响JA调控青蒿素生物合成基因的表达。由于JAZ是负调控蛋白, 其与AaMYC2的相互作用的后果是导致AaMYC2对青蒿素生物合成基因的转录调控作用减弱。在JA信号作用下, JAZ受到诱导后表达量大幅度提高, 其进一步与COI1蛋白结合形成SCFCOI1/JAZ复合体, JAZ被降解后释放MYC2去激活下游相关基因表达[9-12, 25]。在本文鉴定的两个JAZ中, 只有AaJAZ5既能与青蒿COI1 (AaCOI1)相互作用又能与AaMYC2相互作用。但是这并不能充分证明AaJAZ5在调控青蒿素生物合成中具有更重要的作用。在模式植物拟南芥中, 研究人员发现并不是所有能与MYC2相互作用的JAZ都能与COI1相互作用[14]。然而, 除了使用BiFC证明AaJAZ5能与AaCOI1结合, 而AaJAZ6不能与AaCOI1相互作用之外, 对AaJAZ5和AaJAZ6的氨基酸序列的进化树分析、组织表达谱分析和诱导表达谱分析均表明AaJAZ5和AaJAZ6可能存在着功能分化。例如进化树分析表明AaJAZ5和青蒿其他5个JAZ明显不在同一分支上, AaJAZ5与拟南芥JAZ10具有较高同源性, 而现有研究结果表明拟南芥JAZ10是JAZ家族中一个重要成员, 不仅介导了JA信号转导还与JA信号与其他信号途径的相互影响有密切关系。另外拟南芥JAZ10还存在着多个可变剪切的版本[21, 25], 从而在JA信号的负反馈调控中具有重要作用。
综上所述, 本研究从青蒿中克隆了两个新的JAZ基因家族成员, 表明青蒿JAZ家族成员存在着功能分化, AaJAZ5可能是青蒿中真正参与JA信号转导通路的主效基因, 对其在JA信号介导调控青蒿素生物合成的详尽生物学功能有待进一步深入研究。
| [1] | Graham IA, Besser K, Blumer S, et al. The genetic map of Artemisia annua L. identifies loci affecting yield of the antimalarial drug artemisinin[J]. Science, 2010, 327: 328–331. DOI:10.1126/science.1182612 |
| [2] | Weathers PJ, Elkholy S, Wobbe KK. Artemisinin:the biosynthetic pathway and its regulation in Artemisia annua, a terpenoid-rich species[J]. In vitro Cell Dev Biol-Plant, 2006, 42: 309–317. DOI:10.1079/IVP2006782 |
| [3] | Jing F, Zhang L, Li M, et al. Abscisic acid (ABA) treatment increases artemisinin content in Artemisia annua by enhancing the expression of genes in artemisinin biosynthetic pathway.[J]. Biologia, 2009, 64: 319–323. |
| [4] | Xiang L, Zhu S, Zhao T, et al. Enhancement of artemisinin content and relative expression of genes of artemisinin biosynthesis in Artemisia annua by exogenous MeJA treatment[J]. Plant Growth Regul, 2015, 75: 435–441. DOI:10.1007/s10725-014-0004-z |
| [5] | Banyai W, Mii M, Supaibulwatana K. Enhancement of artemisinin content and biomass in Artemisia annua by exogenous GA3 treatment[J]. Plant Growth Regul, 2011, 63: 45–54. DOI:10.1007/s10725-010-9510-9 |
| [6] | Guo XX, Yang XQ, Yang RY, et al. Salicylic acid and methyl jasmonate but not Rose Bengal enhance artemisinin production through invoking burst of endogenous singlet oxygen[J]. Plant Sci, 2010, 178: 390–397. DOI:10.1016/j.plantsci.2010.01.014 |
| [7] | Irfanqureshi M, Israr M, Abdin M, et al. Responses of Artemisia annua L. to lead and salt-induced oxidative stress[J]. Environ Exp Bot, 2005, 53: 185–193. DOI:10.1016/j.envexpbot.2004.03.014 |
| [8] | Tian J, Liu Y, Fang MF. Review on the influence of jasmonates on medicinal plant secondary metabolism[J]. Nat Prod Res Dev, 2015, 27: 185–190. |
| [9] | Vanholme B, Grunewald W, Bateman A, et al. The tify family previously known as ZIM[J]. Trends Plant Sci, 2007, 12: 239–244. DOI:10.1016/j.tplants.2007.04.004 |
| [10] | Browse J. Jasmonate passes muster:a receptor and targets for the defense hormone[J]. Annu Rev Plant Biol, 2009, 60: 183–205. DOI:10.1146/annurev.arplant.043008.092007 |
| [11] | Pauwels L, Barbero GF, Geerinck J, et al. NINJA connects the co-repressor TOPLESS to jasmonate signalling[J]. Nature, 2010, 464: 788–791. DOI:10.1038/nature08854 |
| [12] | Pauwels L, Goossens A. The JAZ proteins:a crucial interface in the jasmonate signaling cascade[J]. Plant Cell, 2011, 23: 3089–3100. DOI:10.1105/tpc.111.089300 |
| [13] | Chico JM, Chini A, Fonseca S, et al. JAZ repressors set the rhythm in jasmonate signaling[J]. Curr Opin Plant Biol, 2008, 11: 486–494. DOI:10.1016/j.pbi.2008.06.003 |
| [14] | Shi M, Zhou W, Zhang J, et al. Methyl jasmonate induction of tanshinone biosynthesis in Salvia miltiorrhiza hairy roots is mediated by JASMONATE ZIM-DOMAIN repressor proteins[J]. Sci Rep, 2016, 6: 20919. DOI:10.1038/srep20919 |
| [15] | Shen Q, Lu X, Yan TX, et al. The jasmonate-responsive AaMYC2 transcription factor positively regulates artemisinin biosynthesis in Artemisia annua[J]. New Phytol, 2016, 210: 1269. DOI:10.1111/nph.13874 |
| [16] | Wang YX, Long SP, Zeng LX, et al. Enhancement of artemisinin biosynthesis in transgenic Artemisia annua L. by overexpressed HDR and ADS genes[J]. Acta Pharm Sin (药学学报), 2014, 49: 1346–1352. |
| [17] | Long SP, Lu Y, Wang YX, et al. Enhancement of tropane alkaloids production in transgenic hair roots of Atropa belladonna by overexpressing endogenous genes AbPMT and AbH6H[J]. Acta Pharm Sin (药学学报), 2013, 48: 243–249. |
| [18] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 2001, 25: 402–408. DOI:10.1006/meth.2001.1262 |
| [19] | Rensing SA, Lang D, Zimmer AD, et al. The physcomitrella genome reveals evolutionary insights into the conquest of land by plants[J]. Science, 2008, 319: 64–69. DOI:10.1126/science.1150646 |
| [20] | Chini A, Fonseca S, Chico JM, et al. The ZIM domain mediates homo-and heteromeric interactions between Arabidopsis JAZ proteins[J]. Plant J, 2009, 59: 77–87. DOI:10.1111/tpj.2009.59.issue-1 |
| [21] | Melotto M, Mecey C, Niu Y, et al. A critical role of two positively charged amino acids in the Jas motif of Arabidopsis JAZ proteins in mediating coronatine-and jasmonoyl isoleucine-dependent interactions with the COI1 F-box protein[J]. Plant J, 2008, 55: 979–988. DOI:10.1111/tpj.2008.55.issue-6 |
| [22] | Kazan K, Manners JM. JAZ repressors and the orchestration of phytohormone crosstalk[J]. Trends Plant Sci, 2012, 17: 22–31. DOI:10.1016/j.tplants.2011.10.006 |
| [23] | Perez AC, Durand AN, Bossche RV, et al. The non-JAZ TIFY protein TIFY8 from Arabidopsis thaliana is a transcriptional repressor[J]. PLoS One, 2014, 9: e84891. DOI:10.1371/journal.pone.0084891 |
| [24] | Chung HS, Koo AJ, Gao X, et al. Regulation and function of Arabidopsis Jasmonate ZIM-domain genes in response to wounding and herbivory[J]. Plant Physiol, 2008, 146: 952–964. DOI:10.1104/pp.107.115691 |
| [25] | Fonseca S, Chini A, Hamberg M, et al. (+)-7-iso-Jasmonayl-L-isoleucine is the endogenous bioactive jasmonate[J]. Nature Chem Biol, 2009, 5: 344. DOI:10.1038/nchembio.161 |
 2018, Vol. 53
2018, Vol. 53






