HIV-1逆转录酶(reverse transcriptase, RT)是一个由p51和p66亚基组成的异二聚体酶, 是HIV-1 pol基因的产物[1]。p51和p66亚基前440个氨基酸序列相同, 但是在空间构象上却有着显著差异, 表现为p51亚基的活性部位序列呈闭合构象而不具有催化功能, 仅起构象调节作用。因此每个p66/p51异二聚体只有一个具有活性的聚合酶功能域, 它位于p66亚基上的N末端部位, p66亚基C末端为RNase H活性部位(图 1)[2]。

|
Figure 1 The crystal structure and sequence structure of RT |
上世纪90年代, RT功能域的晶体结构已得到确证, 从图 1可以看出, 它含有5个β折叠和2个α螺旋结构。RT结构域外形类似于人的右手, 可分为手指、手掌、拇指和连接等4个亚结构域, 其中手掌亚结构域最为保守[2]。聚合酶结构域中包含3个保守的天冬氨酸残基: D110、D185和D186, 该处与引物的3'-OH末端空间上接近, 二者能相互结合, 催化以脱氧核糖核苷三磷酸(dNTP)为原料, 以单链病毒RNA为模板逆转录为双链病毒DNA。研究表明, 上述3个天冬氨酸若发生突变, 聚合酶的活性将会显著降低。此外, 聚合酶结构中的凹槽(cleft)区域, 能引导模板−引物的准确定位。手掌域中的DNA“引物沟”(primer grip)结构域, 对引物末端与聚合酶活性位点的准确契合及后续聚合过程中模板−引物的易位有重要意义[3, 4]。
2 HIV-1逆转录酶聚合酶功能域的功能HIV-1多聚酶主要有两种功能:依赖RNA的DNA多聚酶活性(RDDP)和依赖DNA的DNA多聚酶活性(DDDP)。前者在复制最初阶段以病毒自身的RNA链为模板催化底物生成RNA/DNA杂化双链, 后者则是以新生成的DNA链为模板催化底物生成双链DNA, 就此完成病毒基因由单链RNA转移到双链DNA的逆转录过程[5]。
多聚酶催化的脱氧核苷酸的聚合反应是逆转录过程中至关重要的一步, 此反应是有序、逐步进行的:首先酶与模板引物相结合(step 1);然后适当的底物结合到酶−模板引物复合物上, 在此过程中酶的构象将发生变化(step 2);第三步是金属离子通过亲核进攻使单个的(脱氧)核苷酸结合到延长的DNA链中(step 3), 同时产生磷酸二酯键释放焦磷酸(step 4);最后酶从模板引物中分离出来或是沿着产物DNA易位到下一个新生成的3位末端[即从N位点易位到P位点(step 5), 引物易位前后存在的两个暂时动态中间体分别成为复合物N、复合物P], 开始下一轮的催化反应(图 2)[6]。
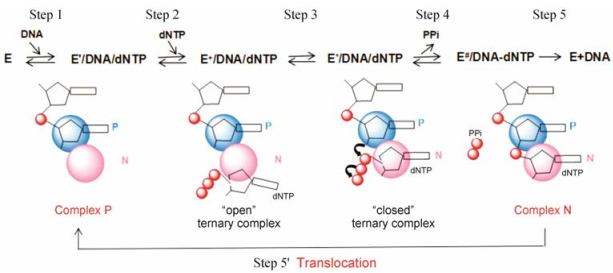
|
Figure 2 Mechanism of action of DNA polymerization |
目前临床应用的HIV-1 RT抑制剂主要分为核苷(酸)类逆转录酶抑制剂(nucleoside/nucleotide RT inhibitors, NRTIs/NtRTIs)和非核苷类逆转录酶抑制剂(nonnucleoside RT inhibitors, NNRTIs)两大类。NRTIs/NtRTIs的结构属于核苷类似物, 可作为天然脱氧核苷酸的类似物嵌入病毒DNA双链, 由于其缺乏3′-OH而导致逆转录反应终止[7]; NNRTIs属于结构多样性的杂环化合物, 它们通过结合在RT活性位点旁边的疏水区诱导逆转录酶构象发生改变进而引起催化部位的结构破坏[8]; 除对传统作用靶点的研究外, 寻找全新作用机制的HIV-1 RT抑制剂也是当前抗艾滋病药物研究的重要任务, 本文重点介绍这方面的研究进展。
3.1 核苷酸竞争性逆转录酶抑制剂核苷酸竞争性逆转录酶抑制剂(nucleotide- competing RT inhibitors, NcRTIs)是一类作用机制独特的抑制剂, 其通过可逆性抑制进入RT活性位点的dNTP底物与酶的结合发挥抑制作用[8]。代表性化合物有吲哚吡啶酮类CBL-4.0 (1)和CBL-4.1 (INDOPY-1, 2)、苯并呋喃并嘧啶类(3~7)、4-二甲氨基-6-乙烯基嘧啶类(DAVPs, 9~11)[9] (图 3)。
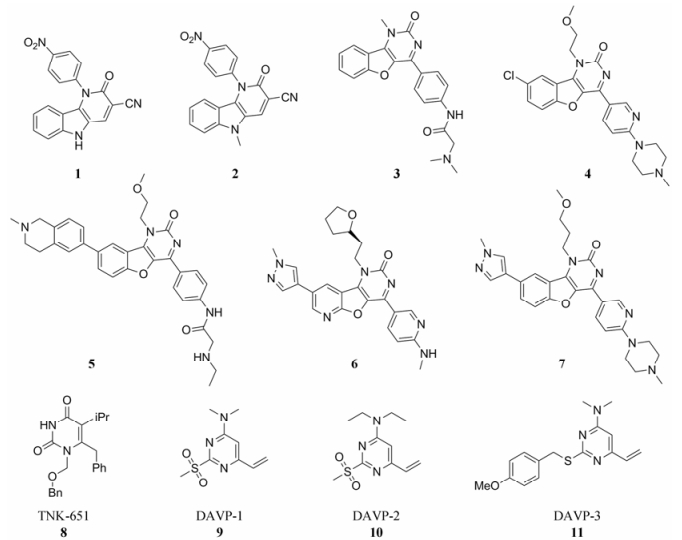
|
Figure 3 Chemical structures of NcRTIs |
Valeant和Tibotec两大公司通过对化合物库高通量筛选得到吲哚吡啶酮类化合物(indolopyridones, INDOPYs) CBL-4.0 (VRX-329747)和CBL-4.1 (INDOPY-1)[9]。这两个化合物具有较好的抗病毒活性, EC50值分别为150~200 nmol·L−1和20~30 nmol·L−1, 且细胞毒性极低(CC50 > 100 μmol·L−1)。机制研究表明两个化合物均作用于HIV-1复制的逆转录过程。INDOPYs结构不同于核苷类似物, 作用机制亦区别于传统的NNRTIs, 主要体现在以下3方面: ① INDOPYs的抗病毒谱与NNRTIs和NRTIs不同。NNRTIs特异性作用于HIV-1而INDOPY-1对HIV-1、HIV-2 (EC50 = 180 nmol·L−1, HIV-2 ROD)和猴免疫缺陷病毒(SIV, EC50 = 210 nmol·L−1, SIV Mac251)均有活性[10]; NRTIs属于广谱抗病毒抑制剂, 而目前报道的INDOPY-1仅作用于慢病毒属[11]; ② INDOPYs对NNRTIs耐药突变株活性影响不同。VRX-329747和INDOPY-1活性几乎不会被NNRTIs选择性突变株影响, 其对NNRTIs代表性的双突变株K103N-L100I RT活性下降为原来的1/4, 而efavirenz对突变株几乎丧失活性[11]; ③ INDOPYs对NRTIs耐药突变株活性影响不同: INDOPY-1的活性几乎不被嘧啶类似物耐药突变(包括M41L/L210W/T215Y和D67N/K70R/T215F/K219Q突变[12])和其他NRTIs诱导的突变影响; 当发生M184V或Y115F单突变时, INDOPY-1的活性下降为原来的1/8~1/3, 而两者发生双突变时INDOPY-1的活性下降约为原来的1/100;发生K65R突变时, INDOPY-1的活性增强2~5倍, 而此突变能够引起除AZT以外所有的NRTIs活性下降[11]。
鉴于INDOPYs特殊的抗耐药性, 研究者对INDOPY-1进行了一系列的机制研究。结果表明, INDOPY-1能够结合并稳固RT-DNA模板二元复合物, 其对DNA聚合反应抑制作用不受外来核苷酸种类的影响(不同于NRTIs的作用机制); 另一个有趣的发现是, 当引物链3'-末端最后一个核苷酸为嘧啶类核苷酸T和C时可以大大提高INDOPY-1的结合力[13]。位点特异性足迹法(site-specific footprinting)表明, INDOPY-1在HIV-1 RT易位后代替核苷酸与RT结合, 诱导形成一个稳固的失活复合物, 从而阻止核苷酸的结合, 若要恢复合成则需要INDOPY-1与RT解离, 即使最后发生解离, INDOPY-1也改变了RT催化合成动力学而发挥抑制作用。
目前尚未得到RT/INDOPY-1的晶体结构, 可能是由于INDOPYs仅特异性作用于RT/模板−引物二元复合物形成的中间过渡态, 这在一定程度上影响了对该类化合物作用机制的理解并限制了对该类化合物的进一步合理修饰。
受INDOPYs启发, Claudio F. Sturino课题组通过高通量筛选的方法发现了苯并呋喃并嘧啶类NcRTIs, 并通过大量结构优化和构效关系探讨得到活性突出的化合物5~7, 与NNRTIs相比, 该类化合物表现出了很好的脂溶性、水溶性和肝微粒体代谢稳定性, 进一步修饰主要针对改善化合物的渗透性和药动学性质[11]。
3.1.2 4-二甲氨基-6-乙烯基嘧啶类(DAVPs)不同于INDOPYs, NcRTIs家族的另一成员DAVPs (9~11)是由组合化学的方法得到。由于DAVPs结构与HEPT衍生物TNK-651 (8)类似, 最初DAVPs被归为NNRTIs, 但随后的酶学研究揭示了DAVPs与核苷酸底物的竞争性抑制机制。不同于特异性作用于RT/模版−引物二元复合物的INDOPYs, DAVPs可以作用于RT/模版−引物二元复合物和游离态RT。以上DAVPs三个化合物中, DAVP-1对野生型酶活性最好(Ki = 8 nmol·L−1), 突变株的活性测试表明, L100I或V179D突变几乎不会影响DAVP-1的活性(Ki = 12和15 nmol·L−1), 而该突变却使3个上市NNRTIs (efavirenz、nevirapine和delavirdine)的活性大大下降[10, 14−16], 并会使etravirine活性中度下降[17−20]。K103N和Y181I突变会使DAVP-1的活性大大下降(Ki = 3.2和39 μmol·L−1)。不同于DAVPs, NcRTIs家族INDOPYs的活性却几乎不会被上述突变所影响[14]。
目前已得到野生型HIV-1 RT/DAVP-1复合物的晶体结构(图 4)[21]。其晶体结构显示, 其作用靶点位于p66亚基拇指和手掌亚结构域交界处, 邻近聚合酶活性位点, 而非NNRTIs结合位点(NNIBP)。NcRTIs抑制剂结合位点(NcIBP)的氨基酸对酶活性的发挥具有重要作用, 该结构域的氨基酸大多属于高度保守的氨基酸, 不易发生突变, 这为设计新型抗耐药性抑制剂提供了有益的启示。

|
Figure 4 The co-crystal complex of DAVP-1/RT (PDB code: 3ISN) |
但是, DAVP-1/RT复合物晶体结构尚不能解释位于NNIBP的K103N和Y181I突变为何会影响DAVP-1的活性。有趣的是, 人们从RT/NNRTIs (PDB: 1TV6、2ZD1、1KLM和2B5J)的晶体结构分析发现, NNIBP和催化活性位点中间有个小的通道使得NNIBP与催化位点相连通。Botta研究组[22]设想DAVP-1与NNIBP结合后经过一系列的相互作用可穿过位于NNIBP和催化位点之间的通道到达NcIBP发挥抑制作用, 随后的分子动力学实验对这一设想进行了验证。这一重要发现让研究者对NNIBP的修饰有了更广阔的思路, 同时, DAVPs特殊的作用机制引起了研究者进一步开发NcRTIs类小分子抑制剂。
3.2 逆转录酶定向突变诱导抑制剂RT定向突变诱导抑制剂整合入病毒DNA后, 可使其发生错配, 增加HIV突变概率, 进一步导致致死性突变, 但是它们不能阻断DNA的合成。这些抑制剂包括部分核苷类衍生物(图 5), 如: 5-OH-dC (12), 其通过细胞激酶催化生成5-OH-dCTP作为核苷酸类似物与鸟嘌呤核苷酸配对改变DNA的复制过程[23]; KP-1212 (14), 属于非链终止的NRTI脱氧胞苷核苷类似物, 由一个脱氧核糖和一个修饰的杂环碱基组成, 它能够通过RT整合进入病毒DNA使碱基发生错配从而增加HIV的突变率, 这种缺陷型病毒会导致HIV致死性突变[24]; KP-1461 (15), 是KP-1212的单磷酸化前药, 使HIV突变率增加, 生存能力大大降低。此外, 5-AZC (13)[25]、5-羟甲基-2'-dU (16)和5-羟甲基-2'-dC (17)[23]也属于核苷类似物突变诱导抑制剂, 新颖独特的抗病毒机制使得这类化合物具有良好的开发前景。

|
Figure 5 Chemical structures of RT-directed mutagenic inducers inhibitors |
引物/模板−竞争性逆转录酶抑制剂有KM-1 (18)[26]、SY-3E4 (19)[27]和N-{2-[4-(胺基磺酰基)苯基]乙基}-2-(2-噻吩)乙酰胺(NAPETA, 20)[28] (图 6)。
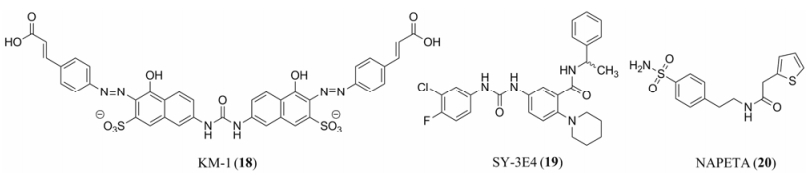
|
Figure 6 Chemical structures of primer/template-competing RT inhibitors |
KM-1类化合物在结构上是高度对称的, 其发挥抑制作用主要是通过替代酶上的DNA, 尽管酶- DNA-抑制剂三元复合物可以形成, 但并不能产生催化作用, 即KM-1的结合位点与模板引物的结合位点是重叠的, KM-1竞争性占据了结合位点使得模板引物无法进入DNA多聚酶的活性位点[26]。SY-3E4能够取代RT的适配子, 选择性抑制DNA依赖的DNA聚合酶, 从而抑制野生型和突变型HIV-1的复制, 但该类抑制剂对RNA依赖的DNA聚合酶活性没有影响[27]。NAPETA抑制剂可以同时抑制DDDP和RDDP, 其IC50值分别为1.2和2.1 μmol·L−1。此外, NAPETA也可以通过干扰RT-DNA复合物的形成发挥抑制作用[28]。
3.4 易位缺陷型抑制剂TDRTIs属于在糖部分做修饰的NRTIs抑制剂, 其主要通过阻止RT易位来发挥作用。其中, 4'位置取代的EFdA活性最好, 属于含3'-OH的核苷酸类似物, 对突变株的活性优于传统的NRTIs (图 7)。例如, EFdA对M184V突变株的EC50为8 nmol·L−1, 对其他突变株甚至更加灵敏。EFdA通过削弱RT从易位前核苷酸结合位点(N site)到易位后引物结合位点(P site)的过程发挥抑制作用。此外, EFdA单磷酸化产物容易作为底物被错误识别, 这也会导致引物链延长失败。研究表明, EFdA的糖环构型与4'-乙炔基在活性发挥中具有重要作用。目前EFdA的研究处于临床Ⅰ期试验阶段[29]。

|
Figure 7 Chemical structure of TDRTIs |
DCTRTIs结构类似于NRTIs, 其dNTPs可作为底物参与DNA链的延长, 进而由于立体位阻干扰RNA: DNA杂合及RT核苷酸结合, 最终封锁DNA链的延长, 此外还可以通过影响碱基配对阻止(+)链DNA的合成[5]。主要包括以下两类化合物(图 8)。
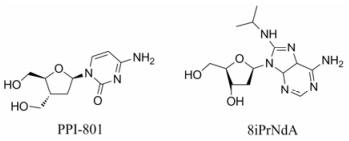
|
Figure 8 Chemical structures of DCTRTIs |
PPI-801三磷酸腺苷可以作为RT的底物参与DNA链的延长, 但不同于其他NRTIs直接作为链的终止剂。相反, 3'-羟甲基取代后仍允许一个额外的dNTP结合, 经过这一步, DNA链在+1的位置发生延迟终止[30]。更重要的是, DNA终止链的倒数第二个核苷酸可以减少PPI-801被RT切除的风险, 正因如此, 该类化合物可以对NRTI突变株产生好的抑制效果[5]。
3.5.2 8-异丙基-氨基-2'-脱氧腺苷8iPrNdA是近几年报道的核苷类似物, 不同于在核糖修饰的NRTIs, 该化合物修饰在腺嘌呤的8位, 修饰后的化合物可能对拇指结构域p66亚基的α螺旋产生立体位阻引起延迟性链终止。同PPI-801相似, 8iPrNdA结合后在+1位终止DNA链合成, 尽管8iPrNdA活性不是特别高, 但是在腺嘌呤的修饰而非核糖部分可以作为NRTIs优化的新方向[5]。
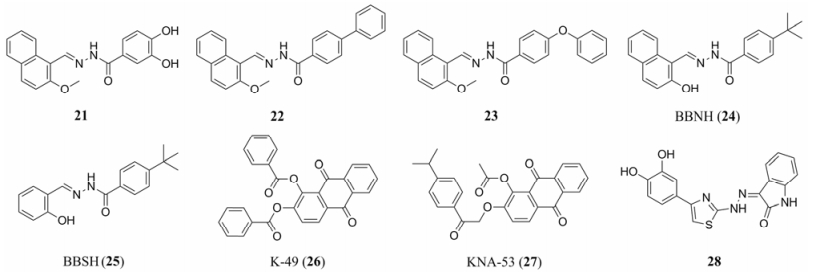
3.6 靶向聚合酶和核糖核酸酶H (RNase H)的双重抑制剂二羟基苯甲酰基萘基腙(DHBNH, 21) (图 9)具有抑制聚合酶和RNase H的活性, 其结合位点距离RNase H活性位点 > 50 Å ,与NNIBP部分重叠。DHBNH可以直接影响聚合酶“引物沟”和拇指结构域的位置来发挥抑制活性(图 10)[31]。对DHBNH进一步修饰得到兼具聚合酶和RNase H抑制活性的化合物22和23。N-(4-叔丁基苯酰基)-2-羟基-1-萘甲醛腙(BBNH, 24)及其类似物N-(4-叔丁基苯酰基)- 2-羟基-1-萘甲醛水杨酰腙(BBSH, 25), 同样是靶向于HIV-1RT的抑制剂, 具有抑制RNase H和DNA聚合酶的活性, 研究发现, 两者还能降低逆转录酶二聚体的稳定性[32]。此外, 化合物K-49 (26)、KNA-53(27)和28也被证实具有双位点抑制活性[33, 34]。化合物28抑制RDDP和RNase H的IC50值分别为2和1.4μmol·L−1[35]。这种多种作用机制可以很好地降低抑制剂的耐药性, 也是今后抗艾滋病药物的研究方向之一。
|
Figure 9 Chemical structures of dual inhibitors of the HIV-1 RT-associated DNA polymerase and RNase H |

|
Figure 10 The co-crystal complex of HIV-1 RT and DHBNH (21) (PDB code: 2I5J) |
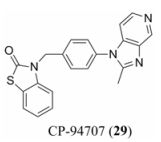
逆转录起始于tRNA模板引物与HIV-1基因的特定序列结合。由于起始过程引物与模板都是RNA, 该过程是逆转录的限速步骤, 因此该过程被认为是新的药物靶点。化合物CP-94707 (图 11, 29)对野生型RT和NNRTIs常见的突变株(Y181、Y188和K103)都有抑制作用, 它结合于RT使其“引物沟”发生扭曲同时影响核苷酸进入速度发挥抑制作用[36]。具有RNase H抑制活性的DHBNH (21)也通过抑制逆转录的起始阶段发挥作用[31]。
|
Figure 11 Chemical structure of CP-94707 |
HIV-1 RT的二聚体结构对于其功能的发挥是必需的, p66和p51亚基的结合部位发生变异会抑制酶的聚合, 从而降低催化活性。因此, 可以通过阻断蛋白间相互作用来抑制RT的功能[37]。目前已经证实一些小分子和肽类化合物具有抑制逆转录酶二聚化的活性, 这类抑制剂具有很好的专一性和较低的耐药性[38]。
3.8.1 肽类抑制剂RT的二聚化过程及其晶体结构表明, p66和p51亚基的主要连接亚结构域中富含色氨酸, 且有文献报道两个亚基连接区域的389~407位氨基酸残基形成的多肽可以在体外抑制HIV RT[39]。Depollier等[37]设计了一个与395~404位氨基酸残基相对应的十肽, 其通过多肽载体转导到HIV-1感染的细胞中, 表现出很高的抑制活性, 且没有毒副作用。此外, RT二聚体的成熟过程也是一个肽类抑制剂的新靶点。Morris等[35]合成了与HIV-1 RT p51亚基的拇指结构域中284~300位氨基酸残基相对应的小分子肽(Pep-A), 其能够与RT二聚体结合, 在不影响二聚体稳定性的前提下, 干扰RT的成熟过程。该类小分子肽类抑制剂与RT连接结构域中对应区域的氨基酸序列相互作用, 而这些氨基酸残基对酶的二聚体的形成是必需的, 因此, 这些小分子肽类能够抑制酶的二聚化, 从而抑制酶的活性。肽类抑制剂不良反应小, 具有较好的研究前景。
3.8.2 非肽类抑制剂TSAO-T衍生物 TSAO-T (叔丁基二甲基甲硅烷基螺氨基氧硫杂环己二烯−胸腺嘧啶, 30)衍生物代表了一类经典的HIV-1 RT抑制剂, 能特异性抑制HIV-1的复制, 对HIV-2无作用。TSAO-T与HIV-1 RT的结合模式类似于中国古代“龙”的形象, 其晶体结构表明(图 12), TSAO-T结合进入NNIBP后使口袋过度膨胀并与大部分氨基酸产生相互作用, 引起p51亚基的β7−β8 loop重排, 重排后很可能通过影响p66/p51二聚化过程产生抑制作用。过去几年, 人们合成了一系列新的TSAO-T类似物。其中4-、5-和6-取代嘧啶类似物的活性和选择性比未被取代的化合物高。在4-或5-取代的1, 2, 3-三唑TSAO类似物中, 5位被氨基、甲胺基或二甲胺基(31)取代的1, 2, 3-三唑TSAO类似物具有很好的抑制HIV-1活性[40]。

|
Figure 12 The crystal structure of TSAO-T (PDB code: 3QO9) |
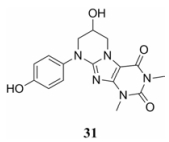
MAS0衍生物 Botta等[41, 42]运用虚拟筛选的方法发现了对聚合酶和RNase H都有抑制作用的化合物MAS0, 显著抑制p51和p66亚基的聚合过程。模拟计算显示(图 13), MAS0的中心稠合环与K395和E399产生极性相互作用; 苯酚结构能够与W402和W410产生π-π堆积作用, 同时与T409形成氢键作用; 体外实验表明该化合物对二聚化的抑制具有剂量依赖性。对MAS0进一步优化筛选得到四氢嘧啶并[2, 1-f]嘌呤二酮可以作为新骨架开发具有新机制及优良抗耐药性的HIV-1 RT抑制剂。
|
Figure 13 Chemical structure of MAS0 |
此外, 前文所述的具有RNase H和DNA聚合酶双重抑制活性的BBNH (24)和BBSH (25)也能通过降低RT二聚体的稳定性发挥抑制作用[32]。
目前, 二聚化抑制剂的研究正处于早期阶段, 但前景非常广阔。如TSAO-T、MAS0, 可以作为先导物, 继续研究新型高效低毒的抗艾滋病药物。顺便指出的是, HIV-1整合酶和蛋白酶均为二聚体, 抑制二聚体的形成也是抗艾滋病研究的新靶点。
3.9 新型双功能非核苷类逆转录酶抑制剂Lai等[43]报道了一系列同时作用于NRTI结合位点和NNIBP的非核苷类化合物。该类双功能小分子最初由Merck公司筛选化合物库发现, 其中化合物32 (图 14)与RT的结合依赖于Mg2+, 对常见的NNRTI突变株(K103N, IC50= 2.5 ± 0.2μmol·L−1; Y181C, IC50 = 0.79 ± 0.14μmol·L−1和K103N/Y181C, IC50 = 0.75 ± 0.29μmol·L−1)表现出了相当或优于野生株(IC50 = 8.5 ± 1.7μmol·L−1)的活性, 而对于rilpivirine和efavirenz常见的三突变株(G190A/Y181C/K103N, IC50 = 9.1μmol·L−1)也表现出了与野生株相当的活性。对31进行深入的构效关系探讨发现了与RT作用无需依赖Mg2+的化合物33。X-射线晶体结构表明, 33能同时作用于催化活性位点的D186, 高度保守的“引物沟”区域的L234和W229及NNIBP中的Y188。该发现揭示了一类新型的抗耐药突变的RT双功能抑制剂, 为克服耐药性的药物研发提供了新的突破点。

|
Figure 14 Chemical structures of bifunctional HIV-1 reverse transcriptase inhibitors |
除上面所述的几种靶向于HIV-1 RT聚合酶功能域的抑制剂, 仍有许多新的结合位点亟待开发, 如“关节”位点、核苷酸进入结合位点、NNRTI相邻位点等[44]。
“关节”位点位于p66手指和手掌亚结构域的连接位点, 毗邻聚合酶活性位点, 该位点中的氨基酸大部分属于保守型氨基酸, 不易发生突变[44]; 核苷酸进入结合位点也位于聚合酶活性位点附近, 由高度保守性氨基酸Y115、F116和E151组成, 不易发生突变, 而且, 小分子抑制剂与口袋结合后直接干扰dNTP的结合, 这两者优点赋予了该位点抑制剂研发以重要意义[44]; NNRTI相邻位点位于p66/p51交界处, 小分子抑制剂通过与口袋周围的氨基酸形成氢键相互作用扭曲YMDD基序的结构发挥抑制作用。值得一提的是, 该口袋与NNIBP距离较近, 可以用来设计包含NNRTI疏水口袋及相邻位点的双位点抑制剂, 以达到提高亲和力及改善耐药性的目的[44]。
此外, 从天然药物及海洋药物中分离得到的具有抗HIV-1 RT活性的化学成分(例如calanolide A及其结构类似物、polycitone A抑制剂、HPH类抑制剂、PAT类抑制剂)的药物也有发展, 这些天然成分具有专属性强、不良反应少、方便有效等优点[45−47], 因此, 从药用植物与海洋生物等天然药物中寻找抗HIV-1 RT活性成分的研究受到越来越多的重视, 现已成为抗HIV-1药物研究的新方向之一。
4 结语多年来, RT因其在HIV-1复制过程中的重要作用被科学家广泛关注。目前研究较为成熟的NRTI和NNRTI已有多个抑制剂上市, 但这两类抑制剂存在的一系列不良反应使得发现具有新型作用机制的抑制剂迫在眉睫。对新机制抑制剂的研究, 将为RT抑制剂的研发提供新的思路:研究者要不断探索RT的新结合位点, 更深入、更全面地了解RT的功能。针对这些结合位点, 运用计算机辅助药物设计技术和药物化学新策略, 开展先导化合物的发现与优化, 为抗艾滋病药物的发现提供物质基础。
| [1] | Liu H, Zhan P, Liu XY. Research progress of dual inhibitors targeting HIV-1 reverse transcriptase and integrase[J]. Acta Pharm Sin (药学学报), 2013, 48: 466–476. |
| [2] | London RE. Structural maturation of HIV-1 reverse transcriptase-a metamorphic solution to genomic instability[J]. Viruses, 2016, 8: 260–294. DOI:10.3390/v8100260 |
| [3] | Sarafianos SG, Clark AD, Das K, et al. Structures of HIV-1 reverse transcriptase with pre-and post-translocation AZTMPterminated DNA[J]. EMBO J, 2002, 21: 6614–6624. DOI:10.1093/emboj/cdf637 |
| [4] | Sarafianos SG, Das K, Hughes SH, et al. Taking aim at a moving target:designing drugs to inhibit drug-resistant HIV-1 reverse transcriptases[J]. Curr Opin Struct Biol, 2004, 14: 716–730. DOI:10.1016/j.sbi.2004.10.013 |
| [5] | Esposito F, Corona A, Tramontano E. HIV-1 reverse transcriptase still remains a new drug target:structure, function, classical inhibitors, and new inhibitors with innovative mechanisms of actions[J]. Mol Biol Int, 2012, 2012: 586401. |
| [6] | Sarafianos SG, Clark AD Jr, Tuske S, et al. Trapping HIV-1 reverse transcriptase before and after translocation on DNA[J]. J Biol Chem, 2003, 278: 16280–16288. DOI:10.1074/jbc.M212911200 |
| [7] | Zhang XQ. The newest developments of the study on antiHIV drugs[J]. Acta Pharm Sin (药学学报), 2015, 50: 509–515. |
| [8] | Wang L, Zhan P, Liu XY. The application of structural optimization strategies in drug design of HIV NNRTIs[J]. Acta Pharm Sin (药学学报), 2012, 47: 1409–1422. |
| [9] | James CA, Deroy P, Duplessis M, et al. Nucleotide competing reverse transcriptase inhibitors:discovery of a series of non-basic benzofurano[3, 2-d]pyrimidin-2-one derived inhibitors[J]. Bioorg Med Chem Lett, 2013, 23: 2781–2786. DOI:10.1016/j.bmcl.2013.02.021 |
| [10] | Geitmann M, Unge T, Danielson UH. Biosensor-based kinetic characterization of the interaction between HIV-1 reverse transcriptase and non-nucleoside inhibitors[J]. J Med Chem, 2006, 49: 2367–2374. DOI:10.1021/jm0504048 |
| [11] | Maga G, Radi M, Gerard MA, et al. HIV-1 RT inhibitors with a novel mechanism of action:NNRTIs that compete with the nucleotide substrate[J]. Viruses, 2010, 2: 880–899. DOI:10.3390/v2040880 |
| [12] | Kang D, Song Y, Chen W, et al. "Old Dogs with New Tricks":exploiting alternative mechanisms of action and new drug design strategies for clinically validated HIV targets[J]. Mol Biosyst, 2014, 10: 1998–2022. DOI:10.1039/C4MB00147H |
| [13] | Marcelin AG, Delaugerre C, Wirden M, et al. Thymidine analogue reverse transcriptase inhibitors resistance mutations profiles and association to other nucleoside reverse transcriptase inhibitors resistance mutations observed in the context of virological failure[J]. J Med Virol, 2004, 72: 162–165. DOI:10.1002/(ISSN)1096-9071 |
| [14] | Menéndez-Arias L. Mutation rates and intrinsic fidelity of retrovial reverse transcriptases[J]. Viruses, 2009, 1: 1137–1165. DOI:10.3390/v1031137 |
| [15] | Elinder M, Nordström H, Geitmann M, et al. Screening for NNRTIs with slow dissociation and high affinity for a panel of HIV-1 RT variants[J]. J Biomol Screen, 2009, 14: 395–403. DOI:10.1177/1087057109333977 |
| [16] | Ludovici DW, De Corte BL, Kukla MJ, et al. Evolution of anti-HIV drug candidates. Part 3:diarylpyrimidine (DAPY) analogues[J]. Bioorg Med Chem Lett, 2001, 11: 2235–2239. DOI:10.1016/S0960-894X(01)00412-7 |
| [17] | deBéthune MP. Non-nucleoside reverse transcriptase inhibitors (NNRTIs), their discovery, development, and use in the treatment of HIV-1 infection:a review of the last 20 years (1989-2009)[J]. Antiviral Res, 2010, 85: 75–90. DOI:10.1016/j.antiviral.2009.09.008 |
| [18] | Katlama C, Haubrich R, Lalezari J, et al. Efficacy and safety of etravirine in treatment-experienced, HIV-1 patients:pooled 48 week analysis of two randomized, controlled trials[J]. AIDS, 2009, 23: 2289–2300. DOI:10.1097/QAD.0b013e3283316a5e |
| [19] | Vingerhoets J, Buelens A, Peeters M, et al. Impact of baseline NNRTI mutations on the virological response to TMC125 in the phase Ⅲ clinical trials DUET-1 and DUET-2[J]. Antivir Ther, 2007, 12: 534. |
| [20] | Johnson VA, Brun-Vézinet F, Clotet B, et al. Update of the drug resistance mutations in HIV-1:2007[J]. Top HIV Med, 2007, 15: 119–125. |
| [21] | Freisz S, Bec G, Radi M, et al. Crystal structure of HIV-1 reverse transcriptase bound to a non-nucleoside inhibitor with a novel mechanism of action[J]. Angew Chem Int Ed Engl, 2010, 49: 1805–1808. DOI:10.1002/anie.v49:10 |
| [22] | Bellucci L, Angeli L, Tafi A, et al. Unconventional plasticity of HIV-1 reverse transcriptase:how inhibitors could open a connection "gate" between allosteric and catalytic sites[J]. J Chem Inf Model, 2013, 53: 3117–3122. DOI:10.1021/ci400414s |
| [23] | EI SY, Paillart JC, Laumond G, et al. 5-Modified-2'-dU and 2'-dC as mutagenic anti HIV-1 proliferation agents:synthesis and activity[J]. J Med Chem, 2010, 53: 1534–1545. DOI:10.1021/jm901758f |
| [24] | Harris KS, Brabant W, Styrchak S, et al. KP-1212/1461, a nucleoside designed for the treatment of HIV by viral mutagenesis[J]. Antiviral Res, 2005, 67: 1–9. DOI:10.1016/j.antiviral.2005.03.004 |
| [25] | Dapp MJ, Clouser CL, Patterson S, et al. 5-Azacytidine can induce lethal mutagenesis in human immunodeficiency virus type 1[J]. J Virol, 2009, 83: 11950–11958. DOI:10.1128/JVI.01406-09 |
| [26] | Wang LZ, Kenyon GL, Johnson KA. Novel mechanism of inhibition of HIV-1 reverse transcriptase by a new nonnucleoside analog, KM-1[J]. J Biol Chem, 2004, 279: 38424–38432. DOI:10.1074/jbc.M406241200 |
| [27] | Yamazaki S, Tan L, Mayer G, et al. Aptamer displacement identifies alternative small-molecule target sites that escape viral resistance[J]. Chem Biol, 2007, 14: 804–812. DOI:10.1016/j.chembiol.2007.06.003 |
| [28] | Herschhorn A, Oz-Gleenberg I, Hizi A. Mechanism of inhibition of HIV-1 reverse transcriptase by the novel broadrange DNA polymerase inhibitor N-{2-[4-(aminosulfonyl)phenyl] ethyl}-2-(2-thienyl)acetamide[J]. Biochemistry, 2008, 47: 490–502. DOI:10.1021/bi7018139 |
| [29] | Salie ZL, Kirby KA, Michailidis E, et al. Structural basis of HIV inhibition by translocation-defective RT inhibitor 4'-ethynyl-2-fluoro-2'-deoxyadenosine (EFdA)[J]. Proc Natl Acad Sci U S A, 2016, 113: 9274–9279. DOI:10.1073/pnas.1605223113 |
| [30] | Cihlar T, Ray AS. Nucleoside and nucleotide HIV reverse transcriptase inhibitors:25 years after zidovudine[J]. Antiviral Res, 2010, 85: 39–58. DOI:10.1016/j.antiviral.2009.09.014 |
| [31] | Himmel DM, Sarafianos SG, Dharmasena S, et al. HIV-1 reverse transcriptase structure with RNase H inhibitor dihydroxy benzoyl naphthylhydrazone bound at a novel site[J]. ACS Chem Biol, 2011, 1: 702–712. |
| [32] | Sluis-Cremer N, Arion D, Parniak MA. Destabilization of the HIV-1 reverse transcriptase dimer upon interaction with N-acyl hydrazoneinhibitors[J]. Mol Pharmacol, 2002, 62: 398–405. DOI:10.1124/mol.62.2.398 |
| [33] | Esposito F, Kharlamova T, Distinto S, et al. Alizarine derivatives as new dual inhibitors of the HIV-1 reverse transcriptase-associated DNA polymerase and RNase H activities effective also on the RNase H activity of non-nucleoside resistant reverse transcriptases[J]. FEBS J, 2011, 278: 1444–1457. DOI:10.1111/j.1742-4658.2011.08057.x |
| [34] | Distinto S, Esposito F, Kirchmair J, et al. Identification of HIV-1 reverse transcriptase dual inhibitors by a combined shape-, 2D-fingerprint-and pharmacophore-based virtual screening approach[J]. Eur J Med Chem, 2012, 50: 216–229. DOI:10.1016/j.ejmech.2012.01.056 |
| [35] | Morris MC, Berducou C, Mery J, et al. The thumb domain of the P51-subunit is essential for activation of HIV reverse transcriptase[J]. Biochemistry, 1999, 38: 15097–15103. DOI:10.1021/bi9914558 |
| [36] | Pata JD, Stirtan WG, Goldstein SW, et al. Structure of HIV-1 reverse transcriptase bound to an inhibitor active against mutant reverse transcriptases resistant to other nonnucleoside inhibitors[J]. Proc Natl Acad Sci U S A, 2004, 101: 10548–10553. DOI:10.1073/pnas.0404151101 |
| [37] | Depollier J, Hourdou ML, Aldrian-Herrada G, et al. Insight into the mechanism of a peptide inhibitor of HIV reverse transcriptase dimerization[J]. Biochemistry, 2005, 44: 1909–1918. DOI:10.1021/bi0484264 |
| [38] | Burke J. The amino acid Asn136 in HIV-1 reverse transcriptase (RT) maintains efficient association of both RT subunits and enables the rational design of novel RT inhibitors[J]. Mol Pharmacol, 2005, 68: 49–60. |
| [39] | Divita G, Restle T, Goody RS, et al. Inhibition of human immunodeficiency virus type 1 reverse transcriptase dimerization using synthetic peptides derived from the connection domain[J]. J Biol Chem, 1994, 269: 13080–13083. |
| [40] | Das K, Bauman JD, Rim AS, et al. Crystal structure of tertbutyldimethylsilyl-spiroaminooxathioledioxide-thymine (TSAO-T) in complex with HIV-1 reverse transcriptase (RT) redefines the elastic limits of the non-nucleoside inhibitor-binding pocket[J]. J Med Chem, 2011, 54: 2727–2737. DOI:10.1021/jm101536x |
| [41] | Tintori C, Corona A, Esposito F, et al. Inhibition of HIV-1 reverse transcriptase dimerization by small molecules[J]. ChemBioChem, 2016, 17: 683–688. DOI:10.1002/cbic.201500668 |
| [42] | Grohmann D, Corradi V, Elbasyouny M, et al. Small molecule inhibitors targeting HIV-1 reverse transcriptase dimerization[J]. ChemBioChem, 2008, 9: 916–922. DOI:10.1002/(ISSN)1439-7633 |
| [43] | Lai MT, Tawa P, Auger A, et al. Identification of novel bifunctional HIV-1 reverse transcriptase inhibitors[J]. J Antimicrob Chemother, 2018, 73: 109–117. DOI:10.1093/jac/dkx332 |
| [44] | Bauman JD, Patel D, Dharia C, et al. Detecting allosteric sites of HIV-1 reverse transcriptase by X-ray crystallographic fragment screening[J]. J Med Chem, 2013, 56: 2738–2746. DOI:10.1021/jm301271j |
| [45] | Fang YU, Liu XY. The structure and functions of HIV-1 polymerase and the development of its inhibitors[J]. Chin J Med Chem (中国药物化学杂志), 2006, 16: 380–386. |
| [46] | Yang Y, Cao YL, Liu HY, et al. Shizukaol F:a new structural type inhibitor of HIV-1 reverse transcriptase RNase H[J]. Acta Pharm Sin (药学学报), 2012, 47: 1011–1016. |
| [47] | Peng ZG, Chen HS, Wang L, et al. Anti-HIV activities of HIV-1 reverse transcriptase inhibitor racemic 11-demethylcalanolide A[J]. Acta Pharm Sin (药学学报), 2008, 43: 456–460. |
 2018, Vol. 53
2018, Vol. 53


