黄酮类化合物是植物体内广泛存在的次生代谢产物, 作为植物天然的“保护衣”, 具有抵御紫外线照射、细菌和真菌侵染[1]、昆虫和食草动物侵害[2-4]等作用。与此同时, 黄酮化合物是许多药用植物的主要药效成分, 在医药健康领域起着重要的作用, 是许多天然药物的重要来源[5]。由于其重要的食用药用价值, 多年来大量研究聚焦于黄酮化合物的合成[6, 7]。在模式植物中对于黄酮化合物合成途径已形成了一个初步框架, 但其中仍有许多问题待研究, 尤其是其他药用植物具有与模式植物不同种类的黄酮, 其合成途径的研究面临许多挑战。
红花是一种重要的传统中药, 具有活血化瘀、消肿止痛的功效[8, 9]。主要成分为羟基红花黄色素A、山柰酚及其苷类、槲皮素及其苷类等黄酮类化合物, 其含量的高低影响了红花药材品质的优劣。为了阐明红花黄酮的生物合成途径, 不少学者已对红花中黄酮合成途径相关酶的特征进行了研究, 如肉桂酸-4-羟化酶(C4H)[10]、油酰磷脂酰胆碱脱氢酶(FAD2)[11], 苯丙氨酸解氨酶(PAL)[12]、查尔酮合酶(CHS)[13]、黄烷酮3-羟化酶(F3H)[14]、UDP糖基转移酶(UGT)[15]等。但到目前为止, 对红花中黄酮类化合物的生物合成途径仍然不清楚。
査尔酮合酶(CHS)是合成黄酮类化合物的关键酶, 也是其限速酶, 在不同植物中CHS催化产生不同种类的黄酮类化合物。自1983年第一个荷兰芹(Petroselinum crispum) CHS基因被报道后[16], 近百种植物CHS基因家族被陆续报道, 如矮牵牛(Petunia hybrida)[17]、豌豆(Pisum sativum)[18]、大豆(Glycine max)[19]、何首乌(Polygonum multiflorum)[20]、小立碗藓(Physcomitrella patens)[21, 22]、葡萄(Vitis vinifera)[23]、大丁草(Gerbera hybrida)[24]、百合(Lilium hybrida)[25]、拟南芥(Arabidopsis thaliana)[26]等。目前红花中已有4个CHS基因被报道[13, 27], 其中CtCHS1 (KY471385)具有促进红花品系中醌式查尔酮合成的作用。但对于CHS基因家族的其他成员在红花黄酮合成途径中的作用尚不明确。因此, 研究红花CHS基因家族的功能对于阐释红花黄酮类的生物合成途径具有重要意义。
JA/MeJA (茉莉酸甲酯)属于植物激素类物质, 具有调控种子发芽、根系生长、开花、果实成熟、衰老等作用[28-30], 并且作为植物信号调节物质可以在植物抵御昆虫、病害、应激UV损伤、植物育性、花青素募集时激活植物体内CHS基因表达[31]。在许多植物中应用JA/MeJA预处理或是后处理可以刺激植物次生代谢通路从而募集多酚类化合物, JA/MeJA预处理黑莓植株, 使黑莓黄酮类化合物大量募集, 提高了黑莓的抗氧化能力[32]; 在葡萄植物中, JA/MeJA通过提高白藜芦醇等花青素类化合物浓度来提高抗氧化能力[33]。课题组前期研究表明JA/MeJA刺激能提高F3H基因的转录水平, 具有促进红花黄酮类成分积累的作用[14]。通过JA/MeJA刺激研究相关基因水平与红花黄酮类成分积累的变化, 能够为探索相关基因在黄酮类成分生物合成中的作用提供依据。
本研究在前期阐明红花的1个CHS基因CtCHS1功能的基础上[13], 作为延续性工作, 继续对红花中的另外2个CHS基因CtCHS2和CtCHS4进行了研究, 以期为阐明红花中黄酮类化合物的生物合成途径积累资料。
材料与方法植物材料 云南巍山红花品系(编号: ZHH0119)来源于新疆农业科学院国家种质资源库并经课题组多代纯化, 由郭美丽教授鉴定为红花(Carthamus tinctorius L.)。材料种植于第二军医大学药学院温室。生长条件设定为25 ℃, 湿度50%~70%, 光照节律为16 h光照/8 h黑暗。精密吸取2.2 mL MeJA (Sigma-Aldrich, 美国, 纯度≥95%)加入1 mL无水乙醇(AR级, 国药化学试剂有限公司, 中国)中, 振摇, 再用水定容至100 mL。在顶球花开花的第一天, 花冠从总苞片中露出时, 以MeJA喷洒顶球花冠(对照组: 0.1%乙醇), 喷洒体积为1 mL, 并立即将200 mL透明塑料袋套在花球上防止MeJA挥发。于喷洒后0 (对照组)、3、6、12 h分别采集不带子房的花冠, 各时间点采集5个样品并立即放入液氮, 保存于-80 ℃。采集时每个花球花冠平分成两份, 一份放液氮保存, 供基因表达分析用, 另一份置50 ℃烘箱内烘干至恒重后用于次生成分的含量测定。
RNA提取及cDNA第一链的合成 按照植物小量RNA提取试剂盒(莱峰公司, 中国)说明书提取花冠总RNA。RNA浓度及纯度通过NanoDrop微量分光光度计和琼脂糖凝胶电泳检测, 保证A260/A280比值在1.9~2.1且A260/A230值大于2.0的RNA样品用于后续实验。并以此为模版使用TransScript® One-Step gDNA Removal and cDNA Synthesis SuperMix试剂盒(北京全式金公司, 中国)合成cDNA第一链, 保存于-20 ℃。
基因全长的获得与生物信息学分析 根据课题组前期得到的CtCHS2、CtCHS4 EST序列信息, 分别设计5'-cDNA片段和3'-cDNA片段的扩增引物(表 1), 使用SMARTerTM RACE cDNA Amplification Kit试剂盒(Clontech, 日本)进行PCR扩增。扩增片段胶回收分别连接载体pMD19-T (TaKaRa公司, 中国)送样于上海美吉生物医药科技有限公司测序。使用DNA MAN拼接测序结果, 并进行BLAST检索比对, 确定为目的基因后依据上述拼接结果, 设计包含完整开放阅读框序列相对应的全长引物, 克隆出目的基因全长(表 1)。
| Table 1 Primers designed for real-time PCR detection |
在NCBI网站上用BLASTX (http://blast.ncbi.nlm.nih.gov/Blast.cgi)对各基因全长序列及其编码蛋白的氨基酸序列进行序列比对及同源性分析; 用ExPASy在线工具SOPMA分析各基因编码蛋白质的二级结构情况; 用ClustalX 2.1软件对目的基因编码蛋白质的氨基酸序列与其同源蛋白的氨基酸序列进行多重比对分析; 并采用相邻节点(NJ, Neighbor-Joining)算法构建系统进化树, 进化树的可靠性采用自展分析法(Bootstrap analysis)进行1 000次重复, 多重比对和构建系统进化树。
qRT-PCR 将0、3、6、12 h采集的花冠第一链cDNA作为模版, 并设计特异性引物(表 1), 利用CFX96 Touch™ Real-Time PCR Detection System (Bio-Rad)和ABI 7500 Real-Time PCR System (Applied Biosystems)进行qPCR反应。以Ct60s (KJ634810)作为qPCR内参基因。
目的基因表达量= 2-ΔCt
Ct60s (60S酸性核糖体蛋白)目的基因的几何平均值RG(Ct), 依据ΔCt =目的基因-参考基因来计算。
黄酮类化合物积累测定 将烘干至恒重的红花花冠样品(0、3、6、12 h)分别精密称取4.0 mg, 置1.5 mL管内, 精密加入60%甲醇1 mL, 密闭浸泡12 h后超声处理40 min, 用0.22 µm微孔滤膜过滤, 采用液相色谱(Agilent 1290 Infinity UHPLC) -串联质谱(Agilent 6538 UHD、Accurate-Mass Q-TOF/MS)进行检测, 条件如表 2。
| Table 2 UPLC/Q-TOF/MS detection analysis conditions |
以山柰酚(Lot#G1324016, Aladdin, 美国)、D-苯丙氨酸(Lot#K1418043, Aladdin, 美国)、芦丁(Lot# B1413082, Aladdin, 美国)、山柰酚-3-O-β-D-葡萄糖苷(Lot#T18O7Z3002, 源叶生物, 中国)、羟基红花黄色素A (Lot#422A022SH8260, Solarbio, 中国)、红花红色素(自制, 编号: 131124, 经HPLC检测, 纯度≥98.0%)、木犀草素(Lot#K1413082, Aladdin, 美国)、槲皮-3-β-D-葡萄糖苷(Lot#B1413082, Aladdin, 美国)、山柰酚-3-O-β-芸香糖苷(自制, 编号: 131211, 经HPLC检测, 纯度≥99.7%)为对照品确定9个指标性成分的荷质比, 评价不同处理组指标化合物含量变化。
CHS重组蛋白诱导表达、纯化 通过无缝克隆试剂盒(Clontech, 日本), 将CtCHS4片段连入经双酶切反应的pMAL-C5X (NEB公司, 新西兰)线性化载体, 转化感受态细胞5α (北京全式金公司, 中国), 筛选阳性克隆。抽提连有目的基因的载体质粒pMAL-C5X-CtCHS4转化诱导菌BL21 (DE3) Plys (北京全式金公司, 中国), 以未连接目的基因的空载体质粒作为对照, 使用IPTG诱导蛋白表达[34]。将上述质粒于含100 mg·L-1氨苄青霉素的LB培养基中37 ℃、200 r·min-1培养至OD600为0.5~0.6, 加入IPTG (终浓度为0.4 mmol·L-1)后继续在37 ℃下孵育4 h后, 以5 000 r·min-1离心10 min收集菌液, 吸出上清液。使细胞重新悬浮在由20 mmol·L-1 Tris-HCl、200 mmol·L-1 NaCl和1 mmol·L-1 EDTA组成的溶液中, 以15 s的脉冲进行机械破碎超声处理。13 000 r·min-1离心20 min, 取上清液SDS-PAGE分析。
蛋白纯化:将淀粉链树脂5 mL通入2.5×10 cm的亲和色谱柱, 以10倍柱体积的缓冲液洗脱, 去除杂质。加入蛋白粗提物10 mL, 以20倍柱体积的缓冲液洗脱。再以10 nmol·L-1的麦芽糖溶液4 mL洗脱可溶性的蛋白, 每毫升收集一次洗脱液, 对洗脱液进行SDS-PAGE分析。采用改良型Bradford法蛋白质浓度测定试剂盒测定可溶性蛋白的浓度。
原核表达蛋白酶促反应 以4-香豆酰辅酶A、丙二酰辅酶A为酶催化底物, 制备方法参考文献[35], 酶促反应的反应体系配置如下: CtCHS4 (10 µg)、p-香豆酰辅酶A (10 nmol)、丙二酰辅酶A (20 nmol), DTT (2 mmol·L-1)调节体系为磷酸盐溶液(pH 7.5) 500 μL, 反应1 h后加入冰醋酸25 µL, 在反应结束时调节pH = 9, 反应10 min。后加入乙酸乙酯500 µL, 减压蒸干后, 加入甲醇250 µL、水250 μL溶解, 上清进行HPLC检测, 条件为反向色谱柱C18, 流动相50%甲醇加入1%甲酸, 流速1 mL·min-1, 289 nm下收集数据, 应用标准品作为对照。
统计分析 实验结果经统计学处理, 计量资料用mean ± SD表示, 各组数据采用ANOVA、Dunnett-t test进行两组间比较。
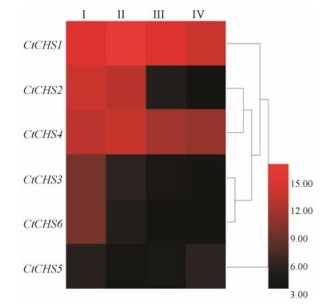
结果 1 CHS基因全长的克隆及生物信息学分析为了研究红花的功能基因, 课题组前期对红花花冠进行了转录组测序和功能注释。依据基因测序及注释信息, 获得6条红花CHS基因, 并制定了红花Agilent 8×15K原位合成芯片[13]。采用HemI软件分析6条CHS基因在不同开花时期(开花前7天、开花前3天, 开花当天和开花第3天)基因相对表达量, 结果显示: CtCHS2、CtCHS4在不同开花期的表达模式与CtCHS1相似(图 1), 提示CtCHS4、CtCHS2可能也参与了红花中醌式查尔酮的生物合成。
|
图 1 Expression pattern of CtCHS at flower different developmental stage. Signal values by gene chip, stage I as control group. The color key was set from 3 to 15 |
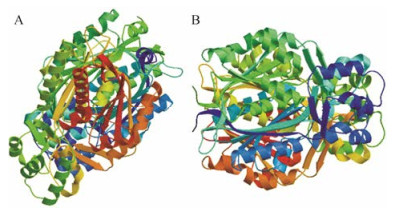
采用RACE技术, 克隆出CtCHS4、CtCHS2基因全长序列为1 222和1 260 bp。ORF FINDER查找CtCHS4、CtCHS2基因ORF并进行翻译结果显示: CtCHS4 ORF 9~1 214 bp, 编码401个氨基酸; CtCHS2 ORF为69~1 253 bp, 编码394氨基酸。将全长序列进行blastx搜索, 结果显示CtCHS4氨基酸序列与菊花(Chryscanthemum boreale) CHS氨基酸序列(AGU91424.1)的同源性最高, 覆盖度达94%, 匹配度达到91%。CtCHS2氨基酸序列与毛果杨(Populus trichocarpa) CHS氨基酸序列(XP006375238.1)的同源性最高, 覆盖度达93%, 匹配度达到82%。Protparam预测CtCHS4编码蛋白质分子质量为43 667.25 Da, 预测理论等电点为5.72, 蛋白质三维结构预测见图 2A。CtCHS2编码的蛋白质分子质量为43 016.36 Da, 预测理论等电点为5.67, 蛋白质三维结构预测见图 2B。
|
Figure 2 The three-dimensional structures of the deduced CtCHS4 and CtCHS2. A: CtCHS4, B: CtCHS2 |
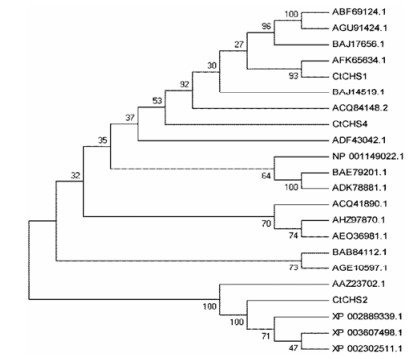
CtCHS2、CtCHS4和CtCHS1核苷酸序列比对显示CtCHS1和CtCHS2基因序列相似性较高(图 3)。系统进化树图谱分析(图 4, 表 3), CtCHS4和CtCHS1在聚类分支上亲缘关系较近, 而与CtCHS2在聚类分支上距离较远, 证明CtCHS4和CtCHS1氨基酸活性有一定的相似性, 而CtCHS4与CtCHS2在氨基酸活性催化功能上存在较大的差异性。

|
Figure 3 Nucleotide sequence alignment of CtCHS4, CtCHS2 and CtCHS1 |
|
Figure 4 Unrooted phylogram comparison of the amino acid sequences of CtCHS2, 4 and other functionally characterized CHS proteins. The phylogenetic tree was constructed by MEGA 5.0, after alignment using Clustal X software. Node support was estimated using neighbor-joining bootstrap analysis (1 000 bootstrap replicates) |
| Table 3 GenBank accession numbers amino acid sequences of CHSs used for construction of the phylogenetic tree |
CHS属于Ⅲ型PKS聚类家族, 具有很强的保守性, 编码长约1.2 kb, 一般由一个内含子间隔, 外显子1约编码57~64个氨基酸, 而外显子2编码约340个氨基酸, 且更为保守。一般科之间的氨基酸同源性在70%~90%, 各种氨基酸在分支上的分布略有不同, 对于大部分科的序列分布形成很近的分支, 只有很少的科的序列分布在相聚很远的分支中。本研究从红花中克隆出的基因CtCHS2和CtCHS4, 从系统发育树来看, 分布较远, 属于少数的相聚在很远分支上的基因。
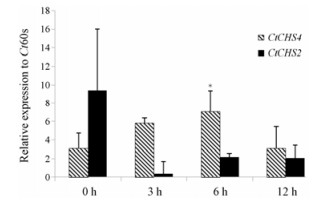
2 MeJA处理刺激了查尔酮合酶基因的表达qPCR分析显示CtCHS2和CtCHS4基因响应MeJA诱导的模式不同, CtCHS4基因的表达量在MeJA诱导后的3和6 h明显升高, 特别在诱导后6 h, 表达量提高了2.3 (P < 0.05), 诱导后12 h基因的表达量则没有明显变化; CtCHS2基因的表达量在诱导后3、6、12 h具有降低的特征(P = 0.07), 见图 5。提示CtCHS2和CtCHS4基因对于MeJA刺激的响应模式不同, 可能在红花黄酮类生物合成途径中的作用不同。
|
Figure 5 CtCHS2 and CtCHS4 relative expression to Ct60s in safflower. *P < 0.05 vs 0 h group |
UPLC-QTOF/MS检测MeJA处理后不同时间点(0、3、6、12 h)红花花冠9个黄酮类成分芦丁、羟基红花黄色素A、D-苯丙氨酸、山柰酚-3-O-β-芸香糖苷、红花红色素、山柰酚、山柰酚-3-O-β-D-葡萄糖苷、木犀草素、槲皮素-3-β-D-葡糖苷的含量积累水平(图 6), 从含量变化折线图可以看出, 9个黄酮成分均受到MeJA的诱导, 且种类和含量积累模式存在一定不同。MeJA刺激诱导了羟基红花黄色素A、D-苯丙氨酸、山柰酚-3-O-β-芸香糖苷、红花红色素积累, 尤其是刺激后6、12 h积累量具有显著提高, 对山柰酚、山柰酚-3-O-β-D-葡萄糖苷、木犀草素、槲皮素-3-β-D-葡萄糖苷的积累则影响不明显。
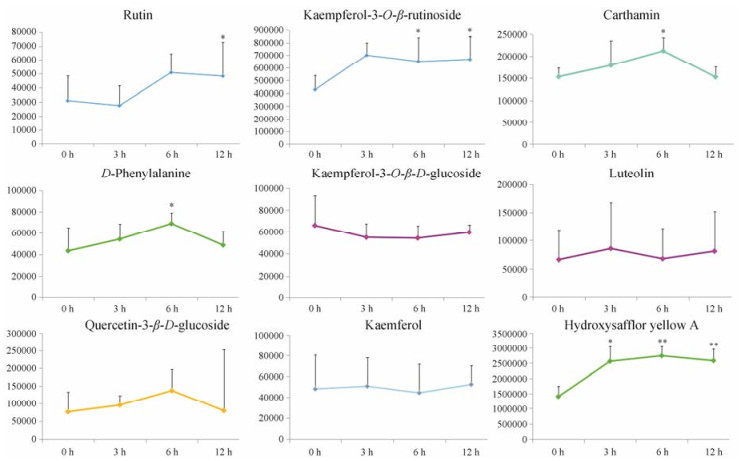
|
Figure 6 Flavonoids content accumulation in safflower. *P < 0.05, **P < 0.01 vs blank group (0 h) |
根据CtCHS2和CtCHS4基因响应MeJA诱导不同时间点(0、3、6、12 h)的表达量(图 5), 计算黄酮类生物合成途径2个CHS的表达与黄酮类化合物积累量的Pearson相关系数r, 可以看出羟基红花黄色素A、D-苯丙氨酸、红花红色素的积累量与CtCHS4的表达均成正相关(表 4)。提示CtCHS4在红花黄酮类成分的积累中起着重要作用, CtCHS4可能是形成羟基红花黄色素A、红花红色素的关键基因, 在醌式查尔酮的合成途径中具有重要作用。
| 表 4 The correlation coefficients between CHSs and flavon oids |
将CtCHS4与pMAL-C5X载体连接, 构建成功的重组质粒导入到诱导表达BL21 (DE3) Plys中进行菌液培养, 应用PCR方法进行阳性菌株鉴定。挑取CtCHS4阳性菌株进行IPTG诱导, 同时应用导入空载体的菌液作为对照诱导, 经SDS-PAGE检测, 发现导入目的基因的阳性菌株CtCHS4有特异性蛋白表达(图 7)。
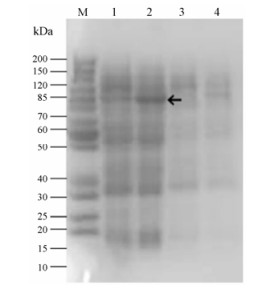
|
Figure 7 CtCHS4 induced expression of target protein SDS-PAGE detection. M: Marker, 1: IPTG induced after precipitation, 2: Induced fusion protein in the supernatant, 3: Without induced by IPTG in the precipitation, 4: Without induced by IPTG in the suspernatant |
针对CtCHS4的融合蛋白进行纯化(图 8)、酶切浓缩处理, 最终从400 mL的菌液中纯化得到约70 µg的可溶性蛋白, 应用4-香豆酰辅酶A和丙二酰辅酶A为底物, 对得到的蛋白进行酶活性检测, 加入CHS蛋白后经过处理除生成含有底物的峰形外, 在10~11 min左右生成了一个新的单峰, 经相同色谱条件应用柚皮素为对照品比较, 鉴定为柚皮素(图 9)。
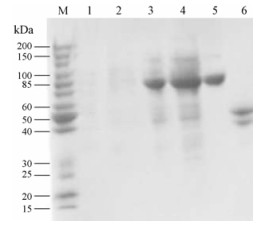
|
Figure 8 SDS-PAGE analysis of purified CtCHS4. M: Marker, 1, 2, 3, 4: pMAL-C5x-CtCHS4 protein purification process, 5: Purified pMAL-C5x-CtCHS4 protein, 6: pMAL-C5x control vector |
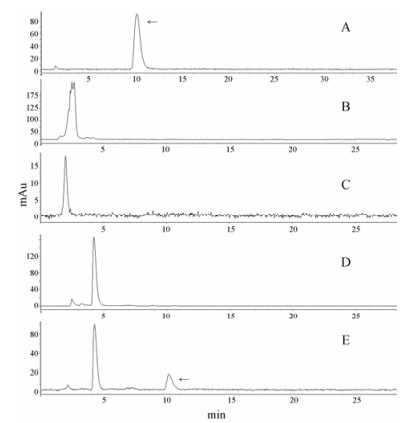
|
Figure 9 Detection of the catalysate by CtCHS4 by HPLC. A: Naringenin standard; B: p-Coumaryol-CoA standard; C: Malony-CoA standard; D: The HPLC pigment of malony-CoA and p-coumaryol-CoA under the condition of 50% methanol (1% formic acid) without CHS; E: The HPLC pigment of naringenin from CHS catalytic substrate p-coumaryol-CoA and malonyl-CoA |
红花 Carthamus tinctorius L.是著名的药用植物, 其花中含有的查尔酮类成分和多种黄酮醇化合物, 如羟基红花黄色素A、槲皮素及其苷类、山柰酚及其苷类等是红花发挥活血化瘀活性的重要物质基础[36]。CHS等作为黄酮类生物代谢关键酶基因对黄酮类化合物合成起到关键性作用[37]。但目前对于红花中黄酮化合物生物合成途径的研究还十分浅显, 尤其是查尔酮类化合物的生物合成, 由各种内源及外源性因素诱导, 且受到大量的转录因子、结构酶基因的共同调控, 但对其分子机制知之甚少, 至今未形成突破[38-40]。由主通道基因入手, 逐步深入探索其生物合成途径的关键基因, 将为最终阐明红花品质形成的分子机制提供重要科学依据。
在植物体中CHS作为植物聚酮类化合物代谢的入口酶, 催化产生黄酮类化合物以参与植物的UV防护、植物生殖器官发育、抗病原体侵染等[41]。对于CHS基因家族的调控和功能在许多植物中已经进行了研究, 大多数植物含有一个较小的CHS基因家族, 如豌豆中有8个CHS基因被报道[42]、番薯中发现了6个CHS基因[18]、矮牵牛中有8个完整的和4个部分被测序的CHS基因[17]。本研究中克隆了2个CHS基因, 与课题组前期报道的CtCHS1以及Shinozak等[27]报道的3个CHS基因组成一个小的CHS基因家族, 这与之前报道的其他植物中CHS基因家族情况吻合。此外, 红花CHS基因与其他植物CHS基因序列比对显示出高度相似性, 表明CHS基因在不同物种中保守度高。
Shinozaki等[27]报道了3个CtCHS基因在体外诱导生成柚皮素的酶活性, 说明其在黄酮合成途径中具有一定的作用。课题组前期对红花中CtCHS1过表达, 提高了红花中重要药效成分羟基红花黄色素A含量的积累, 证明了其在醌式查尔酮合成途径中具有重要作用, 同时在过表达CtCHS1植株中, CtCHS4也表现出转录水平显著提高, 提示其可能在羟基红花黄色素A途径中也具有调控作用[13]。结合基因芯片分析结果, 本研究使用MeJA刺激的方法, 探索CtCHS4、CtCHS2与黄酮类成分积累模式的关系, 证明了CtCHS4的表达量与醌式查尔酮类成分羟基红花黄色素A、红花红色素的积累量存在显著性正相关(r≥0.8), 提示CtCHS4具有与先前报道的CtCHS1相似的功能, 可能参与了醌式查尔酮类成分的生物合成。
CHS基因编码的多基因家族中的每个个体都可以被不同的调控方式所调控, 如组织特异性表达, 由光、诱导处理或病原体侵染而诱导的转录, 及由其他调节基因控制的转录[43, 44]。且细胞内的CHS基因通常处于静止状态, 很难被检测到, 只有在UV或是生物诱导因子作用下在转录水平上被激活[45], 从而行使其催化黄酮类化合物的功能。本课题组前期研究证明了红花CtCHS1在醌式查尔酮类合成途径中的作用[13]。本研究对另外2条查尔酮合酶基因(CtCHS2、CtCHS4)的功能进行研究的结果表明, 2个基因对MeJA刺激具有不同的反应, 而且可能在MeJA刺激红花黄酮类生物合成途径中具有不同的作用, 其中CtCHS4可能是形成羟基红花黄色素A和红花红色素的关键基因, 在红花醌式查尔酮的合成途径中具有重要作用, 而CtCHS2的作用则不明显。
在黄芩(Scutellaria viscidula)[46]、大麻(Cannabis sativa)[47]等植物中已证实CHS以香豆酰辅酶A作为起始底物使中间分子C6和C1位置进行缩合生成查尔酮。同时在酸性条件下, 可以使查尔酮进行闭环反应生成柚皮素。Shinozaki等[27]已报道了3个在体外查尔酮合酶活性诱导香豆酰辅酶A生成柚皮素的CHS基因。本研究在体外构建酶促反应体系验证CtCHS4所编码酶的活性, 用香豆酰辅酶A和丙二酰辅酶A为底物, 诱导产物鉴定为柚皮素, 证实了红花CtCHS4的蛋白活性, 也为进一步研究红花黄酮生物合成途径酶的功能提供了线索。本研究结果为阐明红花品质形成的分子机制、定向调控红花的品质提供了部分依据。
| [1] | Middleton EJ, Kandaswami C, Theoharides TC. The effects of plant flavonoids on mammalian cells:implications for inflammation, heart disease, and cancer[J]. Pharmacol Rev, 2000, 52: 673–751. |
| [2] | El-Wakeil NE. Botanical pesticides and their mode of action[J]. Gesunde Pflanze, 2013, 65: 24. |
| [3] | Ortu o A, Báidez A, Gómez P, et al. Citrus paradisi and Citrus sinensis flavonoids:their influence in the defence mechanism against Penicillium digitatum[J]. Food Chem, 2006, 98: 8. |
| [4] | Steinkellner S, Lendzemo V, Langer I, et al. Flavonoids and strigolactones in root exudates as signals in symbiotic and pathogenic plant-fungus interactions[J]. Molecules, 2007, 12: 1290–1306. DOI:10.3390/12071290 |
| [5] | Sankari SL, Babu NA, Rani V, et al. Flavonoids-clinical effects and applications in dentistry:a review[J]. J Pharm Bioallied Sci, 2014, 6: S26–S29. DOI:10.4103/0975-7406.137256 |
| [6] | Giannasi DE. Systematic aspects of flavonoid biosynthesis and evolution[J]. Bot Rev, 1978, 44: 399–429. DOI:10.1007/BF02860845 |
| [7] | Gutha LR, Casassa LF, Harbertson JF, et al. Modulation of flavonoid biosynthetic pathway genes and anthocyanins due to virus infection in grapevine (Vitis vinifera L.) leaves[J]. BMC Plant Biol, 2010, 10: 187. DOI:10.1186/1471-2229-10-187 |
| [8] | Wang W, Jin M, Tong J, et al. Inhibitory effect of hydroxy-safflor yellow A against PMN activation induced by LPS[J]. Acta Pharm Sin (药学学报), 2011, 46: 153–157. |
| [9] | Wang XF, Jin M, Tong J, et al. Protective effect of hydroxy-safflor yellow A against acute lung injury induced by oleic acid and lipopolysaccharide in rats[J]. Acta Pharm Sin (药学学报), 2010, 45: 940–944. |
| [10] | Sadeghi M, Dehghan S, Fischer R, et al. Isolation and characterization of isochorismate synthase and cinnamate 4-hydroxylase during salinity stress, wounding, and salicylic acid treatment in Carthamus tinctorius[J]. Plant Signal Behav, 2013, 8: e27335. DOI:10.4161/psb.27335 |
| [11] | Cao S, Zhou XR, Wood CC, et al. A large and functionally diverse family of Fad2 genes in safflower (Carthamus tinctorius L.)[J]. BMC Plant Biol, 2013, 13: 5. DOI:10.1186/1471-2229-13-5 |
| [12] | Dehghan S, Sadeghi M, Poppel A, et al. Differential inductions of phenylalanine ammonia-lyase and chalcone synthase during wounding, salicylic acid treatment, and salinity stress in safflower, Carthamus tinctorius[J]. Biosci Rep, 2014, 34: e00114. |
| [13] | Guo DD, Xue YR, Li D, et al. Overexpression of CtCHS1 increases accumulation of quinochalcone in safflower[J]. Front Plant Sci, 2017, 8: 1409. DOI:10.3389/fpls.2017.01409 |
| [14] | Tu YH, Liu F, Guo DD, et al. Molecular characterization of flavanone 3-hydroxylase gene and flavonoid accumulation in two chemotyped safflower lines in response to methyl jasmonate stimulation[J]. BMC Plant Biol, 2016, 16: 132. DOI:10.1186/s12870-016-0813-5 |
| [15] | Guo DD, Liu F, Tu YH, et al. Expression patterns of three UGT genes in different chemotype safflower lines and under MeJA stimulus revealed their potential role in flavonoid biosynthesis[J]. PLoS One, 2016, 11: e0158159. DOI:10.1371/journal.pone.0158159 |
| [16] | Reimold U, Kroger M, Kreuzaler F, et al. Coding and 3' non-coding nucleotide sequence of chalcone synthase mRNA and assignment of amino acid sequence of the enzyme[J]. EMBO J, 1983, 2: 1801–1805. |
| [17] | Koes RE, Spelt CE, van den Elzen PJ, et al. Cloning and molecular characterization of the chalcone synthase multigene family of Petunia hybrida[J]. Gene, 1989, 81: 245–257. DOI:10.1016/0378-1119(89)90185-6 |
| [18] | Durbin ML, McCaig B, Clegg MT. Molecular evolution of the chalcone synthase multigene family in the morning glory genome[J]. Plant Mol Biol, 2000, 42: 79–92. DOI:10.1023/A:1006375904820 |
| [19] | Akada S, Kung SD, Dube SK. Nucleotide sequence of a soybean chalcone synthase gene with a possible role in ultraviolet-B sensitivity, Gmchs6[J]. Plant Physiol, 1993, 102: 699–701. DOI:10.1104/pp.102.2.699 |
| [20] | Hai L. Cloning and sequence analysis of chalcone synthase gene in Polygonum multiflorum Thunb[J]. J Anhui Agric Sci (安徽农业科学), 2009, 15: 129. |
| [21] | Koduri PK, Gordon GS, Barker EI, et al. Genome-wide analysis of the chalcone synthase superfamily genes of Physcomitrella patens[J]. Plant Mol Biol, 2010, 72: 247–263. DOI:10.1007/s11103-009-9565-z |
| [22] | Jiang C, Schommer CK, Kim SY, et al. Cloning and charac-terization of chalcone synthase from the moss, Physcomitrella patens[J]. Phytochemistry, 2006, 67: 2531–2540. DOI:10.1016/j.phytochem.2006.09.030 |
| [23] | Sparvoli F, Martin C, Scienza A, et al. Cloning and molecular analysis of structural genes involved in flavonoid and stilbene biosynthesis in grape (Vitis vinifera L.)[J]. Plant Mol Biol, 1994, 24: 743–755. DOI:10.1007/BF00029856 |
| [24] | Helariutta Y, Elomaa P, Kotilainen M, et al. Chalcone synthase-like genes active during corolla development are differentially expressed and encode enzymes with different catalytic properties in Gerbera hybrida (Asteraceae)[J]. Plant Mol Biol, 1995, 28: 47–60. DOI:10.1007/BF00042037 |
| [25] | Liu YL, Lou Q, Xu WR, et al. Characterization of a chalcone synthase (CHS) flower-specific promoter from Lilium orential 'Sorbonne'[J]. Plant Cell Rep, 2011, 30: 2187–2194. DOI:10.1007/s00299-011-1124-9 |
| [26] | Wang WK, Schaal BA, Chiou YM, et al. Diverse selective modes among orthologs/paralogs of the chalcone synthase (Chs) gene family of Arabidopsis thaliana and its relative A-halleri ssp gemmifera[J]. Mol Phylogen Evol, 2007, 44: 503–520. DOI:10.1016/j.ympev.2007.05.006 |
| [27] | Shinozaki J, Kenmoku H, Nihei K, et al. Cloning and functional analysis of three chalcone synthases from the flowers of safflowers Carthamus tinctorius[J]. Nat Prod Commun, 2016, 11: 787–790. |
| [28] | Diallo AO, Agharbaoui Z, Badawi MA, et al. Transcriptome analysis of an mvp mutant reveals important changes in global gene expression and a role for methyl jasmonate in vernalization and flowering in wheat[J]. J Exp Bot, 2014, 65: 2271–2286. DOI:10.1093/jxb/eru102 |
| [29] | Staswick PE, Su W, Howell SH. Methyl jasmonate inhibition of root growth and induction of a leaf protein are decreased in an Arabidopsis thaliana mutant[J]. Proc Natl Acad Sci USA, 1992, 89: 6837–6840. DOI:10.1073/pnas.89.15.6837 |
| [30] | Concha CM, Figueroa NE, Poblete LA, et al. Methyl jasmonate treatment induces changes in fruit ripening by modifying the expression of several ripening genes in Fragaria chiloensis fruit[J]. Plant Physiol Biochem, 2013, 70: 433–444. DOI:10.1016/j.plaphy.2013.06.008 |
| [31] | Richard S, Lapointe G, Rutledge RG, et al. Induction of chalcone synthase expression in white spruce by wounding and jasmonate[J]. Plant Cell Physiol, 2000, 41: 982–987. DOI:10.1093/pcp/pcd017 |
| [32] | Wang SY, Bowman L, Ding M. Methyl jasmonate enhances antioxidant activity and flavonoid content in blackberries (Rubus sp.) and promotes antiproliferation of human cancer cells[J]. Food Chem, 2008, 107: 1261–1269. |
| [33] | Nopo-Olazabal C, Condori J, Nopo-Olazabal L, et al. Differential induction of antioxidant stilbenoids in hairy roots of Vitis rotundifolia treated with methyl jasmonate and hydrogen peroxide[J]. Plant Physiol Biochem, 2014, 74: 50–69. DOI:10.1016/j.plaphy.2013.10.035 |
| [34] | Tan JF, Tu LL, Deng FL, et al. A genetic and metabolic analysis revealed that cotton fiber cell development was retarded by flavonoid naringenin[J]. Plant Physiol, 2013, 162: 86–95. DOI:10.1104/pp.112.212142 |
| [35] | Beuerle T, Pichersky E. Enzymatic synthesis and purification of aromatic coenzyme a esters[J]. Anal Biochem, 2002, 302: 305–312. DOI:10.1006/abio.2001.5574 |
| [36] | Tu YH, Xue YR, Guo DD, et al. Carthami flos:a review of its ethnopharmacology, pharmacology and clinical applications[J]. Rev Bras Farmacogn, 2015, 25: 553–566. DOI:10.1016/j.bjp.2015.06.001 |
| [37] | Wu YY, Chen CP, Ren CX, et al. Correlation between accumulation of flavoneand expression of functional genes in Carthamus tinctorius[J]. China J Chin Mater Med (中国中药杂志), 2017, 42: 83–87. |
| [38] | Lewis DR, Ramirez MV, Miller ND, et al. Auxin and ethylene induce flavonol accumulation through distinct transcriptional networks[J]. Plant Physiol, 2011, 156: 144–164. DOI:10.1104/pp.111.172502 |
| [39] | Lu Y, Chen Q, Bu Y, et al. Flavonoid accumulation plays an important role in the rust resistance of malus plant leaves[J]. Front Plant Sci, 2017, 8: 1286. DOI:10.3389/fpls.2017.01286 |
| [40] | Cavallini E, Matus JT, Finezzo L, et al. The phenylpropanoid pathway is controlled at different branches by a set of R2R3-MYB C2 repressors in grapevine[J]. Plant Physiol, 2015, 167: 1448–1552. DOI:10.1104/pp.114.256172 |
| [41] | Mierziak J, Kostyn K, Kulma A. Flavonoids as important molecules of plant interactions with the environment[J]. Molecules, 2014, 19: 16240–16265. DOI:10.3390/molecules191016240 |
| [42] | Ito M, Ichinose Y, Kato H, et al. Molecular evolution and functional relevance of the chalcone synthase genes of pea[J]. Mol Gen Genet, 1997, 255: 28–37. DOI:10.1007/s004380050471 |
| [43] | Du F, Fan JM, Wang T, et al. Identification of differentially expressed genes in flower, leaf and bulb scale of Lilium oriental hybrid 'Sorbonne' and putative control network for scent genes[J]. BMC Genomics, 2017, 18: 899. DOI:10.1186/s12864-017-4303-4 |
| [44] | Mariz-Ponte N, Mendes RJ, Sario S, et al. Tomato plants use non-enzymatic antioxidant pathways to cope with moderate UV-A/B irradiation:a contribution to the use of UV-A/B in horticulture[J]. J Plant Physiol, 2017, 221: 32–42. |
| [45] | Merkle T, Frohnmeyer H, Schulze-Lefert P, et al. Analysis of the parsley chalcone-synthase promoter in response to different light qualities[J]. Planta, 1994, 193: 275–282. |
| [46] | Raharjo TJ, Chang WT, Verberne MC, et al. Cloning and over-expression of a cDNA encoding a polyketide synthase from Cannabis sativa[J]. Plant Physiol Biochem, 2004, 42: 291–297. DOI:10.1016/j.plaphy.2004.02.011 |
| [47] | Lei W, Tang SH, Luo KM, et al. Molecular cloning and expression profiling of a chalcone synthase gene from hairy root cultures of Scutellaria viscidula Bunge[J]. Genet Mol Biol, 2010, 33: 285–291. DOI:10.1590/S1415-47572010005000031 |
 2018, Vol. 53
2018, Vol. 53


