组蛋白的翻译后修饰是表观遗传调控的重要组成部分, 主要包括甲基化、乙酰化、泛素化、磷酸化、巴豆酰化和琥珀酰化等[1]。其中组蛋白甲基化是一个可逆的修饰过程[2], 在许多生理过程中起到关键的作用, 主要包括遗传信息的转录、翻译、细胞增殖与细胞分化、信号的转导和胚胎的发育等[3]。目前催化组蛋白甲基化的酶主要分为3类:蛋白精氨酸甲基转移酶(PRMT)家族、含有SET结构域[Su(var), Enhancer of zeste (E(z)), and Trithorax]的赖氨酸甲基转移酶(PKMT)家族和无SET结构域的赖氨酸甲基转移酶。
Dot1 (也称为KMT4)是Miriam S. Singer等在啤酒酵母(Saccharomyces cerevisiae)中筛选影响端粒沉默的因子时首次发现的, 其过表达将导致端粒沉默[4]。在哺乳动物中, Dot1被称作Dot1-like蛋白(DOT1L), 是一种进化上保守的赖氨酸甲基化酶, 也是迄今唯一一个被证实不含SET结构域的组蛋白赖氨酸甲基转移酶, 并且只能特异地催化位于核心球体部位的组蛋白H3赖氨酸79 (H3K79)发生单甲基化、二甲基化和三甲基化, 而不能催化从核小体上游离的组蛋白H3[5]。其催化机制是: DOT1L以S-(5'-腺苷)-L-甲硫氨酸(SAM)作为甲基供体, 使SAM的甲基与底物核小体的H3K79上的ε-NH2发生SN2亲核取代反应, 将SAM的甲基转移到ε-NH2上, 生成甲基化的底物以及S-腺苷-L-高半胱氨酸(SAH) (图 1)。
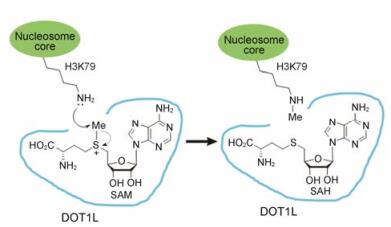
|
Figure 1 Mechanism of catalysis of DOT1L |
在生理条件下, DOT1L在转录调节、细胞周期调节、DNA修复和胚胎发育等过程中发挥着至关重要的作用[6], 而DOT1L的异常调控与多种疾病的发生和发展密切相关, 例如DOT1L的过表达能够催化H3K79异常甲基化, 促进白血病相关基因(如HOXA9和MEISI)的表达, 导致白血病发生和发展[7], 抑制DOT1L的过表达将导致细胞周期停滞以及促进肿瘤细胞的分化和凋亡[8]。近年来, DOT1L已成为治疗肿瘤的重要靶标, 越来越受到人们的关注, 基于此, 本文总结和探讨了关于DOT1L的结构与功能、DOT1L与肿瘤的关系及DOT1L抑制剂等内容, 概述了DOT1L的最新研究进展。
1 结构与生物功能 1.1 hDOT1L的结构hDOT1L蛋白由1 537个氨基酸组成, 分子质量为165 kDa。Min等[9]通过晶体衍射方法研究和分析了hDOT1催化结构域的结构(图 2)。其催化结构域主要位于氨基酸残基1~416之间, 包含: ① N末端结构域, 由5个α螺旋和2对短β折叠结构构成; ② C末端结构域, 该结构域是一个开放的α/β结构, 由位于中央的7个β折叠及外围的5个α螺旋结构构成, 研究表明, 结构上无序的C末端(319~416号氨基酸残基)是一个柔性的正电荷区域, 其通过静电相互作用的方式识别并结合带负电的核小体, 以实现对H3K79的催化作用; ③ S-腺苷酸-L-甲硫氨酸(SAM)结合结构域, 主要由以下5个片段组成: 161~169号氨基酸残基(Ⅰ)、186~191号氨基酸残基(Ⅱ)、239~245号氨基酸残基(Ⅲ)、133~139号氨基酸残基(Ⅳ)和221~224号氨基酸残基(Ⅴ)。其中片段Ⅰ~Ⅲ与其他SAM依赖性甲基转移酶具有相同的氨基酸序列, 片段Ⅳ是一个控制开关, 其开启和关闭可以控制SAM/SAH的进入/离开, 而片段Ⅳ和Ⅴ是DOT1L所特有的。疏水性残基Phe243、Ala244、Val135、Gly137和Trp305构成了一个潜在的赖氨酸结合通道, 该通道最窄部分约为4Å, 可以容纳单、二或三甲基化的赖氨酸。
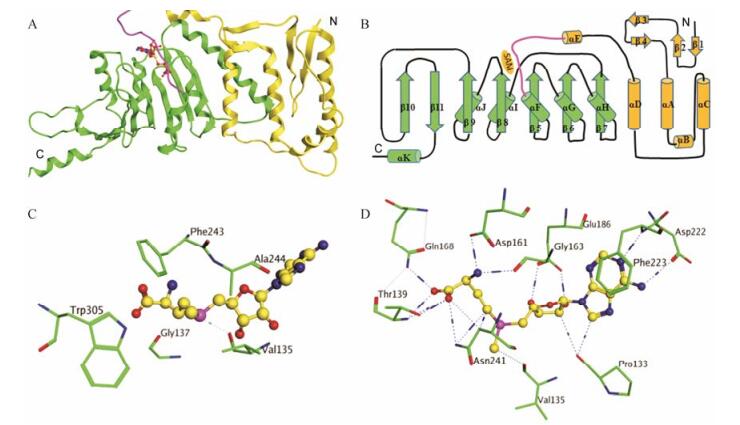
|
Figure 2 (A) Ribbon diagram of the hDOT1L (1-416) structure. The N-terminal region (1-126) is colored yellow, the open α/β structure (141-332) is shown in green, and the loop L-EF connecting the two regions is shown in purple. The bound SAM molecule is shown in a ball and stick model; (B) Topological diagram of hDOT1L (1-416); (C) Close-up view of the lysine binding channel, with the methyl group of SAM shown in ball and stick model; (D) Close-up view of the active site of the DOT1L: SAM structure, with H-bonds shown as dotted lines |
研究表明DOT1L介导H3K79的甲基化水平与基因转录的活跃状态密切相关, Shahbazian等[10]在酵母的基因转录区域均检测到Dot1和甲基化的H3K79。Steger等[11]发现H3K79甲基化与小鼠的基因转录相关。在人CD4+ T细胞中, ChIP-seq实验表明甲基化的H3K79在转录基因上富集[12]。在果蝇中, ChIP芯片分析也揭示了H3K79me2与活性基因转录之间的相关性[13]。
除了在转录中的作用外, DOT1L通过催化H3K79甲基化控制细胞周期, 在哺乳动物中, DOT1L介导H3K79的甲基化被认为是调控G1/S转换过程中细胞周期进程的关键因素。在人NCI-H1299和A549肺癌细胞中, DOT1L缺失导致异常的纺锤体和中心体形成, CDK抑制剂如p18INK4c和p21CIP1/WAF1的上调, 使细胞周期停滞在G1期[14]。此外, 在MLL融合蛋白转导的小鼠细胞中, DOT1L的缺失也可导致p16INK4a的表达上调, 使细胞周期停滞于G1期, 最终导致细胞老化, 细胞增殖受到抑制[15]。可见, DOT1L是细胞周期中重要的表观遗传调节因子。
DOT1L介导的组蛋白修饰对于胚胎发育至关重要, 敲除小鼠的DOT1L基因将引起胚胎发育不良, 包括发育迟缓、心血管发育不良和心脏扩张等[16], 该现象表明DOT1L的功能是胚胎发育所必需的。
此外, H3K79甲基化对维持正常的造血功能具有重要的作用。在小鼠骨髓细胞中敲除DOT1L将导致相关靶基因的异常表达(如GATA2的下调和PU.1/ Sfpi1的过表达), 从而显著地抑制红细胞的形成, 极大地减少了造血干细胞和骨髓祖细胞的数量[17]。
2 DOT1L与肿瘤的关系 2.1 MLL-r白血病MLL (mixed lineage leukemia)基因常在11号染色体上发生各种易位重排, 形成融合基因, 并编码多个onco-MLL融合蛋白(如MLL-AF4、MLL-AF9、MLL-AF10和MLL-ENL)。这些致瘤蛋白质结合到某些MLL靶基因(如Hoxa9)的启动子区[18], 异常招募DOT1L到onco-MLL启动子区引起H3K79超甲基化, 启动和维持基因转录, 促使多个onco-MLL的靶基因(如Hoxa9、Hoxa7和Meis1)过度表达, 最终促进白血病发生、发展[19]。
Bernt等[20]研究表明, DOT1L介导的H3K79异常甲基化是鼠和人MLL-AF9白血病的标志, 在DOT1L敲除的MLL-AF9白血病小鼠模型中, Dot1l的缺失显著抑制了肿瘤细胞的增殖细胞, 同时使大多数细胞停滞于G0/G1期, 部分细胞还出现凋亡及分化现象。Krivtsov等[21]通过siRNA抑制DOT1L的表达, 从而消除MLL-AF10和MLL-AF4的转化能力。这些发现在生物学上证实了DOT1L在MLL-r白血病的发生中起重要作用, 为MLL-r白血病的治疗提供了重要靶标。
2.2 乳腺癌Oktyabri等[22]发现, 过表达的DOT1L催化H3K79发生异常甲基化, 导致支链氨基酸转移酶1 (BCAT1)的表达上调, 最终促进乳腺癌细胞的迁移及乳腺癌干细胞微球体的形成, 此外, DOT1L还通过招募c-Myc蛋白到EMT (epithelial-mesenchymal transition)转录因子的启动子区, 与P300形成转合物共同调控EMT转录因子的表达, 引起EMT形态学的改变, 使E-钙粘蛋白的表达下调, 增强了肿瘤细胞的转移和侵袭能力, 同时也促进CSCs (cancer stem cells)的自我更新能力及肿瘤微球体的形成[23]。
2.3 肺癌DOT1L介导的H3K79甲基化调节酵母、锥虫和小鼠的正常细胞周期进程, 但对肺癌细胞是否有影响尚未被阐明。Kim等[14]报道, 在肺癌细胞系A549中敲除DOT1L基因, 导致不对称的细胞分裂, 造成纺锤体的形成和中心体数目的异常, 从而影响染色体的稳定。另外, DOT1L的缺失也能诱导细胞周期蛋白依赖激酶抑制剂(如P21、P18等)的表达上调, 使细胞周期停滞在G1期, 引起细胞不可逆的早衰, 细胞增殖受到抑制。
2.4 神经母细胞瘤Wong等[24]对88例成神经细胞瘤患者的肿瘤组织分析发现, DOT1L mRNA的表达水平与N-Myc mRNA、ODC1 mRNA和E2F2 mRNA的表达呈正相关; 同时也与患者的总体生存率相关, DOT1L mRNA的表达水平越高, 总体生存率、无事件生存率越低。机制研究表明, 在N-Myc靶基因(如DDC1、E2F2)的启动子区, DOT1L直接与N-Myc的Myc BOX Ⅱ结合域相结合, 形成转录复合物, 诱导启动子区H3K79二甲基化, 激活致瘤基因DDC1、E2F2的表达, 促进神经母细胞瘤的发生、发展和增殖。总之, DOT1L通过激活致瘤基因ODC1、E2F2的表达, 而在N-Myc介导的成神经细胞瘤中扮演着重要角色。
3 抑制剂自从发现DOT1L在MLL白血病中的关键作用以来, 科学家致力于开发具有选择性的DOT1L抑制剂, 其不仅可以作为小分子探针用于研究DOT1L在癌症和其他疾病中的生物学功能, 还有希望被进一步开发为新型抗肿瘤药物。迄今为止, 已有多个DOT1L小分子抑制剂相继被报道, 根据结构类型, 大致可分为: SAH类似物、氨基甲酸酯类、脲/苯并咪唑类和其他类。
3.1 SAH类似物Richon等[25]对人组蛋白甲基化酶与其辅因子SAM相互作用进行研究发现, 其反应产物SAH对多种组蛋白甲基转化酶均具有一定的抑制作用, 其中对DOT1L的Ki值为160 nmol·L-1, 这引起了科学家的高度关注, 希望通过对SAH进行结构修饰, 得到高度选择性的DOT1L抑制剂, 随后一系列的SAH类似物相继被报道(图 3)。

|
Figure 3 SAH-like DOT1L inhibitors |
Anglin等[26]将DOT1L-SAM复合物的晶体结构和其他组蛋白甲基转移酶(PRMT)与SAM复合物的晶体结构进行分析、比较, 设计并合成了一系列选择性抑制DOT1L的N6取代的SAH类似物(如结构2), 其中化合物1对DOT1L的活性最佳(Ki = 290 nmol·L-1), 而其他取代基(包括烯丙基、环丙基、异丙基、丁基和苄基等)对DOT1L的活性相对较弱, Ki值在1.1~8.9 μmol·L-1之间。分子模拟研究显示, 化合物1的N6-甲基能够占据由Phe223、Leu224、Val249、Lys187和Pro133残基构成的疏水空腔, 而其他较大的烷基取代则暴露于疏水空腔之外。
Yao等[27]利用基于机制的药物设计方法发现了另一类选择性DOT1L抑制剂(化合物3~5)。这类化合物的作用机制是在中性pH条件下, 先进行分子内环化, 形成吖啶鎓中间体(如结构式6), 然后再共价结合H3K79的ε-NH2基团进而抑制ε-NH2的甲基化。但化合物3对DOT1L的抑制活性(IC50 = 16 μmol·L-1)远不及化合物4 (IC50 = 38 nmol·L-1)和化合物5 (IC50 = 120 nmol·L-1), 可能是化合物3中C-5'位侧链的两个C-N键键长(约为1.47 Å)比SAM/SAH中C-5'位侧链的两个C-S键键长(约为1.82 Å)短, 未能占据DOT1L活性口袋的最佳结合位点。相比较而言, 化合物4、5分别在C-5'位侧链增加了一个亚甲基, 总体上增加了侧链的长度, 从而能达到最佳结合位点。由于化合物4的N6位没有引入甲基, 所以选择性不及化合物5。
Yu等[28]通过虚拟筛选, 发现化合物7具有抑制DOT1L的作用(IC50 = 18 μmol·L-1), 分子模拟结果表明, 化合物7和SAH的腺苷部分与DOT1L具有相似的结合模式。为了增强化合物7与DOT1L的相互作用, 将SAH的同型半光氨酸侧链取代化合物7的C-5'位侧链的羟基, 同时用溴原子取代C-7位碘原子得到具有高活性、高选择性的化合物8, 其IC50为77 nmol·L-1。分子对接发现, 除了与SAH具有相同的结合模式之外, 溴原子还占据Phe245、Val249、Leu224和Pro133残基形成的特异性疏水性空腔, 但该化合物对肿瘤细胞抑制作用较弱, 可能是由于其细胞膜渗透性差的原因。为了改善化合物8的细胞膜渗透性, Spurr等[29]将氰基取代C-7位的溴原子后得到化合物9, 这一修饰不仅提高了化合物9对DOT1L的抑制作用(IC50 = 26 nmol·L-1), 同时也改善了该化合物的细胞膜渗透性。
3.2 氨基甲酸酯类在生理pH条件下, SAH类似物的极性带电氨基酸部分对其生物利用度有很大影响, 不利于药物透过细胞膜。虽然这类抑制剂的氨基酸部分与DOT1L形成5个氢键, 增强了受体与配体的结合力, 但DOT1L的活性口袋同时也含有几个疏水残基(Phe239、Val169、Thr139和Tyr136), 因此引入亲脂的侧链是有利的。基于此, Anglin等[26]设计并合成了一系列的氨基甲酸酯类DOT1L选择性抑制剂(图 4), 体外活性筛选发现化合物10~12b对DOT1L具有抑制作用, 其中化合物12a活性最佳, Ki值为16 μmol·L-1。
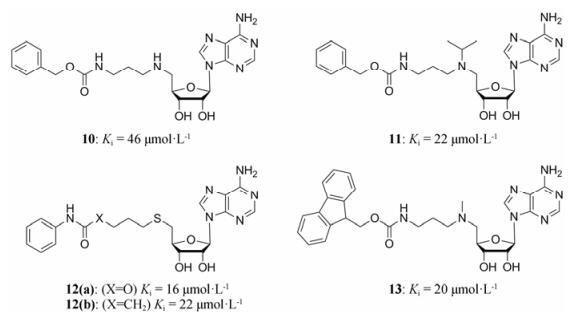
|
Figure 4 Carbamate-containing DOT1L inhibitors |
Anglin等[26]继续对化合物12a进行结构修饰, 衍生了另一系列DOT1L的抑制剂, 如化合物14~17b (图 5), 它们都表现出较好的DOT1L抑制活性, 尤其是化合物17a和17b, 其不仅具有较好的抑制活性(Ki = 0.8 nmol·L-1), 同时也对DOT1L具有较强的选择性。这说明脲基团上的两个-NH-结构是活性所必需的, 这两个-NH-可以作为氢键供体以增加化合物与DOT1L的亲和力。在MLL重排白血病细胞系MV4-11中, 化合物17a和17b(10 μmol·L-1)在给药后的第15天分别显示出61.7%和43.4%的细胞生长抑制率, 到第20天时抑制作用更为显著(抑制率分别为91.7%和77.6%)。
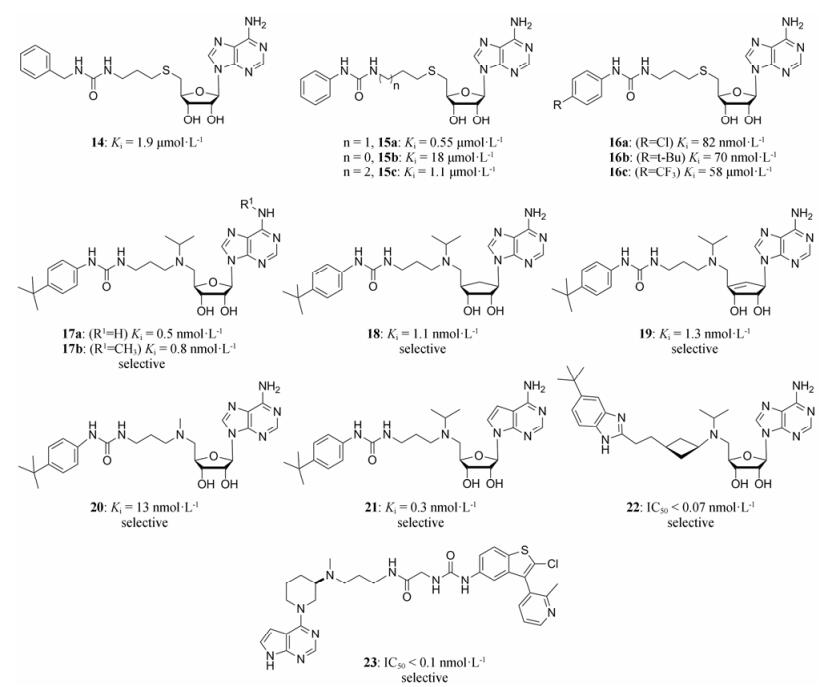
|
Figure 5 Urea/benzimidazole-containing DOT1L inhibitors |
动物模型研究显示, 含腺苷的DOT1L抑制剂都存在一个相同的问题, 即代谢不稳定性, 导致药代动力学差, 例如血浆中的半衰期较短等。主要是由于体内多个酶能够识别腺苷部分, 导致这些化合物被快速分解[30]。Deng等[31]为了提高DOT1L抑制剂在体内的稳定性, 将五元碳环代替腺苷的呋喃糖, 合成了化合物18~19。在MV4-11细胞中, 化合物18和19以剂量依赖的方式阻断H3K79的甲基化。在代谢稳定性方面:化合物18在人血浆和肝微粒体中代谢稳定性较高(CLint = 0.36 μL·min-1·mg-1蛋白), 而化合物19代谢依然不稳定, 这可能是由于化合物19中的C=C双键在体内被一些酶(如细胞色素P450)氧化, 从而使其降解。
Epizyme公司在开发选择性DOT1L抑制剂过程中发现Fmoc保护的氨基甲酸酯化合物13对DOT1L具有潜在的抑制作用(Ki = 20 μmol·L-1), 随后对其进行结构改造, 发现了高效、高选择性的含有脲基团的DOT1L抑制剂20 (Ki = 13 nmol·L-1)和21 (Ki = 0.3 nmol·L-1)。进一步对化合物21进行结构优化, 即化合物21的-CH2CH2CH2-链和4-叔丁基苯基取代的脲基团分别被顺式乙基环丁烷链和5-叔丁基苯并咪唑取代。最终得到化合物22, 显著提高了其抑制作用(Ki < 0.07 nmol·L-1)。这些改变主要是为了降低化合物21的构象柔性和氢键供体数量, 以提高活性和生物利用度, 降低血浆清除率。尽管化合物21在体外对DOT1L和MLL异常相关的多类型白血病细胞的增殖均显示出良好的抑制作用, 但其药代动力学特性较差, 影响了成药性。虽然化合物22在动物模型中的口服生物利用度较低, 但在腹腔和静脉注射给药后能观察到较高的生物利用度和良好的耐受性。目前化合物22用于MLL-r白血病的治疗已进入I期临床试验[32-34]。
化合物23是Möbitz等[35]运用虚拟高通量筛选结合片段拼接的药物设计方法发现的脲类DOT1L抑制剂。在宫颈癌细胞系HeLa和人急性髓性白血病细胞系Molm-13中, 化合物23显著抑制了H3K79的二甲基化(IC50 = 3 nmol·L-1)和HoxA9的表达(IC50 = 17 nmol·L-1)。此外, 化合物23也能有效地抑制MLL-r白血病细胞系MV4-11的增殖(IC50 = 5 nmol·L-1)。研究表明, 化合物23对DOT1L具有良好的选择性(IC50 < 0.1 nmol·L-1), 而对其他PKMTs和PRMTs抑制作用较弱(IC50 = 50 μmol·L-1)。
3.4 其他类Chen等[36]基于结构虚拟筛选的方法发现化合物24 (图 6)对DOT1L具有一定的抑制作用(IC50 = 1.5 μmol·L-1), 体外活性测试发现, 该化合物能选择性抑制MLL-r细胞系MV4-11的增殖(IC50 = 37.1 μmol·L-1), 而对实体瘤细胞和正常体细胞几乎无影响。分子模拟显示该抑制剂能竞争性占据SAH的结合位点。总体而言, 该报道发现了一个全新结构的DOT1L抑制剂, 对后续寻找特异性好, 高效低毒的DOT1L抑制剂具有一定的指导意义。

|
Figure 6 Other DOT1L inhibitors |
Wang等[37]通过虚拟筛选发现了全新的选择性靶向DOT1L的化合物25, 其IC50为8.3 μmol·L-1, 该化合物能选择性抑制MLL-r细胞系MV4-11的增殖, 并促使细胞周期停滞于G0/G1期和细胞凋亡, 对非MLL-r细胞系如K-562、实体瘤细胞系Caki-1以及正常体细胞HK-2无影响。这一发现也为后续DOT1L抑制剂的开发提供了良好的先导化合物。
最近, 诺华公司的Chao Chen小组通过高通量筛选发现化合物26具有潜在的DOT1L抑制作用, 随后对其进行结构改造, 衍生了一系列具有良好抑制DOT1L活性的化合物, 其中化合物27和28对DOT1L的抑制作用最佳, 其IC50分别为1.4和0.4 nmol·L-1。在HeLa细胞中, 化合物27和28都能显著抑制H3K79的二甲基化(IC50值分别为23和16 nmol·L-1), 在MLL-r细胞系MV4-11中, 这两个化合物均显著地抑制肿瘤细胞的增殖(IC50分别为85和128 nmol·L-1)。大鼠实验表明, 化合物27代谢稳定, 具有良好药代动力学性质。另外, 化合物27和28对DOT1L具有较好的选择性(IC50分别为1.4和0.4 nmol·L-1), 而对其他PKMTs和PRMTs的抑制作用较弱或无活性(IC50 > 50 μmol·L-1)[38]。
Scheufler等[39]通过虚拟高通量筛选并结合化学结构改造发现一系列结构新颖的喹唑啉类DOT1L抑制剂, 其中化合物29具有较强的抑制作用(IC50 = 14 nmol·L-1), 该系列抑制剂在结构上与辅因子SAM无关, 并且在SAM结合口袋内没有相互作用, 而是与SAM结合位点相邻的结合口袋相互作用。
4 DOT1L抑制剂的构效关系含腺苷类DOT1L抑制剂的构效关系主要受N-6位、7位和C-5'位影响较大。对于N-6位的修饰, 甲基取代后活性最好, 选择性最高(如化合物1), 较大或较长的烷基(例如异丙基和异戊基等)取代, 活性降低(如化合物2)。主要是由于在N-6位的上方有一个由疏水性残基Leu224、Val249、Lys187和Pro133组成的宽约4Å, 深约5Å的疏水性口袋(图 7A), 化合物1的N6-甲基基团很好地伸到口袋中, 与疏水性口袋形成疏水相互作用[27]。然而, 较大烷基取代的化合物则暴露于溶剂中, 导致结合力降低。
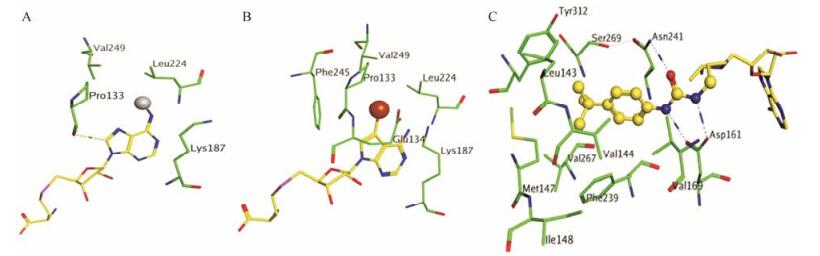
|
Figure 7 (A) Structure of DOT1L-1 showing the binding pocket of the N6-Me (gray sphere); (B) Structure of DOT1L-7 showing the binding pocket of the 7-Br (brown sphere); (C) Structure of DOT1L: 21 showing the binding pocket of the 4-tert-butylphenyl urea group of 21 (in ball and stick model) |
对于7位的修饰, 以-Br和-CN取代时活性较好(如化合物8和9), -I、-Cl取代时活性下降, 图 7B显示了DOT1L-8复合物晶体结构中7-取代基的结合口袋, 该口袋由Phe245、Val249、Leu224、Lys187、Pro133和Glu134构成[40]。这些残基与-Br或-CN形成有利的疏水相互作, 使亲和力增强。
在对C-5'位侧链的修饰中, 4-叔丁基苯基取代的脲基取代时活性最好, 当脲基团被氨基甲酸酯或酰胺替换后活性丢失(如化合物11、12b、21)。可见, 脲基团为活性必须的关键基团。分析DOTlL-21的晶体结构发现, 4-叔丁基苯基位于由Asn241、Ser269、Val267、Tyr312、Met147、Leu143、Val144和Val169新形成的疏水的口袋中, 与之形成较强的疏水相互作用; 同时脲官能团通过与DOT1L形成3个氢键而被识别, 其中羰基O原子与Asn241的侧链氨基具有一个H-键相互作用, 两个-NH-则与Asp161的带负电的羧基形成两个H-键相互作用, 而氨基甲酸酯或酰胺官能团则无此作用(图 7C)[41]。
5 结语近年来, DOT1L作为一种抗肿瘤新靶点受到广泛关注, 它既可以与多种转录调控因子结合, 调节基因的转录过程, 调控肿瘤细胞的增殖, 也可以调控细胞周期, 影响染色质组装与重组。可见, DOT1L与肿瘤的发生、发展密切相关, 但精确的致瘤机制尚不明确, 例如, 已知H3K79的异常甲基化可以导致某些白血病相关基因如Hoxa9和Meis1的过度表达[42], 但白血病相关基因过表达后所引发的下游的具体分子事件目前还不清楚, 所以还需要对DOT1L如何导致肿瘤的发生、发展进行更为深入的研究。目前已有多个的DOT1L抑制剂相继被发现, 但大多都存在代谢不稳定等缺点, 所以开发出代谢较为稳定的DOT1L特异性抑制剂是该研究领域亟待解决的关键问题。
| [1] | Guang LI, Xing XY, Zhang MS, et al. Research progress in epigenetic regulation of myocardial ischemia/reperfusion injury by traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2016, 51: 1047–1053. |
| [2] | Bannister AJ, Kouzarides T. Reversing histone methylation[J]. Nature, 2005, 436: 1103–1106. DOI:10.1038/nature04048 |
| [3] | Zhang Y, Reinberg D. Transcription regulation by histone methylation:interplay between different covalent modifica-tions of the core histone tails[J]. Genes Dev, 2001, 15: 2343–2360. DOI:10.1101/gad.927301 |
| [4] | Singer MS, Kahana A, Wolf AJ, et al. Identification of high-copy disruptors of telomeric silencing in Saccharomyces cerevisiae[J]. Genetics, 1998, 150: 613–632. |
| [5] | Van LF, Gafken PR, Gottschling DE. Dot1p modulates silencing in yeast by methylation of the nucleosome core[J]. Cell, 2002, 109: 745–756. DOI:10.1016/S0092-8674(02)00759-6 |
| [6] | Anglin JL, Song Y. A medicinal chemistry perspective for targeting histone H3 lysine-79 methyltransferase DOT1L:miniperspective[J]. J Med Chem, 2013, 56: 8972–8983. DOI:10.1021/jm4007752 |
| [7] | Muntean AG, Hess JL. The pathogenesis of mixed-lineage leukemia[J]. Annu Rev Pathol, 2012, 7: 283–301. DOI:10.1146/annurev-pathol-011811-132434 |
| [8] | Rau RE, Rodriguez BA, Luo M, et al. DOT1L as a therapeutic target for the treatment of DNMT3A-mutant acute myeloid leukemia[J]. Blood, 2016, 128: 971–981. DOI:10.1182/blood-2015-11-684225 |
| [9] | Min J, Feng Q, Li Z, et al. Structure of the catalytic do-main of human DOT1L, a non-SET domain nucleosomal histone methyltransferase[J]. Cell, 2003, 112: 711–723. DOI:10.1016/S0092-8674(03)00114-4 |
| [10] | Shahbazian MD, Zhang K, Grunstein M. Histone H2B ubiquitylation controls processive methylation but not monomethylation by Dot1 and Set1[J]. Mol Cell, 2005, 19: 271–277. DOI:10.1016/j.molcel.2005.06.010 |
| [11] | Steger DJ, Lefterova MI, Ying L, et al. DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells[J]. Mol Cell Biol, 2008, 28: 2825–2839. DOI:10.1128/MCB.02076-07 |
| [12] | Wang Z, Zang C, Rosenfeld JA, et al. Combinatorial patterns of histone acetylations and methylations in the human genome[J]. Nat Genet, 2008, 40: 897–903. DOI:10.1038/ng.154 |
| [13] | Schübeler D, MacAlpine DM, Scalzo D, et al. The histone modification pattern of active genes revealed through ge-nome-wide chromatin analysis of a higher eukaryote[J]. Genes Dev, 2004, 18: 1263–1271. DOI:10.1101/gad.1198204 |
| [14] | Kim W, Kim R, Park G, et al. Deficiency of H3K79 histone methyltransferase Dot1-like protein (DOT1L) inhibits cell proliferation[J]. Biol Chem, 2012, 287: 5588–5599. DOI:10.1074/jbc.M111.328138 |
| [15] | Nguyen AT, He J, Taranova O, et al. Essential role of DOT1L in maintaining normal adult hematopoiesis[J]. Cell Res, 2011, 21: 1370–1373. DOI:10.1038/cr.2011.115 |
| [16] | Nguyen AT, Taranova O, He J, et al. DOT1L, the H3K79 methyltransferase, is required for MLL-AF9-mediated leukemogenesis[J]. Blood, 2011, 117: 6912–6922. DOI:10.1182/blood-2011-02-334359 |
| [17] | Chou ST, Khandros E, Bailey LC, et al. Graded repression of PU.1/Sfpi1 gene transcription by GATA factors regulates hematopoietic cell fate[J]. Blood, 2009, 114: 983–994. DOI:10.1182/blood-2009-03-207944 |
| [18] | Bitoun E, Oliver PL, Davies KE. The mixed-lineage leukemia fusion partner AF4 stimulates RNA polymerase Ⅱ transcriptional elongation and mediates coordinated chromatin remodeling[J]. Hum Mol Genet, 2006, 16: 92–106. |
| [19] | Okada Y, Feng Q, Lin Y, et al. hDOT1L links histone methylation to leukemogenesis[J]. Cell, 2005, 121: 167–178. DOI:10.1016/j.cell.2005.02.020 |
| [20] | Bernt KM, Zhu N, Sinha AU, et al. MLL-rearranged leukemia is dependent on aberrant H3K79 methylation by DOT1L[J]. Cancer Cell, 2011, 20: 66–78. DOI:10.1016/j.ccr.2011.06.010 |
| [21] | Krivtsov AV, Feng Z, Lemieux ME, et al. H3K79 methyla-tion profiles define murine and human MLL-AF4 leukemias[J]. Cancer Cell, 2008, 14: 355–368. DOI:10.1016/j.ccr.2008.10.001 |
| [22] | Oktyabri D, Ishimura A, Tange S, et al. DOT1L histone methyltransferase regulates the expression of BCAT1 and is involved in sphere formation and cell migration of breast cancer cell lines[J]. Biochimie, 2016, 123: 20–31. DOI:10.1016/j.biochi.2016.01.005 |
| [23] | Cho MH, Park JH, Choi HJ, et al. DOT1L cooperates with the c-Myc-p300 complex to epigenetically derepress CDH1 transcription factors in breast cancer progression[J]. Nat Commun, 2015, 6: 7821–7834. DOI:10.1038/ncomms8821 |
| [24] | Wong M, Tee AE, Milazzo G, et al. The histone methyltransferase DOT1L promotes neuroblastoma by regulating gene transcription[J]. Cancer Res, 2017, 77: 2522–2533. DOI:10.1158/0008-5472.CAN-16-1663 |
| [25] | Richon VM, Johnston D, Sneeringer CJ, et al. Chemogenetic analysis of human protein methyltransferases[J]. Chem Biol Drug Des, 2011, 78: 199–210. DOI:10.1111/jpp.2011.78.issue-2 |
| [26] | Anglin JL, Deng L, Yao Y, et al. Synthesis and structure-activity relationship investigation of adenosine-containing inhibitors of histone methyltransferase DOT1L[J]. J Med Chem, 2012, 55: 8066–8074. DOI:10.1021/jm300917h |
| [27] | Yao Y, Chen P, Diao J, et al. Selective inhibitors of histone methyltransferase DOT1L:design, synthesis, and crystallo-graphic studies[J]. J Am Chem Soc, 2011, 133: 16746–16749. DOI:10.1021/ja206312b |
| [28] | Yu W, Smil D, Li F, et al. Bromo-deaza-SAH:a potent and selective DOT1L inhibitor[J]. Bioorg Med Chem, 2013, 21: 1787–1794. DOI:10.1016/j.bmc.2013.01.049 |
| [29] | Spurr SS, Bayle ED, Yu W, et al. New small molecule inhibitors of histone methyl transferase DOT1L with a nitrile as a non-traditional replacement for heavy halogen atoms[J]. Bioorg Med Chem Lett, 2016, 26: 4518–4522. DOI:10.1016/j.bmcl.2016.07.041 |
| [30] | Lee JE, Smith GD, Horvatin C, et al. Structural snapshots of MTA/AdoHcy nucleosidase along the reaction coordinate provide insights into enzyme and nucleoside flexibility during catalysis[J]. J Mol Biol, 2005, 352: 559–574. DOI:10.1016/j.jmb.2005.07.027 |
| [31] | Deng L, Zhang L, Yao Y, et al. Synthesis, activity and metabolic stability of non-ribose containing inhibitors of histone methyltransferase DOT1L[J]. Medchemcomm, 2013, 4: 822–826. DOI:10.1039/c3md00021d |
| [32] | Lipka DB, Kuck D, Kliem C, et al. Substituted purine and 7-deazapurine compounds as modulators of epigenetic enzymes:a patent evaluation[J]. Expert Opin Ther Pat, 2013, 23: 537–543. DOI:10.1517/13543776.2013.776040 |
| [33] | Chesworth R, Kuntz KW, Olhava E J, et al. Modulators of histone methyltransferase and methods of use thereof: US, 9029343[P]. 2015-5-12. |
| [34] | Chen CW, Armstrong SA. Targeting DOT1L and HOX gene expression in MLL-rearranged leukemia and beyond[J]. Exp Hematol, 2015, 43: 673–684. DOI:10.1016/j.exphem.2015.05.012 |
| [35] | Möbitz H, Machauer R, Holzer P, et al. Discovery of potent, selective, and structurally novel Dot1L inhibitors by a fragment linking approach[J]. ACS Med Chem Lett, 2017, 8: 338–343. DOI:10.1021/acsmedchemlett.6b00519 |
| [36] | Chen S, Li L, Chen Y, et al. Identification of novel disruptor of telomeric silencing 1-like (DOT1L) inhibitors through structure-based virtual screening and biological assays[J]. J Chem Inf Model, 2016, 56: 527–534. DOI:10.1021/acs.jcim.5b00738 |
| [37] | Wang Y, Li L, Zhang B, et al. Discovery of novel disruptor of silencing telomeric 1-like (DOT1L) inhibitors using a target-specific scoring function for the (S)-adenosyl-l-methionine (SAM)-dependent methyltransferase family[J]. J Med Chem, 2017, 60: 2026–2036. DOI:10.1021/acs.jmedchem.6b01785 |
| [38] | Chen C, Zhu H, Stauffer F, et al. Discovery of novel Dot1L inhibitors through a structure-based fragmentation approach[J]. ACS Med Chem Lett, 2016, 7: 735–740. DOI:10.1021/acsmedchemlett.6b00167 |
| [39] | Scheufler C, Möbitz H, Gaul C, et al. Optimization of a fragment-based screening hit toward potent DOT1L inhibitors interacting in an induced binding pocket[J]. ACS Med Chem Lett, 2016, 7: 730–734. DOI:10.1021/acsmedchemlett.6b00168 |
| [40] | Yu W, Chory EJ, Wernimont AK, et al. Catalytic site remodelling of the DOT1L methyltransferase by selective inhibitors[J]. Nat Commun, 2012, 3: 1288. DOI:10.1038/ncomms2304 |
| [41] | Basavapathruni A, Jin L, Daigle SR, et al. Conformational adaptation drives potent, selective and durable inhibition of the human protein methyltransferase DOT1L[J]. Chem Biol Drug Des, 2012, 80: 971–980. DOI:10.1111/jpp.2012.80.issue-6 |
| [42] | Dafflon C, Craig VJ, Méreau H, et al. Complementary activities of DOT1L and menin inhibitors in MLL-rearranged leukemia[J]. Leukemia, 2017, 31: 1269–1277. DOI:10.1038/leu.2016.327 |
 2018, Vol. 53
2018, Vol. 53


