2. 成都中医药大学药学院, 四川 成都 611137
2. Pharmacy College, Chengdu University of Traditional Chinese Medicine, Chengdu 611137, China
红花是菊科一年生草本植物红花(Carthamus tinctorius L.)的干燥花, 自东汉时期张仲景《金匮要略》中“妇人杂病篇”就有使用记载, 其质柔软, 味辛, 性温, 归心、肝经, 具有活血通经、祛瘀止痛的功效, 为活血化瘀常用中药。历史上红花以四川“川红花”、河南“怀红花”最为出名[1]。当前红花在全国各地栽种范围较广, 新疆、云南、四川及河南等地均有种植, 特别是在新疆, 种植面积较大。
红花的化学成分按照结构包括黄酮、生物碱、聚炔、亚精胺、木脂素、倍半萜、有机酸、留醇和烷基二醇等类型。其中黄酮类成分, 包括羟基红花黄色素A (hydroxysafflor yellow A, HSYA)、槲皮素(quercetin)、柚皮素(naringenin)等是红花主要有效成分。研究表明HSYA具有抗凝、抗缺氧、降血压、改善心脑血管供血不足等作用[2, 3], 山奈酚[4]、槲皮素[5]等在活血化瘀上也有较好效果。
MYB转录因子广泛参与调控黄酮类成分的合成, 国内外已有多篇文章进行了综述[6-8]。在模式植物拟南芥中, AtMYB12能调控查尔酮合成酶基因(CHS)、查尔酮异构酶基因(CHI)及黄酮醇羟化酶基因(F3H)等基因表达, 促进调控黄酮类成分的合成[9]。其同源基因AtMYB11和AtMYB111也能调控黄酮类成分的合成[10]。AtMYBL2也被报道参与调控黄酮类成分的合成[11]。而在红花中, 克隆报道的MYB转录因子较少。至今仅见Guan等[12]克隆的CtMYB1一篇报道。
近年来, 为了深入研究红花黄酮类成分的合成调控, 对红花进行了大量的转录组测序工作[13-15]。本研究基于在线二代转录组数据, 利用iTAK软件对MYB转录因子基因进行注释, 设计引物对长片段MYB转录因子基因进行克隆, 其次对克隆到的MYB转录因子基因进行序列分析, 再次利用实时荧光定量PCR (real-time PCR)对克隆的MYB转录因子基因表达进行分析。通过对红花MYB转录因子基因克隆及表达分析, 为进一步解析红花黄酮类成分的调控机制、调控黄酮类成分的合成奠定基础。
材料与方法材料 红花为道地产区四川简阳地方品种, 经成都中医药大学药学院资源与鉴定系严铸云教授鉴定为红花(Carthamus tinctorius L.)。2016年3月底将红花种子种植于成都中医药大学温江校区药用植物园。红花3个时期的管状花混样用于MYB转录因子基因克隆(3个时期见图 1A)。红花根、茎、叶及3个时期的管状花用于MYB转录因子基因表达分析, 根、茎、叶的取样时间是在红花开花第一个时期。田间取样后立即液氮冷冻, 储存于-80 ℃冰箱备用。克隆用大肠杆菌DH5a菌株购自宝生物(TaKaRa, Dalian, China)。

|
Figure 1 Construction of cDNA mixed template. A: The three periods (Ⅰ, Ⅱ and Ⅲ) for flower development; B: RNA electrophoresis for the three periods, M represents 2 000 bp DNA Marker |
仪器与试剂 主要仪器有PCR仪T100TM Thermal Cycler型(Bio-Rad, CA, USA), 高速低温离心机CT15RE型(HITACHI, Japan), 凝胶成像系统Gel DocTM XR+型(Bio-Rad, CA, USA), 实时荧光定量PCR仪Bio-Rad CFX96型(Bio-Rad, CA, USA); 主要试剂有2×TransTaq High Fidelity (HiFi) PCR SuperMix Ⅱ (Transgen, Beijing, China), 琼脂糖凝胶回收试剂盒、质粒提取试剂盒(Tiangen, Beijing, China), TRIzol (Invertrigen, MA, USA)及TRIzol伴侣试剂盒(Tiandz, Beijing, China), 感受态细胞制备试剂盒(TaKaRa, Dalian, China), 克隆载体pMD19-T (TaKaRa, Dalian, China), 引物合成及片段测序送擎科公司(Tsingke, Beijing, China)。
序列获取及引物设计 本研究采用的转录组数据主要参考Huang等[13]发表的在线数据, NCBI登录号SRA048496, 使用百迈克云平台(https://www.biocloud.net/), 选择无参模型进行分析, 利用iTAK软件[16]对调控因子进行注释, 筛选序列较长MYB转录因子基因, 以此序列利用Primer5对克隆引物进行设计。实验中筛选得到8条较长MYB转录因子基因序列, 设计的克隆引物见表 1。
| Table 1 Primer sequences for MYB transcription factor gene cloning |
cDNA混样模板的构建 将冷冻的红花材料液氮研磨, 用TRIzol进行裂解, 利用TRIzol伴侣试剂盒进行RNA提取, 具体实验方法见商品使用说明书。提取的RNA通过凝胶电泳进行完整度检测, 通过NanoDropTM分光光度计ND1000进行浓度测定。利用试剂盒RR047A在PCR仪上进行RNA的反转录。RNA提取和反转录过程中的耗材为去除RNA酶的材料。将3个时期的花RNA同时提取并进行反转录, 将得到的cDNA混样, 用于后续MYB转录因子基因的克隆。
基因克隆 以构建的混样cDNA为模板, 以2×TransTaq High Fidelity (HiFi) PCR SuperMix Ⅱ进行PCR反应。反应体系(20 μL): 2×TransTaq High Fidelity (HiFi) PCR SuperMix 10 μL, ddH2O 7 μL, 上、下游引物各1 μL, cDNA模板1 μL (50 ng·μL-1)。PCR反应条件为: 95 ℃预变性4 min, 95 ℃变性30 s, 56 ℃退火30 s, 72 ℃延伸2 min, 共34个循环, 72 ℃延伸5 min, 12 ℃保温。1%琼脂糖凝胶电泳后切取目的胶块, 利用胶回收试剂盒进行DNA片段回收, 回收片段利用T载体连接试剂盒连接到T载体中, 转化大肠杆菌进行克隆。
序列分析 使用ORFfinder (https://www.ncbi.nlm.nih.gov/orffinder/)对序列开放阅读框及氨基酸进行预测, 使用ProtParam (http://web.expasy.org/protparam/)对蛋白质进行分子量、等电点、平均疏水性进行分析, 使用GO (http://www.geneontology.org/)及Pfam (http://pfam.xfam.org/)对克隆基因进行功能注释。使用IBS1.0对序列结构域进行作图(http://ibs.biocuckoo.org/online.php)。使用DNAMAN软件(版本4.0, Lynnon Biosoft, Quebec, Canada)进行核苷酸序列和蛋白质比对的分析。使用MEGA4软件相邻连接法(neighbor-joining)构建系统进化树, bootstrap检验的重复次数为500次。
MYB转录因子Real-time PCR分析 依据克隆的MYB转录因子序列, 利用Primer 5设计Real-time PCR用特异性引物, 设计参数选择克隆产物大小为200~500 bp, 引物长度为20 ± 2 bp。引物序列见表 2, 其中红花60S rRNA为内参基因。设计的引物通过琼脂糖凝胶电泳测试引物的特异性。使用SYBR PrimeScript RT-PCR试剂盒进行Real-time PCR实验, 实验进行3次重复。反应体系(20 μL): 2×SYBR Ⅱ 10 μL, ddH2O 7 μL, 上、下游引物各1 μL, cDNA模板1 μL (约50 ng·μL-1)。PCR反应条件: 95 ℃预变性3 min, 95 ℃变性10 s, 57 ℃退火延伸30 s, 共43个循环, 65~95 ℃进行熔解曲线绘制。
| Table 2 Primer sequence for Real-time PCR |
高质量的cDNA模板是基因克隆的关键因素之一。为了尽可能对筛选的8个转录因子进行克隆, 实验选择红花不同时期的混样cDNA作为克隆模板。本研究选择了红花3个时期(图 1A), 对3个时期的管状花提取了RNA (图 1B)。结果可知, 3个样品提取的RNA条带清晰无降解。随后分别对3个时期的RNA进行反转录成cDNA, 将3个时期的cDNA样品等量混和, 保证后续基因克隆有高质量的模板。
2 MYB转录因子基因克隆以构建的cDNA混样模板, 对筛选的8个MYB转录因子基因进行克隆(图 2)。由图可知, 6个转录因子基因(CtMYB-TF1、CtMYB-TF2、CtMYB-TF4、CtMYB-TF5、CtMYB-TF6及CtMYB-TF7)有清晰单一条带, 而2个转录因子(CtMYB-TF3及CtMYB-TF8)无明显条带。实验将CtMYB-TF1、CtMYB-TF2、CtMYB-TF4、CtMYB-TF5、CtMYB-TF6及CtMYB-TF7目的条带切胶回收后连接到pMD19-T转入DH5a进行克隆, 对阳性菌液送公司测序。返回结果表明, 实验成功对6个MYB转录因子基因进行了克隆。
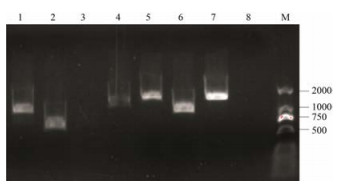
|
Figure 2 Gene cloning of MYB transcription factors. 1-8 represent CtMYB-TF1, CtMYB-TF2, CtMYB-TF3, CtMYB-TF4, CtMYB-TF5, CtMYB-TF6, CtMYB-TF7 and CtMYB-TF8. M represents 2 000 bp DNA marker |
对克隆得到的6个MYB转录因子基因进行了序列分析(表 3)。结果表明, CtMYB-TF1的CDS长度为903 bp、CtMYB-TF2为459 bp、CtMYB-TF4为1 131 bp、CtMYB-TF5为804 bp、CtMYB-TF6为723 bp及CtMYB-TF7为1 233 bp, 都具有DNA结合功能, 属MYB类转录因子。实验利用Pfam对克隆的6个转录因子蛋白序列进行了结构域预测, 发现克隆到的6个MYB转录因子均有MYB类转录因子家族DNA结合区域(图 3)。研究已将克隆到6个MYB转录因子基因相关序列信息提交到NCBI GenBank中, 基因名(GenBank登录号)分别为: CtMYB-TF1 (MF156605)、CtMYB-TF2 (MF156606)、CtMYB-TF4 (MF156607)、CtMYB-TF5 (MF156608)、CtMYB-TF6 (MF156609)及CtMYB-TF7 (MF156610)。
| Table 3 Bioinformatics analysis of MYB transcription factor genes |
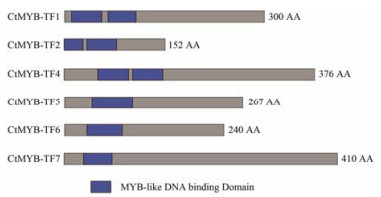
|
Figure 3 Protein domain analysis of MYB transcription factors. The gray frame indicates the full length of the corresponding MYB protein, the length is indicated by a number, and AA repre sent amino acids. The blue box represents the MYB DNA-binding domain |
本实验将克隆得到的转录因子对应氨基酸序列进行了进化分析, 实验选择了红花中已报道的CtMYB1[12]、拟南芥已报道的AtMYB12[9]及AtMYBL2[11], 以及拟南芥转录因子库(http://plntfdb.bio.uni-potsdam.de/v3.0/index.php?sp_id=ALY)中随机3个MYB转录因子共12个转录因子进行了进化分析。结果(图 4)发现, 实验克隆到的MYB转录因子间彼此关系较远, 特别是CtMYB-TF4、CtMYB-TF5、CtMYB-TF6、CtMYB-TF7之间关系, 暗示克隆到转录因子可能具有不同的生物学功能, 特别是CtMYB-TF6同其他克隆到的5个转录因子关系较远。其中仅发现CtMYB-TF7转录因子同AtMYB12和AtMYBL2进化关系相对较近, 暗示CtMYB-TF7可能参与黄酮类成分的调控功能。
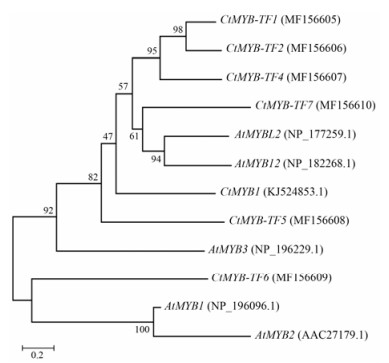
|
Figure 4 Evolution analysis of MYB transcription factors. The gene name is shown in the figure, and the parentheses represent the NCBI accession number |
实验利用Real-time PCR测定了克隆的6个MYB转录因子基因在根、茎、叶及花3个时期中相对表达量(图 5)。CtMYB-TF1在花中3个时期表达量相对较高, 在根、茎及叶中表达量较低; CtMYB-TF2在根和茎中表达量较高, 在叶和花3个时期中表达量较低; CtMYB-TF4在根、茎、叶及花中均有表达, 但表达量较低; CtMYB-TF5、CtMYB-TF6及CtMYB-TF7在根、茎及叶中表达量低, 在花3个时期表达量高。与其他部位和时期比较, CtMYB-TF6在花时期Ⅰ表达最高, CtMYB-TF5及CtMYB-TF7在花时期Ⅱ表达最高。
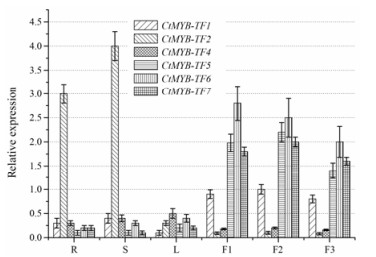
|
Figure 5 Tissue expression analysis of MYB transcription factor genes. The expression level of each gene is indicated by different grid lines, R represents root, S represents stem, L represents leaf, F1-F3 represents the three periods (Ⅰ, Ⅱ and Ⅲ) |
本研究参考Huang等[13]发表的在线数据, 从筛选得到的8个较长片段的MYB转录因子基因序列, 成功克隆了6个MYB转录因子基因, 另外2个MYB转录因子基因没有克隆, 作者推测这2个MYB转录因子在花中表达量较低或不表达, 后面实验将可以构建红花其他部位的混样cDNA, 以此为模板对MYB转录因子基因进行克隆。
进化关系远近可以一定程度反应功能的相关性。实验将克隆到的6个MYB转录因子基因, 连同红花中已报道的CtMYB1[12]和拟南芥中AtMYB12[9]及AtMYBL2[11]等12个MYB转录因子进行了进化分析。结果发现, 克隆MYB转录因子之间彼此关系较远, 其中CtMYB-TF7与已报道的两个黄酮类成分调控转录因子AtMYB12[9]和AtMYBL2[11]关系较近, 这暗示了CtMYB-TF7可能参与了黄酮类成分合成的调控。同时, 实验利用RT-PCR对克隆的6个转录因子进行了组织表达分析, 分析表明CtMYB-TF5、CtMYB-TF6及CtMYB-TF7在根、茎及叶中表达量低, 在花中表达量较高。其中CtMYB-TF6在花时期Ⅰ表达量高, CtMYB-TF5及CtMYB-TF7在花时期Ⅱ表达量高。结合红花黄酮类成分主要在花中积累及进化关系, 表明CtMYB-TF7可能参与了红花黄酮类成分的调控功能, CtMYB-TF5和CtMYB-TF6可能参与红花花中其他生理功能。后续研究将开展相关实验, 验证CtMYB-TF7转录因子是否参与调控红花黄酮类成分的合成。
MYB转录因子家族成员众多, 广泛参与了植物的生长发育各个环节中。模式植物拟南芥已报道的转录因子就有近200个[17]。本实验利用在线二代数据仅分析到8个较长MYB片段, 当前三代测序技术已陆续推出且在中药资源研究方面有较好应用[18-20], 后续研究可以尝试利用三代测序仪器进行测序, 更全面的对红花MYB转录因子基因进行克隆。
| [1] | Peng C. Chinese Genuine Regional Drug Vol. Ⅱ (中华道地药材, 第二册)[M]. Beijing: China Press of Traditional Chinese Medicine, 2011: 1757. |
| [2] | Zang BX, Jin M, Si N, et al. Antagonistic effect of hydroxysafflor yellow A on the platelet activating factor receptor[J]. Acta Pharm Sin (药学学报), 2002, 37: 696–699. |
| [3] | Zhu H, Wang Z, Ma C, et al. Neuroprotective effects of hydroxysafflor yellow A:in vivo and in vitro studies[J]. Planta Med, 2003, 69: 429–433. DOI:10.1055/s-2003-39714 |
| [4] | Zang BX, Jin M, Wu W, et al. Inhibitory effect of kaempferol against binding of platelet activating factor to its receptor[J]. China J Chin Mater Med (中国中药杂志), 2004, 29: 789–791. |
| [5] | Zang BX, Jin M, Wu W, et al. Antagonistic effect of quercetin on the platelet activating factor receptor[J]. J Cardiovasc Pul Dis (心肺血管病杂志), 2004, 23: 105–107. |
| [6] | Lai Y, Li H, Yamagishi M. A review of target gene specificity of flavonoid R2R3-MYB transcription factors and a discussion of factors contributing to the target gene selectivity[J]. Front Biol, 2013, 8: 577–598. DOI:10.1007/s11515-013-1281-z |
| [7] | Liu J, Osbourn A, Ma P. MYB transcription factors as regulators of phenylpropanoid metabolism in plants[J]. Mol Plant, 2015, 8: 689–708. DOI:10.1016/j.molp.2015.03.012 |
| [8] | Xing W, Jin XL. Recent advances of MYB transcription factors involved in the regulation of flavonoid biosynthesis[J]. Mol Plant Breeding (分子植物育种), 2015, 13: 689–696. |
| [9] | Mehrtens F, Kranz H, Bednarek P, et al. The Arabidopsis transcription factor MYB12 is a flavonol-specific regulator of phenylpropanoid biosynthesis[J]. Plant Physiol, 2005, 138: 1083–1096. DOI:10.1104/pp.104.058032 |
| [10] | Stracke R, Ishihara H, Huep G, et al. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling[J]. Plant J, 2007, 50: 660–677. DOI:10.1111/j.1365-313X.2007.03078.x |
| [11] | Dubos C, Gourrierec J, Baudry A, et al. MYBL2 is a new regulator of flavonoid biosynthesis in Arabidopsis thaliana[J]. Plant J, 2008, 55: 940–953. DOI:10.1111/tpj.2008.55.issue-6 |
| [12] | Guan LL, Zhang X, Han YL, et al. Cloning and prokaryotic expression of transcription factor CtMYB1 gene in Carthamus tinctorius[J]. Chin Tradit Herb Drugs (中草药), 2015, 46: 2603–2609. |
| [13] | Huang L, Xiao Y, Pei S, et al. The first illumina-based de novo transcriptome sequencing and analysis of safflower flowers[J]. PLoS One, 2012, 7: e38653. DOI:10.1371/journal.pone.0038653 |
| [14] | Liu X, Dong Y, Yao N, et al. De novo sequencing and analysis of the safflower transcriptome to discover putative genes associated with safflor yellow in Carthamus tinctorius L.[J]. Int J Mol Sci, 2015, 16: 25657–25677. DOI:10.3390/ijms161025657 |
| [15] | Li H, Dong Y, Yang J, et al. De novo transcriptome of safflower and the identification of putative genes for oleosin and the biosynthesis of flavonoids[J]. PLoS One, 2012, 7: e30987. DOI:10.1371/journal.pone.0030987 |
| [16] | Zheng Y, Jiao C, Sun H, et al. iTAK:a program for ge-nome-wide prediction and classification of plant transcription factors, transcriptional regulators, and protein kinases[J]. Mol Plant, 2016, 9: 1667–1670. DOI:10.1016/j.molp.2016.09.014 |
| [17] | Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis[J]. Trends Plant Sci, 2010, 15: 573–581. DOI:10.1016/j.tplants.2010.06.005 |
| [18] | Li QS, Li Y, Song JY, et al. High-accuracy de novo assembly and SNP detection of chloroplast genomes using a SMRT circular consensus sequencing strategy[J]. New Phytol, 2014, 204: 1041–1049. DOI:10.1111/nph.12966 |
| [19] | Xu Z, Peters RJ, Weirather J, et al. Full-length transcriptome sequences and splice variants obtained by a combination of sequencing platforms applied to different root tissues of Salvia miltiorrhiza and tanshinone biosynthesis[J]. Plant J, 2015, 82: 951–961. DOI:10.1111/tpj.12865 |
| [20] | Xu Z, Luo H, Ji A, et al. Global identification of the full-length transcripts and alternative splicing related to phenolic acid biosynthetic genes in Salvia miltiorrhiza[J]. Front Plant Sci, 2016, 7: 100–110. |
 2018, Vol. 53
2018, Vol. 53


