2. 天津大学 天津市现代药物传递与功能高效化重点实验室, 天津 300072
2. Tianjin Key Laboratory for Modern Drug Delivery and High-Efficiency, Tianjin University, Tianjin 300072, China
2009年, 报道了在印度新德里的一家医院里, 医务人员从尿道感染的患者的尿液和粪便中取样得到了一种新型耐药菌——克雷伯杆菌(Klebsiella pneumoniae), 这种耐药菌可产出能够水解近乎所有β-内酰胺类抗生素的酶, 人们以其首次发现的城市将其命名为新德里金属β-内酰胺酶(New Delhi metallo-β-lactmase-1, NDM-1), 产NDM-1的微生物被称为“超级细菌”[1]。NDM-1由位于质粒的blaNDM-1基因编码, 由质粒介导可以在多种细菌之间传播, 可引起诸如泌尿道感染、肺部感染、腹膜炎、软组织感染、植入设备引起的感染、败血症等复杂多样的疾病[2]。含有blaNDM-1基因的质粒通常可以与其他耐药基因相整合, 导致携带blaNDM-1基因的细菌对常用的β-内酰胺类、大环内酯类、四环菌素和磺胺类抗生素等产生耐药性[3]。据巴基斯坦一家医院临床数据统计发现, 患者的住院时间越长, 则在其粪便中分离到NDM-1阳性细菌的几率越大; 但是反过来, 并不能通过临床病症来判断出是否有超级细菌感染[4]。超级细菌的发现, 给当下这个严重依赖抗生素的医疗体系带来巨大的威胁, 超级细菌感染的治疗面临着重重挑战。
2 超级细菌NDM-1的结构特点在过去的几十年里, 金属β-内酰胺酶家族一直都是人类健康的最大威胁, NDM-1是B类金属β-内酰胺酶家族(metallo-β-lactamases, MBLs)中新成员之一[5, 6]。NDM-1是由270个氨基酸残基组成的单链蛋白, 拥有金属β-内酰胺酶家族中典型的三级结构, 即ɑβ/βɑ三明治型结构。蛋白外侧是α螺旋, 两组β折叠被包在中部。一组有4条反向β折叠组成, 另一组由8条反向β折叠组成(图 1)。活性位点呈宽而浅的凹槽状, 处于蛋白表层, 包含两个二价锌离子, 可与溶剂直接接触。两个锌离子的距离在3.2 ~4.6 之间不等[7]。目前蛋白数据库(Protein Data Bank)中已经公布了的NDM-1相关晶体已有43种, 其中包含数十种结合着被水解了的抗生素的NDM-1的晶体结构:氨苄青霉素(ampicilin)、苄甲青霉素(benzylpenicilin)、卡托普利(L-captopril)、甲氧青霉素(methicillin)、美罗培南(meropenem)、苯唑青霉素(oxacillin)等[8-10]; 同时也有结合着可成为潜在抑制剂的NDM-1的晶体结构[11, 12]。将不同的几种NDM-1晶体结构的RMSD值作比较发现, 除了无规卷曲L3 (第63~73号残基)区域的灵活性最大, 蛋白骨架的其他部分都相对稳定[13]。这些复合物晶体结构的存在揭示了β-内酰胺与其底物之间的构效关系, 对于探究NDM-1水解抗生素的机制提供了基础, 从而为探索寻找新型抗生素起到重要作用。
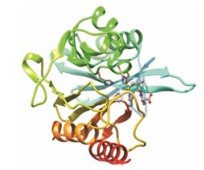
|
Figure 1 The crystal structure of NDM-1 |
活性状态的NDM-1在水溶液中以单链形式存在[14]。然而一些NDM-1晶体由于疏水性和范德华力能够以二聚体形式存在, 例如在嵌入细胞膜状态和纯化状态下的NDM-1。以二聚体形式存在可能是其独特耐药性的原因之一[15]。NDM-1的活性口袋包含两个二价锌离子(图 2), 二价锌离子对酶活性的发挥起着至关重要的作用[16]。将与His120、His122和His189配位的锌离子编号为Zn1, 与His250、Cys208和Asp124配位的锌离子编号为Zn2, 这两个锌离子位于蛋白表面, 可完全与周围溶剂接触[17, 18]。将锌离子定义为活性中心, 是因为锌离子在底物识别过程中起着主要的作用, 它可以络合β-内酰胺环的羧基和羰基。Zn1的作用更主要体现在保持羟基以正确的方向对羰基碳进行亲核进攻, 而Zn2则与β-内酰胺环上的氮相互作用[16]。有实验结论推测NDM-1中的Zn1比Zn2表现出对底物更强的结合能力[15, 19]。然而目前并没有十分精确的理论来解释这种耐药机制。
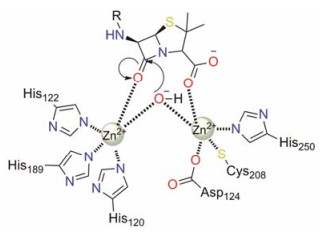
|
Figure 2 Proposed mechanism for the NDM-1-catalyzed hydrolysis of β-lactamase |
Zhang等[7]使用量子力学和分子力学(QM/MM)的方法为NDM-1的耐药机制提出有关于中间态的两种假设。在β-内酰胺环被水解的过程中, 与两个锌离子共同络合的羟基作为亲核基团进攻内酰胺环上的羰基碳原子, 之后的水解有两种可能的中间状态:其一是, 酰胺键在羟基进攻羰基碳之后立即断裂, 从而分别生成-COO-和氮阴离子中间体, 该中间体的阴离子从亲核基团羟基上获得氢[20]; 其二是, 酰胺键的断裂与氮原子的质子化同时发生。两种途径的共同之处是酰胺键断裂, 内酰胺环的氮原子从亲核基团的羟基得到一个质子。然而, NDM-1水解抗生素时的催化状态转瞬即逝, 以上假设提到的中间态并没有得到结构上的证实。此外, 参与水解β-内酰胺环的是亲核基团还是一般碱性基团, 科学界意见并不统一。一种说法是来自周围水环境中大量的水分子与锌离子络合, 贡献出一个质子给活性位点后产生羟基(碱性基团), 这个羟基亲核进攻β-内酰胺环的羰基(图 3); 另一种说法是活性位点附近的水分子把质子贡献给Asp124[21], 或者给了水环境中其他水分子, 这种情况下也能生成羟基来亲核进攻β-内酰胺环的羰基碳原子, 生成羧基中间体, 氮原子得到羟基中的氢原子被质子化。从能垒上来看, 前者能垒约80 kJ·mol-1, 后者需至少200 kJ·mol-1, 因此前者发生的可能性更大[22]。
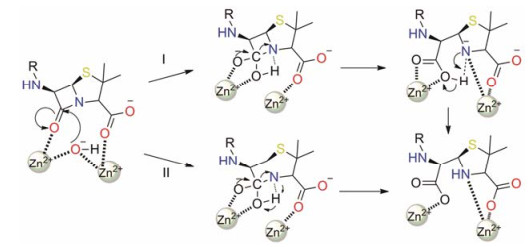
|
Figure 3 The hydrolyzing mechanism of β-lactamase |
近年来, 找到新型有效的金属β-内酰胺酶抑制剂显得十分迫切, 各国科学家对新型抗生素的研究如火如荼地开展着[23]。靶向NDM-1抑制剂的设计研究策略主要包括以下两种思路:一是与碳青霉烯类抗生素(如亚胺培南)联合用药, 试图找到既能保护β-内酰胺环抗生素药效结构不被破坏, 又能起到协同作用从而增强临床用药效果[24], 理论上这种抑制剂应该属于广谱金属β-内酰胺酶抗生素[25]; 二是设计出不被NDM-1水解的新型抗生素, 这是个从无到有、需要更多研发精力去创新和探索的思路, 任重而道远。
4.1 基于已有抗生素发现NDM-1抑制剂目前NDM-1阳性细菌对所有β-内酰胺酶类(包括碳青霉烯类)药物耐药, 仅对多黏菌素、替加环素、氟喹诺酮类等少数抗菌药物敏感[26]。多黏菌素对超级细菌有一定的抗菌活性而被视为抗生素“最后一道防线”, 然而其肾毒性等不良反应, 导致在使用多黏菌素治疗超级细菌感染时受到限制[26]。Kumarasamy等[27]指出硫酸黏菌素(colistin)对从英国、印度清奈、印度哈雅纳三地取样的产NDM-1的大肠杆菌分别达到89%、94%和100%的抑制率, 呈现出较高的抑制活性。然而, 由于硫酸黏菌素自身的神经毒性和肾脏毒性而不能成为理想的治疗NDM-1感染的药物[28]。替加环素和磷霉素(fosfomysin)对抗NDM-1感染的应用也受到限制。替加环素只能用于并发皮肤和皮肤组织感染、并发腹腔感染等。由于替加环素在体内的首过效应显著, 不宜单独用于治疗泌尿道感染[29]。磷霉素不宜单独使用治疗重度感染, 在很多国家它只用于单一治疗泌尿道感染[30]。使用抗生素治疗NDM-1感染时, 主要以微生物药敏试验结果为依据, 然而Bercot等[31]发现硫酸黏菌素、替加环素和磷霉素对产NMD-1的样本菌株只有33%的抑制率。超级细菌不同的病原体可能会影响药敏试验结果重现性。
4.2 基于已知药物分子结构设计发现新型NDM-1抑制剂从发现超级细菌到现在的近10年里, 科研工作者从天然产物[32-34]出发, 抑或从药物的合理设计出发[12, 35-40], 得到了多种新型的NDM-1抑制剂结构。1988年诺贝尔医学奖得主James W. Black爵士曾说过:“新药研发最有效的办法就是从已成功上市的药物开始!”[41]。巯基是非常好的锌离子络合剂[42]。上市药物卡托普利(captopril)是血管紧张素转化酶抑制剂, 分子结构中含有一个巯基, 也已被证实无论是哪种构型(L型或D型)都能一定程度上抑制NDM-1的活性(图 4化合物1)[43]。D-卡托普利结合NDM-1的能力强于L型异构体, 抑制活性IC50值能达到7.9 μmol·L-1, 而L型卡托普利的抑制活性IC50值仅为39.0 μmol·L-1[1, 44]。但是由于临床用药的制约而不能推广用于治疗NDM-1感染[26]。King等[9]已经得到L型卡托普利与NDM-1复合物的晶体数据, 证实了卡托普利的巯基与两个锌离子配位结合。Li等[36]通过药物合理设计的方法得到了IC50为1 μmol·L-1的先导化合物3-巯基-2-甲基丙酮酸结构2 (图 4)。将脯氨酸部分或巯基侧链部分做结构修饰而得到卡托普利的衍生物, 其衍生物在一定程度上也能抑制NDM-1的活性。但是, 将苄胺代替卡托普利中脯氨酸部分得到的衍生物却对NDM-1的抑制活性很弱甚至没有。因此推测巯基和酰胺结构在络合锌离子中起到主要作用, 从而抑制了NDM-1的活性。保留卡托普利结构中的巯基和酰胺, 进一步结构修饰后, 得到IC50值分别为15和10 μmol·L-1的巯基丙醇类化合物3、4 (图 4)。2, 3-二巯基丙醇在临床上用作金属中毒解毒剂, 在此被发掘出抗菌的可能性, 有潜力成为新型抗生素的一员。遗憾的是这项研究并未得到二巯基丙醇与NDM-1复合物的晶体数据, 不能直接说明是巯基络合了NDM-1活性位点中的锌离子。
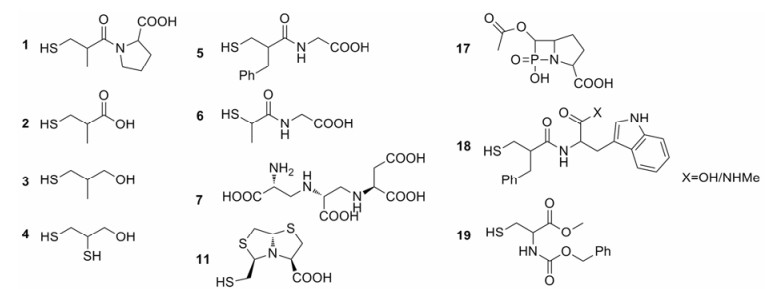
|
Figure 4 NDM-1 inhibitors with captopril analogues |
Klingler等[12]从11个含巯基的已上市药物小分子出发, 研究了这些小分子对金属β-内酰胺酶: NDM-1、VIM-1 (Verona integrin encoded MBLs)和IMP-7 (imipenemase-7)的抑制能力的强弱。尽管这些小分子都含有可以络合锌离子的巯基, 但并不是所有分子都能够抑制金属β-内酰胺酶。因为要达到对金属β-内酰胺酶的抑制不仅要络合活性位点的锌离子, 也要求底物能够很好地结合到活性位点上。通过检测荧光、IC50值和热漂移(thermal shift)找到了最有潜力的NDM-1抑制剂小分子:即2, 3-二巯基丙醇(化合物4, 图 4)、消旋卡多曲的活性代谢物D/L-硫芬(化合物5, 图 4)、D/L-卡托普利和硫普罗宁(化合物6, 图 4)。尽管硫普罗宁抑制NDM-1的效果不如抑制VIM-1和IMP-7, 但是他们得到了硫普罗宁与NDM-1复合物晶体数据(PDB: 5A5Z), 直接证明了硫普罗宁中的硫原子与NDM-1中的锌离子络合, 这为探究底物的抑制机制提供了有力的证据。
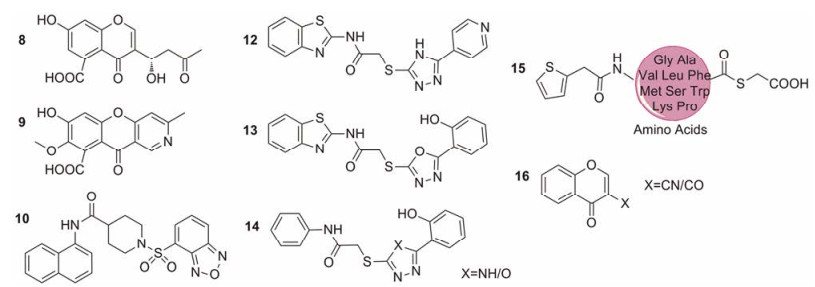
4.3 基于经典的天然产物发现新型NDM-1抑制剂血管紧张素转化酶与金属β-内酰胺酶在某些功能上有相似之处, 都是依赖锌离子的金属蛋白酶[45]。天然化合物曲霉明A (aspergillomarasmine A, AMA) (化合物7, 图 4)能够抑制血管紧张素转化酶, King等[32]从变色曲霉菌(Aspergillus versicolor)的提取物中发现了曲霉明A, 推测曲霉明A可能也会通过与锌离子的相互作用抑制NDM-1。该研究通过以小鼠为模型的动物实验证实了曲霉明A的确能够“捕捉” NDM-1里的锌离子, 在联合用药情况下能够恢复碳青霉烯类抗生素药物的杀菌活性, 进而杀死表达NDM-1的大肠杆菌。曲霉明A的全合成路线最早于2016年由北京大学雷晓光课题组Liao等[46]发布, 并且证实了构型翻转的曲霉明A (R, R, S-AMA)同样能够抑制NDM-1。Gan等[33]从新疆喀纳斯湖真菌Penucillium sp I09F484中粗提得到两种色酮类化合物: IC50分别为94 μmol·L-1的色酮化合物8和89 μmol·L-1的色酮化合物9 (图 5)。Thakur等[34]选取35个天然抗菌药物作为配体库, 与NDM-1 (PDB: 3Q6X)进行对接, 根据底物结合能和IC50模拟值推测印苦楝内酯(nimbolide)和楝树油活性成分(isomargololone)有可能抑制NDM-1活性。然而该团队后续并没有提供相关体外/体内活性实验数据来支持这两个三环化合物的抗NDM-1活性的能力。
|
Figure 5 NDM-1 inhibitors (chromone and oxazole) |
目前, 利用计算机辅助药物设计(computer-aided drug design)方法, 通过虚拟筛选、动力学模拟等手段获得低毒性、新结构、高活性的NDM-1抑制剂, 并指导NDM-1抑制剂分子的设计与改造已取得了一定的成效。计算机辅助NDM-1药物设计, 可以极大的缩短研发时间并降低成本。利用计算机辅助药物设计的核心思想通过建立配体库, 再利用高通量筛选或者药效团筛选等方法寻找在理论模拟上有潜在抑制活性的小分子, 是当下寻找新型先导化合物的最高效的方法。利用现有的小分子数据库来搭建配体库, 例如NCBI PubMed、ChemSpide、ZINC等, 受体则是来自PDB数据库中晶体结构, 通过不同计算方法缩小配体库, 最终找到理论上最有潜力的NDM-1抑制剂的结构。
Wang等[39]从ZINC数据库选出28 000个类药性小分子。受体分子选择氨苄西林。然后进行基于形状互补的对接, 在对接结果中限制一定的绝对能量值、相对能量值、对接打分值等, 从而将配体库逐步精简到298个。基于配体在对接过程中的得分、结构多样性、与受体之间相互作用、是否市售可得和极性、非极性与活性位点互补结合的总体情况, 挑选出44种化合物进入下一步体外生物活性实验。最终得到3个对NDM-1有一定抑制活性的磺胺类化合物, 其中最具潜力的是含有苯并二噁唑结构的磺胺类化合物10 (图 5)。用分子动力学模拟抑制剂与NDM-1之间的相互作用, 理论结果显示苯并二噁唑和His122的芳香环之间有π-π堆积作用, 磺胺结构与Zn2、Asn220、His120、His122、His189、起关键作用的水分子/羟基都可发生相互作用。
作者在探索NDM-1抑制剂的研究中也运用了计算机辅助药物设计的方法:卡托普利是目前公认的对NDM-1有抑制作用的上市药物, 故选取了结合有L-卡托普利的NDM-1晶体数据(PDB: 4EXS)来构建基于受体和配体之间相互作用模式的药效团模型。在构建药效团模型时, 将活性口袋中的锌离子视作氢键供体, 并保留这两个氢键供体矢量(feature)。建立测试集来缩减初次模型中的特征矢量, 最终得到含氢键供体、氢键受体、疏水作用等7个特征矢量的NDM-1抑制剂药效团模型。以廉价易得的天然氨基酸脯氨酸为母核, 基于组合化学理念创建了配体库。利用Lipinski类药五原则对庞大的配体库进行初次筛选, 摒除那些不适合成为药物的分子, 缩小筛选的范围并降低研发成本。然后利用药效团进行复筛, 最终得到了一些有望成为NDM-1抑制剂的磺胺类小分子。结合生物学实验结果的反馈进行结构修饰和优化, 期望得到更优良的NDM-1抑制剂。
基于含有巯基[25, 47]、硫代磷酸[48]、青霉烷[49]结构的化合物对金属β-内酰胺酶有广谱抑制活性, Gonzalez等[11]根据金属β-内酰胺酶识别底物的作用机制推测含有双环结构的底物更容易结合到金属β-内酰胺酶上, 从而设计出类似苄基青霉素(benzylpenicillin)和亚胺培南(imipenem)的杂原子双环系列化合物, 最终找到了IC50值达23 μmol·L-1的bisthiazolidine A (图 4化合物11), 并且得到了bisthiazolidine A与NDM-1复合物的晶体结构(PDB: 4U4L), 再次证明了巯基在“捕捉”活性位点的锌离子的重要作用。Saiz等[50]发现具有将bisthiazolidine A杂原子双环结构引入氰基而得到的衍生物也有对NDM-1抑制作用。
Zhang等[37]在探索广谱抗菌小分子研究中发现一些含三唑(thioazoles)结构的化合物能抑制金属β-内酰胺酶活性。据此他们设计并合成了18个双芳基取代的唑基硫代乙酰胺(diaryl-substituted azolylthioacetamides)小分子, 选取了4个代表性的金属β-内酰胺酶亚型: CcrA (B1a)、NDM-1 (B1b)、ImiS (B2)和L1 (B3)来测定候选小分子的生物活性。结果得到三唑化合物12~14 (图 5)。对这4种金属β-内酰胺酶都有广谱抗菌效果, 其中三唑化合物14 (X=NH)抑制NDM-1的效果最好。在杨科武课题组后续的研究中, Liu等[38]发现巯基乙酸硫酯(2-thiopheneacetyl mercaptoacetic acid thioesters)结构15 (图 5)对包括NDM-1在内的金属β-内酰胺酶L1类有显著抑制作用。
Christopeit等[51]结合了表面离子共振(surface plasmon resonance, SPR)技术和酶活测定, 利用正交筛选法组建了配体库, 从中发现了具有抑制NDM-1活性的结构片段结构——含氰基的色酮16(图 5, 化合物16, X=CN)。利用电喷射离子化-质谱联用(electrospray ionization-mass spectrometry, ESI-MS)和单一的定点突变(single site directed mutagenesis)方法证实了将醛基代替氰基得到的化合物(图 5, 化合物16, X=CO)通过与活性位点的Lys224形成共价键而抑制NDM-1活性[52]。这些发现为设计共价抑制剂奠定了研究基础。
5 检测NDM-1抑制剂生物活性的方法 5.1 生物学测定方法在定量分析β-内酰胺类抗生素的水解程度时可利用紫外吸收光谱法来衡量, 因为β-内酰胺环的水解即羰基氧原子与氮原子之间的化学键的断裂导致结构的变化而引起吸光度的变化[53]。头孢硝噻吩(nitrocefin)在波长380 nm附近呈现黄色, 被水解后在500 nm附近呈现红色, 利用这一现象可以定性分析头孢硝噻吩是否被水解的情况[54]。所以头孢硝噻吩是做定性定量分析最佳候选者。为保护抗生素不被水解, 可将已有抗生素和候选抑制剂与NDM-1共同培养, 测定已有抗生素的最低抑制浓度(MIC)或者IC50, 或者被水解的情况[55]。
Liao等[46]在测定两种构型的曲霉明A的活性(R, R, S-AMA; S, S, S-AMA)时, 将同一浓度的头孢硝噻吩加入到预培养的NDM-1与不同浓度的曲霉明A样品中, 进而测定IC50值。有趣的是R, R, S-AMA并没有抑制活性, S, S, S-AMA的抑制效果很高, IC50为8.1 μmol·L-1。Yang等[56]用磷原子代替β-内酰胺环中的羰基氧原子得到β-phospholactam (图 4, 化合物17), 通过检测头孢硝噻吩在485 nm的紫外吸收光度值定量分析β-phospholactam对IMP-1, CcrA和L1, Bla2和NDM-1的抑制率。虽然β-phospholactam对NDM-1抑制效果并不是很好(抑制率53%), 并且很有可能受培养时间影响, 但这可能为研发新型骨架的抗生素起到了抛砖引玉的作用。这些检测方法的本质仍是利用经典的抗菌生物学测定方法, 在定量衡量抑制剂生物活性的时候选取最低抑制浓度(MIC)或者半数致死率(IC50)。然而, 经典的抗菌生物学测定方法不仅操作繁琐, 检测周期较为严格并且都需要一定的培养时间, 检测周期长, 同时容易受到外界的干扰。
5.2 化学分析方法鉴于经典的生物学抗菌测定方法在实际操作过程中的影响因素颇多, 从而降低了测定结果的可靠性, 很多科学家开发出了1H NMR波谱、质谱等化学分析法测定NDM-1抑制剂生物活性。通过测定抗生素被水解的情况或者抗生素小分子从产NDM-1菌株中解离出来的情况, 间接反映NDM-1抑制剂在联合用药过程中的活性。
Ma等[57]设计出一种1H NMR波谱分析法来监测产NDM-1的大肠杆菌水解美罗培南(meropenem)的过程。当美罗培南被水解后, C4侧链和甲基的化学位移因此发生变化, 进而能反映出底物被水解的情况。利用这种监测方法, 确定了L型卡托普利和螯合剂乙二胺四乙酸(EDTA)对NDM-1的抑制作用。通过该监测方法预测出含有巯基的胺类是潜在的NDM-1抑制剂, 他们发现了化合物卡托普利骨架衍生物(图 4, 化合物18)。Gonzalez等[11]正是利用的这种检测方法证实了Bisthiazolidine A的生物活性。1H NMR波谱分析法不仅可以运用到细菌细胞实验中, 也可以运用到更广泛复杂的生物基质中例如真核细胞。在膜蛋白的体外实验中, 经典的准备步骤(例如表面活化、纯化、折叠与重建)经常会减弱膜蛋白的活性。而运用1H NMR波谱分析法则会保留原本的膜蛋白完整性, 直接在样本的自然状态下测量数据, 且不需要其他的实验操作。这种情况下得到的核磁共振数据与生理环境下的生物学功能十分接近。1H NMR波谱分析法也可以应用到基于靶标的细胞筛选法来发现NDM-1抑制剂中。与一般的表型全细胞筛选相比, 1H NMR波谱分析法不需要靶标预解析, 只需通过对底物/酶复合物直接的波谱分析即可检测目标酶的活性。
Chen等[35]开发了基于质谱的分析方法鉴定NDM-1新配体, 并得到了含巯基的NDM-1抑制剂(图 4, 化合物19)。该法分直接法和间接法。所谓直接法就是将孵化的蛋白和抑制剂底物的复合物, 直接进行纳升电离(nanoelectrospray ionization, nanoESI)然后进行质谱分析, 通过量化纯蛋白和结合底物蛋白的比例, 计算出解离常数, 解离常数对应着底物对蛋白的结合力和相互作用信息[58, 59]。所谓间接法, 是将蛋白和抑制剂的复合物通过超滤而纯化出来, 然后做液相色谱-质谱(LC-MS)分析。间接法建立在液质联用监测底物从复合物中解离出来的情况, 从而评估底物结合的特异性。通过超滤[60-62]、透析[63]、亲和纯化、排阻色谱[64-66]等方法从蛋白-底物复合物中分离出游离的底物。然而这两种方法各有利弊。直接法运用化学计量学的核心想法在很多工作中都有研究使用, 例如筛选可作为治疗剂的小分子、短肽和碳水化合物[61, 67-69]。缺点在于检测底物和蛋白结合的条件需要与后续质谱分析的条件相同, 例如缓冲溶液、pH值等。另外在纳升电离过程中复合物的气相解离和非特异性结合都可造成假阴性或者假阳性结果。间接法虽然不涉及化学计量学, 但其可以完全保留水溶液中底物跟蛋白之间的相互作用[70, 71], 以高效且高通量的方式筛选组合化学库和天然产物[60, 61]。然而间接法在辨别假阴性(短暂或者共价结合)和假阳性(底物结合在非活性位点)结果时就显不足[72]。
6 结语在抗微生物化学治疗中最大的威胁就是由NDM-1引起了广谱抗生素的耐药性, 人类急需尽快研发出有效的抗生素来对抗超级细菌。锌离子络合剂可以捕获生物膜上的锌离子, 进而降解细菌生物膜, 达到杀死细菌的目的, 成为了研发NDM-1的抑制剂重要的出发点[73, 74]。由于人类蛋白约三分之一都是含金属蛋白, 因此, 在从锌离子络合剂角度出发研发NDM-1抑制剂时, 要考虑到抑制剂的选择性和非特异性, 以及可能引起的生物学效应。除了从天然产物提取分离等传统方法, 近年来迅速发展的计算机辅助药物虚拟筛选方法更为NDM-1抑制剂的研发提供了有力的辅助, 大大提高了新药研发的效率[75]。当所得到的NDM-1晶体结构分辨率越高, 越有利于快速而准确地分析配体分子与受体之间的相互作用, 进而更加便于利用计算机辅助药物设计的方法快速高效地找到潜在的新型抗生素[76]。科学技术的发展和检测手段的升级势必为研发NDM-1抑制剂起到助力作用。同时, 先导化合物的发现到临床实验的转化等诸多问题亟待解决。
| [1] | Yong D, Toleman MA, Giske CG, et al. Characterization of a new metallo-beta-lactamase gene, bla(NDM-1), and a novel erythromycin esterase gene carried on a unique genetic structure in Klebsiella pneumoniae sequence type 14 from India[J]. Antimicrob Agents Chemother, 2009, 53: 5046–5054. DOI:10.1128/AAC.00774-09 |
| [2] | Day KM, Ali S, Mirza IA, et al. Prevalence and molecular characterization of Enterobacteriaceae producing NDM-1 carbapenemase at a military hospital in Pakistan and evaluation of two chromogenic media[J]. Diagn Micr Infec Dis, 2013, 75: 187–191. DOI:10.1016/j.diagmicrobio.2012.11.006 |
| [3] | Wailan AM, Paterson DL, Caffery M, et al. Draft genome sequence of NDM-5-producing Escherichia coli sequence type 648 and genetic context of blaNDM-5 in Australia[J]. Genome Announc, 2015, 3: e00194–00115. |
| [4] | Bush K, Fisher JF. Epidemiological expansion, structural studies, and clinical challenges of new beta-lactamases from Gram-negative bacteria[J]. Ann Rev Microbiol, 2011, 65: 455–478. DOI:10.1146/annurev-micro-090110-102911 |
| [5] | Rolain JM, Parola P, Cornaglia G. New Delhi metallo-beta-lactamase (NDM-1):towards a new pandemia?[J]. Clin Microbiol Infect, 2010, 16: 1699–1701. DOI:10.1111/j.1469-0691.2010.03385.x |
| [6] | Cornaglia G, Giamarellou H, Rossolini GM. Metallo-β-lactamases:a last frontier for β-lactams?[J]. Lancet Infect Dis, 2011, 11: 381–393. DOI:10.1016/S1473-3099(11)70056-1 |
| [7] | Zhang H, Hao Q. Crystal structure of NDM-1 reveals a common beta-lactam hydrolysis mechanism[J]. FASEB J, 2011, 25: 2574–2582. DOI:10.1096/fj.11-184036 |
| [8] | Kim Y, Tesar C, Mire J, et al. Structure of apo-and mono-metalated forms of NDM-1-a highly potent carbapenem-hydrolyzing metallo-beta-lactamase[J]. PLoS One, 2011, 6: e24621. DOI:10.1371/journal.pone.0024621 |
| [9] | King DT, Worrall LJ, Gruninger R, et al. New Delhi met-allo-beta-lactamase:structural insights into beta-lactam recognition and inhibition[J]. J Am Chem Soc, 2012, 134: 11362–11365. DOI:10.1021/ja303579d |
| [10] | Feng H, Ding J, Zhu D, et al. Structural and mechanistic insights into NDM-1 catalyzed hydrolysis of cephalosporins[J]. J Am Chem Soc, 2014, 136: 14694–14697. DOI:10.1021/ja508388e |
| [11] | Gonzalez MM, Kosmopoulou M, Mojica MF, et al. Bisthiazolidines:a substrate-mimicking scaffold as an inhibitor of the NDM-1 carbapenemase[J]. ACS Infect Dis, 2015, 1: 544–554. DOI:10.1021/acsinfecdis.5b00046 |
| [12] | Klingler FM, Wichelhaus TA, Frank D, et al. Approved drugs containing thiols as inhibitors of metallo-beta-lactamases:strategy to combat multidrug-resistant bacteria[J]. J Med Chem, 2015, 58: 3626–3630. DOI:10.1021/jm501844d |
| [13] | Green VL, Verma A, Owens RJ, et al. Structure of New Delhi metallo-beta-lactamase 1(NDM-1)[J]. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2011, 67: 1160–1164. DOI:10.1107/S1744309111029654 |
| [14] | Guo Y, Wang J, Niu G, et al. A structural view of the antibiotic degradation enzyme NDM-1 from a superbug[J]. Protein Cell, 2011, 2: 384–394. DOI:10.1007/s13238-011-1055-9 |
| [15] | King D, Strynadka N. Crystal structure of New Delhi met-allo-beta-lactamase reveals molecular basis for antibiotic resistance[J]. Protein Sci, 2011, 20: 1484–1491. DOI:10.1002/pro.697 |
| [16] | Wang Z, Fast W, Valentine AM, et al. Met-allo-beta-lactamase:structure and mechanism[J]. Curr Opin Chem Biol, 1999, 3: 614–622. DOI:10.1016/S1367-5931(99)00017-4 |
| [17] | Page MI, Badarau A. The mechanisms of catalysis by metallo beta-lactamases[J]. Bioinorg Chem Appl, 2008, 3: 249–259. |
| [18] | Jacquin O, Balbeur D, Damblon C, et al. Positively cooperative binding of zinc ions to Bacillus cereus 569/H/9 beta-lactamase Ⅱ suggests that the binuclear enzyme is the only relevant form for catalysis[J]. J Mol Biol, 2009, 392: 1278–1291. DOI:10.1016/j.jmb.2009.07.092 |
| [19] | Thomas PW, Zheng M, Wu S, et al. Characterization of purified New Delhi metallo-beta-lactamase-1[J]. Biochemistry, 2011, 50: 10102–10113. DOI:10.1021/bi201449r |
| [20] | Wang Z, Fast W, Benkovic SJ. On the mechanism of the metallo-beta-lactamase from Bacteroides fragilis[J]. Biochemistry, 1999, 38: 10013–10023. DOI:10.1021/bi990356r |
| [21] | Zheng M, Xu D. New Delhi metallo-beta-lactamase I:substrate binding and catalytic mechanism[J]. J Phys Chem B, 2013, 117: 11596–11607. DOI:10.1021/jp4065906 |
| [22] | Kim Y, Cunningham MA, Mire J, et al. NDM-1, the ultimate promiscuous enzyme:substrate recognition and catalytic mechanism[J]. FASEB J, 2013, 27: 1917–1927. DOI:10.1096/fj.12-224014 |
| [23] | Buynak JD. beta-Lactamase inhibitors:a review of the patent literature (2010-2013)[J]. Expert Opin Ther Pat, 2013, 23: 1469–1481. DOI:10.1517/13543776.2013.831071 |
| [24] | Livermore DM, Mushtaq S, Morinaka A, et al. Activity of carbapenems with ME1071(disodium 2, 3-diethylmaleate) against Enterobacteriaceae and Acinetobacter spp. With carbapenemases, including NDM enzymes[J]. J Antimicrob Chemother, 2013, 68: 153–158. DOI:10.1093/jac/dks350 |
| [25] | Lienard BM, Garau G, Horsfall L, et al. Structural basis for the broad-spectrum inhibition of metallo-beta-lactamases by thiols[J]. Org Biomol Chem, 2008, 6: 2282–2294. DOI:10.1039/b802311e |
| [26] | Rogers BA, Sidjabat HE, Silvey A, et al. Treatment options for New Delhi metallo-beta-lactamase-harboring Enterobacteriaceae[J]. Microb Drug Resist, 2013, 19: 100–103. DOI:10.1089/mdr.2012.0063 |
| [27] | Kumarasamy KK, Toleman MA, Walsh TR, et al. Emergence of a new antibiotic resistance mechanism in India, Pakistan, and the UK:a molecular, biological, and epidemiological study[J]. Lancet Infect Dis, 2010, 10: 597–602. DOI:10.1016/S1473-3099(10)70143-2 |
| [28] | Li J, Nation RL, Turnidge JD, et al. Colistin:the re-emerging antibiotic for multidrug-resistant Gram-negative bacterial infections[J]. Lancet Infect Dis, 2006, 6: 589–601. DOI:10.1016/S1473-3099(06)70580-1 |
| [29] | Livermore DM, Warner M, Mushtaq S, et al. What remains against carbapenem-resistant Enterobacteriaceae? Evaluation of chloramphenicol, ciprofloxacin, colistin, fosfomycin, minocycline, nitrofurantoin, temocillin and tigecycline[J]. Int J Antimicrob Agents, 2011, 37: 415–419. DOI:10.1016/j.ijantimicag.2011.01.012 |
| [30] | Nordmann P, Poirel L, Walsh TR, et al. The emerging NDM carbapenemases[J]. Trends Microbiol, 2011, 19: 588–595. DOI:10.1016/j.tim.2011.09.005 |
| [31] | Bercot B, Poirel L, Dortet L, et al. In vitro evaluation of antibiotic synergy for NDM-1-producing Enterobacteriaceae[J]. J Antimicrob Chemother, 2011, 66: 2295–2297. DOI:10.1093/jac/dkr296 |
| [32] | King AM, Reid-Yu SA, Wang W, et al. Aspergillo-marasmine A overcomes metallo-beta-lactamase antibiotic resistance[J]. Nature, 2014, 510: 503–506. DOI:10.1038/nature13445 |
| [33] | Gan M, Liu Y, Bai Y, et al. Polyketides with New Delhi metallo-beta-lactamase 1 inhibitory activity from Penicillium sp[J]. J Nat Prod, 2013, 76: 1535–1540. DOI:10.1021/np4000944 |
| [34] | Thakur PK, Kumar J, Ray D, et al. Search of potential inhibitor against New Delhi metallo-beta-lactamase 1 from a series of antibacterial natural compounds[J]. J Nat Sci Biol Med, 2013, 4: 51–56. DOI:10.4103/0976-9668.107260 |
| [35] | Chen X, Li L, Chen S, et al. Identification of inhibitors of the antibiotic-resistance target New Delhi met-allo-beta-lactamase 1 by both nanoelectrospray ionization mass spectrometry and ultrafiltration liquid chromatography/mass spectrometry approaches[J]. Anal Chem, 2013, 85: 7957–7965. DOI:10.1021/ac401732d |
| [36] | Li N, Xu Y, Xia Q, et al. Simplified captopril analogues as NDM-1 inhibitors[J]. Bioorg Med Chem Lett, 2014, 24: 386–389. DOI:10.1016/j.bmcl.2013.10.068 |
| [37] | Zhang YL, Yang KW, Zhou YJ, et al. Diaryl-substituted azolylthioacetamides:inhibitor discovery of New Delhi metallo-beta-lactamase-1(NDM-1)[J]. ChemMedChem, 2014, 9: 2445–2448. DOI:10.1002/cmdc.201402249 |
| [38] | Liu XL, Shi Y, Kang JS, et al. Amino acid thioester deriva-tives:a highly promising scaffold for the development of metallo-beta-lactamase L1 inhibitors[J]. ACS Med Chem Lett, 2015, 6: 660–664. DOI:10.1021/acsmedchemlett.5b00098 |
| [39] | Wang X, Lu M, Shi Y, et al. Discovery of novel New Delhi metallo-beta-lactamases-1 inhibitors by multistep virtual screening[J]. PLoS One, 2015, 10: e0118290. DOI:10.1371/journal.pone.0118290 |
| [40] | Worthington RJ, Bunders CA, Reed CS, et al. Small mole-cule suppression of carbapenem resistance in NDM-1 producing Klebsiella pneumoniae[J]. ACS Med Chem Lett, 2012, 3: 357–361. DOI:10.1021/ml200290p |
| [41] | Raju TNK. The Nobel chronicles[J]. Lancet, 2000, 356: 81. |
| [42] | Marrs EC, Day KM, Perry JD. In vitro activity of mecil-linam against Enterobacteriaceae with NDM-1 carbapenemase[J]. J Antimicrob Chemother, 2014, 69: 2873–2875. DOI:10.1093/jac/dku204 |
| [43] | Bebrone C. Metallo-beta-lactamases (classification, ac-tivity, genetic organization, structure, zinc coordination) and their superfamily[J]. Biochem Pharmacol, 2007, 74: 1686–1701. DOI:10.1016/j.bcp.2007.05.021 |
| [44] | Bonomo RA. New Delhi metallo-beta-lactamase and mul-tidrug resistance:a global SOS?[J]. Clin Infect Dis, 2011, 52: 485–487. DOI:10.1093/cid/ciq179 |
| [45] | English WR, Corvol P, Murphy G. LPS activates ADAM9 dependent shedding of ACE from endothelial cells[J]. Biochem Biophys Res Commun, 2012, 421: 70–75. DOI:10.1016/j.bbrc.2012.03.113 |
| [46] | Liao D, Yang S, Wang J, et al. Total synthesis and structural reassignment of aspergillomarasmine A[J]. Angew Chem Int Ed Engl, 2016, 55: 4291–4295. DOI:10.1002/anie.201509960 |
| [47] | Mollard C, Moali C, Papamicael C, et al. Thiomandelic acid, a broad spectrum inhibitor of zinc beta-lactamases:kinetic and spectroscopic studies[J]. J Biol Chem, 2001, 276: 45015–45023. DOI:10.1074/jbc.M107054200 |
| [48] | Lassaux P, Hamel M, Gulea M, et al. Mercapto-phosphonate compounds as broad-spectrum inhibitors of the metallo-beta-lactamases[J]. J Med Chem, 2010, 53: 4862–4876. DOI:10.1021/jm100213c |
| [49] | Johnson JW, Gretes M, Goodfellow VJ, et al. Cyclobutanone analogues of beta-lactams revisited:insights into conformational requirements for inhibition of serine-and metallo-beta-lactamases[J]. J Am Chem Soc, 2010, 132: 2558–2560. DOI:10.1021/ja9086374 |
| [50] | Saiz C, Villamil V, Gonzalez MM, et al. Enantioselective synthesis of new oxazolidinylthiazolidines as enzyme inhibittors[J]. Tetrahedron-Asymmetr, 2017, 28: 110–117. DOI:10.1016/j.tetasy.2016.11.002 |
| [51] | Christopeit T, Leiros HK. Fragment-based discovery of inhibitor scaffolds targeting the metallo-beta-lactamases NDM-1 and VIM-2[J]. Bioorg Med Chem Lett, 2016, 26: 1973–1977. DOI:10.1016/j.bmcl.2016.03.004 |
| [52] | Christopeit T, Albert A, Leiros HK. Discovery of a novel covalent non-beta-lactam inhibitor of the met-allo-beta-lactamase NDM-1[J]. Bioorg Med Chem, 2016, 24: 2947–2953. DOI:10.1016/j.bmc.2016.04.064 |
| [53] | Yang KW, Zhou Y, Ge Y, et al. Real-time activity moni-toring of New Delhi metallo-beta-lactamase-1 in living bacterial cells by UV-Vis spectroscopy[J]. Chem Commun (Camb), 2017, 53: 8014–8017. DOI:10.1039/C7CC02774E |
| [54] | O'Callaghan CH, Morris A, Kirby SM, et al. Novel method for detection of beta-lactamases by using a chromogenic cephalosporin substrate[J]. Antimicrob Agents Chemother, 1972, 1: 283–288. DOI:10.1128/AAC.1.4.283 |
| [55] | Drawz SM, Bonomo RA. Three decades of beta-lactamase inhibitors[J]. Clin Microbiol Rev, 2010, 23: 160–201. DOI:10.1128/CMR.00037-09 |
| [56] | Yang KW, Feng L, Yang SK, et al. New beta-phospholactam as a carbapenem transition state analog:synthesis of a broad-spectrum inhibitor of metallo-beta-lactamases[J]. Bioorg Med Chem Lett, 2013, 23: 5855–5859. DOI:10.1016/j.bmcl.2013.08.098 |
| [57] | Ma J, Mcleod S, Maccormack K, et al. Real-time monitoring of New Delhi metallo-beta-lactamase activity in living bacterial cells by 1H-NMR spectroscopy[J]. Angew Chem Int Ed Engl, 2014, 53: 2130–2133. DOI:10.1002/anie.201308636 |
| [58] | Duffell KM, Hudson SA, McLean KJ, et al. Nanoelectros-pray ionization mass spectrometric study of Mycobacterium tuberculosis CYP121-ligand interactions[J]. Anal Chem, 2013, 85: 5707–5714. DOI:10.1021/ac400236z |
| [59] | Xu N, Yang H, Cui M, et al. High-performance liquid chromatography-electrospray ionization-mass spectrometry ligand fishing assay:a method for screening triplex DNA binders from natural plant extracts[J]. Anal Chem, 2012, 84: 2562–2568. DOI:10.1021/ac202796v |
| [60] | Mulabagal V, Calderon AI. Development of an ultrafiltra-tion-liquid chromatography/mass spectrometry (UF-LC/MS) based ligand-binding assay and an LC/MS based functional assay for Mycobacterium tuberculosis shikimate kinase[J]. Anal Chem, 2010, 82: 3616–3621. DOI:10.1021/ac902849g |
| [61] | Choi Y, Jermihov K, Nam SJ, et al. Screening natural products for inhibitors of quinone reductase-2 using ul-trafiltration LC-MS[J]. Anal Chem, 2011, 83: 1048–1052. DOI:10.1021/ac1028424 |
| [62] | Liu D, Guo J, Luo Y, et al. Screening for ligands of human retinoid X receptor-alpha using ultrafiltration mass spectrometry[J]. Anal Chem, 2007, 79: 9398–9402. DOI:10.1021/ac701701k |
| [63] | Orsak T, Smith TL, Eckert D, et al. Revealing the allos-terome:systematic identification of metabolite-protein interactions[J]. Biochemistry, 2012, 51: 225–232. DOI:10.1021/bi201313s |
| [64] | Moy FJ, Haraki K, Mobilio D, et al. MS/NMR:a structure-based approach for discovering protein ligands and for drug design by coupling size exclusion chromatography, mass spectrometry, and nuclear magnetic resonance spectroscopy[J]. Anal Chem, 2001, 73: 571–581. DOI:10.1021/ac0006270 |
| [65] | Wabnitz PA, Loo JA. Drug screening of pharmaceutical discovery compounds by micro-size exclusion chromatography/mass spectrometry[J]. Rapid Commun Mass Sp, 2002, 16: 85–91. DOI:10.1002/(ISSN)1097-0231 |
| [66] | Blom KF, Larsen BS, McEwen CN. Determining affinity-selected ligands and estimating binding affinities by online size exclusion chromatography/liquid chromatography-mass spectrometry[J]. J Comb Chem, 1999, 1: 82–90. DOI:10.1021/cc980010k |
| [67] | Mulabagal V, Calderon AI. Development of binding assays to screen ligands for Plasmodium falciparum thioredoxin and glutathione reductases by ultrafiltration and liquid chromatography/mass spectrometry[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2010, 878: 987–993. DOI:10.1016/j.jchromb.2010.02.030 |
| [68] | Johnson BM, Nikolic D, van Breemen RB. Applications of pulsed ultrafiltration-mass spectrometry[J]. Mass Spectrom Rev, 2002, 21: 76–86. DOI:10.1002/(ISSN)1098-2787 |
| [69] | Clark SM, Konermann L. Determination of ligand-protein dissociation constants by electrospray mass spectrome-try-based diffusion measurements[J]. Anal Chem, 2004, 76: 7077–7083. DOI:10.1021/ac049344o |
| [70] | van Breemen RB, Huang CR, Nikolic D, et al. Pulsed ultrafiltration mass spectrometry:a new method for screening combinatorial libraries[J]. Anal Chem, 1997, 69: 2159–2164. DOI:10.1021/ac970132j |
| [71] | Zhao YZ, van Breemen RB, Nikolic D, et al. Screening solution-phase combinatorial libraries using pulsed ultrafiltration/electrospray mass spectrometry[J]. J Med Chem, 1997, 40: 4006–4012. DOI:10.1021/jm960729b |
| [72] | Li X, Gianoulis TA, Yip KY, et al. Extensive in vivo metabolite-protein interactions revealed by large-scale systematic analyses[J]. Cell, 2010, 143: 639–650. DOI:10.1016/j.cell.2010.09.048 |
| [73] | Drawz SM, Papp-Wallace KM, Bonomo RA. New beta-lactamase inhibitors:a therapeutic renaissance in an MDR world[J]. Antimicrob Agents Chemother, 2014, 58: 1835–1846. DOI:10.1128/AAC.00826-13 |
| [74] | Martti V. Agents that increase the permeability of the outer membrane[J]. Microbiol Rev, 1992, 56: 395–411. |
| [75] | Randhawa V, Jamwal R. Molecular modeling and virtual screening studies of NDM-1 beta lactamase for identification of a series of potent inhibitors[J]. Int Res J Biochem Bioinform, 2011, 1: 95–102. |
| [76] | Oelschlaeger P, Ai N, Duprez KT, et al. Evolving carbap-enemases:can medicinal chemists advance one step ahead of the coming storm?[J]. J Med Chem, 2010, 53: 3013–3027. DOI:10.1021/jm9012938 |
 2018, Vol. 53
2018, Vol. 53


