近年来, 基因编辑领域取得了飞速的发展, 锌指核酸酶(zinc finger nucleases, ZFNs)[1]、转录激活因子样效应物核酸酶(transcription activator-like effector nucleases, TALENs)[2, 3]和CRISPR/Cas (clus tered regularly interspaced short palindromic repeats/CRISPR-associated)系统大大改变了科研人员在哺乳动物系统中研究基因及其功能的方式。相较于ZFN和TALEN采用蛋白质作为靶标识别物, CRISPR/Cas9采用特殊小向导RNA (small guide RNA, sgRNA)靶向目标基因, 操作简单快捷, 成本低廉, 脱靶效应较低[4], 已经成为基因功能研究领域强有力的武器。在新药研发过程中, 高效的靶点筛选和验证是极其关键的步骤。CRISPR/Cas9技术的出现实现了对靶基因的定点编辑, 使得构建动物模型或细胞系模型更加便捷, 极大地加速了药物靶点的筛选验证及新药的研发。
1 CRISPR/Cas9系统的发现历史和作用机制基因编辑技术CRISPR/Cas9被《科学》杂志列为2013年年度十大科技进展之一, 被称为“魔剪”和“上帝之手”, 它的迅速崛起引发了生物医学研究的革命。CRISPR是规律间隔性成簇短回文重复序列的简称, Cas是CRISPR相关蛋白的简称[5]。早在1987年, 日本科学家在对K12大肠杆菌碱性磷酸酶基因编码区进行研究时发现附近有串联间隔重复序列[6], 但功能尚不清楚。后来的研究发现, 这种重复序列广泛存在于细菌和古细菌中, 并于2002年正式命名[7]。随着测序技术和生物信息学的发展, 2005年, 3个研究组同时发现间隔序列和感染细菌的病毒或噬菌体基因序列高度同源[8−10], 从而推测这一系统可能是细菌抵御病毒和质粒入侵的一种方式。2007年, DANISCO公司的科学家通过增加和敲除CRISPR位点中间的重复序列调节了嗜热链球菌(Streptococcus thermophilus)对噬菌体的敏感性[11]。2008年, Marraffini等[12]发现细菌CRISPR系统能阻止外源质粒的转移, 首次用实验验证了该系统的功能。
基于重复序列结构和Cas蛋白亚类, CRISPR/Cas系统可以分为3类[13]: Ⅰ型、Ⅱ型和Ⅲ型。相较Ⅰ型和Ⅲ型系统, Ⅱ型CRISPR/Cas系统(即CRISPR/Cas9系统)成分最简单, 已经被改造成为理想的基因编辑工具。2012年, Wiedenheft等[14]阐明了Ⅱ型系统的机制。CRISPR/Cas9系统包含3个关键性组分: CRISPR RNA (crRNA)、反式激活crRNA (transactivating crRNA, tracrRNA)及Cas9核酸内切酶。crRNA的一部分序列可与tracrRNA通过碱基互补配对, 结合形成嵌合RNA (tracrRNA/crRNA), 另一部分则可与靶标DNA位点进行碱基互补配对。这种嵌合RNA能够识别特定的NGG序列, 即PAM位点, 并引导Cas蛋白复合物结合到这个特定的位点上对DNA双链进行切割[15]。通过对tracrRNA/crRNA进行改造, 可以构建成一个具有引导作用的sgRNA, 从而将Cas9系统进一步简化为Cas9核酸内切酶和sgRNA两个组分。
当Cas9对靶标DNA进行切割时, 会造成DNA双链断裂(double strand break, DSB), 随即细胞启动DNA修复机制。在非同源末端连接(non-homologous end joining, NHEJ)修复机制下能够形成随机的多个碱基插入或缺失, 使编码蛋白的基因发生移码和错义等严重突变, 阻碍蛋白行使正常功能[16, 17]; 而在同源重组(homologous recombination, HR)修复机制下则可以实现精确的基因敲入遗传修饰, 如点突变、插入和表位标记等[18]。因此, 通过这两种修复机制, CRISPR/Cas9技术即可实现基因敲除和基因敲入的基因组改造[19] (图 1)。
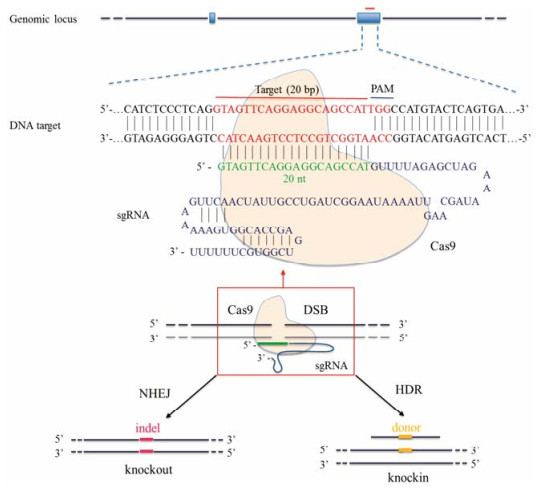
|
Figure 1 Schematic of the RNA-guided Cas9 nuclease and two types of double strand break (DSB)-induced repair mechanism |
CRISPR/Cas9技术主要包含引物设计、sgRNA表达质粒的构建、靶细胞转染、混合pool的筛选与鉴定和单克隆的分离与验证几大步骤, 大致流程如图 2。
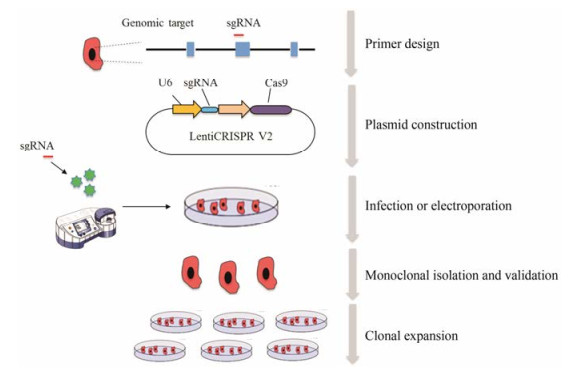
|
Figure 2 Overview of experiments |
首先需确定待敲除基因的靶位点, 找到该基因CDS (coding sequence)区, 明确外显子部分。一般来说, 尽量选择靠前的外显子。对于蛋白编码基因, 若该蛋白具有重要结构功能域, 可以将基因敲除位点设计在编码该结构域的外显子上。如果不能明确基因产物的性质, 可以选择将待敲除位点设计在起始密码子(ATG)后的外显子上。确定靶位点之后, 即可选择一段23~250 bp的外显子序列输入在线设计软件中, 自动输出sgRNA序列, 也可手动进行选择。将sgRNA序列克隆到sgRNA scaffold backbone (BB)质粒上, 与表达Cas9蛋白的质粒共同转染到靶细胞中, 通过单克隆分选、扩增和检测, 便可得到稳定的基因敲除或特定点突变的细胞系。
若是为了实现准确的基因敲入, 则需要将一个同源重组修复模板与sgRNA、Cas9蛋白同时转入细胞中以介导同源重组修复。这个同源重组修复模板可以是质粒模板(plasmid donor template)、双链线性DNA模板(double-stranded DNA template)或单链寡核苷酸链(single-stranded donor oligonucleotides)。同源重组修复模板的设计应遵循以下原则:左右两端同源臂长度至少40 bp, 以90 bp为最佳; 链的方向以与滞后链(lagging strand)结合为最好; 突变位点距切割位点越近越好, 且最好在PAM位置的5'端。以BTKC481S点突变为例, 在包含点突变的sgRNA序列两端各加上90 bp的同源臂, 得到一段203 bp的序列, 并在sgRNA序列中引入同义突变以插入一个BamHI酶切位点, 即可通过酶切验证阳性, 如图 3所示。
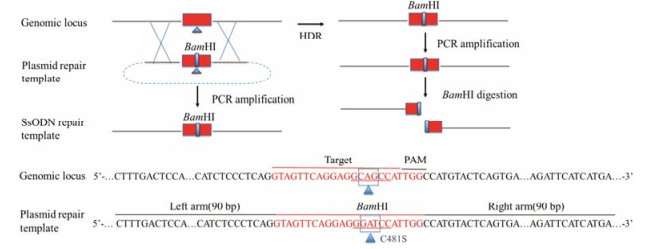
|
Figure 3 HDR donor-induced BTKC481S mutation |
药物靶点的筛选和鉴定是新药研发中的关键一环, 也是耗资巨大的过程。因此, 药物开发需要一个良好的药物靶点的发现与验证的平台。早期, 这个平台一直建立在“基因敲除”(genetic knockout)的模式动物上, 这项技术虽然能够对染色体上的单个基因进行敲除, 但是不能进行“剂量效应”(dose-response effects)的研究, 也不能敲除对胚胎发育至关重要或者致死的基因, 且周期长、价格高, 极大限制了药物研发进程。RNA干扰技术(RNAi)的出现大大缩短了筛选药物靶标和先导化合物的时间, 利用这项技术可以有效且高选择性地靶向靶基因的mRNA, 抑制靶基因表达。但RNAi技术存在重现性差和脱靶效应较严重等问题, 有时甚至会导致完全错误的结果。CRISPR/Cas9因其强大的基因编辑功能, 已被用于功能基因的筛选和编辑、药物靶点的筛选与验证、动物模型的构建等多项领域, 为新型药物研发或先导化合物的开发奠定了良好的基础。
3.1 CRISPR/Cas9技术在功能基因筛选中的应用人类基因组的功能尚未完全揭示, 许多疾病尚未找到合适的药用靶点。基因组的不方便操作性极大地制约了疾病机制的揭示及药物靶点的筛选研究。CRISPR/Cas9基因编辑技术只需要通过建立一个sgRNAs文库导入细胞中, 再通过相关表型的评估, 即可实现对基因组的大规模定点编辑, 进而揭示基因的生理功能, 为新药研发提供可靠的靶标。2013年, 来自英国桑格研究所的科学家们创建了一个可以靶向小鼠基因组内全部基因的sgRNAs文库, 利用慢病毒将这些sgRNAs引入组成型表达Cas9蛋白的小鼠胚胎干细胞中以创造出突变的干细胞库, 并用梭状芽胞杆菌细菌毒素α对这个突变的干细胞库进行遗传筛选, 从中发现27个已知的抗性基因和4个未被报道的抗性基因[20]。
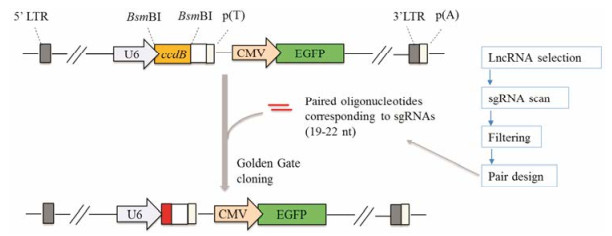
对于转录非编码RNA的基因来说, 仅有插入或缺失突变很可能对转录产物的表型没有显著影响, 因此筛选起来较为困难。2014年, 北京大学的魏文胜研究组[21]开发了一种基于CRISPR/Cas9系统的慢病毒聚焦型人源细胞文库和功能性基因筛选平台(图 4), 并结合深度测序技术, 成功鉴定出了对炭疽和白喉毒素毒性至关重要的宿主基因, 这将为新型抗菌药物的研发提供靶点。此外, 他们联合哈佛大学的刘小乐教授[22], 采用基于配对向导RNA (pgRNA)的方式, 以慢病毒为载体构建出pgRNA库(图 5), 在全基因组范围内对人源肝癌细胞系Huh7.5OC中的近700个与癌症或其他疾病相关的长链非编码RNA (lncRNA)进行了功能筛选。这种方法是利用CRISPR/ Cas9系统在两种分别靶向目标基因不同位点的sgRNA组合成的pgRNA指导下, 对同一基因造成两处DNA双链断裂, 两个断裂点之间的基因片段可能会被DNA修复过程“遗漏”, 最终不会出现在修复完毕的基因中。其基本策略可被用于对其他非编码RNA功能的分析中, 具有巨大的潜在应用场景。
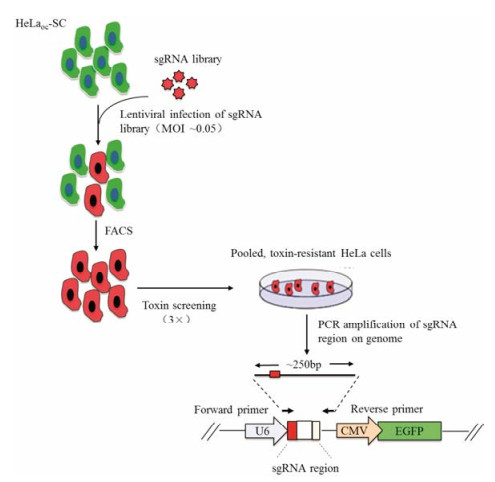
|
Figure 4 Schematic of lentiviral-delivered sgRNA library construction and functional screening[21] |
|
Figure 5 pgRNA library design[22] |
2016年底, Fulco等[23]分别证明了基于CRISPR/ Cas技术可以用于识别疾病相关基因的关键调控元件。前者利用包含98 000个sgRNA序列的文库分析了MYC和GATA1基因周围1.29 MB序列, 他们利用失活形式的Cas9与KRAB蛋白相互融合从而沉默靶基因, 在上百个调控元件中发现了2个调控GATA1基因的增强子元件和7个调控MYC表达的增强子元件。后者则采用包含18 000个sgRNA序列的文库分析了vemurafenib耐药性相关基因(CUL3、NF1和NF2)周围100个碱基范围, 发现了影响黑色素瘤患者耐药性基因的非编码调控元件[24]。这两项研究延伸了CRISPR/Cas9技术在非编码基因组研究中的应用。
3.2 CRISPR/Cas9技术在药物靶点筛选与验证中的应用在新药研发中, 药物与靶点之间相互作用的验证是必不可少的。找到耐药性突变可谓是确定靶点的黄金准则, 而在野生型背景的细胞中引入耐药性突变则是靶点验证最后的关键一步[25], 如rapamycin-TOR1这对重要的药物靶点组合的发现[26]。将CRISPR/Cas9技术与全基因组测序及耐药性突变筛选结合, 能够使得靶点验证更加高效[27]。
2014年, Wang等[28]利用包含73 000个sgRNA序列的文库对HL60和KBM7两株细胞进行筛选, 验证了靶基因MSH2、MSH6、MLH1和PMS2能修复6-TG引起的DNA损伤, 及靶基因TOP2A能抵抗依托泊苷(etoposide)的毒性。随后, Shalem等[29]建立了靶向18 080个基因的包含64 751个sgRNA序列的全基因组范围的CRISPR/Cas9敲除(GeCKO)文库, 该文库能在人类细胞中进行正向和负向选择性筛选。他们利用GeCKO文库鉴定了对癌细胞和多能干细胞细胞活力必不可少的基因, 并在A375黑色素瘤模型中筛选出了与维罗非尼耐药性有关的基因, 如先前发现的NF1和MED12, 及未被发现的NF2、CUL3、TADA2B和TADA1。同年, 剑桥大学的Smurnyy等[25]通过CRISPR/Cas9基因编辑技术和耐药性克隆测序技术验证了6-TG-HPRT1和triptolide-ERCC3这两对药物靶标的配对关系, 他们将CRISPR/Cas9技术应用于单倍体细胞模型KBM7细胞中, 表明这项技术对显性耐药性基因和隐性耐药性基因的验证都有效。Neggers等[30]则利用CRISPR/Cas9剪切之后的同源重组修复(homology-dorected repair, HDR)机制在急性T细胞白血病细胞Jurkat中精确地引入了XPO1C528S点突变, 证实selinexor (KPT-330)能够靶向并阻断核输出蛋白受体XPO1。2017年4月发表的一项研究中, 研究人员组合使用CRISPR/Cas9技术和iPSC技术构建出特定的巨噬细胞模型, 证明免疫系统中的IRF5基因和IL-10RA基因在抵抗衣原体感染中起到关键性作用, 这为治疗由衣原体引起的疾病鉴定出了新的药物靶标[31]。
目前, 利用CRISPR/Cas9技术进行筛选的策略主要是靶向诱导候选基因(candidate genes)的5'外显子产生突变, 这种方法的缺陷在于容易产生保留部分功能的框内突变体(in-frame variants), 即使存在很强的遗传相关性也可能观察不到显著的表型差异。为了克服这个问题, 美国冷泉港实验室的研究人员[32]针对性地突变编码蛋白功能域的外显子, 导致更多的无效突变(null mutation), 从而大幅提高了阴性选择的效率。在这项研究中, 他们筛选了小鼠MLL-AF9/NrasG12D急性髓系白血病细胞的192个染色质调节性区域, 结果找到了6个已知的药物作用靶点及19个潜在的药物靶点, 表明CRISPR/Cas9技术在癌症药物靶点的筛选与验证上还有很大的优化空间。
3.3 CRISPR/Cas9技术在动物模型构建和疾病治疗中的应用传统小鼠模型的建立主要依赖于同源重组和抗生素的筛选, 通常需要采用胚胎干细胞作为过渡, 过程繁琐且耗时, 而CRISPR/Cas9技术大大缩减了构建动物模型的时间和经费。李大力和刘明耀课题组[33]利用RNA注射法将CRISPR/Cas9系统导入小鼠受精卵中, 相比DNA注射, 该方法能更为有效地在小鼠胚胎中产生定点突变, 并且不受小鼠遗传品系的限制。迄今为止, 研究者已利用CRISPR/Cas9系统构建出人肝癌、肺腺癌等小鼠癌症模型[34]及B型血友病、心脏衰竭等小鼠疾病模型。利用CRISPR/Cas9系统, 研究者可以清除小鼠模型中潜伏感染的病毒基因组, 如乙型肝炎病毒; 也可以修复遗传基因缺陷, 如囊性纤维化等。2015年12月, 《科学》杂志发表了3项关于CRISPR/Cas9技术在小鼠模型中的实验[35-37]。在这3篇文章中, 研究者均通过非致病性腺相关病毒(adeno-associated virus, AAV)成功移除杜氏肌营养不良症(Dunchenne’s muscular dystrophy, DMD)模型小鼠的肌萎缩蛋白基因第23号外显子, 从而改善了其肌肉的生化特性, 使其骨骼肌和心肌萎缩蛋白的功能得到部分恢复。这些突破增长了研究者攻克其他危害人类健康的疾病及难治愈癌症的信心。
2015年3月, 美国加州拉西瑞亚大学的研究人员通过设计针对人类免疫缺陷病毒(human immunodeficiency virus, HIV)的sgRNA序列, 引导Cas9蛋白在特定位置上对HIV病毒的DNA双链进行剪切, 使得人体18%~72%感染细胞内的HIV病毒失活[38]。同年12月, 美国杜克大学的研究者利用CRISPR/Cas9技术构建特定的转录激活子, 诱导感染HIV的人类细胞表达所缺乏的限制因子APOBEC3G和APOBEC3B, 从而阻断细胞内Vif缺陷型HIV-1的复制, 并部分抑制野生型HIV-1的传染性[39]。2016年3月, Kaminski等[40]则利用一种特定的CRISPR/Cas9系统, 特异性地靶向HIV-1前病毒DNA, 有效且安全地将HIV病毒从体外培养的人CD4+ T细胞中清除, 这些根除HIV-1 DNA的T细胞可以正常地生长和发挥功能。这些研究成果有望应用于HIV感染的临床治疗中。
除了艾滋病, CRISPR/Cas9系统对治疗其他疾病也有巨大的潜力。2015年9月, Canver等[41]利用CRISPR/Cas9技术移除了控制人类造血干细胞中分子开关BCL11A的红系增强子, 使造血干细胞成熟后胎儿型血红蛋白(HbF)明显增加, 成人型血红蛋白(HbA)相对减少, 而HbF能够对抗镰状细胞突变效应。2016年11月15日, Cyranoski[42]报道了由四川大学华西医院卢铀教授所主持的全球首个CRISPR技术的人体应用, 卢铀教授研究团队通过CRISPR/Cas9技术敲除T细胞中抑制免疫功能的PD-1基因, 并在体外进行T细胞扩增, 当细胞达到一定数量后, 再回输至非小细胞肺癌患者体内进行肿瘤治疗。10月28日, 首名患者接受了该项治疗。此后, 四川大学的研究人员[43]采用多功能高分子RGD-R8-PEG-HA对人工病毒进行修饰, 将一种安全性较高的靶向乳腺癌中MTH1基因的CRISPR/Cas9系统RRPHC/Cas9-hMTH1输送至小鼠肿瘤模型中, 并有效地抑制了肿瘤的生长, 这项研究提供了一种崭新的体内导入CRISPR/Cas9质粒的方式。这些研究表明, 利用CRISPR/Cas9技术靶向癌症或其他疾病的药物靶标, 将为人类疾病的治疗带来巨大的帮助。
4 CRISPR/Cas9技术的缺陷与改进虽然CRISPR/Cas9技术十分高效且应用广泛, 但它也存在一定的限制性, 只有当目标位点附近存在PAM序列时, Cas9蛋白才能进行准确切割[44, 45], 而它的脱靶效应可能会使目标基因以外的其他基因产生变化。这些问题的存在深深影响着该技术在治疗应用中的安全性和有效性, 因此仍需不断完善。目前, 减轻CRISPR/Cas9脱靶效应的常规方式包括改变sgRNA的二级结构[46], 缩短sgRNA序列的长度[47], 利用FokI-Cas9融合核酸酶、纯化的Cas9核糖核蛋白、配对的催化突变Cas9切口酶等[48], 这些方法能实现较低的脱靶率, 但也在一定程度上降低打靶效率。自CRISPR/Cas9技术诞生以来, 科学家们为它的改进和完善做出了不断的探索, 并取得了显著的成果。
许多人类遗传疾病都是由点突变引起, 而当前的基因组编辑方法不能高效地校正细胞中的突变, 而且会发生随机的核苷酸插入或删除(indel)。CRISPR/Cas9技术的基因编辑作用基于sgRNA介导的Cas9酶切割造成的双链断裂, 能校正单核苷酸突变的同源重组修复发生率很低, 只有0.5%~20%, 且只发生在细胞周期的S期和G2期[49-51]。为了提高修正点突变的效率, 同时减少indel的频率, 哈佛大学的Komor等[52]让Cas9部分失活, 使它不能切割DNA双链, 但仍能结合到目标DNA序列上。同时, 通过将Cas9与胞苷脱氨酶(鼠源APOBEC1)偶联在一起, 直接将胞嘧啶(C)转换成尿嘧啶(U)。因此, 在细胞基因组靶位点上会产生一对错配的碱基对, 即一条链上新产生的U与另一条链上的初始碱基G错配, 从而引发错配修复, 最终正确的碱基校正率可达到15%~75%, 同时只有1%以下的序列发生indel。
2016年4月, 美国格拉斯通研究所的研究人员[53]将CRISPR/Cas9技术应用于诱导性多能干细胞(iPSCs), 将编码dCas9的基因和编码KRAB抑制结构域的基因融合在一起, 形成dCas9-KRAB融合基因并将其导入AAV病毒载体中, 再转导iPSCs和源自iPSCs的心肌细胞, 从而在宿主细胞中表达这种融合蛋白, 结果表明这种CRISPRi (CRISPR interfereence)系统比CRISPR/Cas9系统更加高效。剑桥大学和惠康基金会桑格研究所的研究人员[54]则将一种一体化的Cas9 D10A切口酶载体与荧光激活的细胞分选富集后相结合, 基于高通量基因型和表型单克隆筛选, 在满足低脱靶率的同时实现了高效率的基因敲除和敲入。此外, 科学家们还将四环素诱导系统和CRISPR/Cas9技术结合, 建立了一种高效可控的基因编辑平台sOPTiKO/sOPTiKD, 可以在细胞发育任何阶段的任何细胞类型中精密调控基因表达水平[55]。
RNAi技术部分抑制基因表达, CRISPR/Cas9技术可以完全抑制基因表达。当涉及药物开发时, 这两者的区别变得非常重要, 因为对任何一种药物而言, 实现对靶标100%的抑制是非常罕见的。将CRISPR与RNAi结合起来验证靶标可能会是一种较为理想的方法。2015年, 哈佛医学院的一个研究小组[56]结合CRISPR/Cas9技术及RNAi技术, 鉴定出了3个对肿瘤生长十分重要的基因mRNA-cap、Pitslre和CycT (对应人源RNGTT、CDK11和CCNT1)。他们利用CRISPR/Cas9技术建立了TSC1或TSC2缺陷的突变果蝇细胞系, 再用RNAi方法筛查其所有的激酶和磷酸酶, 结果发现这3个基因的敲低会引起TSC1/TSC2缺陷型细胞生长速度减慢, 而对野生型细胞没有影响。随后, 研究人员在哺乳动物TSC2缺陷型细胞系中敲低这些基因, 也发生了类似的生长抑制现象, 这也说明这种跨物种的筛选策略可用于药物靶点的鉴定。2017年, Liu等[57]开发出一种CRISPRi平台, 也可以实现基因的部分抑制。他们靶向7种不同细胞系中的16 401个lncRNA位点, 并鉴定出其中499个lncRNA位点是细胞生长所需要的, 而很多lncRNA位点在细胞增殖中发挥重要作用, 这可能最终对靶向癌症治疗产生影响。
CRISPR技术还有许多奥秘尚待挖掘。2016年, Pawluk等[58]发现了CRISPR/Cas9的“关闭开关” (off-switches), 鉴定出了3个天然存在的、能够抑制Cas9活性的蛋白质家族, 从而可以实现对CRISPR/ Cas9的自由关闭。科学家们还鉴定出了一种能够靶向和降解RNA的RNA导向酶(RNA-guided enzyme) C2c2, CRISPR/C2c2系统可以实现细胞基因组短暂的改变, 并且能够帮助细菌对抗病毒感染[59]。近期, 关于CRISPR/C2c2系统的最新研究表明, C2c2对双链DNA的切割会产生7 nt的黏性末端, 这是目前所有用于基因组编辑的CRISPR/Cas系统中所能产生的最长黏性末端[60]。此外, 研究人员还找到了Cas9的一种潜在替代者——Cpf1酶。CRISPR/Cpf1也能在crRNA的引导下在人类细胞中剪切目标DNA, 而且还能对RNA进行加工[61]。随后, CRISPR/Cpf1的晶体结构也被解析[62]。同时, Burstein等[63]通过对不同地点采集到的微生物进行宏基因组分析, 发现了两种新型的Ⅱ型CRISPR/Cas9系统, 包括在古细菌中发现的一种Cas9酶和在细菌中发现的两种小Cas酶(CasX和CasY)。将CRISPR/CasX或CRISPR/CasY引入大肠杆菌中都可以阻止遗传物质进入细胞, 但其是否具有基因组编辑功能还有待于进一步研究。这些新的发现也表明寻找新形式的CRISPR还有巨大的可能性。
5 结语与展望CRISPR技术已成为基因编辑最有效的手段之一, 它在发现和验证药物靶标方面具有巨大潜力, 也为癌症及其他疾病的治疗提供了新思路。利用CRISPR/ Cas9技术靶向编码蛋白功能结构域的外显子造成突变, 可对维持癌细胞生长存活及发育等过程具有重要作用的蛋白或结构域进行大规模筛选, 这对寻找合适的药物作用靶点及开发癌症治疗药物具有重要意义。本课题组已利用CRISPR/Cas9技术在特定癌症细胞系中构建激酶的点突变, 作为药物靶点筛选与验证的细胞模型。CRISPR/Cas9技术不仅在基础研究中发挥重要作用, 在制药业也渐渐崭露头角。如福泰制药(Vertex)与CRISPR技术公司CRISPR Therapeutics签约4年合作开发针对已经有明确人类基因靶点的CRISPR/Cas9药物, 主要针对囊肿性肺纤维化和镰刀型贫血症。
CRISPR/Cas9系统在广受推崇的同时, 它在人类生殖细胞或胚胎上的使用也引发了全球持续性争议。如英国伦敦弗朗西斯克里克机构的研究团队已获得英国人类受精与胚胎管理局(HEFA)审核通过, 即将开始编辑体外受精(IVF)的胚胎基因, 该研究很可能制造出第一个基因改造的人类胚胎, 而这也使得关于基因编辑的伦理争论更加激烈。
一方面, 随着CRISPR/Cas9技术的不断完善, 它势必得到更广泛的应用, 对人类健康领域做出重大贡献; 另一方面, 对于基因编辑技术, 研究者最为需要的是认真思考, 制定出合理的规范, 使其发挥出最积极的效用。
| [1] | Porteus MH, Carroll D. Gene targeting using zinc finger nucleases[J]. Nat Biotechnol, 2005, 23: 967–973. DOI:10.1038/nbt1125 |
| [2] | Joung JK, Sander JD. TALENs:a widely applicable tech-nology for targeted genome editing[J]. Nat Rev Mol Cell Biol, 2013, 14: 49–55. DOI:10.1038/nrm3486 |
| [3] | Gaj T, Gersbach CA, Barbas CF 3rd. ZFN, TALEN and CRISPR/Cas-based methods for genome engineering[J]. Trends Biotechnol, 2013, 31: 397–405. DOI:10.1016/j.tibtech.2013.04.004 |
| [4] | Pauwels K, Podevin N, Breyer D, et al. Engineering nucle-ases for gene targeting:safety and regulatory considerations[J]. N Biotechnol, 2014, 31: 18–27. DOI:10.1016/j.nbt.2013.07.001 |
| [5] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineer-ing using CRISPR/Cas systems[J]. Science, 2013, 339: 819–823. DOI:10.1126/science.1231143 |
| [6] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the IAP gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. J Bacteriol, 1987, 169: 5429–5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [7] | Jansen R, Embden JD, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43: 1565–1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [8] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J]. Microbiology, 2005, 151: 2551–2561. DOI:10.1099/mic.0.28048-0 |
| [9] | Mojica FJ, Díez-Villaseñor C, García-Martínez J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. J Mol Evol, 2005, 60: 174–182. DOI:10.1007/s00239-004-0046-3 |
| [10] | Pourcel C, Salvignol G, Vergnaud G, et al. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 151: 653–663. DOI:10.1099/mic.0.27437-0 |
| [11] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315: 1709–1712. DOI:10.1126/science.1138140 |
| [12] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA[J]. Science, 2008, 322: 1843–1845. DOI:10.1126/science.1165771 |
| [13] | Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nat Rev Microbiol, 2011, 9: 467–477. DOI:10.1038/nrmicro2577 |
| [14] | Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482: 331–338. DOI:10.1038/nature10886 |
| [15] | Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471: 602–607. DOI:10.1038/nature09886 |
| [16] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337: 816–821. DOI:10.1126/science.1225829 |
| [17] | Mali P, Yang L, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339: 823–826. DOI:10.1126/science.1232033 |
| [18] | Sung P, Klein H. Mechanism of homologous recom-bination:mediators and helicases take on regulatory functions[J]. Nat Rev Mol Cell Biol, 2006, 7: 739–750. DOI:10.1038/nrm2008 |
| [19] | Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8: 2281–2308. DOI:10.1038/nprot.2013.143 |
| [20] | Koikeyusa H, Li Y, Tan EP, et al. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library[J]. Nat Biotechnol, 2014, 32: 267–273. DOI:10.1038/nbt.2800 |
| [21] | Zhou Y, Zhu S, Cai C, et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J]. Nature, 2014, 509: 487–491. DOI:10.1038/nature13166 |
| [22] | Zhu S, Li W, Liu J, et al. Genome-scale deletion screening of human long non-coding RNAs using a paired-guide RNA CRISPR-Cas9 library[J]. Nat Biotechnol, 2016, 34: 1279–1286. DOI:10.1038/nbt.3715 |
| [23] | Fulco CP, Munschauer M, Anyoha R, et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference[J]. Science, 2016, 354: 769–773. DOI:10.1126/science.aag2445 |
| [24] | Sanjana NE, Wright J, Zheng K, et al. High-resolution interrogation of functional elements in the noncoding genome[J]. Science, 2016, 353: 1545–1549. DOI:10.1126/science.aaf7613 |
| [25] | Smurnyy Y, Cai M, Wu H, et al. DNA sequencing and CRISPR-Cas9 gene editing for target validation in mammalian cells[J]. Nat Chem Biol, 2014, 10: 623–625. DOI:10.1038/nchembio.1550 |
| [26] | Huang J, Dibble CC, Matsuzaki M, et al. The TSC1-TSC2 complex is required for proper activation of mTOR complex 2[J]. Mol Cell Biol, 2008, 28: 4104–4115. DOI:10.1128/MCB.00289-08 |
| [27] | Kasap C, Elemento O, Kapoor TM. DrugTargetSeqR:a genomics-and CRISPR/Cas9-based method to analyze drug targets[J]. Nat Chem Biol, 2014, 10: 626–628. DOI:10.1038/nchembio.1551 |
| [28] | Wang T, Wei JJ, Sabatini DM, et al. Genetic screens in human cells using the CRISPR-Cas9 system[J]. Science, 2014, 43: 80–84. |
| [29] | Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343: 84–87. DOI:10.1126/science.1247005 |
| [30] | Neggers JE, Vercruysse T, Jacquemyn M, et al. Identifying drug-target selectivity of small-molecule CRM1/XPO1 inhibitors by CRISPR/Cas9 genome editing[J]. Chem Biol, 2015, 22: 107–116. DOI:10.1016/j.chembiol.2014.11.015 |
| [31] | Yeung ATY, Hale C, Lee AH, et al. Exploiting induced pluripotent stem cell-derived macrophages to unravel host factors influencing Chlamydia trachomatis pathogenesis[J]. Nat Commun, 2017, 8: 15013. DOI:10.1038/ncomms15013 |
| [32] | Shi J, Wang E, Milazzo JP, et al. Discovery of cancer drug targets by CRISPR-Cas9 screening of protein domains[J]. Nat Biotechnol, 2015, 33: 661–667. DOI:10.1038/nbt.3235 |
| [33] | Li D, Qiu Z, Shao Y, et al. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31: 681–683. DOI:10.1038/nbt.2661 |
| [34] | Platt RJ, Chen S, Zhou Y, et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling[J]. Cell, 2014, 159: 440–455. DOI:10.1016/j.cell.2014.09.014 |
| [35] | Nelson CE, Hakim CH, Ousterout DG, et al. In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy[J]. Science, 2016, 351: 403–407. DOI:10.1126/science.aad5143 |
| [36] | Tabebordbar M, Zhu K, Cheng JK, et al. In vivo gene editing in dystrophic mouse muscle and muscle stem cells[J]. Science, 2016, 351: 407–411. DOI:10.1126/science.aad5177 |
| [37] | Long C, Amoasii L, Mireault AA, et al. Postnatal genome editing partially restores dystrophin expression in a mouse model of muscular dystrophy[J]. Science, 2016, 351: 400–403. DOI:10.1126/science.aad5725 |
| [38] | Liao HK, Gu Y, Diaz A, et al. Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells[J]. Nat Commun, 2015, 6: 6413. DOI:10.1038/ncomms7413 |
| [39] | Bogerd HP, Kornepati AV, Marshall JB, et al. Specific induction of endogenous viral restriction factors using CRISPR/Cas-derived transcriptional activators[J]. Proc Natl Acad Sci U S A, 2015, 112: E7249–E7256. DOI:10.1073/pnas.1516305112 |
| [40] | Kaminski R, Chen Y, Fischer T, et al. Elimination of HIV-1 genomes from human T-lymphoid cells by CRISPR/Cas9 gene editing[J]. Sci Rep, 2016, 6: 22555. DOI:10.1038/srep22555 |
| [41] | Canver MC, Smith EC, Sher F, et al. BCL11A enhancer dissection by Cas9-mediated in situ saturating mutagenesis[J]. Nature, 2015, 527: 192–197. DOI:10.1038/nature15521 |
| [42] | Cyranoski D. CRISPR gene-editing tested in a person for the first time[J]. Nature, 2016, 539: 479. DOI:10.1038/nature.2016.20988 |
| [43] | Li L, Song L, Liu X, et al. Artificial virus delivers CRISPR-Cas9 system for genome editing of cells in mice[J]. ACS Nano, 2017, 11: 95–111. DOI:10.1021/acsnano.6b04261 |
| [44] | Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nat Biotechnol, 2013, 31: 839–843. DOI:10.1038/nbt.2673 |
| [45] | Duan J, Lu G, Xie Z, et al. Genome-wide identification of CRISPR/Cas9 off-targets in human genome[J]. Cell Res, 2014, 24: 1009–1012. DOI:10.1038/cr.2014.87 |
| [46] | Dang Y, Jia G, Choi J, et al. Optimizing sgRNA structure to improve CRISPR-Cas9 knockout efficiency[J]. Genome Biol, 2015, 16: 280. DOI:10.1186/s13059-015-0846-3 |
| [47] | Fu Y, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nat Biotechnol, 2014, 32: 279–284. DOI:10.1038/nbt.2808 |
| [48] | Slaymaker IM, Gao L, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351: 84–88. DOI:10.1126/science.aad5227 |
| [49] | Yang H, Wang H, Shivalila CS, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 154: 1370–1379. DOI:10.1016/j.cell.2013.08.022 |
| [50] | Wang H, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153: 910–918. DOI:10.1016/j.cell.2013.04.025 |
| [51] | Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proc Natl Acad Sci U S A, 2012, 109: E2579–E2586. DOI:10.1073/pnas.1208507109 |
| [52] | Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533: 420–424. DOI:10.1038/nature17946 |
| [53] | Mandegar MA, Huebsch N, Frolov EB, et al. CRISPR interference efficiently induces specific and reversible gene silencing in human iPSCs[J]. Cell Stem Cell, 2016, 18: 541–553. DOI:10.1016/j.stem.2016.01.022 |
| [54] | Chiang TW, le Sage C, Larrieu D, et al. CRISPR-Cas9(D10A) nickase-based genotypic and phenotypic screening to enhance genome editing[J]. Sci Rep, 2016, 6: 24356. DOI:10.1038/srep24356 |
| [55] | Bertero A, Pawlowski M, Ortmann D, et al. Optimized inducible shRNA and CRISPR/Cas9 platforms for in vitro studies of human development using hPSCs[J]. Development, 2016, 143: 4405–4418. DOI:10.1242/dev.138081 |
| [56] | Housden BE, Valvezan AJ, Kelley C, et al. Identification of potential drug targets for tuberous sclerosis complex by synthetic screens combining CRISPR-based knockouts with RNAi[J]. Sci Signal, 2015, 8: rs9. DOI:10.1126/scisignal.aab3729 |
| [57] | Liu SJ, Horlbeck MA, Cho SW, et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells[J]. Science, 2017. DOI:10.1126/science.aah7111 |
| [58] | Pawluk A, Amrani N, Zhang Y, et al. Naturally occurring off-switches for CRISPR-Cas9[J]. Cell, 2016, 167: 1829–1838. DOI:10.1016/j.cell.2016.11.017 |
| [59] | Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J]. Science, 2016, 353: aaf5573. DOI:10.1126/science.aaf5573 |
| [60] | Yang H, Gao P, Rajashankar KR, et al. PAM-dependent target DNA recognition and cleavage by C2c1 CRISPR-Cas endonuclease[J]. Cell, 2016, 67: 1814–1828. |
| [61] | Fonfara I, Richter H, Bratovič M, et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA[J]. Nature, 2016, 532: 517–521. DOI:10.1038/nature17945 |
| [62] | Dong D, Ren K, Qiu X, et al. The crystal structure of Cpf1 in complex with CRISPR RNA[J]. Nature, 2016, 532: 522–526. DOI:10.1038/nature17944 |
| [63] | Burstein D, Harrington LB, Strutt SC, et al. New CRISPR-Cas systems from uncultivated microbes[J]. Nature, 2017, 542: 237–241. |
 2018, Vol. 53
2018, Vol. 53


