2. 福建农林大学 福建农林大学作物遗传育种与综合利用教育部重点实验室, 福建 福州 350002;
3. 河南农业大学农学院, 河南 郑州 450002
2. Key Laboratory of Ministry of Education for Genetics, Breeding and Multiple Utilization of Crops, Fujian Agriculture and Forestry University, Fuzhou 350002, China;
3. Agricultural College, Henan Agricultural University, Zhengzhou 450002, China
地黄(Rehmannia glutinosa Libosch)是玄参科地黄属多年生草本植物, 栽培历史悠久, 是我国常用大宗药材之一。然而, 在地黄的生产中连作障碍问题却十分严重, 连作地黄植株表现为生长不良、病虫害加重、块根不能正常膨大, 其产量和品质明显下降, 甚至绝收, 且每茬收获后须隔8~10年方可再种[1], 严重制约了道地药材地黄资源的可持续利用和区域经济的发展。目前关于栽培植物连作障碍的研究虽有大量报道, 但其具体形成机制仍不明晰, 越来越多的证据表明:连作地黄根际化感物质的积累及其所诱导根际微生态群体失衡是连作障碍形成的根本原因[2-7]。然而, 地黄的根际环境恶化仅伤害其自身, 对后茬其他作物并无伤害, 这表明地黄体内有独特的信号感知系统, 响应了连作地黄的化感自毒物质或失衡的微生态环境[8, 9]。为了揭示连作植物对根际灾变环境的分子响应机制, 课题组前期通过连作和头茬地黄差异表达基因、差异表达蛋白和相关的实验均发现, 钙信号系统在地黄连作障碍形成过程中扮演着重要的角色[10, 11]。同时, 在水稻、黄瓜、人参等作物的连作障碍的研究中同样发现钙信号可以被连作特异地诱导[12-14]。因此, 钙信号体系与栽培植物连作障碍的形成可能具有密切的关系。
钙信号体系主要包括钙编码系统和钙信号感知与传递系统。钙信号编码系统是当植物遭受外界胁迫因子或其他刺激条件时, 细胞质内的钙离子在时间、空间及浓度上会产生特异性的变化, 形成特征性“钙峰”[15-19], 这些特征信号往往蕴含着重要的“外界刺激”信息。植物为了将这些具有标志性的信息解码并传递给下游信号通路, 需要一套重要信号传递受体和蛋白。钙调素(CaM)、钙依赖蛋白激酶(CDPK)、钙磷脂酶(CBL)是迄今已经被证明的分布最广、分子结构高度保守的钙靶蛋白, 每种钙感知蛋白均包含着大量的家族成员, 每个家族成员又可以激活大量的下游转录因子。此外, 钙信号和其他信号转导通路之间亦存在着大量的交叉, 可以相互激活、相互反馈、相互调节[20, 21]。正是由于这些多样化的钙信号传导蛋白家族成员的存在, 植物体内信号传递网络才会更加复杂、多样, 也才能保证植物细胞能将多样化的刺激进行精准传递。在连作条件下, 植物遭受着根际化感自毒物质、有害病原微生物等多种因素的直接或间接胁迫, 并且多种胁迫因子交叉在一起形成连作所特有的复杂胁迫逆境。前期课题组通过设置不同逆境胁迫条件研究连作胁迫的属性, 发现连作胁迫包含了不同胁迫的特征属性[22, 23], 可以推论地黄体内的钙信号刺激响应可能是不同复合作用的结果, 或者不同胁迫因子诱发钙信号交叉结果。因此, 如何筛选连作特异的钙信号或者化感自毒物质、连作特异有害微生物诱发的特异信号对于了解地黄连作障碍形成分子机制和实行彻底的分子治疗具有重要的意义。虽然不同的逆境胁迫因子引发的钙信号会在变化幅度、时间及空间分布上有所不同(即产生了不同特征的“钙信号”), 但现有技术手段对此还无法区分, 也就无法利用这些特征将响应自毒物质的特异信号提取出来。然而钙信号体系的传递蛋白往往具有特定胁迫响应特征和属性, 可以通过钙信号的解码蛋白对连作特异的信号转导途径进行鉴定。因此, 对钙信号传递蛋白通路中的各个成员进行精细鉴定, 研究其在连作障碍形成过程中的分子行为, 对于理解连作障碍形成复杂胁迫背景下钙信号的作用具有重要意义。
本课题组在前期大容量转录组文库基础上研究其在连作障碍形成过程中的分子行为, 对于理解连作障碍形成复杂胁迫背景下钙信号的作用具有重要意义。详细鉴定地黄所有钙信号感知与传递系统蛋白的基因, 并详细分析其在地黄连作障碍形成中的表达模式, 筛选出连作胁迫下地黄体内钙信号基因的响应状况。同时, 进一步通过焦锑酸盐钙离子原位沉淀法和钙荧光指示剂法测定钙离子浓度以直接确证连作地黄中钙信号的作用。本研究为揭示钙信号体系相关基因在地黄连作障碍形成过程中的作用机制, 完善连作障碍形成的关键分子证据链提供重要的技术支撑。
材料与方法材料 本研究以河南温县广泛种植的“温85-5”地黄品种(经河南省中医药研究院张留记研究员鉴定, 为Rehmannia glutinosa L.)为供试材料, 在福建农林大学中药材GAP研究所野外生态定位观测站(温县基地)选择试验田种植地黄, 以头茬地黄为对照、连作地黄为处理, 各设立3个重复。在地黄苗期、拉线期、块根膨大前期、块根膨大中期、块根膨大后期共5个不同生长时期[24], 分别采集头茬与连作地黄根系鲜样, 液氮速冻, -80 ℃保存, 用于钙离子浓度及位置分布的检测。
地黄中钙信号传递系统相关基因的筛选和鉴定 在拟南芥数据库(https://www.arabidopsis.org/)中搜索并筛选出拟南芥CaM家族、CBL家族、CDPK家族、CIPK家族、CML家族、CRK家族的完整的核酸和氨基酸序列。以拟南芥钙信号传递蛋白家族基因的编码序列作为探针序列, 用Blastn工具在地黄转录组文库中进行同源比对检索(E值设为1×10-20), 筛选出地黄中钙信号传递蛋白家族相关的候选编码基因。将上述所获取的地黄钙信号候选基因再次与地黄转录组原始数据进行比对, 筛选出具有同源性片段, 使用CAP3 (http://doua.prabi.fr/software/cap3)对原始数据重新组装去冗余, 获得相对完整的核苷酸序列。
地黄中钙信号体系相关基因的生物信息学分析 将筛选出的地黄钙信号体系相关基因序列通过ORF finder工具(https://www.ncbi.nlm.nih.gov/orffinder/)分析其氨基酸和开放阅读框长度。同时, 用MEGA7.0对其进行家族分析(采用Neighbor-Joining算法, Bootstrap检测1 000次[25]); 用InterPro (http://www.ebi.ac.uk/interpro/)和IBS1.0 (http://www.mybiosoftware.com/ibs-illustrator-of-biological-sequences/)预测其结构域并绘制蛋白结构图; 用Target P1.1 (http://www.cbs.dtu.dk/services/TargetP/)预测其所处的细胞位置。
地黄中钙信号体系相关基因表达量分析 基于前期课题组所构建的连作和头茬地黄不同时期RNA-Seq测序结构[26], 对本研究所获取的地黄钙信号体系相关基因的表达量进行重新统计, 获取连作和头茬地黄钙信号体系相关基因在苗期、拉线期、膨大前期、膨大中期、膨大后期等5个生长时期的表达量, 并将数据统一标准化(获取RPKM值)。为了对比分析钙信号体系相关基因在连作和头茬地黄中的表达模式差异, 分别以苗期地黄作为对照, 按照log2 (拉线期/苗期)、log2 (膨大前期/苗期)、log2 (膨大中期/苗期)、log2 (膨大后期/苗期)方式获取不同时期的表达量。随机选择12个钙信号相关基因, 用荧光实时定量PCR (qRT-PCR)验证钙信号相关基因在连作和头茬地黄发育过程中表达水平。利用Beacon Designer 8.0软件(http://beacon-designer.software.informer.com/8.0/)设计荧光定量引物(表 1)。
| Table 1 Primer sequences used in this study |
用Trizol法提取连作和头茬地黄不同时期根系的总RNA, 使用TaKaRa公司的PrimeScript™RT reagent Kit反转录试剂盒合成cDNA。用实时荧光定量PCR (qRT-PCR)方法检测所选基因在连作和头茬地黄不同时期的表达水平。各cDNA样品分别用对应基因(表 1)和内参基因进行荧光实时定量PCR, 且每个反应重复3次。反应体系为25 μL: SYBR Premix Ex Taq Ⅱ (Tli RNase H Plus) 12.5 μL, cDNA模板2 μL, ddH2O 8.5 μL, 引物各1 μL[27]。PCR反应程序: 95.0 ℃ 2 min; 95.0 ℃ 5 s, 60.0 ℃ 30 s, 72 ℃ 30 s进行39个循环; 熔解曲线检测条件是95 ℃ 1 min, 60 ℃ 1 min, 然后以0.5 ℃·s-1的速度提高到95 ℃, 连续检测荧光信号。基因的表达量以参照基因18S (DQ469606)作为标准进行相对定量分析, 相对定量方法采样普遍使用的是2-ΔΔCt (Livak)方法[28]。进行目的基因表达模式分析时, 计算同一个样品目的基因的Ct值, 取3个平行孔校正后Ct值的平均数。分别以连作与头茬地黄苗期样品的Ct值的平均数设为1, 进行基因表达分析。以内参18S进行校正, 数据处理使用2-ΔΔCt法计算相对表达量。
利用钙离子荧光染料法测定头茬与连作地黄根尖组织内钙离子浓度 分别采集头茬与连作地黄拉线期的根尖, 清水轻洗干净, 剪取2 cm左右根尖, 放入含50 μmol·L-1 Fluo-3/AM、50 mmol·L-1山梨糖醇、1%皂素的溶液中, 4 ℃下避光孵育2 h, 水洗后转入50 mmol·L-1山梨糖醇溶液, 20 ℃避光孵育2 h。将标记有Fluo-3/AM的根尖临时压片, 以未孵育Fluo-3/AM的根尖为对照, 在LSM510共聚焦荧光显微镜下进行观察拍照(激发波长为488 nm, 发射波长为535 nm)。
利用焦锑酸钾沉淀法测定头茬与连作地黄根尖组织内钙离子浓度 分别采集头茬和连作地黄拉线期的块根, 在解剖显微镜下将块根的分裂形成层组织切成1 mm × 1 mm × 2 mm的横切小块。经初固定、再固定、脱水、透明、树脂浸透、树脂包埋与聚合、切片、染色、镜检(JEM-1400型透射电镜)后, 进行对照处理, 将在电镜下已确定由焦锑酸钾沉淀的定位切片漂浮在100 mmol·L-1 EGTA (pH 8.0)溶液中, 60 ℃处理1 h, 使EGTA与Ca2+螯合, 脱去原沉淀中的钙离子, 再置于电镜下观察与拍照。
结果与分析 1 头茬与连作地黄根尖及块根组织内钙离子的浓度检测分别采集地黄拉线期的头茬和连作根尖及块根, 利用共聚焦荧光显微技术在组织水平对钙离子在头茬和连作根尖区细胞内的分布进行了观察。在共聚焦荧光显微镜下观察到, 连作地黄根尖的荧光强度明显高于头茬地黄根尖的荧光强度(如图 1A~1D)。并且在头茬地黄根尖细胞内, 荧光信号只集中在细胞内的部分区域, 而在连作地黄细胞内的大部分区域都有强烈的荧光强度, 表明连作地黄细胞内出现了大量的钙离子积累。通过透射电镜观察焦锑酸钾钙离子原位沉淀超薄切片, 发现在头茬地黄拉线期的块根分裂形成层组织细胞内, 只有很少量的钙离子黑色沉淀颗粒, 多数都分布在细胞膜和液泡膜等膜结构上, 钙沉淀颗粒小而分散; 而在同期的连作地黄块根分裂形成层组织细胞内, 则存在大量的钙离子沉淀黑色颗粒, 除了在膜结构上分布有大量钙沉淀颗粒, 在细胞质和液泡内也存在有大量钙沉淀颗粒散乱分布, 钙沉淀颗粒大而集聚; 含有钙离子黑色沉淀颗粒的切片经EGTA处理之后, 在电镜下观察到在原有钙沉淀的地方出现空洞, 说明电镜下观察到的黑色颗粒为钙离子沉淀(如图 1a~1d)。超薄切片电镜观察结果表明在连作地黄细胞内钙离子浓度增加, 细胞质内出现大量游离钙离子积累。两种钙离子浓度的检测方法均说明连作胁迫的影响促使地黄细胞内钙离子大量积累, 导致钙离子介导的信号通路出现紊乱。

|
Figure 1 Calcium fluorescence of root tip cells and calcium ion concentration of tuberous root cells in normal growth (one-year) and continuous cropped R. glutinosa. The comparison of calcium ion concentration in root tip cells and tuberous root cells between one-year (A, B, a, b) and continuous cropping (C, D, c, d) planted R. glutinosa. Calcium fluorescence: A-D; pyroantimonate precipitation: a-d; A-D: 10×; a, c: 3 000×; b, d: 5 000× |
利用生物信息学方法在地黄转录组共筛选得到84条地黄钙信号感知与传递蛋白基因, 其中, 包括5个CaM家族基因、12个CBL家族基因、21个CDPK家族基因、21个CIPK家族基因、16个CML家族基因、9个CRK家族基因(表 2)。序列长度分析表明, 地黄CaM家族基因和拟南芥基因的平均相似度为83%, 基因平均长度为1 024 bp, 开放阅读框(ORF)长度均值为451 bp, 编码蛋白长度均值为149个氨基酸(aa); CBL家族基因和拟南芥基因的平均相似度为83%, 基因平均长度为1 429 bp, ORF长度均值为685 bp, 编码蛋白长度均值为226 aa; CDPK家族基因和拟南芥基因的平均相似度为79%, 基因平均长度为2 349 bp, ORF长度均值为1 619 bp, 编码蛋白长度均值为539 aa; CIPK家族基因和拟南芥基因的平均相似度为82%, 基因平均长度为2 382 bp, ORF长度均值为1 335 bp, 编码蛋白长度均值为444 aa; CML家族基因和拟南芥基因的平均相似度为88%, 基因平均长度为1 097 bp, ORF长度均值为597 bp, 编码蛋白长度均值为198 aa; CRK家族基因和拟南芥基因的平均相似度为80%, 基因平均长度为2 399 bp, ORF长度均值为1 732 bp, 编码蛋白长度均值为576 aa。采用Target P1.1分析其亚细胞定位预测结果表明, 有6个CBLs位于SP (signal peptide)、2个CMLs位于SP、2个CBLs位于mTP (mitochondrial targeting peptide)、3个CDPKs位于mTP、5个CIPKs位于mTP、1个CML位于mTP、6个CDPKs位于cTP (chloroplast transit peptide)、1个CIPK位于cTP、5个CMLs位于cTP、2个CRKs位于cTP。但仍有51个钙信号相关蛋白无法预测其相应的亚细胞位置。
| Table 2 The genes related to calcium signaling system identified in R. glutinosa. S: Signal peptide; M: Mitochondrial targeting peptide; C: Chloroplast transit peptide |
通过对所有钙信号相关蛋白进行聚类分析发现, 所有地黄钙信号体系相关基因可被明显聚类为两个部分, 一类是CML、CaM、CBL, 拥有典型的EF-hand结构域, 另一类是CRK、CDPK、CIPK, 这两类的主要区别在于蛋白结构域中是否有蛋白激酶域。进一步对钙信号相关蛋白的保守结构域进行分析发现, (如图 2B)地黄中CaM、CBL和CML家族蛋白均只含有EF-hand结构域, CRK家族和CDPK家族大部分同时含有EF-hand域和protein kinase-like域, 而CIPK家族基因均含3个或4个结构域, 除含有EF-hand域和protein kinase-like域外, 个别蛋白同时还拥有NAF域和NAF/FISL域。
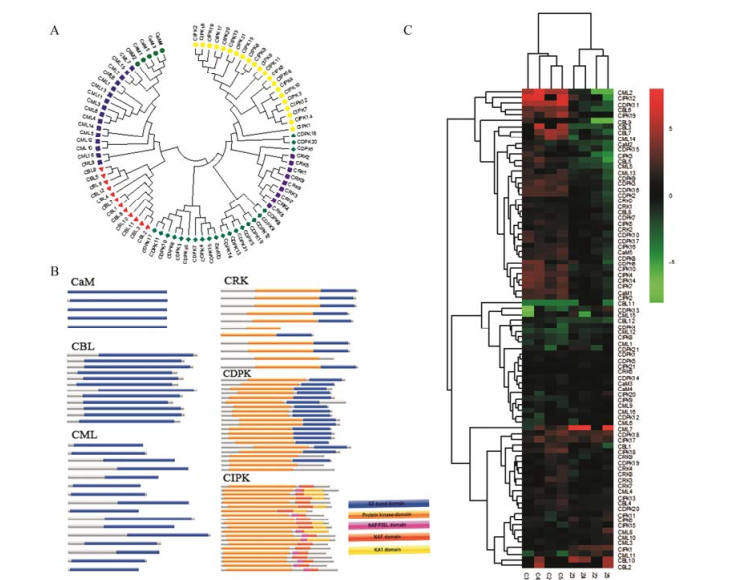
|
Figure 2 The phylogenetic relationship and conserved domains of calcium signaling related proteins, and expression pattern of calcium signaling related genes between first years and continuous planting R. glutinosa. A: Phylogenetic analysis; B: Conserved domain; C: Expression profiles. Note: C2-4 and Z2-4 indicated different developmental stages for continuous planting and one-year R. glutinosa, in which 2-4 represent elongation, pre-expanding, mid-expanding, late-expanding stage, respectively. The colors and its depth in legend show gene expression levels in different developmental stages. The reds and greens represent for regulated and downregulated expression in special stages in comparison to stages |
为了进一步阐明连作地黄钙信号相关基因对地黄连作胁迫的响应特征, 作者在前期RNA-seq数据的基础上, 对钙信号相关基因在连作和头茬中的表达模式进行重新定量。结果如图 2C表明, 在连作/头茬地黄不同生育阶段, 钙信号相关基因中, 有表达的基因共84个。对所有钙信号相关基因的表达模式聚类, 可以明显看出3种截然不同的表达模式, 第一种表达模式:在地黄发育进程中, 连作响应基因显著高于头茬, 此类基因共有37个; 第二种表达模式:在头茬和连作时期基因表达量均下调, 此类基因共有22个。第三种表达模式:基因表达量在头茬和连作不同时期存在上调情况但并不显著, 此类基因共有25个。此外, 第一个模式中的基因在连作条件下的响应和头茬条件下差异最大, 可能是响应连作特异的钙信号感知蛋白, 也是阐述地黄连作障碍机制的重要候选蛋白。
5 地黄中钙信号感知蛋白关键基因的表达模式分析为了深入分析验证钙信号体系相关基因在连作中的功能方式, 本研究从上述钙信号体系相关基因中选取CaM1、CaM3、CBL4、CBL6、CDPK11、CDPK20、CIPK2、CIPK10、CIPK20、CML2、CML6和CRK3这12个基因, 用qRT-PCR方法详细研究其在地黄连作形成中的功能表达模式(图 3)。结果从连作和头茬地黄不同发育时期整体来看, 部分钙信号基因随发育时期其表达量相对增强, 而部分钙信号基因的表达量随着发育进程的推进表达量则逐渐降低。从连作和头茬地黄在不同时期的表达量来看绝大部分钙信号基因在连作地黄不同时期的表达量显著高于头茬地黄, 但是部分钙信号相关基因在特定时期在连作地黄中表达量相对低于头茬。比如, CaM1的表达量在连作地黄的4个时期均上调表达, CBL6、CML2、CIPK10和CBL4在拉线期、膨大前期和膨大后期在连作中的表达量均显著高于头茬; CIPK2在膨大前期、膨大中期和膨大后期在连作中的表达量均高于头茬; CRK3在连作条件下在拉线期和膨大后期上调表达; CaM3和CML6在膨大后期连作地黄中表达量显著高于头茬地黄; CIPK20在连作条件下除膨大中期以外, 其余3个时期均下调表达; CDPK11在连作地黄拉线期表达量上调, 其余时期均下调; CDPK20则在膨大中期连作地黄中表达量显著高于头茬地黄, 其余时期均下调。从钙信号相关基因在连作和头茬地黄的整体表达趋势来看, 不同钙信号编码蛋白可能在地黄连作障碍形成过程的不同阶段承担相应角色, 一些钙信号基因可能在连作障碍形成整个过程深入参与, 一些钙信号基因可能只在连作障碍形成的某个特定时期起作用, 这些钙信号更像钙信号主要转导途径的候补角色, 但整体而言这些钙信号的蛋白相互补充和相互增益显著介导和促进了地黄连作障碍的形成。
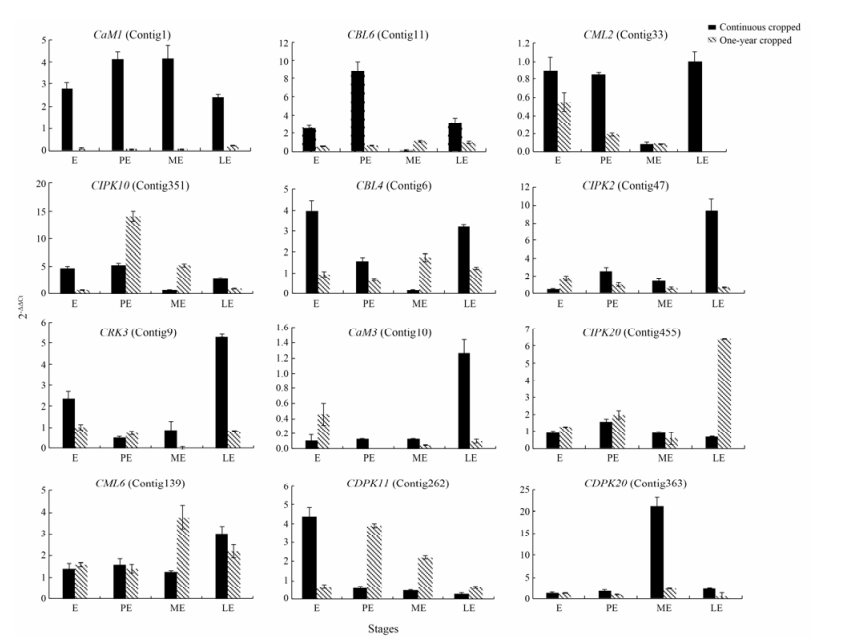
|
Figure 3 Expression analysis of 12 selected calcium signal related genes. E: Elongation; PE: Pre-elongation; ME: Mid-elongation; LE: Late-elongation |
在植物的生长发育过程中, 经常会遭受逆境胁迫的影响。植物在面对逆境胁迫时, 细胞膜会感知外界胁迫信号因子, 再通过膜受体的一系列磷酸化反应激活膜上的钙离子通道, 引发钙离子从钙库释放到细胞质中, 进而导致细胞质内钙离子浓度增加, 产生钙信号[29, 30]。如Dias等[31]发现在高温下小麦根中出现Ca2+积累, Tracy等[32]发现NaCl的胁迫水平与钙离子的升高程度有着密切关系, Bita等[33]发现热激能诱导拟南芥细胞内钙离子瞬时剧烈升高。因此, 越来越多的研究表明:钙信号传递系统是响应胁迫的重要系统, 而胞内钙离子浓度的增加是胁迫响应的一个主要标志[34-36]。连作障碍形成及加重发生的原因不是单一或孤立的, 而是“栽培植物-土壤-微生物”系统内多种因素综合作用的结果, 其主要根源在自毒物质诱导下的根际微生态失衡[37]。前期的大量研究表明药用植物化感自毒物质及其所诱发的环境灾变机制是连作障碍形成基本原因, 包含了连作植物体本身、微生物群体失衡和化感自毒物质影响等多元的胁迫因素。因此, 连作障碍就其本质而言同样是一种胁迫, 其体内的钙信号体系很有可能被扰动[37-39]。比如: Biedrzycki等[40]发现化感物质焦儿茶酸能引起拟南芥和白花蛇舌草细胞内钙离子浓度震荡, 并能引起钙信号系统调控基因的表达。Chi等[12]发现化感物质胡桃醌(Juglone)能引起水稻根尖细胞内的钙积累, 显著诱导根部CDPK的上调表达。本研究利用焦锑酸钾沉淀法和钙信号荧光染色法详细研究了连作障碍形成过程中钙离子浓度的状态变化, 结果显示在连作地黄根尖组织细胞内, 钙离子浓度显著大于头茬, 并且钙离子在细胞内的分布散乱, 证实了连作条件下诱发了地黄根细胞内钙离子浓度的增加, 表明连作地黄体内的钙离子被大量释放。
外界胁迫所诱发的钙离子信号, 只有通过细胞内的钙调素(CaM)、钙依赖蛋白激酶(CDPK)、钙磷脂酶(CBL)等一系列靶蛋白才能最终诱发植物体内的一系列胁迫响应。钙靶蛋白又通过和其他靶蛋白分子结合而启动基因表达, 从而构成钙信号转导的复杂体系, 传递各种外界信号刺激。虽然课题组前期明确证实了钙信号参与地黄连作障碍的形成[7, 41], 但地黄中究竟是哪些钙信号传递了连作的伤害, 目前仍未被证实。本研究通过生物信息学、高通量测序的方法鉴定了共84个在地黄中特异表达的钙信号体系相关基因, 其中包括5个CaMs家族基因、12个CBLs家族基因、21个CDPKs家族、21个CIPKs家族基因、16个CMLs家族、9个CRKs家族基因。根据钙信号相关基因在连作和头茬地黄发育进程中的表达模式差异, 可以将地黄钙信号相关基因分为三种功能模式。第一种钙信号相关基因在连作地黄的发育进程中显著高于头茬, 这些钙信号基因很可能参与了地黄连作胁迫进程, 在连作障碍的形成中起着重要的作用; 第二种是钙信号相关基因表达量在头茬和连作地黄不同发育时期随着发育进程推进显著上调表达, 这些钙信号体系相关基因可能同时参与连作胁迫和地黄植株生理发育的过程; 第三种钙信号相关基因在连作和头茬地黄不同发育时期表达模式基本相似, 并且在不同发育时期其表达丰度变化不大, 这些基因可能在地黄发育进程中更多的是一种组成性表达, 可能与植物基本发育有关。
对于本研究而言, 更多关注的是第一类地黄钙信号基因, 这些基因表达与地黄连作障碍形成症状具有明显的契合性、同步性。对这类基因的详细解析可以进一步为深入地理解钙信号在连作障碍形成过程中的具体机制提供重要参考。本研究共获取了37个连作障碍中显著上调钙信号基因, 这些基因中值得注意的是CaM1、CBL6、CML2和CIPK10, 这4个基因在不同作物中已经有了较为明晰的功能。其中, CaM1在拟南芥细胞中与钙信号传递相关, 主要存在于保卫细胞、维管束和叶中, 有束缚钙离子的作用; CBL6在拟南芥全株中都有表达, 在花粉和胚的发育等过程中都有参与, 还有着束缚钙离子的作用; CML2在拟南芥的花和叶中有表达, 有束缚钙离子的功能; CIPK10在拟南芥的花、茎、根中都有表达, 能编码丝氨酸、苏氨酸蛋白激酶, 在甘露醇影响下、低温胁迫、盐害条件下有响应作用。此外, 在第二种模式下, 连作和头茬地黄钙信号基因均为上调表达, 可能在地黄连作形成中具有双向的功能, 一方面参与了连作胁迫, 另一方面可能与植物正常发育进程密切相关。例如:在此类中CIPK13在拟南芥中有着结合ATP和调节激酶活性等作用, CIPK10既有结合钙离子的功能, 在信号转导过程中响应, 也能应答脱落酸和调节激酶活性等。此外, 前期实验证明钙信号除了参与连作胁迫响应外, 还与地黄的块根启动和发育密切相关[42]。不同研究也证明了钙信号广泛参与了植物根系等进程, 虽然通过钙离子荧光信号检测发现头茬地黄的根系内钙离子荧光较低, 但仍然能够明显看见一些钙信号。可以推测并非所有的钙信号均对连作响应, 而部分钙信号承担着植物的基本生长发育功能, 因此, 这部分钙信号基因具有典型的双向调节功能, 适当的钙离子浓度可能促进了植物的发育, 而过多钙离子可能响应胁迫毒害。
| [1] | Wen XS, Yang SL, Wei JH, et al. Textual research on planting history of Rehmannia glutinosa and its cultivated varieties[J]. Chin Herb Drugs (中草药), 2002, 33: 946–949. |
| [2] | Zhang ZY, Chen H, Yang YH, et al. Effects of continuous cropping on bacterial community diversity in rhizosphere soil of Rehmannia glutinosa[J]. Chin J Appl Ecol (应用生态学报), 2010, 21: 2843–2848. |
| [3] | Chen H, Hao HR, Xiong J, et al. Effects of successive cropping Rehmannia glutinosa on rhizosphere soil microbial flora and enzyme activities[J]. Chin J Appl Ecol (应用生态学报), 2007, 18: 2755–2759. |
| [4] | Guo LC, Liu CH, Zuo GS, et al. Mechanism analysis of continuous cropping obstacle of Rehmannia glutinosa[J]. Chin Agric Sci Bull (中国农学通报), 2012, 28: 227–230. |
| [5] | Du JF, Yin WJ, Li J, et al. Dynamic change of phenolic acids in soils around rhizosphere of Replanted Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2009, 34: 948–952. |
| [6] | Liu HY, Wang F, Wang YP, et al. The causes andcontrol of continuous cropping barrierin Dihuang (Rehmannia glutinosa Libosch.)[J]. Acta Agric Boreali-Sin (华北农学报), 2006, 21: 131–132. |
| [7] | Zhang ZY, Li MJ, Chen XJ, et al. Research advancement and control strategy of consecutive monoculture problem of Rehmannia glutinosa L[J]. Mod Chin Med (中国现代中药), 2013, 15: 38–44. |
| [8] | Yang YH, Li MJ, Wang PF, et al. Transcriptome-wide identification of the genes responding to replanting disease in Rehmannia glutinosa L[J]. Mol Biol Rep, 2015, 42: 881–892. DOI:10.1007/s11033-014-3825-y |
| [9] | Yang YH, Li MJ, Wang PF, et al. De novo characterization of the Rehmannia glutinosa leaf transcriptome and analysis of gene expression associated with replanting disease[J]. Mol Breeding, 2014, 34: 905–915. DOI:10.1007/s11032-014-0084-5 |
| [10] | Fan HM, Li MJ, Zheng HY, et al. Spatiotemporal expression and analysis of responding consecutive monoculture genes in Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2012, 37: 3029–3035. |
| [11] | Guo GY, Li MJ, Wang PF, et al. Abnormal change of calcium signal system on consecutive monoculture problem of Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 1471–1478. |
| [12] | Chi WC, Fu SF, Huang TL, et al. Identification of transcriptome profiles and signaling pathways for the allelochemical juglone in rice roots[J]. Plant Mol Biol, 2011, 77: 591–607. DOI:10.1007/s11103-011-9841-6 |
| [13] | Xie LJ. Effects of Ca2+ on Some Physiological Metabolisms of the Cucumber under Cinnamic Acid Stress (钙对苯丙烯酸胁迫下黄瓜若干生理指标的影响)[D]. Fuzhou:Fujian Agriculture and Forestry University, 2007. http://d.wanfangdata.com.cn/Thesis/Y1175380 |
| [14] | Wu B, Long Q, Gao Y, et al. Comprehensive characterization of a time-course transcriptional response induced by autotoxins in Panax ginseng, using RNA-Seq[J]. BMC Genomics, 2015, 16: 1–15. DOI:10.1186/1471-2164-16-1 |
| [15] | Shang ZL, Mao GH, Sun DY. The specifity of calcium signaling in plant cells[J]. Plant Physiol J, 2003, 39: 93–100. |
| [16] | Zheng GH, Pan DM, Niu XQ, et al. Changes in cell Ca2+ distribution in loquat leaves and its effects on cold tolerance[J]. Korean J Hort Sci Technol, 2014, 32: 607–613. DOI:10.7235/hort.2014.13009 |
| [17] | Yang WX. Effects of Calcium on Heat Stress Signal Transduction and Physiological Response in Leaves of Pinellia ternate (钙转导半夏高温信号及其生理响应)[D]. Chongqing:Southwest University, 2015. http://d.wanfangdata.com.cn/Thesis/Y2812185 |
| [18] | Zhao G, Shi Q, Yu H, et al. The physiological and biochemical responses of a medicinal plant (Salvia miltiorrhiza L.) to stress caused by various concentrations of NaC[J]. PLoS One, 2014, 9: e89624. DOI:10.1371/journal.pone.0089624 |
| [19] | Lu JH, Lv X, Liang YC, et al. Salt tolerance of Glycyrrhiza inflata seedlings in Xinjiang and its ion response to salt stress[J]. Chin J Plant Ecol (植物生态学报), 2013, 37: 839–850. |
| [20] | Kudla J, Batistic O, Hashimoto K. Calcium signals:the lead currency of plant information processing[J]. Plant Cell, 2010, 22: 541–563. DOI:10.1105/tpc.109.072686 |
| [21] | Dodd AN, Kudla J, Sanders D. The language of calcium signaling[J]. Ann Rev Plant Biol, 2010, 61: 593–620. DOI:10.1146/annurev-arplant-070109-104628 |
| [22] | Wang FQ, Tian YH, Li MJ, et al. Molecular cloning and expression analysis of an Aux/IAA gene (RgIAA1) from Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 4033–4039. |
| [23] | Wu LK, Chen J, Wu HM, et al. Comparative proteomics analysis of R. glutinosa tuber root in response to consecutive monoculture[[J]. Acta Agron Sin (作物学报), 2016, 42: 243–254. DOI:10.3724/SP.J.1006.2016.00243 |
| [24] | Wang PF, Li XY, Li MJ, et al. Observation of prime position and driving zones in process of tuberous root expanding and expression analysis of phytohormone relative genes in Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 3245–3253. |
| [25] | Qi LJ, Yuan Y, Wu C, et al. Bioinformatics analysis of DNA demethylase genes in Lonicera japonica Thunb[J]. Acta Pharm Sin (药学学报), 2015, 50: 367–371. |
| [26] | Feng FJ. The Screening and Functional Analysis of the Key Genes Responding to Continuous Monoculture Problems during the Development of Rehmannia glutinosa Libosch (地黄连作障碍形成过程中关键响应基因的筛选与功能分析)[D]. Fuzhou:Fujian Agriculture and Forestry University, 2015. |
| [27] | Wang FQ, Zhou Y, Huang Y, et al. Cloning and expression analysis of the expansin gene RgEXPA10 in Rehmannia glutinosa[J]. Acta Pharm Sin (药学学报), 2015, 50: 233–240. |
| [28] | Livak KJ, Sehmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△CT method[J]. Methods, 2001, 25: 402–408. DOI:10.1006/meth.2001.1262 |
| [29] | Alvarez ME, Nota F, Cambiagno DA. Epigenetic control of plant immunity[J]. Mol Plant Pathol, 2010, 11: 563–576. DOI:10.1111/j.1364-3703.2010.00621.x |
| [30] | Hashimoto K, Kudla J. Calcium decoding mechanisms in plants[J]. Biochimie, 2011, 93: 2054–2059. DOI:10.1016/j.biochi.2011.05.019 |
| [31] | Dias AS, Lidon FC, Ramalho JC.. I. Heat stress in triticum:kinetics of Ca2+ and Mg2+ accumulation[J]. Braz Soc Plant Physiol, 2009, 21: 123–124. DOI:10.1590/S1677-04202009000200005 |
| [32] | Tracy FE, Gilliham M, Dodd AN, et al. NaCl-induced changes in cytosolic free Ca2+ in Arabidopsis thaliana are heterogeneous and modified by external ionic composition[J]. Plant Cell Environ, 2008, 31: 1063–1073. DOI:10.1111/pce.2008.31.issue-8 |
| [33] | Bita CE, Gerats T. Plant tolerance to high temperature in a changing environment:scientific fundamentals and production of heat stress-tolerant crops[J]. Front Plant Sci, 2013, 4: 273. |
| [34] | Bose J, Pottosin Ⅱ, Shabala SS, et al. Calcium efflux systems in stress signaling and adaptation in plants[J]. Front Plant Sci, 2011, 2: 85. |
| [35] | Whalley HJ, Sargeant AW, Steele JF, et al. Transcriptomic analysis reveals calcium regulation of specific promoter motifs in Arabidopsis[J]. Plant Cell, 2011, 23: 4079–4095. DOI:10.1105/tpc.111.090480 |
| [36] | Krasensky J, Jonak C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks[J]. J Exp Bot, 2012, 63: 1593–1608. DOI:10.1093/jxb/err460 |
| [37] | Chen AG, Li MJ, Zhang B, et al. Review on catastrophe mechanism of medicinal plant and its rhizosphere microecosystem mediated by consecutive monoculture[J]. Mod Chin Med (中国现代中药), 2016, 18: 239–245. |
| [38] | Wu L, Wang J, Huang W, et al. Corrigendum:plant-microbe rhizosphere interactions mediated by Rehmannia glutinosa root exudates under consecutive monoculture[J]. Sci Rep, 2015, 6: 19101. |
| [39] | Li M, Yang Y, Feng F, et al. Differential proteomic analysis of replanted Rehmannia glutinosaroots by iTRAQ reveals molecular mechanisms for formation of replant disease[J]. BMC Plant Biol, 2017, 17: 116. DOI:10.1186/s12870-017-1060-0 |
| [40] | Biedrzycki ML, Venkatachalam L, Bais HP. Transcriptome analysis of Arabidopsis thaliana plants in response to kin and stranger recognition[J]. Plant Signal Behav, 2011, 6: 1515–1524. DOI:10.4161/psb.6.10.16525 |
| [41] | Guo GY, Li MJ, Wang PF, et al. Abnormal change of calcium signal system on consecutive monoculture problem of Rehmannia glutinosa[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 1471–1478. |
| [42] | Li MJ, Yang YH, Li XY, et al. Analysis of integrated multiple 'omics' datasets reveals the mechanisms of initiation and determination in the formation of tuberous roots in Rehmannia glutinosa[J]. J Exp Bot, 2015, 66: 5837–5851. DOI:10.1093/jxb/erv288 |
 2017, Vol. 52
2017, Vol. 52


