肿瘤作为全球范围内发病率和死亡率都很高的一种疾病, 严重威胁着人类健康, 成为世界各国面临的重要社会问题之一。大量的研究发现肿瘤的发生、发展与一些致癌蛋白的过度表达密切相关, 针对这些致癌蛋白的小分子抑制剂的研发, 让肿瘤的靶向治疗取得了很大的进步。然而, 小分子抑制剂类抗肿瘤药物的研发也面临着诸多问题:靶蛋白容易发生结构突变或者激活其他致癌蛋白, 产生耐药性[1, 2]; 部分蛋白抑制其活性后会反馈性上调蛋白表达, 限制其抗肿瘤活性[3, 4]; 多数蛋白没有足够大的结合口袋, 如转录因子等非酶蛋白, 开发小分子抑制剂难度较大[5]; 可逆性小分子抑制剂往往需要较大剂量, 以保证足够的结合率维持疗效, 易出现脱靶效应, 产生不良反应[6]。
与小分子抑制剂通过结合致癌蛋白活性位点抑制其活性不同, 调控这些致癌蛋白的降解是抗肿瘤药物研发的另一策略。RNA干扰[7]、CRISPR/Cas9[8]等技术是近年来发展起来的基因敲除技术, 因其能够特异而有效地阻断靶基因的表达, 已被广泛用于基因功能和疾病治疗研究。但是由于核苷类物质的体内不稳定性、易产生机体免疫应答、体内脱靶效应等问题, 限制了其临床应用[9, 10]。研发可特异性降解致癌蛋白的小分子化合物, 成为目前抗肿瘤研发的热点领域之一。
1 PROTACs降解蛋白机制泛素介导的蛋白质降解是细胞内蛋白质最主要的负向调节方式。泛素化降解途径可以降解细胞内80%~90%泛素化的蛋白质, 参与调节细胞周期、增殖、凋亡、转移、基因表达、信号传递等几乎一切生命活动。该过程是在泛素激活酶E1、泛素结合酶E2和泛素连接酶E3的协同作用下进行, 底物蛋白被泛素化后, 在蛋白酶体中被降解, 依靠泛素连接酶E3对底物蛋白特异的识别能力, 决定了泛素介导的蛋白降解具有特异性。
2001年, Deshaies课题组[11]首次报道了一种利用泛素-蛋白酶体降蛋白质的方法:蛋白水解靶向嵌合分子(protein proteolysis-targeting chimeras, PROTACs)。PROTACs本质上是一种杂合双功能小分子化合物, 其结构中含有两种不同配体:一个是泛素连接酶E3配体, 另一个是与细胞中目标靶蛋白结合配体, 两个配体之间通过连接基相连。PROTACs通过将目标靶蛋白和细胞内的E3拉近, 形成靶蛋白-PROTACs-E3三元聚合体, 通过E3泛素连接酶给目标靶蛋白加上泛素化蛋白标签, 启动细胞内强大的泛素化水解过程, 利用泛素-蛋白酶体途径特异性地降解靶蛋白(图 1)。Ciulli研究小组[12]利用靶蛋白-PROTACs-E3三元聚合体的共晶结构和等温滴定量热法实验, 证实PROTAC可以通过自身折叠将靶蛋白与E3拉近, 三者之间通过相互协同作用埋藏在蛋白质表面, 形成特异性的蛋白-蛋白相互作用, 让靶蛋白-PROTACs-E3三元聚合物比靶蛋白-PROTACs和PROTACs-E3二元复合物具有更好的稳定性。
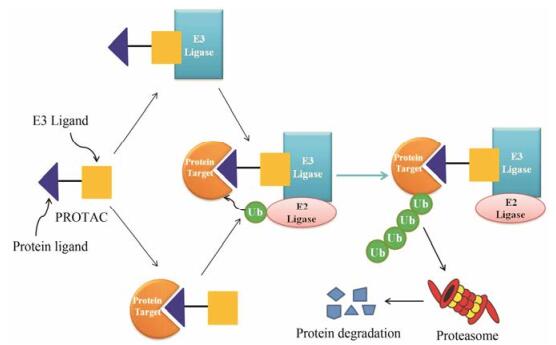
|
Figure 1 Proposed model of PROTACs-induced protein degradation |
2001年, Deshaies课题组[11]报道了第一个PROTAC分子PROTAC-1 (图 2), 首次清晰地展示了PROTAC的概念。PROTAC-1通过连接基将血管生成抑制剂卵假散囊菌素(ovalicin, OVA)与核因子κB抑制蛋白α (NF-κB inhibitor α, ⅠκBα)结构中的由10个氨基酸残基构成的磷酸肽相连。OVA可以与蛋氨酸氨肽酶2 (amino peptidase-2, MetAP-2)活性位点中第231位组氨酸共价结合并抑制其活性, ⅠκBα可以与SCFβ-TRCP E3泛素连接酶蛋白复合体中的β-TRCP结合。PROTAC-1通过将MetAP-2与SCFβ-TRCP拉近, 利用泛素-蛋白酶体系统泛素化降解MetAP-2。
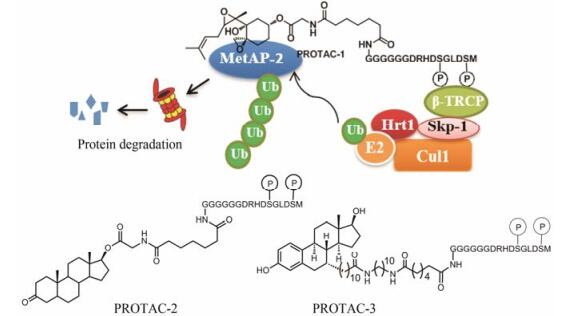
|
Figure 2 Reported peptidic PROTACs that recruit the SCFβ-TRCP E3 ligase |
Deshaies课题组[13]随后又利用该策略设计合成了降解雄激素受体(androgen receptor, AR)的PROTAC-2与降解雌激素受体α (estrogen receptor α, ERα)的PROTAC-3。AR与ERα均属于核受体超家族成员, 参与机体多种生理病理过程的调控, 与肿瘤的发生、发展密切相关。PROTAC-2通过连接基将AR小分子配体二氢睾酮与ⅠκBα磷酸肽相连, PROTAC-3则由ERα小分子配体雌二醇、连接基和ⅠκBα磷酸肽组成。
实验结果证明PROTAC1-3确实可以泛素-蛋白酶体依赖性地降解目标靶蛋白, 但是由于结构中存在磷酸肽结构, PROTAC1-3的细胞通透性较差, 且易于被磷酸酶水解, 导致降解细胞内目标靶蛋白的能力很弱。
2.2 基于CRL2VHL E3的多肽类PROTACs希佩尔-林道蛋白(von Hippel-Lindau protein, pVHL)是CRL2VHL E3泛素连接酶的底物受体。在细胞处于正常氧分压条件下时, 缺氧诱导因子1α (hypoxia-inducible factor 1alpha, HIF-1α)的Pro402和Pro564残基被脯氨酰羟化酶羟基化后与VHL蛋白结合, 进而被CRL2VHL E3泛素化降解。
Sakamoto等[14]利用HIF-1α蛋白中可以与CRL2VHL E3结合的羟基化五肽结构, 设计合成了PROTAC-4和PROTAC-5 (图 3)。PROTAC-4由AR小分子配体二氢睾酮、连接基和HIF-1α羟基化五肽组成。用12.5 μmol·L-1 PROTAC-4处理LNCaP前列腺癌细胞72 h后可以降低细胞内AR表达水平, 使细胞周期阻滞在G1期, 抑制细胞的增殖(72 h IC50 = 12.5 μmol·L-1, 144 h IC50 = 1.52 μmol·L-1), 而对不表达AR的PC-3和DU-145前列腺癌细胞, PROTAC-4则不能抑制细胞的增殖, 显示出对AR的靶向降解。PROTAC-5由ERα小分子配体雌二醇、连接基和HIF-1α羟基化五肽组成。50 μmol·L-1 PROTAC-5可以明显降低雌激素依赖的MCF-7和T47D乳腺癌细胞内ERα表达水平, 下调Cyclin D1, 阻滞细胞周期, 抑制细胞增殖。对雌激素非依赖的SKBr3细胞株, PROTAC-5不具有抗增殖活性。

|
Figure 3 Reported peptidic PROTACs that recruit the CRL2VHL E3 ligase |
Crews等[15]设计了一个活性受磷酸化调节的PROTAC分子ErbB2PPPI3K。ErbB2PPPI3K由一段源自受体酪氨酸激酶ErbB2的氨基酸序列、VHL结合氨基酸序列与连接基组成。在神经生长素的作用下, ErbB2PPPI3K结构中的ErbB2氨基酸序列发生磷酸化, 与ErbB2底物蛋白PI3K结合, 进而招募CRL2VHL E3泛素化降解PI3K, 抑制激酶信号通路。在OVCAR8卵巢癌细胞裸鼠移植瘤模型中, 腹腔注射10 mg·kg-1 ErbB2PPPI3K 47天可以有效抑制肿瘤的生长, 抑瘤率达到40%。
3 小分子PROTACs的研究进展在过去的十几年中, 由于缺乏高亲和性、特异性的E3小分子配体, 设计合成的PROTACs分子都是多肽类化合物, 很难穿透细胞膜, 降解靶蛋白的效果不佳, 限制了PROTACs技术的应用。近几年, CRL4CRBN[16, 17]、CRL2VHL[18]、cIAP[19]等E3泛素连接酶特异性小分子配体的发现, 让PROTACs技术取得了巨大的突破。多个研究小组利用PROTACs实现了对溴结构域蛋白4 (BRD4)、BCR-ABL、ERα、雌激素相关受体α (ERRα)、丝氨酸苏氨酸激酶2 (RIPK2)和转录相关酸性卷曲蛋白3 (TACC3)等多种癌症相关蛋白的降解, 成为抗肿瘤药物研发领域的热点之一, 有望在肿瘤靶向药物研究方面实现新的突破[20-26]。
3.1 基于MDM2 E3的小分子PROTACs2008年, Crews课题组[27]报道了第一个小分子PTOTAC: PROTAC_AR (图 4)。PROTAC_AR通过聚乙二醇连接基将MDM2 E3泛素连接酶小分子抑制剂nutlin与非甾体雄激素受体小分子配体相连。用10 μmol·L-1 PROTAC_AR处理人宫颈癌HeLa细胞7 h, 可以降低细胞内AR水平。而如果用蛋白酶体抑制剂环氧霉素预处理HeLa细胞, 则PROTAC_AR不能够降解HeLa细胞内AR, 说明PROTAC_AR是通过蛋白酶体泛素化降解AR。虽然PROTAC_AR降解AR的效率很低, 但是PROTAC_AR的报道为小分子PTOTAC化合物的研究提供了思路。
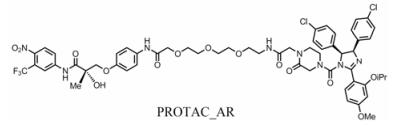
|
Figure 4 Reported small-molecule PROTAC that recruits the MDM2 E3 ligase |
细胞凋亡抑制蛋白1 (cellular inhibitor of apoptosis protein 1, cIAP1)是高度保守的内源性抗细胞凋亡因子, 主要包含BIR、CARD和RING 3个结构域, 其中位于碳端的RING结构域具有E3泛素连接酶活性。乌苯美司(bestatin)是一个氨肽酶抑制剂, 研究发现其甲酯化合物(MeBS)可以通过乌苯美司结构端与cIAP1的BIR3结构域结合, 激活cIAP1的E3泛素连接酶活性, 诱导cIAP1发生自泛素化, 然后被蛋白酶体降解[19, 28]。利用这一特点, 多个研究小组在乌苯美司甲酯化合物的甲酯端通过连接基连接不同的靶蛋白小分子抑制剂, 得到了多个基于cIAP1 E3的PROTACs (图 5)。
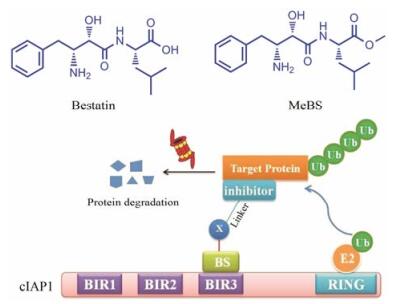
|
Figure 5 PROTAC mechanism of action utilizing the E3 ubiquitin ligase cIAP1 |
细胞视黄酸结合蛋白Ⅱ (cellular retinoic acid-binding protein Ⅱ, CRABP-Ⅱ)是细胞内视黄酸的结合蛋白, 将视黄酸转运至细胞核, 使其与受体作用调节下游基因表达。CRABP-Ⅱ的异常表达与成神经细胞瘤、肾母细胞瘤、头颈鳞状细胞癌的发生发展密切相关。2010年, Hashimoto研究组[29]报道了第一个基于cIAP1 E3降解CRABP-Ⅱ的PROTAC分子SNIPER-(2) (图 6)。SNIPER-(2)通过连接基将全反式视黄酸与乌苯美司甲酯相连。由于乌苯美司甲酯可以诱导cIAP1自身发生泛素化降解, 因此SNIPER-(2)在泛素化降解CRABP-Ⅱ的同时会泛素化降解cIAP1, 在很大程度上限制了其降解CRABP-Ⅱ的效率。通过对乌苯美司进一步的结构改造发现, 乌苯美司的酰胺衍生物保留了与cIAP1的结合活性, 并且不会引起cIAP1的自身泛素化降解。2011年, Mikihiko等[30]在此基础上设计合成了SNIPER-(4)。10 μmol·L-1 SNIPER-(4)可以明显降低人神经母细胞瘤IMR-32细胞内CRABP-Ⅱ蛋白, 而对cIAP1的蛋白水平没有影响。SNIPER-(4)通过降解CRABP-Ⅱ下调MycN蛋白水平, 激活caspase-3/7, 抑制细胞的增殖。对不表达MycN蛋白的人成纤维肉瘤HT1080细胞和人乳腺癌MCF-7细胞, SNIPER-(4)虽然可以降低细胞内CRABP-Ⅱ蛋白水平, 但是对细胞的增殖几乎没有抑制作用。2013年, Mikihiko研究小组[31]将ERα调节剂4-羟基他莫昔芬通过连接基与乌苯美司酰胺衍生物相连, 合成得到了SNIPER (ERα)。10 μmol·L-1 SNIPER (ERα)可以显著降低人乳腺癌MCF-7细胞内ERα表达量, 抑制雌激素依赖的pS2基因的表达。用30 μmol·L-1 SNIPER (ERα)处理MCF-7细胞6 h后, 细胞内ROS水平显著升高, 细胞发生明显坏死。
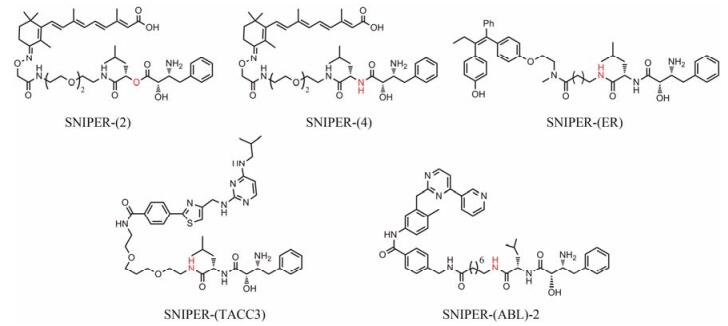
|
Figure 6 Reported small-molecule PROTACs that recruit the cIAP1 E3 ligase |
TACC3是细胞有丝分裂过程中重要的纺锤体形成调控蛋白。TACC3在宫颈癌、乳腺癌、肺癌、淋巴瘤、胶质母细胞瘤等癌症中高表达, 与肿瘤的发生密切相关。抑制TACC3的活性或者降低其表达量可以抑制细胞分裂、诱导细胞凋亡, 是目前抗肿瘤药物研发的一个重要靶点。30 μmol·L-1 SNIPER (TACC3) 6 h内可以明显降低人纤维肉瘤HT1080细胞、人乳腺癌MCF-7细胞和人骨肉瘤U2OS细胞内的TACC3表达水平。用10 μmol·L-1 SNIPER (TACC3)处理HT1080或者MCF-7细胞48 h, 可以裂解PARP, 引起caspase-3活化, 诱导细胞凋亡, 抑制细胞的增殖。SNIPER (TACC3)对高表达TACC3蛋白的肿瘤细胞具较好抑制活性, 而对人正常纤维原细胞抑制活性较差, 显示出较好的选择性[32]。
SNIPER (ABL)-2由BCR-ABL抑制剂伊马替尼、连接基和乌苯美司酰胺衍生物组成。30 μmol·L-1 SNIPER (ABL)-2处理人髓性白血病K562细胞8 h, 可以明显降低细胞内BCR-ABL蛋白水平, 下调BCR-ABL下游的STAT5和CrkL蛋白水平, 抑制K562细胞的增殖[33]。
3.3 基于CRL2VHL E3的小分子PROTACsHIF-1α的Pro402和Pro564残基被脯氨酰羟化酶羟基化后可以与VHL蛋白结合, 其中羟基化的Pro564残基在HIF-1α与VHL的结合中起着关键的作用, Ciulli等[18, 34]在4-羟基脯氨酸的基础上经过结构改造, 得到了能与VHL特异性结合的小分子配体(图 7)。
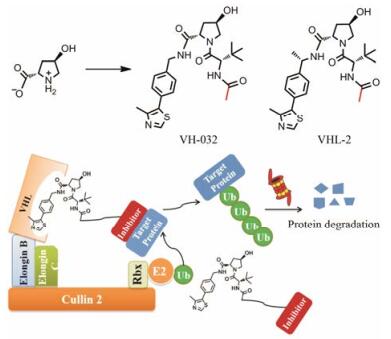
|
Figure 7 PROTAC mechanism of action utilizing the E3 ubiquitin ligase CRL2VHL |
化合物VH032与VHL蛋白的共晶结构显示, VH032末端乙酰基的甲基处于溶剂暴露区, 是PROTACs连接基的合适连接位置。2015年, Ciulli研究小组[35]采用聚乙二醇连接基将JQ1的羧基与VH032末端乙酰基的甲基相连, 得到了通过招募CRL2VHL E3降解BRD4的PROTACs: MZ1和MZ2 (图 8)。MZ1显示了比MZ2更加高效的BRD4降解活性, 说明连接基的长短对PROTACs的活性有着重要影响。100 nmol·L-1 MZ1可以显著降解人宫颈癌HeLa细胞与人骨肉瘤U2OS细胞内的BRD4, 下调c-Myc的表达。虽然JQ1为BET非选择性抑制剂, MZ1却在较低浓度时对BRD2、BRD3显示出较好的选择性。

|
Figure 8 Reported small-molecule PROTACs that recruitthe CRL2VHL E3 ligase |
2016年, Coleman等[36]利用JQ1与VHL小分子配体VHL-2设计合成了PROTAC分子ARV-771。ARV-771可以快速降解去势抵抗性前列腺癌22Rv1、LnCaP95和VCaP细胞中BRD4蛋白(DC50 < 5 nmol·L-1), 抑制c-Myc的表达(IC50 < 1 nmol·L-1)。虽然ARV-771、JQ1与BRD4的Kd值相近, 但是ARV-771下调c-Myc表达水平的活性是JQ1的10余倍, 显示出了催化降解蛋白的能力。300 nmol·L-1 ARV-771可以显著诱导22Rv1细胞内PARP蛋白裂解, 激活capase-3/7, 诱导细胞凋亡, 其抗细胞增殖活性是JQ1的10余倍。在人前列腺癌AR-V7阳性22Rv1细胞Nu/Nu裸鼠皮下移植瘤模型中, ARV-771可以下调BRD4和AR-V7蛋白水平, 相比OTX015 80%的抑瘤率, ARV-771可以使肿瘤消退, 部分小鼠的肿瘤完全消失。
ERRα在雌激素依赖性肿瘤如乳腺癌及非雌激素依赖性肿瘤如结直肠癌中均高表达, ERRα是乳腺癌预后较差的一个分子标记物。PROTAC_ERRα通过连接基将ERRα噻唑烷二酮类小分子配体与CRL2VHL E3小分子配体相连。PROTAC_ERRα可以显著降低人乳腺癌MCF-7细胞内ERRα蛋白水平, DC50为100 nmol·L-1。在人乳腺癌MDA-MB-231细胞裸鼠移植瘤模型中, PROTAC_ERRα可以降低肿瘤组织中50%的ERRα表达[37]。
3.4 基于CRL4CRBN E3的小分子PROTACs沙利度胺及其类似物泊马度胺、来那度胺是目前常用的免疫调节药物, 并且对多发性骨髓瘤具有较好疗效(图 9)。研究发现, 沙利度胺类药物可以通过其戊二酰亚胺结构与CUL4-RBX1-DDB1-Cereblon (CRL4CRBN) E3泛素连接酶复合物的底物受体Cereblon蛋白结合, 促进转录因子IKZF1/3与Cereblon的结合, 诱导IKZF1/3发生泛素化降解[38-40]。近几年, 利用沙利度胺及其类似物可以与CRL4CRBN E3特异性结合的特点, 将不同的靶蛋白小分子抑制剂通过连接基与沙利度胺或其类似物连接, 设计合成了多个基于CRL4CRBN E3的PROTACs。
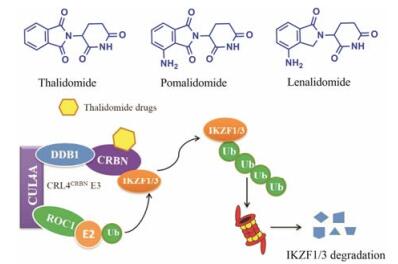
|
Figure 9 Reported CRL4CRBN E3 small-molecule ligands |
BRD4是含溴结构域和超末端结构(bromodomain and extraterminal domain, BET)蛋白家族成员。BRD4通过溴结构域结合组蛋白乙酰化赖氨酸残基, 在调节细胞基因转录、细胞周期等生物过程中发挥重要作用。BRD4的过度表达与急性髓性白血病、乳腺癌和黑色素瘤等多种恶性肿瘤的发生发展密切相关, 是目前抗肿瘤药物研发的热点靶标之一, 目前已经有多个小分子抑制剂进入临床试验(图 10)。BRD4小分子抑制剂通过和BRD4溴结构域结合, 阻断其与组蛋白乙酰化赖氨酸残基的相互作用, 调节下游基因表达。但是用小分子抑制剂抑制BRD4活性后会反馈性上调BRD4基因的表达, 造成对下游信号通路抑制不够充分, 限制了其疗效。构效关系研究发现BRD4小分子抑制剂的羧基结构[41]和沙利度胺的苯环结构为可修饰结构[39]。多个研究小组利用不同的连接基将BRD4小分子抑制剂羧基端与沙利度胺的苯环端相连, 得到了多个靶向降解BRD4的PROTACs。
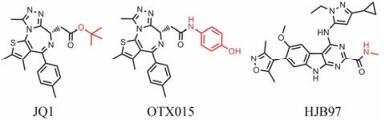
|
Figure 10 Reported BRD4 small-molecule inhibitors |
2015年, Bradner等[42]报道了靶向降解BRD4的PROTAC小分子dBET1 (图 11)。dBET1通过连接基将BRD4抑制剂JQ1与CRL4CRBN E3小分子配体沙利度胺相连。与JQ1抑制BRD4活性后反馈性引起细胞内BRD4表达上调不同, 100 nmol·L-1 dBET1在2 h内可以完全降解急性髓性白血病MV4:11细胞中的BRD4, 显示出比JQ1更加显著持久的下调c-Myc基因、诱导细胞凋亡和抑制细胞增殖能力。在小鼠MV4:11白血病后肢移植瘤模型中, 与JQ1相比, dBET1可以更加有效地抑制肿瘤生长。单独用JQ1、沙利度胺或者二者同时处理MV4:11细胞不能下调BRD4蛋白水平; 用蛋白酶体抑制剂处理MV4:11细胞可以抑制dBET1下调BRD4蛋白水平的活性; 在Cereblon蛋白缺失的多发性骨髓瘤MM1.S-CRBN-/-细胞中, dBET1不能降低BRD4蛋白水平, 说明dBET1是通过招募CRL4CRBN E3泛素化降解BRD4蛋白。
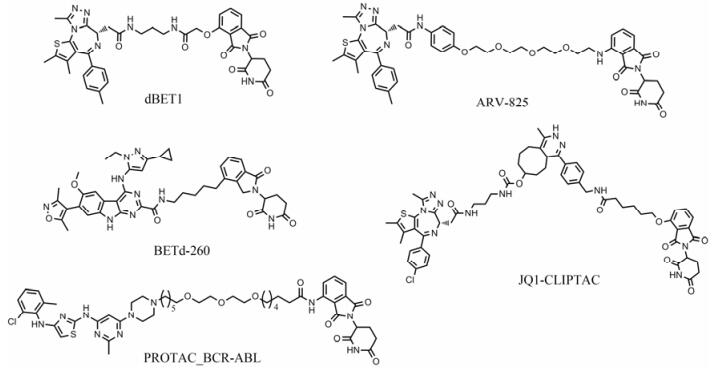
|
Figure 11 Reported small-molecule PROTACs that recruit the CRL4CRBN E3 ligase |
Crews等[43]利用同一策略设计合成了ARV-825, ARV-825与dBET1的区别仅仅在于连接基的不同。虽然ARV-825与BRD4、CRL4CRBN的Kd值分别是29 nmol·L-1与3 μmol·L-1, 但是其降解BRD4的DC50却小于1 nmol·L-1, 显示出亚化学计量的降解能力。10 nmol·L-1 ARV-825于6 h内几乎可以完全降解Burkitt淋巴瘤细胞中的BRD4蛋白, 抗肿瘤活性明显强于同等浓度的BRD4小分子抑制剂JQ1和OTX015。ARV-825还可以有效降解患者源性急性髓系白血病细胞中的BRD4, 下调c-Myc、CDK4/6和JAK2等致癌基因, 诱导细胞凋亡的能力明显强于OTX015, 且对卢佐替尼耐药的UKE1细胞保持较强抑制活性[44]。
2017年, 王少萌课题组[45]设计合成了由BDR4抑制剂HJB97与CRL4CRBN E3小分子配体泊马度胺组成的PROTAC分子BETd-260。BETd-260在低至30 pmol·L-1的浓度, 3 h内可以有效降解RS4;11人急性淋巴白血病细胞内的BRD4蛋白。0.1 nmol·L-1 BETd-260就可以显著下调RS4;11细胞内c-Myc基因表达, 是BRD4抑制剂HJB97的1 000多倍。BETd-260在纳摩尔浓度可以诱导RS4;11和MOLM-13白血病细胞发生凋亡, 抑制细胞的增殖, IC50分别是51 pmol·L-1和2.3 nmol·L-1。在RS4;11白血病细胞裸鼠移植瘤模型中, 5 mg·kg-1 BETd-260可以有效降解肿瘤组织内BDR4, 大于90%的小鼠体内的肿瘤发生消退。在三阴性人乳腺癌细胞裸鼠移植瘤模型中, BETd-260也可以显著地降解BDR4, 抑制肿瘤生长。
Heightman课题组[46]利用四嗪标记的沙利度胺与反式环辛烯标记JQ1之间的生物正交反应, 在细胞内原位生成PROTAC分子JQ1-CLIPTAC。3.0 μmol·L-1 JQ1-CLIPTAC在18 h内可以完全降解人宫颈癌HeLa细胞内的BRD4蛋白。
BCR-ABL是Bcr基因与Abl基因融合后产生的致癌酪氨酸激酶, 是导致慢性髓细胞白血病的主要原因。尽管目前已经有伯舒替尼、达沙替尼等多个小分子抑制剂上市, 但是部分患者会逐渐产生耐药性。2016年, Crews小组[47]报道了首个靶向酪氨酸激酶BCR-ABL的PROTAC分子PROTAC_BCR-ABL。1 μmol·L-1 PROTAC_BCR-ABL可以降解90%的c-ABL和BCR-ABL, 对BCR-ABL驱动的人髓性白血病K562细胞EC50达到4.4 nmol·L-1, 而对非BCR-ABL驱动的人乳腺癌HEK293T细胞和SK-BR-3细胞活性较差, 具有很好的选择性。
4 影响PROTAC降解活性的因素虽然PROTAC技术得到了越来越多药物化学家的关注, 但是目前报道的PROTACs分子大都是随机合成, 没有进行系统的构效关系研究。如何合理设计, 筛选得到降解活性最优的PROTAC分子将是本领域的一个研究重点。
PROTAC分子由E3配体、靶蛋白抑制剂和连接基三部分构成, 不同的E3配体、靶蛋白抑制剂和连接基的组合, 对PROTAC的降解活性有着十分关键的作用。Crews研究小组[47]将BCR-ABL抑制剂伊马替尼、伯舒替尼、达沙替尼与VHLE3配体、CRBN E3配体两两组合, 设计合成了6个类型的PROTACs, 活性评价结果显示, 不同组合产生的PROTAC分子之间, 降解活性有着很大的差别。PROTAC伯舒替尼-VHL对c-ABL和BCR-ABL没有降解活性, 而1 μmol·L-1 PROTAC达沙替尼-VHL在24 h内可以降解65%的c-ABL, 但是对BCR-ABL蛋白没有影响, 1 μmol·L-1 PROTAC达沙替尼-CRBN 24 h内则可以同时降解细胞内85%的c-ABL和60%的BCR-ABL。
连接基的结构类型、长度以及连接基与连接分子之间的结合位点对PROTAC分子的活性也有着重要影响, 合适的连接基及结合位点使其两端连接的E3与靶蛋白之间的相互作用更加有效。Kim研究小组[48]设计合成了连接基长度分别为9、12、16、19和21个碳原子的PROTACs, 降解活性评价结果显示, 当连接基长度为16个碳原子时, PROTAC降解AR的活性最高。ARV-825与dBET1的区别仅仅在于连接基的结构不同, 但是ARV-825降解BRD4的活性是dBET1的10多倍[43]。Kim等[49]设计合成了连接基分别与雌二醇的17位O原子、16位C原子及7α位C原子结合的PROTACs, 活性评价结果显示, 当连接基结合位点在7α位C原子时, PROTAC的蛋白降解活性最好。
5 结论与展望PROTACs利用泛素-蛋白酶体系统对靶蛋白进行翻译后降解, 与小分子抑制剂类抗肿瘤药物相比, PROTACs展现出了独特的优势: PROTACs不需要与目标靶蛋白长时间和高强度的结合, 可以降解转录因子等“不可药靶蛋白”发挥抗肿瘤作用; 致癌蛋白被降解后需要重新合成才能恢复功能, 因此降解致癌蛋白比抑制其活性显示出更加高效、持久的抗肿瘤作用; PROTACs降解靶蛋白过程类似于催化反应, 可循环结合、降解靶蛋白, 不需要等摩尔量的药物, 实现亚化学计量用药。
虽然PROTACs技术取得了巨大的进步, 但是仍面临着诸多的问题: PROTACs往往具有较大的分子质量, 导致部分PROTACs药代动力学性质不佳, 口服生物利用度较差; 可利用的E3泛素连接酶及其小分子配体种类有限; E3小分子配体的体内稳定性不够, 导致PROTACs分子半衰期较短, 限制了其亚化学计量用药特性; 目前报道的PROTACs大都没有进行系统的构效关系研究, 如连接基的长短、不同的E3泛素连接酶等对PROTACs活性的影响。
PROTACs技术已经吸引了药物化学家和国际医药公司的广泛关注, 随着越来越多的E3特异性小分子配体的发现和对PROTACs系统的构效关系研究, PROTACs目前面临的问题将会逐渐被解决, 成为小分子抑制剂、单克隆抗体之后另一重要的肿瘤治疗手段。
| [1] | Ma CX, Reinert T, Chmielewska I, et al. Mechanisms of aromatase inhibitor resistance[J]. Nat Rev Cancer, 2015, 15: 261–275. DOI:10.1038/nrc3920 |
| [2] | He QY. Tumor heterogeneity and drug resistance of targeted antitumor agents[J]. Acta Pharm Sin (药学学报), 2016, 51: 197–201. |
| [3] | Hatzivassiliou G, Song K, Yen I, et al. RAF inhibitors prime wild-type RAF to activate the MAPK pathway and enhance growth[J]. Nature, 2010, 464: 431–435. DOI:10.1038/nature08833 |
| [4] | Shimamura T, Chen Z, Soucheray M, et al. Efficacy of BET bromodomain inhibition in Kras-mutant non-small cell lung cancer[J]. Clin Cancer Res, 2013, 19: 6183–6192. DOI:10.1158/1078-0432.CCR-12-3904 |
| [5] | Russ AP, Lampel S. The druggable genome:an update[J]. Drug Discov Today, 2005, 10: 1607–1610. DOI:10.1016/S1359-6446(05)03666-4 |
| [6] | Adjei AA. What is the right dose? The elusive optimal biologic dose in phase Ⅰ clinical trials[J]. J Clin Oncol, 2006, 24: 4054–4055. DOI:10.1200/JCO.2006.07.4658 |
| [7] | Matzke M, Matzke AJ, Kooter JM. RNA:guiding gene silencing[J]. Science, 2001, 293: 1080–1083. DOI:10.1126/science.1063051 |
| [8] | Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas 9[J]. Science, 2014, 346: 1258096. DOI:10.1126/science.1258096 |
| [9] | Burnett JC, Rossi JJ. RNA-based therapeutics:current progress and future prospects[J]. Chem Biol, 2012, 19: 60–71. DOI:10.1016/j.chembiol.2011.12.008 |
| [10] | Bruce A, Sullenger SN. From the RNA world to the clinic[J]. Science, 2016, 352: 1417–1420. DOI:10.1126/science.aad8709 |
| [11] | Sakamoto KM, Kim KB, Kumagai A, et al. Protacs:chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation[J]. Proc Natl Acad Sci U S A, 2001, 98: 8554–8559. DOI:10.1073/pnas.141230798 |
| [12] | Gadd MS, Testa A, Lucas X, et al. Structural basis of PROTAC cooperative recognition for selective protein degradation[J]. Nat Chem Biol, 2017, 13: 514–521. DOI:10.1038/nchembio.2329 |
| [13] | Sakamoto KM, Kim KB, Verma R, et al. Development of PROTACS to target cancer-promoting proteins for ubiquitination and degradation[J]. Mol Cell Proteom, 2003, 2: 1350–1358. DOI:10.1074/mcp.T300009-MCP200 |
| [14] | Rodriguez-Gonzalez A, Cyrus K, Salcius M, et al. Targeting steroid hormone receptors for ubiquitination and degradation in breast and prostate cancer[J]. Oncogene, 2008, 27: 7201–7211. DOI:10.1038/onc.2008.320 |
| [15] | Hines J, Gough JD, Corson TW, et al. Posttranslational protein knockdown coupled to receptor tyrosine kinase activation with phosphoPROTACs[J]. Proc Natl Acad Sci U S A, 2013, 110: 8942–8947. DOI:10.1073/pnas.1217206110 |
| [16] | Matyskiela ME, Lu G, Ito T, et al. A novel cereblon modulator recruits GSPT1 to the CRL4(CRBN) ubiquitin ligase[J]. Nature, 2016, 535: 252–257. DOI:10.1038/nature18611 |
| [17] | Kronke J, Fink EC, Hollenbach PW, et al. Lenalidomide induces ubiquitination and degradation of CK1alpha in del (5q) MDS[J]. Nature, 2015, 523: 183–188. DOI:10.1038/nature14610 |
| [18] | Buckley DL, Van Molle I, Gareiss PC, et al. Targeting the von Hippel-Lindau E3 ubiquitin ligase using small molecules to disrupt the VHL/HIF-1alpha interaction[J]. J Am Chem Soc, 2012, 134: 4465–4468. DOI:10.1021/ja209924v |
| [19] | Sekine K, Takubo K, Kikuchi R, et al. Small molecules destabilize cIAP1 by activating auto-ubiquitylation[J]. J Biol Chem, 2008, 283: 8961–8968. DOI:10.1074/jbc.M709525200 |
| [20] | Crews CM, Georg G, Wang S. Inducing protein degradation as a therapeutic strategy[J]. J Med Chem, 2016, 59: 5129–5130. DOI:10.1021/acs.jmedchem.6b00735 |
| [21] | Crunkhorn S. Anticancer drugs:selectively targeting proteins for degradation[J]. Nat Rev Drug Discov, 2015, 14: 459. DOI:10.1038/nrd4670 |
| [22] | Deshaies RJ. Protein degradation:prime time for PROTACs[J]. Nat Chem Biol, 2015, 11: 634–635. DOI:10.1038/nchembio.1887 |
| [23] | Ohoka N, Shibata N, Hattori T, et al. Protein knockdown technology:application of ubiquitin ligase to cancer therapy[J]. Current Cancer Drug Targets, 2016, 16: 136–146. DOI:10.2174/1568009616666151112122502 |
| [24] | Runcie AC, Chan KH, Zengerle M, et al. Chemical genetics approaches for selective intervention in epigenetics[J]. Curr Opin Chem Biol, 2016, 33: 186–194. DOI:10.1016/j.cbpa.2016.06.031 |
| [25] | Chi KR. Drug developers delve into the cell's trash-disposal machinery[J]. Nat Rev Drug Discov, 2016, 15: 295–297. DOI:10.1038/nrd.2016.86 |
| [26] | Ottis P, Crews CM. Proteolysis-targeting chimeras:induced protein degradation as a therapeutic strategy[J]. ACS Chem Biol, 2017, 12: 892–898. DOI:10.1021/acschembio.6b01068 |
| [27] | Schneekloth AR, Pucheault M, Tae HS, et al. Targeted intracellular protein degradation induced by a small molecule:En route to chemical proteomics[J]. Bioorg Med Chem Lett, 2008, 18: 5904–5908. DOI:10.1016/j.bmcl.2008.07.114 |
| [28] | Sato S, Aoyama H, Miyachi H, et al. Demonstration of direct binding of cIAP1 degradation-promoting bestatin analogs to BIR3 domain:synthesis and application of fluorescent bestatin ester analogs[J]. Bioorg Med Chem Lett, 2008, 18: 3354–3358. DOI:10.1016/j.bmcl.2008.04.031 |
| [29] | Itoh Y, Ishikawa M, Naito M, et al. Protein knockdown using methyl bestatin-ligand hybrid molecules:design and synthesis of inducers of ubiquitination-mediated degradation of cellular retinoic acid-binding proteins[J]. J Am Chem Soc, 2010, 132: 5820–5826. DOI:10.1021/ja100691p |
| [30] | Okuhira K, Ohoka N, Sai K, et al. Specific degradation of CRABP-Ⅱ via cIAP1-mediated ubiquitylation induced by hybrid molecules that crosslink cIAP1 and the target protein[J]. FEBS Lett, 2011, 585: 1147–1152. DOI:10.1016/j.febslet.2011.03.019 |
| [31] | Okuhira K, Demizu Y, Hattori T, et al. Development of hybrid small molecules that induce degradation of estrogen receptor-alpha and necrotic cell death in breast cancer cells[J]. Cancer Sci, 2013, 104: 1492–1498. DOI:10.1111/cas.2013.104.issue-11 |
| [32] | Ohoka N, Nagai K, Hattori T, et al. Cancer cell death induced by novel small molecules degrading the TACC3 protein via the ubiquitin-proteasome pathway[J]. Cell Death Dis, 2014, 5: 5e1513. |
| [33] | Demizu Y, Shibata N, Hattori T, et al. Development of BCR-ABL degradation inducers via the conjugation of an imatinib derivative and a cIAP1 ligand[J]. Bioorg Med Chem Lett, 2016, 26: 4865–4869. DOI:10.1016/j.bmcl.2016.09.041 |
| [34] | Galdeano C, Gadd MS, Soares P, et al. Structure-guided design and optimization of small molecules targeting the protein-protein interaction between the von Hippel-Lindau (VHL) E3 ubiquitin ligase and the hypoxia inducible factor (HIF) alpha subunit with in vitro nanomolar affinities[J]. J Med Chem, 2014, 57: 8657–8663. DOI:10.1021/jm5011258 |
| [35] | Zengerle M, Chan KH, Ciulli A. Selective small molecule induced degradation of the bet bromodomain protein BRD4[J]. ACS Chem Biol, 2015, 10: 1770–1777. DOI:10.1021/acschembio.5b00216 |
| [36] | Raina K, Lu J, Qian YM, et al. PROTAC-induced BET protein degradation as a therapy for castration-resistant prostate cancer[J]. Proc Natl Acad Sci U S A, 2016, 113: 7124–7129. DOI:10.1073/pnas.1521738113 |
| [37] | Bondeson DP, Mares A, Smith IE, et al. Catalytic in vivo protein knockdown by small-molecule PROTACs[J]. Nat Chem Biol, 2015, 11: 611–617. DOI:10.1038/nchembio.1858 |
| [38] | Chamberlain PP, Lopez-Girona A, Miller K, et al. Structure of the human Cereblon-DDB1-lenalidomide complex reveals basis for responsiveness to thalidomide analogs[J]. Nat Struct Mol Biol, 2014, 21: 803–809. DOI:10.1038/nsmb.2874 |
| [39] | Fischer ES, Bohm K, Lydeard JR, et al. Structure of the DDB1-CRBN E3 ubiquitin ligase in complex with thalidomide[J]. Nature, 2014, 512: 49–53. |
| [40] | Martiniani R, Di Loreto V, Di Sano C, et al. Biological activity of lenalidomide and its underlying therapeutic effects in multiple myeloma[J]. Adv Hematol, 2012, 2012: 842945. |
| [41] | Anders L, Guenther MG, Qi J, et al. Genome-wide localization of small molecules[J]. Nat Biotechnol, 2014, 32: 92–96. |
| [42] | Winter GE, Buckley DL, PaulkJ, et al. Drug Development. Phthalimide conjugation as a strategy for in vivo target protein degradation[J]. Science, 2015, 348: 1376–1381. DOI:10.1126/science.aab1433 |
| [43] | Lu J, Qian Y, Altieri M, et al. Hijacking the E3 ubiquitin ligase cereblon to efficiently target BRD4[J]. Chem Biol, 2015, 22: 755–763. DOI:10.1016/j.chembiol.2015.05.009 |
| [44] | Saenz DT, Fiskus W, Qian Y, et al. Novel BET protein proteolysis-targeting chimera exerts superior lethal activity than bromodomain inhibitor (BETi) against post-myeloproliferative neoplasm secondary (s) AML cells[J]. Leukemia, 2017, 31: 1951–1961. DOI:10.1038/leu.2016.393 |
| [45] | Zhou B, Hu JT, Xu FM, et al. Discovery of a small-molecule degrader of bromodomain and extra-terminal (BET) proteins with picomolar cellular potencies and capable of achieving tumor regression[J]. J Med Chem, 2017. DOI:10.1021/acs.jmedchem.6b01816 |
| [46] | Lebraud H, Wright DJ, Johnson CN, et al. Protein degradation by in-cell self-assembly of proteolysis targeting chimeras[J]. ACS Cent Sci, 2016, 2: 927–934. DOI:10.1021/acscentsci.6b00280 |
| [47] | Lai AC, Toure M, Hellerschmied D, et al. Modular PROTAC design for the degradation of oncogenic BCR-ABL[J]. Angew Chem Int Ed Engl, 2016, 55: 807–810. DOI:10.1002/anie.201507634 |
| [48] | Cyrus K, Wehenkel M, Choi EY, et al. Impact of linker length on the activity of PROTACs[J]. Mol Biosyst, 2011, 7: 359–364. DOI:10.1039/C0MB00074D |
| [49] | Cyrus K, Wehenkel M, Choi EY, et al. Jostling for position:optimizing linker location in the design of estrogen receptor-targeting PROTACs[J]. ChemMedChem, 2010, 5: 979–985. DOI:10.1002/cmdc.v5:7 |
 2017, Vol. 52
2017, Vol. 52


