以ITS为核心序列的DNA条形码技术为药用植物的分子鉴定提供了便利。DNA条形码技术流程通常包括样品处理、样品DNA提取、PCR扩增、DNA测序、序列拼接、鉴定分析六个环节[1]。由于药用植物种类繁多, 不同组织器官中通常含有多糖、脂类、色素、酚类等次生代谢产物, 严重影响DNA提取质量和产量, 进而影响PCR扩增结果。因此, 获取高质量的样品DNA被认为是DNA条形码技术的必要前提和关键环节[2]。
Luo等[2]以含多糖较多的麦冬药材为对象, 对比了5种常用植物DNA提取方法, 筛选出试剂盒法和改良的CTAB法为佳, 其中改良的CTAB法相对最为经济有效, 以该方法提取的10种不同中药材DNA, ITS序列PCR扩增成功率为90%。无论是试剂盒法还是改良的CTAB法, 均需对材料进行破碎或研磨, 然后经过组织裂解、萃取、RNA去除、沉淀或吸附DNA等步骤, 单个样品DNA制备需要约1 h才能完成。由此可见, DNA提取是DNA条形码技术中工作量最多、人工耗时最长的环节, 特别是样品量较多时, 这一环节工作尤为繁重, 成为限制分子鉴定的主要因素。
直接PCR技术通过对微量组织样品的裂解物为DNA模板, 借助于灵敏且抗干扰能力强的TaqDNA聚合酶进行PCR扩增, 避免了传统DNA提取方法的大量繁琐工作[3]。本研究以ITS序列为目标产物, 探索直接PCR技术在药用植物分子鉴定中的应用, 以期提高工作效率, 为基于ITS序列的药用植物样本快速处理及分子鉴定提供了新的技术思路。
材料与方法 实验材料种植于上海中医药大学百草园中的苘麻(Abutilon theophrasti Medicus)、佩兰(Eupatorium fortunei. Turcz)、白花前胡(Peucedanum praeruptorum Dunn)、细叶远志(Polygala tenuifolia willd.)等80种植物。详见表 1。
| Table 1 Plant species and Direct-PCR amplification and Blast results. Note: A total of 80 plant species belong to 27 families and 71 genus |
Eppendorf移液器、排枪; Eppendorf vapo.protect PCR扩增仪; DYY-6C型双稳定时电泳仪(北京市六一仪器厂); Syngene凝胶成像系统(G: BOX, Syngene英国); 台式紫外透射仪(UVP, 美国); 5415R型高速冷冻离心机(Eppendorf, 德国); Votex-Genie涡旋振荡器(Coleparmer, 美国)。
试剂植物组织直接PCR试剂盒(PDP, 包括样品处理液Solution A、Solution B, Direct-PCRmix)、优化PCR mix、安全核酸染料、DNA marker购自上海创英生物科技有限公司; 琼脂糖购自Amersco公司; TAE电泳缓冲液自配, 所用Tris碱、冰醋酸、乙二胺四乙酸二钠盐(Na2EDTA)购自上海国药集团公司。
样品DNA制备剪取上述植物不同组织部位材料少许, 置于200μL PCR扩增用八连管中(图 1A), 参照植物组织直接PCR试剂盒说明, 每个PCR管中加入90 μL Solution A, 于PCR扩增仪中运行95 ℃ 15 min; 取出八连管, 每管加入10 μL Solution B, 混匀, 简短离心, 上清液即为样品DNA (图 1B), 直接用作模板进行PCR扩增。

|
Figure 1 Samples from different plant species. A: Samples before treatment; B: Samples after treatment. Number in the figure indicate the sample numbers, which are consistent with those in Table 1 |
ITS序列扩增选用两对引物: 18S-a (5'-CGTAACAAGGTTTCCGTAGGTGA AC-3')/26S-b (5'-TTATTGATATGCTTAAACTCAGCG GG-3')[4]; ITSF (5'-ATGCGATACTTGGTGTGAAT-3')/ ITS3R (5'-GACGCTTCTCCAGACTACAAT-3')[5]。18S-a/ 26S-b为本实验室常用引物, 扩增产物包含ITS1、5.8S和ITS2区域, 预期片段750 bp左右[4]; ITS2F/ITS3R为ITS2扩增通用引物, 预期片段500 bp左右[5]。20 μL PCR反应体系中含有Direct PCRmix 10 μL、正反向引物(10 μmol·L-1)各0.5 μL、样品DNA溶液2 μL, 无菌超纯水补至20 μL; 扩增程序为95 ℃ 2 min; 35 X [95 ℃ 30 s; 55 ℃ (18S-a/26S-b)或51 ℃ (ITSF/ITS3R) 30 s; 72 ℃ 1 min]; 72 ℃ 5 min。取5 μL PCR产物用于琼脂糖凝胶电泳检测, 条带单一且清晰的样品送上海生工生物工程有限公司测序。
直接PCR反应体系优化扩增首次PCR扩增没有清晰条带的样品, 一部分按原有体系进行再次扩增; 对另一部分颜色较深的样品DNA溶液用无菌水稀释50倍后为模板, 参照原有体系进行PCR扩增; 或者重新采集材料, 以ITS2F/ITS3R为引物, 以植物组织直接PCR试剂盒处理后样品DNA原液或稀释50倍后为模板, 再次进行扩增, 反应程序同上。对所有PCR产物进行琼脂糖凝胶电泳检测, 条带单一且清晰的样品送公司测序。
序列处理及分析对测得的序列, 结合测序源文件, 去除两侧读序不确定区域, 获得ITS序列进行Blast在线分析。
结果与分析 1 样品DNA获得苘麻(A. theophrasti)、佩兰(E. fortunei)、白花前胡(P. praeruptorum)、细叶远志(P. tenuifolia)等植物不同组织部位材料(图 1A), 经植物组织直接PCR试剂盒中Solution A/B处理后, 裂解液即样品DNA溶液呈现不同颜色(图 1B), 其中12、15、16、17、18、19、21、23、29、37、44、58、59、60、61、63、67、72、80号共19个材料的样品DNA溶液颜色较深, 尤以17、18、44、59、61、80号样品为甚, 呈现不透明的深褐色或红褐色。对比图 1A可以看出, 这些样品与其他材料在处理前颜色和取材大小差异并不明显。样品DNA溶液中颜色差异, 是由于不同植物材料裂解后释放出不同的代谢产物。
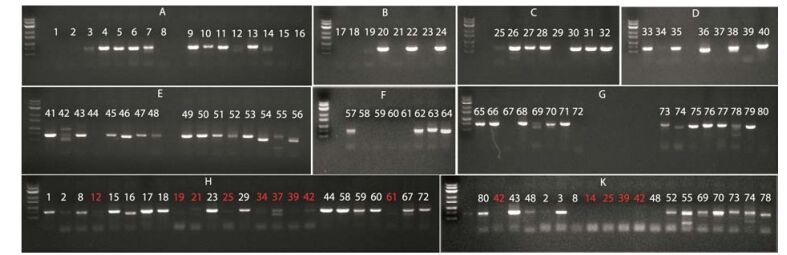
2 直接PCR首次扩增首次PCR扩增以18S-a/26S-b为引物, 琼脂糖凝胶电泳结果如图 2A~G所示。
|
Figure 2 Agarose gel electrophoresis results of Direct-PCR amplified ITS products. A-G: 1st PCR results; H, K: 2nd PCR results. Numbers in the figure indicate the sample numbers, which are consistent with those in Figure 1 and Table 1. Daily 1 kB DNA ladder (HerogenBio) was used to measure the size of PCR products |
样品DNA溶液颜色较深且不透明的19个样品中, 29、63号材料出现明显扩增, 其余17个材料首次扩增没有出现预期的单一条带; 样品DNA溶液颜色较深但澄清的8个样品中有6个样品(20、22、26、30、33、35、38号材料)的PCR产物条带单一且明晰, 两个(34、39号样品)材料没有扩增。由此推测, 样品DNA溶液中过多杂质会影响PCR扩增效果, 使样品DNA溶液颜色变深且浑浊的物质不利于PCR扩增。
3 直接PCR二次扩增对首次扩增无条带、条带弱或有杂带的15个样品, 采用同样体系再次进行PCR扩增, 其中8个样品PCR扩增情况明显改善, 出现清晰条带(图 2K, 2-78);样品DNA溶液颜色较深的24个样品(1、2、8、12、15、16、17、18、19、21、23、25、29、34、37、39、42、44、58、59、60、67、72、80号材料)稀释50倍后为模板进行扩增, PCR产物进行琼脂糖凝胶电泳, 结果如图 2H, K (2-80)所示: 24个样品中有14个样品出现预期ITS产物, 且条带单一、清晰, 表明通过稀释模板可以有效降低杂质对PCR扩增影响, 提高PCR扩增效率。
4 直接PCR反应体系优化扩增对二次扩增没有显著目标片段的10个样品(图 2H, K, 红色数字样品)重新采集材料(图 3A), 以Solution A/B处理后样品DNA溶液为模板, 以ITS2F/ ITS3R为引物进行PCR扩增, 结果如图 3C所示: 10个样品中, 7个扩增到预期大小的ITS片段, 其中6个扩增产物较多, 产物条带清晰明亮; 12号(丹参)、37号(夏枯草)、61号(龙芽草)无扩增产物。从样品DNA溶液图片(图 3B)可以看出, 12、37、61号样品DNA溶液颜色较深, 含有较多杂质。对这3个样品DNA溶液稀释50倍后为模板, 连同37号样品DNA原液, 用Direct-PCRmix及优化PCR mix中PR2、PR3、PR4 3种PCRmix进行扩增, 结果显示:以37号样品DNA原液为模板的4种PCR反应体系中没有得到目的基因产物; 而以样品DNA溶液稀释50倍后为模板的4种PCR反应体系, 12、37、61号样品ITS目的基因均得到高效扩增。以Direct-PCRmix和PR2为反应体系的扩增结果如图 3D, E所示。

|
Figure 3 Re-collected 10 samples Direct-PCR amplified ITS products. A: Samples before treatment; B: Samples after treatment; C-E: Agarose gel electrophoresis results with different PCR reactions. Direct-PCRmix with original DNA samples (C). Direct-PCRmix (D) and Sure-PCRmix-PR2 (E) with original DNA sample of No. 37 and 1/50 dilution DNA samples of No. 37, 61 and 12. Numbers in the figure indicate the sample numbers, which are consistent with those in Figure 1, 2 and Table 1. DM3000 DNA ladder (HerogenBio) was used to measure the size of PCR products |
值得注意的是, 这10个植物材料中有3个属于唇形科:丹参、夏枯草和益母草。由于同属物种在次生代谢产物上具有类似性, 以上结果进一步说明:通过稀释样品DNA可有效提高直接PCR扩增效率。
综合以上结果可以看出, 采用植物组织直接PCR技术, 以80种植物不同组织的微量样品为起始材料, 通过调整PCR反应体系, 80个样品ITS片段均得到有效扩增(表 1), PCR扩增成功率达100%。
5 测序结果分析以上所得的80个样品的PCR扩增产物送公司测序。对所得到序列在NCBI网站进行Blast在线分析, 80个样品中78个样品的ITS序列与GenBank中同物种或同属物种序列相同性在96%~100% (表 1), 可以准确地对物种进行鉴别。
GenBank数据库中没有收录单花莸[Caryopteris nepetaefolia (Benth.) Maxim.表 1样品39] ITS序列, Blast结果显示其与唇形科植物林石蚕(Teucrium scorodonia) ITS序列具有92%同源性, 为本研究新发现序列。水苏(Stachys japonica Miq)样品(表 1样品34)的ITS序列与一枝黄花(Solidago virgaurea)显示出98%同源性。由于实验所用水苏材料为地下根, 而一枝黄花为生命力极强的广布生长植物, 推测这一结果可能是由于取样时误取了一枝黄花的地下根部分造成的。
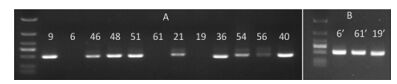
6 直接PCR技术可重复性验证为检测所建立方法的可重复性, 从上面80种植物材料中, 挑选了12个样品(包括首次扩增即成功的6、9、36、40、46和54号样品, 首次扩增条带较弱的48、51和56号样品, 以及经PCR反应体系优化后得到条带的19、21和61号样品), 重新取样, 再次进行样品处理、PCR扩增、电泳检测, 结果如图 4所示。可以看出, 前面实验中首次即成功的6个样品中5个样品再次实验也成功扩增; 条带较弱的3个样品再次实验中两个样品目的基因条带明显, 56号样品依然较弱; 前面实验需要进行PCR反应体系优化的3个样品中21号样品首次即出现扩增; 未出现扩增的3个样品(6、61和19号), 以其1/50稀释的样品DNA为模板再次扩增, 也获得ITS序列目的基因条带。以上结果说明, 本研究所建立的直接PCR方法具有可重复性。
|
Figure 4 Repeated Diret-PCR results of ITS PCR amplification. A: Agarose gel electrophoresis results of ITS PCR products using original DNA samples as template. B: PCR products using 1/50 dilution DNA samples as template. 6-61: Sample numbers as in Table 1. 6', 61' and 19': The same samples correspond to 6, 61 and 19, respectively, but using 1/50 dilution DNA samples as template. DM3000 DNA ladder was used to measure the size of PCR products |
PCR作为一项经典技术, 已广泛应用于生物材料的分子检测鉴定。在确保结果准确、稳定的前提下, 提高PCR方法效率, 缩短检测时间, 是新方法开发时需要考虑的重要指标[6]。直接PCR技术可以快速有效地同时处理大量样本, 在以基因组DNA为模板的PCR扩增中, 具有很大优势和应用潜力。近年来, 在临床样本快速鉴定检测[7]、转基因玉米筛选[6]等方面得到探索和应用。以药用植物为材料的直接PCR研究尚未见报道。
本研究中, 随机选择了80种常见药用植物, 通过调整PCR反应体系, 建立了批量样本处理及分子鉴定的直接PCR方法。与传统的需要提取制备DNA相比, 本研究所建立的方法具有如下特点:
根据所用植物组织直接PCR试剂盒说明, 植物材料以1~3 mg为宜。实际操作中, 可通过估测取样。本实验用植物材料均来自本单位百草园, 取材时, 直接剪取少许植物材料, 并没有严格称量。从PCR扩增结果来看, 样品量最少的13、53、54、62、63号样品, 均在首次PCR扩增中即得到显著的目的基因条带; 样品量中等的如30、31、32、65、66号样品, 也在首次扩增时得到清晰的目的片段; 样品量相对最多的23号样品, 首次扩增没有预期片段, 采用同样体系再次进行PCR扩增时, 也得到了预期的目的基因。这说明微量植物材料即可取得预期扩增结果, 因而对植物影响也就很小, 可用于不同发育阶段、不同组织器官药用植物的无损鉴定, 尤其是特殊发育时期如种子萌发、幼苗期等生物量较少时的生物材料。
采用传统的提取制备DNA方法, 首先要建立针对特定药材的DNA提取方法, 以去除多糖、酚酸等影响PCR扩增的化学成分, 特别是含有较多次生代谢产物的药用植物如丹参[8, 9]、夏枯草[10]、茴香[11]; 有时, 即使以提取纯化的DNA为模板, 也并非一次就能扩增成功, 需要对PCR反应体系进行优化, 以获得理想结果, 如夏枯草RAPD分子鉴定[10]、桔梗的ITS2序列扩增[12]等。
与之相比, 本研究可将多种药用植物材料同时进行处理, 所有样品DNA可在15 min内一次性获得。对于丹参、夏枯草、桔梗、羊蹄等含有较多杂质的24个样品DNA, 使用50倍稀释液为模板, 所有材料均得到有效扩增。实验结果说明, 通过简单的稀释, 即可降低杂质对PCR扩增的影响, 显著提高PCR扩增效率。从植物材料处理、PCR扩增、电泳检测, 到获得ITS序列的PCR扩增产物, 80个样品可以在8 h左右完成, 工作效率大大提高。
本研究所用80种植物材料分属27科71属, 包括酚酸类化合物含量较多的唇形科植物丹参、紫苏、夏枯草等; 取材组织部位包括叶51份、茎22份、根6份、花1份, 采用本研究所建立的直接PCR方法, 其ITS序列都得到了有效扩增。
值得一提的是, 本研究还随机选择了地肤子、白芍、川菊、百两金等7种干药材进行了针对ITS序列的直接PCR扩增, 结果表明3种药材在首次PCR扩增中即有目的条带。可以预见, 直接PCR技术应用于干药材的分子鉴定也将是可行的。
综上所述, 直接PCR技术以微量样品为起始材料, 高效扩增药用植物ITS基因片段, 节省了时间和人力, 可以显著提高工作效率。当有大量样品需要处理时, 这一优势更为明显, 为基于ITS序列的药用植物分子鉴定提供了更为便捷的技术思路。
| [1] | Chen S, Yao H, Han J, et al. Principles for molecular identification of traditional Chinese materia medica using DNA barcoding[J]. China J Chin Mater Med (中国中药杂志), 2013, 38: 141–148. |
| [2] | Luo K, Ma P, Yao H, et al. Study on DNA extraction method for Chinese herbs[J]. World Sci Technol/Mod Tradit Chin Med Mater Med (世界科学技术——中医药现代化), 2012, 14: 1432–1439. |
| [3] | Hwang H, Bae SC, Lee S, et al. A rapid and simple genotyping method for various plants by Direct-PCR[J]. Plant Breed Biotechnol, 2013, 1: 290–297. DOI:10.9787/PBB. |
| [4] | Ye Z, Lu Y, Wang Z, et al. Identification of Dendrobii Caulis basing on ITS sequence[J]. China J Chin Mater Med (中国中药杂志), 2014, 39: 3928–3935. |
| [5] | Xiang L, Zhang W, Chen S. Literature study and DNA barcoding of traditional Chinese medicine Qinghao (Artermisia annua L.)[J]. Acta Pharm Sin (药学学报), 2016, 51: 486–495. |
| [6] | Xing Z, Dong L, Liu N, et al. Rapid screening of genetically modified maize be Direct PCR[J]. J Maize Sci (玉米科学), 2017, 1: 29–33. |
| [7] | Liu R, Yin L, Pu Y, et al. MTHFR C677T polymorphism rapidly detected with direct amplification protocol in peripheral blood samples[J]. J Environ Occup Med (环境与职业医学), 2007, 24: 21–24. |
| [8] | Wang C, Liang Z, Li D, et al. Optimization of DNA extrac-tion methods from Salvia miltiiorrhiza Bge[J]. Pharm Biotechnol (药物生物技术), 2007, 14: 29–33. |
| [9] | Song Z, Wang M, Wang J, et al. Comparison of genomic DNA extraction methods for Salvia miltiorrhiza[J]. Chin Wild Plant Resour (中国野生植物资源), 2008, 27: 54–57. |
| [10] | Lai J, Li P, Yuan Y, et al. Extraction of genomic DNA and optimization of RAPD reaction condition for Prunella vulgaris L[J]. Nat Prod Res Dev (天然产物研究与开发), 2007, 19: 123–126, 134. |
| [11] | Li H. Comparison of genomic DNA extraction methods for Foeniculum vulgare Mill[J]. Northern Horticulture (北方园艺), 2008, 3: 189–192. |
| [12] | Liu J, Pang M, Zhang G, et al. Identification of Platycodonis Radix seeds by using ITS2 DNA barcode[J]. World Sci Technol/Mod Tradit Chin Med Mater Med (世界科学技术——中医药现代化), 2016, 18: 173–178. |
 2017, Vol. 52
2017, Vol. 52


