艾滋病是严重威胁人类生命健康的传染病。截至2016年底, 全球有约3 670万HIV携带者, 其中2016年新增感染人数约180万, 死于艾滋病的人数为100万[1]。随着艾滋病在世界范围的泛滥, 目前我国HIV/AIDS流行已经进入快速增长期, 感染人数已超过70万。因此, 对HIV/AIDS的预防已成为我国不容忽视的社会问题, 研发有效的抗艾滋病药物是今后我国医药发展的一项重大战略。目前, 上市的药物面临如下几方面的挑战: ①由于长期的临床应用催生了对现有药物不敏感的耐药突变株; ②服用药物带来的肝肾毒性等不良反应对患者造成巨大痛苦; ③联合用药给患者带来诸多不便, 影响依从性[2]。HIV-1的核壳体蛋白7 (nucleocapsid protein 7, NCp7)在病毒的逆转录过程与整合过程都起到重要作用。1998年, Huang等[3]对NCp7核苷酸序列中的单个氨基酸残基或几个氨基酸残基进行突变实验, 研究表明NCp7具有高度的保守性。因此, 针对此靶点的抑制剂不易因NCp7发生基因突变而产生耐药性, 成为近年来备受关注的抗病毒药物研究的新作用靶点之一[4]。近10年来, 鲜有对NCp7抑制剂的综述报道, 本文主要介绍HIV-1 NCp7的结构与功能, 以及近10年来针对此靶点的抑制剂研究新进展。
1 HIV-1核壳体蛋白NCp7锌指受体的结构与功能成熟的HIV-1病毒中大约有2 000个核壳体蛋白(nucleocapsid protein, NCp), 分子质量约为7 kDa。它们与基因组的70S RNA、整合酶、逆转录酶(RT)以及蛋白酶分子密切相关[5]。NCp15蛋白在成熟病毒中最终分化成NCp7、基质、衣壳和p6蛋白等。
1994年, Morellet等[6]通过核磁共振谱确证NCp7由72个氨基酸残基组成, 具有2个锌指结构。其中心结构域由2个CCHC (Cys-X2-Cys-X4-His-X4-Cys)类锌指结构组成, 且通过基本序列29RAPRKKG35连接[7] (图 1上)。1998年, Morellet等[8]报道了通过核磁共振谱确证的(12-53) NCp7结构(图 1下)。
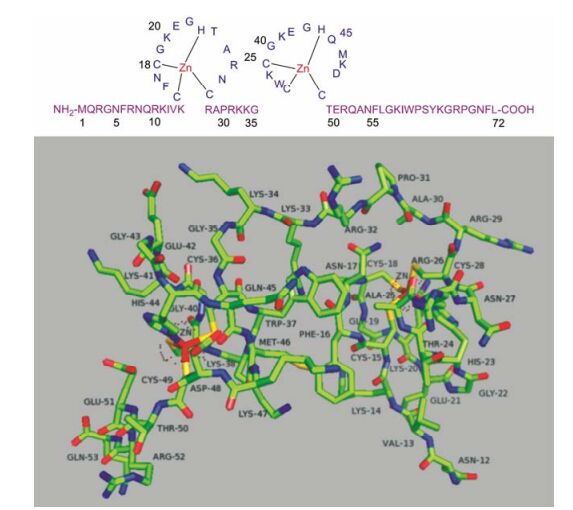
|
Figure 1 The amino acid sequence and the crystal structure of HIV-1 NCp7. PDB: 1BJ6; the cofactor zinc was shown in red |
NCp7在HIV逆转录和整合过程中都具有重要作用。在HIV逆转录起始复合物的形成阶段需要逆转录酶(RT) p66-p51与启动子tRNAlys3的高亲和, 以及RT与NCp7的相互作用; 复制过程中NCp7可能加速RT-RNase H介导的病毒RNA降解, 并降低RNA断口处的非特异性复制发生频率, 减少复制错误的发生[9, 10]。RT-RNase H降解RNA序列时, 允许NCp7作为分子伴侣来伴随两条互补PBS DNA的复性[11-13]。在前病毒DNA合成的完成阶段, NCp7和整合酶(integrase, IN)一起连接到前病毒DNA, 两者共同确保病毒DNA保持构象并且整合到宿主基因组中[14]。研究发现, 在体外的DNA模型中NCp7能够显著增强IN催化的整合过程, 但是其体内的作用还需进一步证实[15]。
2 HIV-1核壳体蛋白NCp7抑制剂的研究基于NCp7的结构特征及其在HIV-1生命周期中的作用机制, 目前已经设计合成出大量针对此靶点的化合物, 显示出很高的体外抗病毒活性。HIV-1核壳体蛋白NCp7抑制剂机制主要分两种: ①通过抑制剂与NCp7作用“逐出”锌指结构里的锌离子, 从而改变NCp7的结构, 并导致其丧失原有的功能, 最终抑制HIV-1的复制; ②通过直接干扰核酸与核壳体蛋白结合抑制HIV-1的复制。
2.1 基于锌离子“逐出”机制的抑制剂 2.1.1 苯甲酰胺硫酯类化合物化合物1 (S-acyl-2-mercaptobenzamide thioester, SAMT, 图 2)只与锌指的C端结合, 具有较高的选择性。抑制剂首先通过亲核作用乙酰化Cys39, 然后通过分子内酰基转移到Lys33和Lys38, 最终使锌指结构发生改变并逐出锌离子[16]。另外, SAMT通过分子内酰基转移后得到化合物2 (2-mercaptobenzamide thioester, MT), 再次通过细胞内乙酰辅酶A乙酰化得到SAMT; 同时, 化合物3a和3b (MT前药)通过酯酶水解、自发消除得到MT。活性结果表明:化合物2在CEM-SS细胞系中的抗HIV活性EC50为2.3 μmol·L-1, 细胞毒性TC50为741 μmol·L-1; 化合物1在CEM-SS细胞系中的抗HIV活性EC50为0.6 μmol·L-1, 细胞毒性TC50大于100 μmol·L-1, 其活性大约提高了4倍; 化合物3a在CEM-SS细胞系中抗HIV活性EC50为0.2 μmol·L-1, 其抗病毒活性大约提高了9倍, 且细胞毒性大于100 μmol·L-1。2016年, Hartman等[17, 18]设计合成的MT前药3b也表现了较好的靶向NCp7活性, 结果表明: 3b抗HIV活性优于原药2, 其中化合物2在单核巨噬细胞和PBMC细胞系中的抗HIV活性EC50分别为2.5~19 μmol·L-1和1.44~10.7 μmol·L-1; 化合物3b在单核巨噬细胞和PBMC细胞系中的抗HIV活性EC50分别为0.6~20 μmol·L-1和0.87~12.3 μmol·L-1。
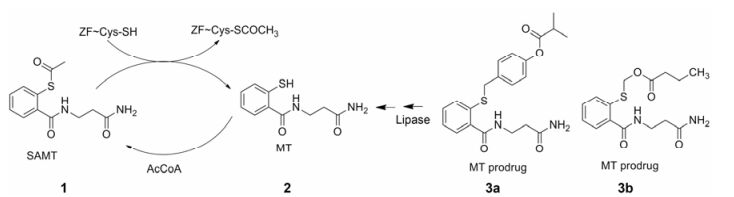
|
Figure 2 The structures of compounds1, 2, 3a and 3b |
化合物4 ([SP-4-2]-[PtCl (9-EtGua)(NH3)(quin)]+, 图 3)是铂配合物类化合物, 通过铂酸盐碱基与色氨酸以非共价键π-π堆积作用分子识别, 然后通过铂与蛋白复合物靶向结合将锌离子从核壳体蛋白锌指的碳端结构域逐出。其体外抑制HIV复制的EC50值为41.9 μmol·L-1, CC50>200 μmol·L-1[19]。
2.1.3 噻二唑类化合物硫酯类化合物通过与核壳体蛋白共价键结合起作用, 而噻二唑类化合物通过与锌离子的螯合作用使锌离子逐出。其中, 化合物5 (NV038, 图 3)是广谱的抗病毒抑制剂, 对HIV-1 (EC50 = 17 ± 3μmol·L-1)、HIV-2 (EC50 = 26 ± 7 μmol·L-1)和猴免疫缺陷病毒(SIV) (EC50 = 17 ± 4 μmol·L-1)都有一定的抑制作用[20], 且其细胞毒性较小(CC50>297.5 μmol·L-1), 可作为先导化合物进一步优化。另外, 化合物6 (WDO-217, 图 3)也是一类较好的广谱抗病毒抑制剂(HIV-1 (ⅢB) (EC50: 5 ± 3 μmol·L-1)、HIV-2 (ROD) (EC50: 2.3 ± 0.3 μmol·L-1)和SIV (Mac251) (EC50: 5 ± 1 μmol·L-1), 细胞毒性CC50 = 72 μmol·L-1)[21]。研究结果表明, 它们都可以逐出两个锌指结构中的锌离子, 且不受NCp7与寡核苷酸结合的影响[17]。
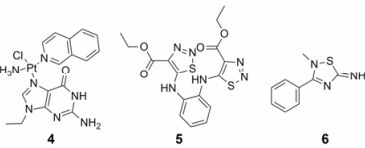
|
Figure 3 The structures of compounds 4-6 |
近年来, 除了基于锌离子“逐出”机制的抑制剂, 一些以非共价方式与核酸结合的NCp7抑制剂也相继被报道。该类抑制剂具有高选择性和低毒性的特点, 可作为理想的先导化合物进行药物开发。目前, 基于核酸结合的抑制剂主要分为两种机制: ①与核酸以非共价键竞争性结合核壳体蛋白; ②与核壳体蛋白的核酸伴侣非共价键结合。
2.2.1 竞争性结合核壳体蛋白抑制剂化合物7 ~11 (图 4)从4 800小分子中通过高通量筛选得到, 并经质谱和荧光各向异性分析确证, 其抑制常数Ki = 8.5~15 μmol·L-1, 遗憾的是, 没有测定该类化合物细胞水平的抗病毒活性。基于分子的对接模型研究表明:这些化合物在疏水性口袋中通过π-π作用与核酸重要结合残基Trp37作用[17, 22]。
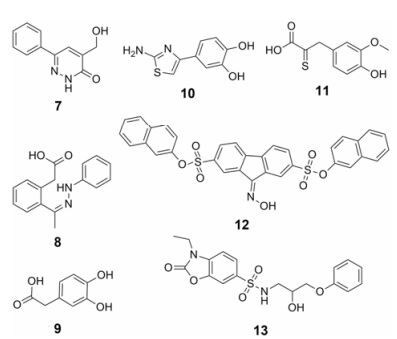
|
Figure 4 The structures of compounds 7-13 |
2012年, Mori等[23, 24]优化虚拟筛选模型, 并对Asinex数据库中的390 000个化合物进行了虚拟筛选, 然后通过质谱分析和细胞活性测定, 得到了2个新的NCp7抑制剂12和13 (图 4)。接着对NC(11-55)进行不同位点氨基酸荧光标记实验, 结果表明化合物12对NCp7的结合作用更强, 其结合力常数为5.6 ± 0.9 μmol·L-1; 进一步的活性测定结果显示化合物12抑制HIV-1复制的EC50约为2 μmol·L-1。
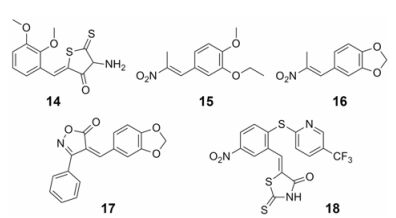
2012年, Breuer等[24, 25]利用荧光偏振分析法从含有14 400个小分子的Maybridge化合物库中筛选出了101个小分子, 这些化合物能够取代由羧基荧光素标记并与壳体蛋白作用的SL2 DNA (pSL-2)。对其进行NC-pSL-2筛选后得到36个化合物, 再次经过不同的荧光扫描实验和β-半乳糖苷酶等酶活评价得到5个与NCp7亲和力强的小分子化合物14 ~18 (图 5), 其亲和力常数Ki分别为: 18 ± 14、14 ± 7、22 ± 9、20 ± 5和73 ± 50 nmol·L-1。进一步活性研究结果表明, 化合物14和15具有较小的细胞毒性(1 μmol·L-1时, 对正常细胞的抑制率小于3%), 且在CD4 T细胞水平的抗HIV-1活性筛选中, 表现出较好的抗HIV-1 DNA复制活性, 其EC50分别为3和0.32 μmol·L-1; 然而, 化合物16 ~18在1 μmol·L-1时, 对正常细胞的抑制率大于70%, 具有一定的细胞毒性。
|
Figure 5 The structures of compounds 14-18 |
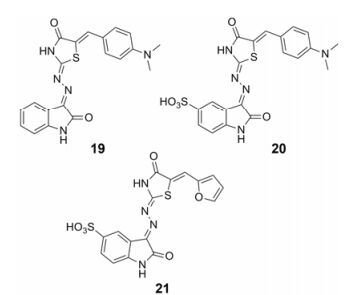
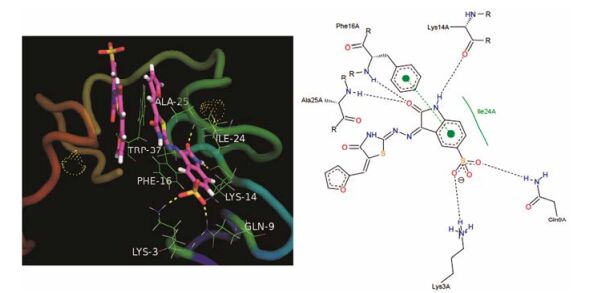
2013年, Goudreau等[26-28]通过高通量筛选得到壳体蛋白装配抑制剂19 (图 6), 其IC50为1.7 μmol·L-1, 抑制HIV复制的EC50为2.6 μmol·L-1, CC50为16.2 μmol·L-1, 进一步研究机制表明: 19抑制NCp7与DNA的相互作用, 与核酸竞争性结合NCp7, 其IC50为0.59 μmol·L-1。通过合理药物设计得到化合物20和21, NCp7-寡核苷酸亲和活性测定结果表明其活性相当, IC50分别为0.11和0.95 μmol·L-1, 且水溶性也有所提高, 但其抗病毒活性明显下降。光谱分析和核磁共振研究表明:两分子抑制剂连接到一个核壳体蛋白NC (1-55)的两个锌指活性位点, 形成三元复合物。从共晶结合模式图中可以看到, 化合物21与Ala25、Lys24、Lys3和Gln9形成较强的氢键作用, 同时与Phe16、Trp37和Ile24形成一定的疏水作用(图 7)。
|
Figure 6 The structures of compounds 19-21 |
|
Figure 7 The binding mode of compound 21 in HIV-1 NCp7 (PDB code: 2M3Z) |
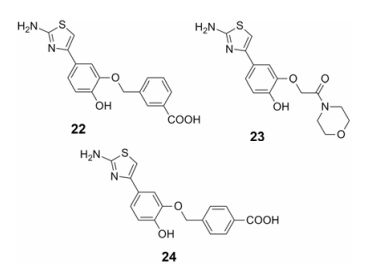
化合物10属于邻苯二酚类化合物, 对氧敏感, 容易被氧化成醌或者半醌自由基而影响与NCp7或者核酸的作用, Mori等[29]试图用其他官能团取代苯二酚, 同时提高其脂溶性。首先从ZINC化合物库中筛选出950个苯环上含有取代基的2-氨基-4-苯噻唑结构化合物, 然后对其进行虚拟筛选, 对接打分, 最后选择化合物22 ~24 (图 8)进行NC-cTAR亲和实验。结果显示:在100 μmol·L-1浓度下, 化合物22 ~24对诱导Rh6G释放的抑制率分别为7%、10%和28%。其中, 化合物24的活性最高, IC50为140 ± 90 μmol·L-1, Ki为50 ± 30 μmol·L-1。对活性最好的化合物24再次进一步研究, 通过等温滴定量热法(isothermal titration calorimetry, ITC)实验确证化合物24是竞争性抑制NCp7与核酸的结合, 与核壳体蛋白NC (11-55)的亲和常数Kd值大约为100 μmol·L-1, 抑制病毒复制的EC50为95 μmol·L-1。
|
Figure 8 The structures of compounds22-24 |
SL3 (stem-loop 3 RNA)是一个比较保守的基因序列, 存在于不同的病毒中, 对病毒的包裹具有重要作用。Warui等[30]于2009年通过基于对接的虚拟筛选得到了一些具有较高亲和力的小分子化合物, 这些化合物能够与SL3RNA作用, 并通过影响NCp7与ψ-RNA的作用, 进而抑制病毒的复制。其中, 化合物25和26 (图 9)的选择性最高, 与SL3RNA的解离常数KD值分别为11和98 μmol·L-1。2012年, Warui等[31]再次通过高通量筛选, 得到16个与SL3RNA亲和活性在微摩尔水平的化合物。但只有4个化合物27 ~30 (图 9)表现出较弱的抑制SL3-NCp7相互作用的活性, 其IC50值分别为110、20、200和1 140 μmol·L-1。然而, 这些化合物尚未进行抗病毒活性筛选。

|
Figure 9 The structures of compounds25-30 |
小分子化合物与TAR和cTAR的结合会影响NCp7功能, 进而干扰HIV病毒的复制。蛋白复合物晶体研究发现, 化合物31 (图 10)中的赖氨酸氨基与TAR RNA链上氨基酸的骨架磷酸酯离子键的相互作用至关重要[32], Sosic等[33, 34]推测化合物31可能与TAR RNA和cTAR结合, 而增加赖氨酸与芳香环的距离会减弱与核酸的非经典结构作用, 因此选择了不同长度的阳离子设计了一系列化合物进行活性研究。实验结果表明改变阳离子的距离可以提高与核酸的作用和对NCp7的抑制作用, 其中N端鸟氨酸化合物32 (图 10)对核壳体蛋白的作用最强, 其IC50为21.9 ± 0.46 μmol·L-1。核壳体退火电泳实验表明, 增加链长, 连接其他的氨基酸同样可以提高对核壳体蛋白的作用, 其中化合物33 (图 10)活性较好, 对全长核壳体蛋白以及NC (11-55)蛋白的IC50分别为20.3 ± 0.13和13.2 ± 0.35 μmol·L-1[35, 36]。

|
Figure 10 The structures of compounds 31-33 |
Kim等[37]基于抑制核壳体蛋白与ψ-RNA相互作用的细胞活性筛选方法发现化合物34 (图 11)与HIV核壳体蛋白的作用较强, 抑制HIV病毒复制的EC50大约为1 μmol·L-1。实验结果表明:化合物34对cTAR的稳定有一定的破坏作用, 进而抑制HIV病毒的复制。
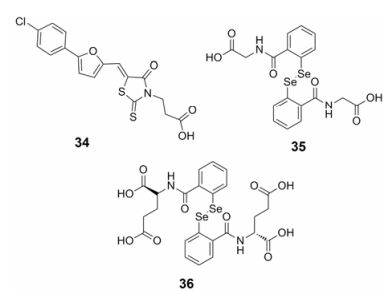
|
Figure 11 The structures of compounds 34-36 |
2015年, Sancineto等[38]对核壳体蛋白抑制剂二硫代苯甲酰胺类化合物进行修饰, 用硒原子代替先导化合物中的硫原子, 设计了一类2, 2'-二硒双苯酰胺(diselenobisbenzamides, DISeBAs)化合物。这类化合物抑制前体Gap的加工, 具有广谱的抗病毒作用, 对于一些HIV耐药株[HIV-1RES056、HIV-1ⅢB/RIT、HIV-1L6、HIV-1B12、HIV-1ADP/141、HIV-1 (ⅢB)、HIV-2 (ROD)]和猴免疫缺陷病毒(SIVMac251)都有一定的抑制作用, 其中化合物35和36 (图 11)在急慢性感染的细胞中抑制HIV病毒的选择性最高。其在MT-4细胞系对HIV-1、HIV-2的抑制活性和细胞毒性见表 1。
| Table 1 Anti-HIV-1/-2 activity and cytotoxicity of DISeBAs in MT-4 cells |
HIV-1NCp7中的锌指结构为研制抗艾滋病药物提供了新的靶点。近年来针对该靶点的小分子抑制剂已经取得了显著进展, 一部分化合物已经进入临床试验。如二氮二羰基酰胺类(azadicarbonamide, ADA)、吡啶烷酰基硫醚类化合物(pyridinioalkanoylthiolesters, PATEs)等, 开拓了抗HIV-1病毒药物研究的新领域。且化合物的构效关系研究提供了进一步结构优化的重要依据。由于NCp7蛋白的高度保守性, 此类抑制剂引起耐药性的可能性较低。因此, 对于已经对逆转录酶抑制剂和蛋白酶抑制剂产生耐药性的患者, 将会有很好的疗效。针对锌指受体的抑制剂能够抑制病毒的再感染, 这样通过联合用药将会达到最大的临床治疗效价, 有望彻底杀死体内的HIV病毒, 使抗AIDS的临床治疗有新的突破。
在基于锌离子逐出机制的抑制剂研究中, 可通过晶体学研究发现锌离子结合到NCp7活性位点的氨基酸残基, 通过计算机辅助药物设计进行基于结构的合理药物设计; 另一方面, 基于蛋白-蛋白、蛋白-核酸相互作用的抑制剂的研究中, 可通过基于靶标的高通量筛选从小分子化合物库发现苗头化合物, 再通过基于结构及性质的优化来完善该分子的活性及理化性质, 同时还可以通过X-射线晶体学、核磁共振以及同源建模等方法来获取或预测蛋白-配体复合物的结合模式, 以有效地指导新一轮的结构优化, 经几轮“设计-合成-生物活性”的循环研究, 最终获得药物先导化合物乃至候选药物[39]。总之, 基于NCp7的靶标结构生物学信息, 并综合运用药物化学新策略进行合理药物设计, 将是该领域的重要研究方向。
| [1] | World Health Organization (WHO) Data of HIV/AIDS (Up-dated June 2017)[EB/OL]. http://wwwwhoint/mediacentre/factsheets/fs360/en/. |
| [2] | Liu H, Zhan P, Liu XY. Research progress of dual in-hibitors targeting HIV-1 reverse transcriptase and integrase[J]. Acta Pharm Sin (药学学报), 2013, 48: 466–476. |
| [3] | Huang M, Maynard A, Turpin JA, et al. Anti-HIV agents that selectively target retroviral nucleocapsid protein zinc fingers without affecting cellular zinc finger proteins[J]. J Med Chem, 1998, 41: 1371–1381. DOI:10.1021/jm9708543 |
| [4] | Turpin JA. The next generation of HIV/AIDS drugs:novel and developmental anti-HIV drugs and targets[J]. Expert Rev Anti Infect Ther, 2003, 1: 97–128. DOI:10.1586/14787210.1.1.97 |
| [5] | Kankia BI, Barany G, Musier-Forsyth K. Unfolding of DNA quadruplexes induced by HIV-1 nucleocapsid protein[J]. Nucleic Acids Res, 2005, 33: 4395–4403. DOI:10.1093/nar/gki741 |
| [6] | Morellet N, Rocquigny HD, Mély Y, et al. Conformational behaviour of the active and inactive forms of the nucleocapsid NCp7 of HIV-1 studied by 1H NMR[J]. J Mol Biol, 1994, 235: 287–301. DOI:10.1016/S0022-2836(05)80033-6 |
| [7] | Mark-Danieli M, Laham N, Kenan-Eichler M, et al. Single point mutations in the zinc finger motifs of the human immunodeficiency virus type 1 nucleocapsid alter RNA binding specificities of the gag protein and enhance packaging and infectivity[J]. J Virol, 2005, 79: 7756–7767. DOI:10.1128/JVI.79.12.7756-7767.2005 |
| [8] | Morellet N, Déméné H, Teilleux V, et al. Structure of the complex between the HIV-1 nucleocapsid protein NCp7 and the single-stranded pentanucleotide d(ACGCC)[J]. J Mol Biol, 1998, 283: 419–434. DOI:10.1006/jmbi.1998.2098 |
| [9] | Belfetmi A, Zargarian L, Tisne C, et al. Insights into the mechanisms of RNA secondary structure destabilization by the HIV-1 nucleocapsid protein[J]. RNA, 2016, 22: 506–517. DOI:10.1261/rna.054445.115 |
| [10] | Hong MK, Harbron EJ, O'Connor DB, et al. Nucleic acid conformational changes essential for HIV-1 nucleocapsid protein-mediated inhibition of self-priming in minus-strand transfer[J]. J Mol Biol, 2003, 325: 1–10. DOI:10.1016/S0022-2836(02)01177-4 |
| [11] | Chang Y, Liu XY. Progress ofinhibitors targeting HIV-1 NCp7[J]. Chin Pharm J (中国药学杂志), 2007, 42: 9–12. |
| [12] | Golinelli MP, Hughes SH. Self-priming of retroviral mi-nus-strand strong-stop DNAs[J]. Virology, 2001, 285: 278–290. DOI:10.1006/viro.2001.0970 |
| [13] | Rajendran A, Endo M, Hidaka K, et al. HIV-1 nucleo-capsid proteins as molecular chaperones for tetramolecular antiparallel G-quadruplex formation[J]. J Am Chem Soc, 2013, 135: 18575–18585. DOI:10.1021/ja409085j |
| [14] | Poljak L, Batson SM, Ficheux D, et al. Analysis of NCp7-dependent activation of HIV-1 cDNA integration and its conservation among retroviral nucleocapsid proteins[J]. J Mol Biol, 2003, 329: 411–421. DOI:10.1016/S0022-2836(03)00472-8 |
| [15] | Buckman JS, Bosche WJ, Gorelick RJ. Human immunodeficiency virus type 1 nucleocapsid Zn2+ fingers are required for efficient reverse transcription, initial inte-gration processes, and protection of newly synthesized viral DNA[J]. J Virol, 2003, 77: 1469–1480. DOI:10.1128/JVI.77.2.1469-1480.2003 |
| [16] | Jenkins LMM, Ott DE, Hayashi R, et al. Small-molecule inactivation of HIV-1 NCp7 by repetitive intracellular acyl transfer[J]. Nat Chem Biol, 2010, 6: 887–889. DOI:10.1038/nchembio.456 |
| [17] | Hartman TL, Yang L, Helfrick AN, et al. Preclinical evaluation of a mercaptobenzamide and its prodrug for NCp7-targeted inhibition of human immunodeficiency virus[J]. Antiviral Res, 2016, 134: 216–225. DOI:10.1016/j.antiviral.2016.08.022 |
| [18] | Garg D, Torbett BE. Advances in targeting nucleocapsid-nucleic acid interactions in HIV-1 therapy[J]. Virus Res, 2014, 193: 135–143. DOI:10.1016/j.virusres.2014.07.004 |
| [19] | Quintal SM, Depaula QA, Farrell NP. Zinc finger proteins as templates for metal ion exchange and ligand reactivity. Chemical and biological consequences[J]. Metallomics Integr Biometal Sci, 2011, 3: 121–139. DOI:10.1039/c0mt00070a |
| [20] | Pannecouque C, Szafarowicz B, Volkova N, et al. In-hibition of HIV-1 replication by a bis-thiadiazolbenzene-1, 2-diamine that chelates zinc ions from retroviral nucleocapsid zinc fingers[J]. Antimicrob Agents Chemother, 2010, 54: 1461–1468. DOI:10.1128/AAC.01671-09 |
| [21] | Vercruysse T, Basta B, Dehaen W, et al. A phenyl-thiadia-zolylidene-amine derivative ejects zinc from retroviral nucleocapsid zinc fingers and inactivates HIV virions[J]. Retrovirology, 2012, 9: 2264–2265. |
| [22] | Shvadchak V, Sanglier S, Rocle S, et al. Identification by high throughput screening of small compounds inhibiting the nucleic acid destabilization activity of the HIV-1 nucleocapsid protein[J]. Biochimie, 2009, 91: 916–923. DOI:10.1016/j.biochi.2009.04.014 |
| [23] | Mori M, Schult-Dietrich P, Szafarowicz B, et al. Use of virtual screening for discovering antiretroviral compounds interacting with the HIV-1 nucleocapsid protein[J]. Virus Res, 2012, 169: 377–387. DOI:10.1016/j.virusres.2012.05.011 |
| [24] | Breuer S, Chang MW, Yuan J, et al. Identification of HIV-1 inhibitors targeting the nucleocapsid protein[J]. J Med Chem, 2012, 55: 4968–4977. DOI:10.1021/jm201442t |
| [25] | Torbett BE, Goodsell DS, Richman DD. The Future of HIV-1 Therapeutics[M]. Springer International Publishing, 2015. |
| [26] | Goudreau N, Hucke O, Faucher AM, et al. Discovery and structural characterization of a new inhibitor series of HIV-1 nucleocapsid function:NMR solution structure determination of a ternary complex involving a 2:1 inhibitor/NC stoichiometry[J]. J Mol Biol, 2013, 425: 1982–1998. DOI:10.1016/j.jmb.2013.02.022 |
| [27] | Stierand K, Maass PC, Rarey M. Molecular complexes at a glance:automated generation of two-dimensional complex diagrams[J]. Bioinformatics, 2006, 22: 1710–1716. DOI:10.1093/bioinformatics/btl150 |
| [28] | http://poseview.zbh.uni-hamburg.de/pdb_files/search?name=2m3ze790bd81-d697-4d0f-8b02-2d1c8374ba00. |
| [29] | Mori M, Nucci A, Lang MC, et al. Functional and structural characterization of 2-amino-4-phenylthiazole inhibitors of the HIV-1 nucleocapsid protein with antiviral activity[J]. ACS Chem Biol, 2014, 9: 1950–1955. DOI:10.1021/cb500316h |
| [30] | Warui DM, Baranger AM. Identification of specific small molecule ligands for stem loop 3 ribonucleic acid of the packaging signal Psi of human immunodeficiency virus-1[J]. J Med Chem, 2009, 52: 5462–5473. DOI:10.1021/jm900599v |
| [31] | Warui DM, Baranger AM. Identification of small molecule inhibitors of the HIV-1 nucleocapsid-stem-loop 3 RNA complex[J]. J Med Chem, 2012, 55: 4132–4141. DOI:10.1021/jm2007694 |
| [32] | Zagotto G, Sissi C, Lucatello L, et al. Aminoacyl-anthraquinone conjugates as telomerase inhibitors:synthesis, biophysical and biological evaluation[J]. J Med Chem, 2008, 51: 5566–5574. DOI:10.1021/jm800160v |
| [33] | Sosic A, Frecentese F, Perissutti E, et al. Design, synthesis and biological evaluation of TAR and cTAR binders as HIV-1 nucleocapsid inhibitors[J]. MedChemComm, 2013, 4: 1388–1393. DOI:10.1039/c3md00212h |
| [34] | Frecentese F, Sosic A, Saccone I, et al. Synthesis and in vitro screening of new series of 2, 6-dipeptidyl-anthraquinones:influence of side chain length on HIV-1 nucleocapsid inhibitors[J]. J Med Chem, 2016, 59: 1914–1924. DOI:10.1021/acs.jmedchem.5b01494 |
| [35] | Sosic A, Cappellini M, Scalabrin M, et al. Nucleocapsid annealing-mediated electrophoresis (NAME) assay allows the rapid identification of HIV-1 nucleocapsid inhibitors[J]. J Vis Exp, 2015, 95: 52474. |
| [36] | Sosic A, Sinigaglia L, Cappellini M, et al. Mechanisms of HIV-1 nucleocapsid protein inhibition by lysyl-peptidyl-anthraquinone conjugates[J]. Bioconjugate Chem, 2016, 27: 247–256. DOI:10.1021/acs.bioconjchem.5b00627 |
| [37] | Kim MJ, Kim SH, Park JA, et al. Identification and charac-terization of a new type of inhibitor against the human immunodeficiency virus type-1 nucleocapsid protein[J]. Retrovirology, 2015, 12: 90. DOI:10.1186/s12977-015-0218-9 |
| [38] | Sancineto L, Mariotti A, Bagnoli L, et al. Design and synthesis of diselenobisbenzamides (DISeBAs) as nucleocapsid protein 7(NCp7) inhibitors with anti-HIV activity[J]. J Med Chem, 2015, 58: 9601–9614. DOI:10.1021/acs.jmedchem.5b01183 |
| [39] | Wang L, Zhan P, Liu XY. The application of structural optimization strategies in drug design of HIV NNRTIs[J]. Acta Pharm Sin (药学学报), 2012, 47: 1409–1422. |
 2017, Vol. 52
2017, Vol. 52


