2. 中南大学临床药理研究所, 遗传药理学湖南省重点实验室, 湖南 长沙 410078;
3. 复旦大学遗传与发育协同创新中心, 上海 200433;
4. 湖南省分子靶标新药研究协同创新中心, 湖南 衡阳 421001
2. Institute of Clinical Pharmacology, Central South University, Hunan Key Laboratory of Pharmacogenetics, Changsha 410078, China;
3. Collaborative Innovation Center for Genetics and Development, Fudan University, Shanghai 200433, China;
4. Hunan Province Cooperative Innovation Center for Molecular Target New Drug Study, Hengyang 421001, China
药物基因组学致力于利用基因组学及分子生物学手段, 发现个体间用药疗效及毒副反应差异与基因之间关联并对其机制进行阐明, 其研究策略包括候选基因方法和全基因组关联分析(genome-wide association study, GWAS)方法等[1]。GWAS等基因与性状关联性研究发现大量与用药后表型相关基因多态性, 然而这些位点大多处于非编码序列区域, 对这些非编码位点的机制的解释和阐明需借助多种手段。
表达数量性状位点(eQTL)是基因组内与基因表达水平连锁或关联的遗传变异位点。eQTL的变异类型既可能是单核苷酸多态性(SNP)、拷贝数变异(CNV)也可能是插入缺失(InDel)。药物反应的个体差异表型不仅取决于个体的基因型, 还取决于个体间体内药物应答相关蛋白(包括药物作用通路相关蛋白、代谢酶和转运体等)的表达量。eQTL能够通过mRNA稳定性、DNA甲基化程度、基因间互相作用和调控等来影响这些基因的转录水平, 进而改变关键蛋白量直至表型。研究表明, 大多数GWAS分析发现的性状相关SNP位点大都可能是eQTL[2]。传统的药物基因组学研究方法结合GWAS与eQTL, 为阐明非编码区遗传变异影响个体表型的机制提供了桥梁。将eQTL分析应用于药物基因组学领域, 能够为药物临床疗效研究提供更多有效手段, 促进更多的相关生物标志物位点的发现[3]。本文就eQTL在药物基因组学中的应用及其前景进行综述。
1 eQTL分析方法 1.1 药物基因组学eQTL分析方法eQTL分析方法由Jansen和Nap[4]于2001年提出。其通过将整个基因组中所有基因多态性与组织中基因的mRNA表达水平定量作图, 进而发现特定组织对特定的基因表达存在影响的基因多态性, 即为该组织中这些基因的eQTL。由于mRNA表达在各个组织中存在差异, 因而eQTL也存在组织特异性, 相同基因在不同组织的eQTL不同。药物基因组学eQTL分析中的一类模式是采用淋巴母细胞系(lymphoblastoid cell lines, LCLs, 通过EBV病毒感染捐赠者的B淋巴细胞使其永生化得到)进行(流程如图 1所示), 对LCL细胞进行药物处理, 处理后的与未处理的细胞mRNA表达水平采用高通量的微阵列芯片(microarray)或者RNA测序技术(RNA-seq)[5]测定, 其用药表型通过对应方法测定(如细胞毒性测定细胞凋亡等)。通过分析药物处理前后LCL细胞药物相应表型、mRNA表达水平与基因组位点之间的关联(eQTL作图), 从而得到药物疗效相关eQTL。另一类模式则是关注药物基因组学相关关键蛋白的eQTL (如肝脏CYP450酶的eQTL), 研究这些eQTL对重要蛋白的影响。eQTL按照多态性与表达基因位置的关系, 分为在该基因转录起始位置(transcription start sites, TSS) 1 Mb内的顺式(cis或local) eQTL和基因TSS 1 Mb外的反式(trans或distal) eQTL。
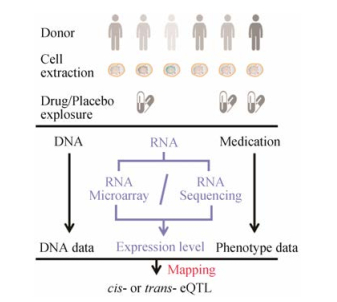
|
Figure 1 The process of pharmacogenomics expression quantitative trait loci (eQTL) analysis by LCL cell |
Lalonde等[6]通过对成熟RNA与未加工的RNA进行RNA-seq发现:约75% eQTL与转录前mRNA水平相关, 剩余约25%的eQTL与共转录或转录后mRNA水平相关。eQTL中经过机制验证的SNP为eSNP, eSNP影响转录前mRNA水平的机制包括(图 2中1~4): ① 调控序列变异, 进而影响包括DNA的甲基化程度、乙酰化程度以及和转录因子等在内的调节蛋白的结合, 直接影响转录。② eSNP位于DNA与核小体结合的区域, 会使得与核小体结合减弱甚至不结合核小体, 直接影响转录; 此类变异大多为cis-eQTL。③ eSNP (cis-eQTL)与蛋白产物间反馈影响mRNA水平。④ eSNP (trans-eQTL)通过基因转录调节网络的直接或间接作用影响一个或多个下游基因的表达。eSNP影响转录后mRNA水平的机制包括(图 2中5~8): ① eSNP (cis-eQTL)改变了转录后的mRNA稳定性, 但并不直接影响转录。② eSNP (cis-eQTL)影响mRNA的剪接, 通过可变剪接(AS)影响mRNA水平。③ miRNA相关eSNP (包括miRNA结合的靶基因3' UTR区变异和miRNA编码基因变异)能够通过影响miRNA与mRNA的结合及miRNA自身的稳定性进而改变靶基因的表达水平, 其既可能是cis-eQTL也可能是trans-eQTL, 如Wei等[7]发现14个外源化学物代谢酶和转运体(XMET)基因的27个3' UTR区变异中的26个均与miRNA结合相关。④ ceRNA相关eSNP, 使得circRNA或lncRNA对miRNA的内源性竞争结合能力发生改变, 进而影响miRNA对mRNA的抑制作用。
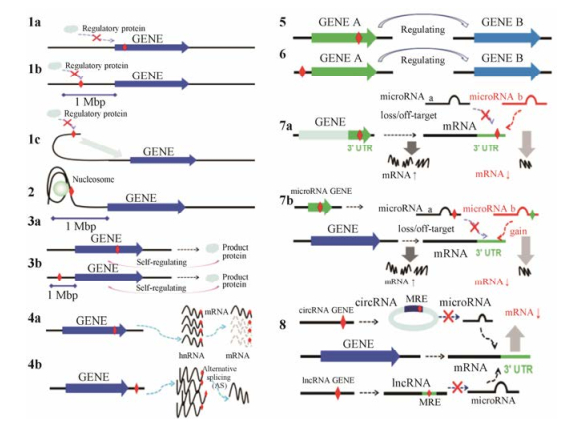
|
Figure 2 Mechanisms of eQTL affect the level of mRNA expression. The red diamonds mean eSNP (e-single nucleotide polymor-phism) |
顺铂等化疗药物耐药及其毒性是造成肿瘤治疗失败的重要原因, 其疗效及毒性与遗传间关联是药物基因组学关注的重点之一。eQTL rs17115814与顺铂耐药相关的宿主基因PRPF39的表达水平显著相关[8]。Huang等[9]通过三步模型发现与顺铂半数抑制浓度(IC50)相关的多个eQTL, 其与DDIT4、NEK2及MYC等mRNA水平相关, 影响化疗药物顺铂所致毒性。Wen等[10]将顺铂、卡铂、紫杉醇、5'-DFUR、阿糖胞苷和培美曲塞等3大类6种化疗药物引起的细胞凋亡作为表型, 发现了19个与细胞毒性和细胞凋亡相关的eQTL, 其与291个基因的表达水平相关; CTTN和ZMAT3表达水平与培美曲塞表型显著相关, 受到37个eQTL的调控[11]。Wheeler等[12]将GWAS和LCL细胞模型整合, 分析得到rs7254081等多个eQTL通过调控RFX2的mRNA水平, 与紫杉醇细胞毒性和患者的外周感觉神经病变相关。Duan等[13]的研究发现一条抗肿瘤药物通路——叶酸通路相关基因FPGS的表达水平与eQTL rs9990951相关。这些研究表明eQTL能够通过影响化疗药物相关基因的mRNA表达量进而影响药物表型。这些与化疗药物临床疗效及毒性关联的eQTL, 具有成为预测化疗药物疗效及毒性的生物标志物的潜力, 能够为实施化疗药物临床个体化用药提供桥梁。
2.2 糖尿病治疗药物应答eQTL糖尿病是一类全球范围的发病率高、治疗个体差异大的重大疾病。与糖尿病相关的GWAS研究发现了大量的关联基因及变异, 而新的eQTL研究也揭示了GWAS研究所未能阐明的基因多态性调控表型的机制[14]。糖尿病治疗一线用药二甲双胍存在较大个体差异, 前期药物基因组学研究发现, 相关转运体基因变异能够影响其吸收转运并影响疗效; rs8192675等cis-eQTL能够改变转运体SLC2A2基因mRNA表达水平, 并影响二甲双胍疗效[15]。噻唑烷二酮(TZD)类降糖药主要通过靶向PPARγ蛋白发挥作用, TMEM170B基因的表达SNP (eSNP) rs568867能够显著改变该基因与转录调控蛋白PPARγ结合水平, 并影响TZD类药物糖尿病疗效[16]。将以上eQTL应用于糖尿病药物的个体化用药, 有助于提高糖尿病治疗的有效率。
2.3 他汀类疗效及所致肌病相关eQTL他汀类是一类临床常用的降脂药, 然而其可能引起严重的肌肉疾病, 导致横纹肌溶解和急性肾功能衰竭。前期研究证明, 他汀类的疗效和毒副反应受遗传因素的影响, 存在显著个体差异。通过发现他汀类药物的药物
基因组学标志物, 有助于减少不良反应的发生并提高治疗效果。Postmus等[17]通过GWAS、eQTL研究以及对多个类似研究meta分析发现, SORT1/CELSR2/ PSRC1上的rs646776、rs12740374和SLCO1B1基因上的rs2900478与他汀类疗效(如低密度脂蛋白应答)相关。Mangravite等[18]发现肌酸合成限速酶GATM的cis-eQTL rs9806699等6个eQTL与辛伐他汀降脂效果及他汀类所致肌病的发生相关。
2.4 哮喘治疗药糖皮质激素类疗效相关eQTL哮喘作为一种顽固性慢性气管疾病, 其治疗常采用吸入性糖皮质激素疗法, 糖皮质激素疗效受遗传因素影响。对糖皮质激素的药物基因组学eQTL研究发现, rs7850103能够影响与疗效显著相关的TNC基因mRNA表达水平, 因此造成地塞米松累积, 并影响吸入性糖皮质激素疗效[19, 20]。此外, eSNP rs6504666和rs1380657能够影响SPATA20表达水平, 与使用吸入性糖皮质激素的患者呼吸道表型相关联[21]。
2.5 药代动力学相关蛋白eQTL肝脏作为体内药物代谢的重要器官, 其蛋白基因的遗传变异能够明显影响药物在体内的反应, 这些重要蛋白的基因型是药物基因组学的重要生物分子标志物。但是只有13%的药物相关表型与遗传变异之间联系可以被蛋白质结构变异所解释; 除蛋白结构变异外, 药物相关表型还与ADME基因表达水平相关, 遗传变异能够影响包括基因转录、可变剪切、转录稳定、翻译速率及蛋白表达等[22]。Schadt等[23-26]的多个针对肝脏eQTL的研究, 确定了一系列肝脏eQTL; Fu等[27]将其与PharmGKB数据库结合, 总结出与COMT、CYP2D6等“Very Important Pharmacogenes (VIP)”相关的eSNPs。Bonder等[28]研究了婴儿及成人肝脏基因表达相关的eQTL和甲基化数量性状位点(meQTL), 这些eQTL和meQTL能够影响基因表达水平和甲基化水平, 为肝脏药物反应及肝脏相关疾病病因提供新的见解。Matsson等[29]发现24个与ABC族转运体表达水平有关的local-eQTL, 这些SNP通过调控ABC转运体表达水平, 能够影响相关疾病的易感性和药物反应。Schröder等[25]对ADME相关mRNA表达表型进行了全基因组关联分析, 重复出21个、新发现74个与酶、转运体、转录调节及其他基因相关的eQTL。由于大多数药物均通过肝脏代谢, 这些药物应答相关蛋白的肝脏eQTL, 可能具有成为药物临床应答相关的生物标志物的潜力。
3 eQTL应用于药物基因组学的不足 3.1 研究人群缺乏代表性目前进行的药物应答相关eQTL研究, 其选取人群(表 1)多为人类多态性研究中心(the Centre d’Etude du Polymorphisme Humain, CEPH) HapMap计划的西北欧洲后裔人群(CEU)和非洲尼日利亚约鲁巴人群(YRI), 而大多缺少东方人群的代表——中国北京汉族人群(CHB)。这使得这些研究缺少东方人特有的变异对基因组mRNA水平的影响, 其研究结果应用于东方人群的个体化用药存在缺陷。
| Table 1 Pharmacogenomics related eQTL studies. CEU: Utah residents with Northern and Western European ancestry from the CEPH (the Centre d'Etude du Polymorphisme Humain) collection; YRI: Yoruba in Ibadan, Nigeria (West Africa) |
人体的众多基因在不同的组织和器官的表达是不同的, 因而各组织器官的mRNA表达水平也不一致。特定的eQTL只在某一组织或者器官影响其相应的基因表达, 而非普遍性的。LCL细胞系作为一种单一的细胞系模型, 无法模拟体内众多类型的组织器官。例如, LCL细胞系无法表达肝脏常见的CYP450酶, 因而无法模拟肝脏细胞许多药物基因组学重要酶mRNA的表达情况, 也无法得到CYP450酶基因的eQTL。使用肝脏组织细胞直接进行eQTL研究, 虽然能够发现许多重要的药物基因组学相关基因的eQTL, 但却缺乏针对具体药物疗效和不良反应相关的研究。
4 展望eQTL应用于药物基因组学, 能够促进药物疗效及不良反应相关遗传多态性的发现, 阐明众多前期GWAS等发现的非编码区域位点的机制。近年来, 蛋白质QTL (pQTL)也被广泛地用于分析遗传与表型关联。相较于直接对承载生理蛋白质定量, mRNA表达量并不能够完全反映表型的水平(图 3)。但由于mRNA定量微芯片技术和测序技术的成熟, 相对于采用质谱等复杂手段的pQTL, eQTL具有开展便捷、通量高、技术成熟和成本低等优点。与GWAS研究和全转录组关联分析(TWAS)联合(图 3), 有助于提高对药物基因组学相关生物标志物的发现能力, 为更多的相关临床个体化精准用药的实施提供依据。
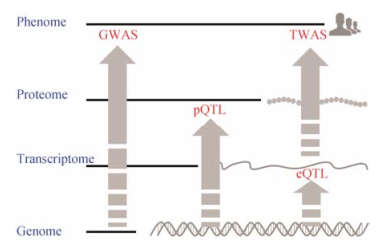
|
Figure 3 eQTL, pQTL (protein quantitative trait loci), GWAS (genome-wide association study) and TWAS (transcriptome-wide association study) research levels |
| [1] | Zhou Q, Yu LS, Zeng S. Personalized dosing from perspective of pharmacogenomics of drug metabolizing enzymes and transporters[J]. Acta Pharm Sin (药学学报), 2017, 52: 1–7. |
| [2] | Gamazon ER, Huang RS, Cox NJ, et al. Chemotherapeutic drug susceptibility associated SNPs are enriched in expression quantitative trait loci[J]. Proc Natl Acad Sci U S A, 2010, 107: 9287–9292. DOI:10.1073/pnas.1001827107 |
| [3] | Hormozdiari F, van de Bunt M, Segrè AV, et al. Colocalization of GWAS and eQTL signals detects target genes[J]. Am J Hum Genet, 2016, 99: 1245–1260. DOI:10.1016/j.ajhg.2016.10.003 |
| [4] | Jansen RC, Nap JP. Genetical genomics:the added value from segregation[J]. Trends Genet, 2001, 17, 7: 388–391. |
| [5] | Pickrell JK, Marioni JC, Pai AA, et al. Understanding mechanisms underlying human gene expression variation with RNA sequencing[J]. Nature, 2010, 464: 768–772. DOI:10.1038/nature08872 |
| [6] | Lalonde E, Ha KCH, Wang Z, et al. RNA sequencing reveals the role of splicing polymorphisms in regulating human gene expression[J]. Genome Res, 2011, 21: 545–554. DOI:10.1101/gr.111211.110 |
| [7] | Wei R, Yang F, Urban TJ, et al. Impact of the interaction between 3'-UTR SNPs and microRNA on the expression of human xenobiotic metabolism enzyme and transporter genes[J]. Front Genet, 2012, 3: 248. |
| [8] | Stark AL, Delaney SM, Wheeler HE, et al. Functional consequences of PRPF39 on distant genes and cisplatin sensitivity[J]. Hum Mol Genet, 2012, 21: 4348–4355. DOI:10.1093/hmg/dds266 |
| [9] | Huang RS, Duan S, Shukla SJ, et al. Identification of genetic variants contributing to cisplatin-induced cytotoxicity by use of a genomewide approach[J]. Am J Humn Genet, 2007, 81: 427–437. DOI:10.1086/519850 |
| [10] | Wen Y, Gorsic LK, Wheeler HE, et al. Chemotherapeuticinduced apoptosis:a phenotype for pharmacogenomics studies[J]. Pharmacogenet Genomics, 2011, 21: 476–488. DOI:10.1097/FPC.0b013e3283481967 |
| [11] | Wen Y, Gamazon ER, Bleibel WK, et al. An eQTL-based method identifies CTTN and ZMAT3 as pemetrexed susceptibility markers[J]. Hum Mol Genet, 2012, 21: 1470–1480. DOI:10.1093/hmg/ddr583 |
| [12] | Wheeler HE, Gamazon ER, Wing C, et al. Integration of cell line and clinical trial genome-wide analyses supports a polygenic architecture of paclitaxel-induced sensory peripheral neuropathy[J]. Clin Cancer Res, 2013, 19: 491–499. DOI:10.1158/1078-0432.CCR-12-2618 |
| [13] | Duan S, Huang RS, Zhang W, et al. Expression and alternative splicing of folate pathway genes in HapMap lymphoblastoid cell lines[J]. Pharmacogenomics, 2009, 10: 549–563. DOI:10.2217/pgs.09.8 |
| [14] | Das SK, Sharma NK. Expression quantitative trait analyses to identify causal genetic variants for type 2 diabetes susceptibility[J]. World J Diabetes, 2014, 5: 97–114. |
| [15] | Zhou K, Yee SW, Seiser EL, et al. Variation in the glucose transporter gene SLC2A2 is associated with glycemic response to metformin[J]. Nat Genet, 2016, 48: 1055–1059. DOI:10.1038/ng.3632 |
| [16] | Soccio RE, Chen ER, Rajapurkar SR, et al. Genetic variation determines PPARγ function and anti-diabetic drug response in vivo[J]. Cell, 2015, 162: 33–44. DOI:10.1016/j.cell.2015.06.025 |
| [17] | Postmus I, Trompet S, Deshmukh HA, et al. Pharmacogenetic meta-analysis of genome-wide association studies of LDL cholesterol response to statins[J]. Nat Commun, 2014, 5: 5068. DOI:10.1038/ncomms6068 |
| [18] | Mangravite LM, Engelhardt BE, Medina MW, et al. A statindependent QTL for GATM expression is associated with statininduced myopathy[J]. Nature, 2013, 502: 377–380. DOI:10.1038/nature12508 |
| [19] | Park HW, Tantisira KG, Weiss ST. Pharmacogenomics in asthma therapy:where are we and where do we go?[J]. Annu Rev Pharmacol Toxicol, 2015, 55: 129–147. DOI:10.1146/annurev-pharmtox-010814-124543 |
| [20] | Grundberg E, Adoue V, Kwan T, et al. Global analysis of the impact of environmental perturbation on cis-regulation of gene expression[J]. PLoS Genet, 2011, 7: e1001279. DOI:10.1371/journal.pgen.1001279 |
| [21] | Qiu W, Rogers AJ, Damask A, et al. Pharmacogenomics:novel loci identification via integrating gene differential analysis and eQTL analysis[J]. Hum Mol Genet, 2014, 23: 5017–5024. DOI:10.1093/hmg/ddu191 |
| [22] | Glubb DM, Dholakia N, Innocenti F. Liver expression quantitative trait loci:a foundation for pharmacogenomic research[J]. Front Genet, 2012, 3: 153. |
| [23] | Schadt EE, Molony C, Chudin E, et al. Mapping the genetic architecture of gene expression in human liver[J]. PLoS Biol, 2008, 6: e107. DOI:10.1371/journal.pbio.0060107 |
| [24] | Innocenti F, Cooper GM, Stanaway IB, et al. Identification, replication, and functional fine-mapping of expression quantitative trait loci in primary human liver tissue[J]. PLoS Genet, 2011, 7: e1002078. DOI:10.1371/journal.pgen.1002078 |
| [25] | Schröder A, Klein K, Winter S, et al. Genomics of ADME gene expression:mapping expression quantitative trait loci relevant for absorption, distribution, metabolism and excretion of drugs in human liver[J]. Pharmacogenomics J, 2013, 13: 12–20. DOI:10.1038/tpj.2011.44 |
| [26] | Greenawalt DM, Dobrin R, Chudin E, et al. A survey of the genetics of stomach, liver, and adipose gene expression from a morbidly obese cohort[J]. Genome Res, 2011, 21: 1008–1016. DOI:10.1101/gr.112821.110 |
| [27] | Fu J, Wolfs MGM, Deelen P, et al. Unraveling the regulatory mechanisms underlying tissue-dependent genetic variation of gene expression[J]. PLoS Genet, 2012, 8: e1002431. DOI:10.1371/journal.pgen.1002431 |
| [28] | Bonder MJ, Kasela S, Kals M, et al. Genetic and epigenetic regulation of gene expression in fetal and adult human livers[J]. BMC Genomics, 2014, 15: 860. DOI:10.1186/1471-2164-15-860 |
| [29] | Matsson P, Yee SW, Markova S, et al. Discovery of regulatory elements in human ATP-binding cassette transporters through expression quantitative trait mapping[J]. Pharmacogenomics J, 2012, 12: 214–226. DOI:10.1038/tpj.2011.8 |
 2017, Vol. 52
2017, Vol. 52


